Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista bio ciencias
On-line version ISSN 2007-3380
Revista bio ciencias vol.10 Tepic 2023 Epub Feb 22, 2024
https://doi.org/10.15741/revbio.10.e1395
Artículo Original
Evaluación de compuestos bioactivos de plantas de uso medicinal en México, para predecir posibles inhibidores del SARS-CoV-2: análisis entre dos servidores de acoplamiento molecular.
1 Facultad de Ciencias Químicas. Universidad Autónoma de Coahuila. 25280, Saltillo, Coahuila,México.
2 Centro de Investigación y Jardín Etnobiológico. Universidad Autónoma de Coahuila 27480, Viesca Coahuila, México.
3 Departamento de Bioquímica. Centro de Investigación Biomédica. Universidad Autónoma de Coahuila. 27000, Torreón, Coahuila, México.
4 Centro de Investigación y Estudios Interdisciplinados. Universidad Autónoma de Coahuila. 25280, Arteaga, Coahuila, México
Las plantas utilizadas en la medicina tradicional podrían ser una alternativa frente al SARS-CoV-2. En este estudio se evaluó la energía de unión de los bioactivos de 10 plantas de uso en México contra la COVID-19 mediante acoplamiento molecular con dos servidores que están disponibles en línea: COVID-19 Docking Server y DockThor. Remdesivir se usó como control. Los resultados mostraron que Cypellocarpin B, Cypellocarpin C, Luteolin 7-glucoside y Syringetin glucopyranoside mostraron la mayor afinidad hacia las proteínas Mpro y RdRp con respecto a Remdesivir. La comparación de los servidores de acoplamiento molecular mostró diferencias en los motores de acoplamiento y las proteínas disponibles en cada servidor. Los compuestos bioactivos evaluados son capaces de actuar como inhibidores potenciales frente a las proteínas Mpro y RdRp de COVID-19 según el acoplamiento molecular in silico.
PALABRAS CLAVE: Compuestos fenólicos; Acoplamiento molecular; DockThor; COVID-19; SARS-CoV-2
Plants traditionally used for medicinal use could be an alternative against SARS-CoV-2. In this study, the binding energy of 10 bioactive compounds of plants used in Mexico against COVID-19 was evaluated by molecular docking with two online servers: COVID-19 Docking Server and DockThor. Remdesivir was used as a control. The results showed that Cypellocarpin B, Cypellocarpin C, Luteolin 7-glucoside, and Syringetin glucopyranoside showed the highest binding energy towards Mpro and RdRp proteins with respect to Remdesivir. The comparison of molecular docking servers showed differences in the docking motors and proteins available on each server. The bioactive compounds evaluated can act as potential inhibitors against Mpro and RdRp proteins of COVID-19, according to in silico molecular docking.
KEY WORDS: compounds; Molecular docking; DockThor; COVID-19; SARS-CoV-2
Introducción
La enfermedad por Coronavirus 2019 (COVID-19), causada por la infección de SARS-CoV-2, se ha propagado rápidamente en todo el mundo y se ha convertido en una emergencia de salud pública mundial (Yang et al., 2020). El coronavirus está relacionado con el Síndrome Respiratorio Agudo Severo (SARS), tipo 2 (Yoshimoto, 2020). Algunas de las principales proteínas virales del SARS-CoV-2 son la proteína espiga (S; Spike), la principal proteína estructural en la invasión celular mediada por los receptores ACE2, y la proteína de la célula huésped TMPRSS2. La proteína S es actualmente la diana terapéutica más investigada. Otra proteína importante es la proteasa principal no estructural Mpro (también conocida como 3CLPro) que facilita el procesamiento proteolítico de las poliproteínas (Da Silva et al., 2020; Paraiso et al., 2020). La ARN polimerasa dependiente de ARN (RdRp) juega un papel crucial en el ciclo viral. RdRp es la región más conservada y accesible de los virus de ARN; apuntar a esta región para inhibir la replicación viral puede ser un enfoque terapéutico práctico (Aftab et al., 2020).
Además del desarrollo y uso de vacunas, se ha propuesto el uso de medicamentos preexistentes. Sin embargo, la efectividad de estos es limitada. El uso de los antivirales existentes también se ha reasignado para reducir el tiempo y el costo en comparación con el desarrollo de nuevos medicamentos. Algunos medicamentos existentes evaluados frente al SARS-CoV-2 son Remdesivir, Lopinavir/Ritonavir (Yang et al., 2020).
Efecto y mecanismo de acción de Remdesivir
Los análogos de nucleósidos son agentes antivirales que han demostrado eficacia contra virus, como coronavirus, VIH, hepatitis B y C (Eastman et al., 2020). Remdesivir es un profármaco análogo de nucleósido que se metaboliza dentro de las células y se convierte en un metabolito activo de nucleósido trifosfato (NTP) (Moneriz & Castro-Salguedo, 2020). El metabolito activo se dirige a la maquinaria de replicación del ARN viral y actúa como sustrato para RdRp, donde compite con el ATP para incorporar nuevas hebras para formar la cadena de ARN. La incorporación de remdesivir interrumpe los procesos moleculares posteriores (Malin et al., 2020). La RdRp viral es la proteína diana del metabolito activo, aunque los estudios in silico también han demostrado que remdesivir puede unirse fuertemente a la proteína Mpro (Nguyen et al., 2020).
Compuestos bioactivos
En frutas, verduras y plantas, compuestos bioactivos como las vitaminas, fitoquímicos y los compuestos fenólicos (flavonoides y carotenoides) tienen beneficios para la salud. Estos compuestos tienes propiedades biológicas como actividad antioxidante, anticancerígena y antimicrobiana (Torres-León et al., 2017). Las propiedades antimicrobianas incluyen actividad antiviral. Los componentes antivirales de varias frutas y plantas pueden actuar sobre los virus y las células huésped para prevenir infecciones virales (Mukhtar et al., 2008; Bright & Gilling, 2016; Ben-Shabat et al., 2019).
Ensayos in silico
Los ensayos in silico se han investigado para evaluar la posible interacción y afinidad de los compuestos bioactivos contra el SARS-CoV-2. Se ha informado a través de estudios in silico e in vitro que algunos polifenoles tienen el potencial de inhibir RdRP y las proteasas virales de SARS-CoV-2 (Torres-León et al., 2020; Singh et al., 2020). Los polifenoles son compuestos prometedores para tener afinidad con las proteasas virales involucradas en la replicación viral (Paraiso et al., 2020; Singh et al., 2020). Se han realizado estudios in silico sobre algunas plantas medicinales ricas en taninos hidrolizables, que pueden ser utilizadas para tratar el SARS-CoV-2 (Khalifa et al., 2020). Otros estudios sugieren que los glucósidos de flavonoides pueden desempeñar un papel clave como inhibidores del SARS-CoV-2 en Mpro y RdRp (Da Silva et al., 2020). Por lo tanto, es relevante evaluar los compuestos bioactivos presentes en plantas, frutas y vegetales a través de un enfoque de acoplamiento molecular para inhibir las proteínas del SARS-CoV-2 (Tallei et al., 2020).
Los programas de software para acoplamiento molecular están disponibles comercialmente. Sin embargo, algunos son bastante costosos, lo que limita su uso y algunas licencias deben renovarse cada año (Benfenati et al., 2010). Algunos programas de software gratuitos están disponibles en línea, donde los costos son cero y pueden usarse fácilmente para evaluar cualquier producto químico. Este tipo de programas pueden ayudar a reducir el tiempo en la selección de compuestos bioactivos y facilitar su análisis in vitro o in vivo.
En México, existe una gran cantidad de plantas medicinales que se utilizan en la medicina tradicional. El estudio de estas plantas ha contribuido al descubrimiento de nuevas sustancias con actividad biológica. Por ello, en este trabajo se han seleccionado diversas plantas tales como Parthenium argentatum A.Gray, Turnera diffusa Willd. ex Schult., Larrea tridentata (Sessé & Moc. ex DC.) Coville, Taraxacum officinale (L.) Weber ex F.H.Wigg, Moringa oleifera L., Eucalyptus camaldulensis Dehnh., y Bougainvillea glabra Choisy. Estas plantas se encuentran distribuidas en todo el territorio mexicano y han sido utilizadas como plantas medicinales. Las moléculas de cada planta fueron seleccionadas de acuerdo con una investigación previa sobre la presencia de actividad viral o biológica. Los compuestos bioactivos contra las proteínas SARS-CoV-2, Mpro (PDB; 6LU7) y RdRp (PDB; 7BV2) se probaron mediante acoplamiento molecular utilizando dos servidores disponibles en línea.
Material y Métodos
Plantas y moléculas
Las plantas, y sus moléculas entre paréntesis: Parthenium argentatum A. Gray (Neochlorogenic acid; CID: 5280633), Turnera diffusa Willd. ex Schult. (Luteolin 7-glucoside; CID: 5280637, Syringetin glucopyranoside; CID: 16109838), Larrea tridentata (Sessé & Moc. ex DC.) Coville (Nordihydroguayretic acid; CID: 4534), Moringa oleifera Lam. (Kaempferol; CID: 5280863, Apigenine 7 O-glucóside; CID: 5280704), Eucalyptus camaldulensis Dehnh. (Cypellocarpin B; CID: 10506563, Cypellocarpin C; CID: 10625791), Taraxacum officinale (L.) Weber ex F.H.Wigg. (Betulin; CID: 72326) y Bougainvillea glabra Choisy (Vitexin; CID: 5280441) fueron seleccionados para este estudio. Remdesivir (CID: 121304016) se utilizó como control positivo. Esta molécula ha sido aprobada por la FDA para su uso en pacientes con COVID-19 (FDA, 2020), y tiene una amplia investigación como posible tratamiento contra COVID-19 (Beigel et al., 2020; Ko et al., 2020; Ma et al., 2021; Wang, Y. et al., 2020; Yang et al., 2020). Las moléculas en 2D se obtuvieron de PubChem (https://pubchem.ncbi.nlm.nih.gov/) en formato SDF.
Determinación de la lipofilicidad
Se utilizó la herramienta web SwissADME (http://www.swissadme.ch/index.php) para predecir la lipofilicidad (LogP). La lipofilicidad es un factor importante para la absorción, distribución en el cuerpo, penetración a través de membranas vitales y barreras biológicas, metabolismo y excreción de un compuesto (propiedades ADME).
El logP de un compuesto destinado a la administración oral debe ser <5. Por lo tanto, se evaluaron las moléculas seleccionadas para determinar cuáles cumplen con esta regla.
Acoplamiento molecular in silico
Las moléculas se evaluaron como ligandos potenciales para las proteínas SARS-CoV-2, Mpro (PDB: 6LU7) y RdRp (PDB: 7BV2), utilizando dos servidores de acoplamiento molecular en línea (ambos servidores están disponibles para su uso gratuito). Las moléculas descargadas de PubChem no fueron modificadas antes del análisis.
Actualmente, existe una gran cantidad de servidores para hacer acoplamiento molecular; algunos de ellos deben instalarse en una computadora y complementarse con el uso de más programas. Además, la mayoría de estos programas tienen que ser comprados. Sin embargo, también existen programas gratuitos para acoplamiento molecular. Los programas gratuitos en línea tienen ventajas económicas (costo cero), de infraestructura (no requieren computadoras de alta calidad), de almacenamiento (la información que generan estos programas se almacena en sus servidores) y de análisis (las proteínas de interés ya están disponibles). Las desventajas son las opciones limitadas para cambiar detalles y la menor disponibilidad de recursos computacionales para experimentos de detección virtual de alto rendimiento. Sin embargo, los servidores en línea para acoplamiento molecular son útiles para buscar compuestos bioactivos contra el SARS-CoV-2.
En el presente estudio, utilizamos dos servidores disponibles en línea y gratuitos. Para la selección de los dos servidores se ingresó a la base de datos BIOPEP-UWM de la Universidad de Mazury en Olsztyn, Polonia (https://biochemia.uwm.edu.pl/en/docking-2/). La base de datos cuenta con 23 programas en línea para acoplamiento molecular: herramienta B-AceP, AMMOS2, AutoDock Vina, CB-Dock, BINANA, ClusPro, CovalentDock Cloud, COVID-19 Docking Server, DockThor, EDock, FitDock, GalaxyPEPDOCK, HawkDock, HPEPDOCK, Hex, InstaDock, PIPER-FlexPepDock, ProteinsPlus, SwissDock, systemsDock, servidor UNRES, Webina, ZDOCK. De todos los programas, seleccionamos dos de forma no aleatoria: COVID-19 Docking Server (CDS) y DockThor (DT).
CDS
CDS (https://ncov.schanglab.org.cn/index.php) (Kong et al., 2020) tiene proteínas de SARS-CoV-2 disponibles para acoplamiento molecular; esto facilita la evaluación de las afinidades y modos de unión entre proteínas y ligandos (moléculas pequeñas, péptidos y anticuerpos). Las proteínas utilizadas para realizar el acoplamiento molecular fueron Mpro y RdRp (sitio RTP). Las moléculas para acoplamiento se cargaron en formato SDF.
Autodock Vina se utiliza como motor de acoplamiento en CDS. La caja de acoplamiento se define como el centro de la coordenada del ligando nativo con una longitud de 30 Å × 30 Å × 30 Å para incluir residuos de toda la cavidad. CDS utiliza MGLTools para agregar hidrógenos y preparar proteínas y ligandos. El nivel de exhaustividad fue el predeterminado (12). El análisis se llevó a cabo con las proteínas Mpro (PDB: 6LU7) y RdRp (PDB: 7BV2). Se seleccionó la proteína de interés y se agregó la molécula a analizar. Se utilizó el valor de puntuación del modelo 1 (en kcal/mol).
DT
DT (https://dockthor.lncc.br/v2/) (da Silveira et al., 2019; Santos et al., 2020) tiene proteínas virales de SARS-CoV-2 específicas para realizar acoplamiento molecular. Por lo tanto, solo se carga la molécula de interés y se selecciona la proteína a evaluar. Las estructuras 2D de las moléculas en formato SDF se convirtieron a formato PDB con el convertidor en línea SMILES (https://cactus.nci.nih.gov/translate/). Para las proteínas se utilizaron las estructuras de tipo salvaje Nsp5-Mpro (PDB: 6LU7) y Nsp12-RdRp (PDB:7BV2).
Como parámetros de acoplamiento, se definió el sitio de unión catalítica de ambas proteínas y el tamaño de la cuadrícula en 20 Å × 20 Å × 20 Å. Se utilizó la precisión del algoritmo estándar (1,000,000 de evaluaciones, tamaño de población de 750 y 24 ejecuciones).
Análisis de unión proteína-ligando
Los resultados del servidor DT que tuvieron resultados similares o mejores que el control Remdesivir (Cypellocarpins B y C, Luteolin 7-glucoside y Syringetin glucopyranoside) se descargaron para analizar las interacciones de unión ligando-proteína utilizando Discovery Studio Visualizer. Se generaron imágenes 2D que representan la interacción molécula-proteína.
Análisis de los datos
El análisis se realizó mediante métodos descriptivos. Los resultados obtenidos corresponden a la energía de enlace (kcal/mol) y el tipo de enlace entre el compuesto y la proteína. Los valores de energía de unión indican afinidad entre los compuestos y las proteínas. Los valores de energía de unión indican afinidad entre compuestos y proteínas. Una energía de enlace negativa significa que el compuesto tiene afinidad por la proteína y una energía positiva indica que el compuesto no tiene afinidad por la proteína. El valor de energía de enlace negativo indica una reacción espontánea y un sistema estable que permite la formación de enlaces.
Resultados y Discusión
Determinación de la lipofilicidad
Los resultados obtenidos mostraron que 10 moléculas tienen valores de LogP dentro del rango (<5), Remdesivir (1.50), Neochlorogenic acid (-0.38), Luteolin 7-glucoside (0.16), Syringetin glucopyranoside (-1.55), Nordihydroguayretic acid (3.29), Kaempferol (1.58), Apigenine 7 O-glucoside (0.55), Cypellocarpin B (0.70), Cypellocarpin C (1.04), Betulin (6.36), Vitexin (-0.07). De todas las moléculas, solo Betulin mostró un valor superior de LogP. Esto indica que la mayoría de las moléculas son similares a los fármacos, y su estudio como posibles tratamientos contra el SARS-CoV-2 es apropiado.
Análisis de acoplamiento molecular de compuestos bioactivos
La Tabla 1 muestra el acoplamiento entre las moléculas y las estructuras proteicas de SARS-CoV-2, Mpro y RdRp. Estos valores representan la energía de enlace (kcal/mol) obtenida con los servidores CDS y DT. En el acoplamiento entre los compuestos bioactivos y la proteína RdRp, CDS obtuvo una energía de enlace superior a la mostrada por DT, entre -8,10 y -10.60 kcal/mol, mientras que los resultados de DT están entre -6.511 y -7,542 kcal/mol. Además, el acoplamiento con la proteína Mpro mostró valores en un rango similar en ambos servidores.
Respecto a la proteína Mpro: Luteolin 7-glucoside (CDS: -8.30 kcal/mol; DT: -9.040 kcal/mol) y Syringetin glucopyranoside (CDS: -8.20 kcal/mol; DT: -8.191 kcal/mol) pertenecientes a Turnera diffusa, así como Cypellocarpin B (CDS: -9.00 kcal/mol; DT: -8.100 kcal/mol) y Cypellocarpin C (CDS: -8.60 kcal/mol; DT: -7.056 kcal/mol) de Eucalyptus camaldulensis, mostraron energía de unión superior a Remdesivir (CDS: -8.30 kcal mol; SD: -7.919 kcal/mol) y al resto de compuestos en general. Para RdRp: Luteolin 7-glucoside (CDS: -10.00 kcal/mol; DT: -6.511 kcal/mol), Syringetin glucopyranoside (CDS: -10.60 kcal/mol; DT: -7.274 kcal/mol), Cypellocarpin B (CDS: -10.10 kcal/mol; DT: -7.296 kcal/mol) y Cypellocarpin C (CDS: -9.80 kcal/mol; DT: -7.478 kcal/mol), obtuvieron una energía de unión superior a Remdesivir (CDS: -9.20 kcal/mol; DT: -7.330 kcal/mol) y a los demás compuestos. Dado que los compuestos mencionados presentaron mejor energía de unión obtenida por Remdesivir y los demás compuestos, se realizó un análisis detallado de estas moléculas.
Cypellocarpin B y C
Cypellocarpin B (Mpro: -9.00 kcal/mol; RdRp: -10.10 kcal/mol) y Cypellocarpin C (Mpro: -8.60 kcal/mol; RdRp: -9.80 kcal/mol) con CDS mostraron una mayor energía de unión a Remdesivir (Mpro: -8.30 kcal/mol; RdRp: -9.20 kcal/mol) para Mpro y RdRp. Mientras que para DT, solo Cypellocarpin B (-8.100 kcal/mol) tuvo una mayor energía de unión que Remdesivir (-7.919 kcal/mol) con la proteína Mpro. En el acoplamiento con la proteína RdRp, solo Cypellocarpin C (-7.478 kcal/mol) tuvo una puntuación superior a Remdesivir (-7.330 kcal/mol).
La literatura ha reportado que las cipelocarpinas tienen un efecto similar al (-) galato de epigaloilcatequina al inhibir la activación del antígeno del virus de Epstein-Barr, la principal causa de la mononucleosis infecciosa aguda (Brezáni & Karel, 2013). Los estudios in vitro han demostrado que Cypellocarpin B y C suprimen la cancerogénesis en células de piel de ratón (Goodger & Woodrow, 2013). Además, Cypellocarpin C es un potente agente antitumoral y tiene un efecto mayor que el aciclovir en el tratamiento del virus HSV-2 responsable del herpes genital (Treml et al., 2020).
Se han realizado diferentes estudios in silico en los que se evalúan compuestos bioactivos del género Eucalyptus como posibles inhibidores del virus SARS-CoV-2. Según nuestro conocimiento, no se han encontrado investigaciones que incluyan la evaluación de las cipelocarpinas B y C. Fitriani et al. (2020), evaluaron la energía de unión entre la proteína Mpro y la Cypellocarpin A con AutoDock tool 4. El resultado del acoplamiento mostró que la Cypellocarpin A (-6.60 kcal/mol) tiene una energía de unión más baja que el remdesivir (-7.63 kcal/mol). La diferencia entre Cypellocarpin B y C, y Cypellocarpin A puede deberse al número de hidrógenos que pueden donar; cypellocarpin C puede donar cinco enlaces de hidrógeno, cypellocarpin B dona 6 y la cypellocarpin A dona 7. También, la proteína utilizada para el acoplamiento en el trabajo de Fitriani et al. (2020) fue diferente. La proteína Mpro utilizada fue 3CLpro- X77 (PDB: 6W63), y en nuestro trabajo se utilizó Mpro (PDB: 6LU7), la preparación de ligandos y proteínas en el trabajo de Fitriani et al. (2020) se realizó con el programa Chimera 1.13.1. Se agregaron hidrógenos a la molécula y los parámetros de la cuadrícula se establecieron utilizando AutoDock Tools (ADT).
Tabla 1 Resultados del acoplamiento molecular entre moléculas bioactivas con CDS y DT.
| Fuente | PubChem CID | Molécula | Peso molecular (g/mol) | CDS | DT | ||
|---|---|---|---|---|---|---|---|
| Mpro valor (kcal/mol) | RdRp valor (kcal/mol) | Mpro valor (kcal/mol) | RdRp valor (kcal/mol) | ||||
| Chemical | 121304016 | Remdesivir (Control) | 602.6 | -8.30 | -9.20 | -7.919 | -7.330 |
| Parthenium argentatumA.Gray | 5280633 | Neochlorogenic acida | 354.31 | -7.40 | -9.30 | -7.253 | -7.075 |
| Turnera diffusa Willd. ex Schult. | 5280637 | Luteolin 7-glucosideb | 448.4 | -8.30 | -10.00 | -9.040 | -6.511 |
| 16109838 | Syringetin glucopyranosideb | 670.6 | -8.20 | -10.60 | -8.191 | -7.274 | |
| Larrea tridentata (Sessé & Moc. ex DC.) Coville | 4534 | Nordihydroguayretic acidb | 302.4 | -7.60 | -9.50 | -7.305 | -6.702 |
| Moringa oleífera L. | 5280863 | Kaempferolc | 286.24 | -7.80 | -9.30 | -8.209 | -6.641 |
| 5280704 | Apigenine 7 O-glucosidec | 432.4 | -8.00 | -9.60 | -8.910 | -7.128 | |
| Eucalyptus camaldulensis Dehnh. | 10506563 | Cypellocarpin Bd | 538.5 | -9.00 | -10.10 | -8.100 | -7.296 |
| 10625791 | Cypellocarpin Cd | 520.5 | -8.60 | -9.80 | -7.056 | -7.478 | |
| Taraxacum officinale (L.) Weber ex F.H.Wigg. | 72326 | Betuline | 442.7 | -7.10 | -9.10 | -8.886 | -7.542 |
| Bougainvillea glabra Choisy | 5280441 | Vitexinf | 432.4 | -7.90 | -8.10 | -7.596 | -7.427 |
aPiluzza et al., (2020), bGovea-Salas et al., (2017), cSaucedo-Pompa et al., (2018), dHakki et al., (2010), eDíaz et al., (2018), Abarca -Vargas & Petricevich (2018).
Los resultados de este estudio muestran que las Cypellocarpins B y C tienen mejor afinidad contra la proteína Mpro del SARS-CoV2 que la Cypellocarpin A y Remdesivir, por lo que pueden considerarse para estudios in vitro o in vivo.
Luteolin 7-glucoside
Las energías de unión de Luteolin 7-glucoside con Mpro, en CDS y DT fueron -8.30 kcal/mol y -9.040 kcal/mol, respectivamente. Remdesivir fue -8.30 kcal/mol y -7.919 kcal/mol, respectivamente. En CDS, la puntuación de Luteolin 7-glucoside es similar al control Remdesivir. Con DT se muestran resultados superiores al control, lo que indica una mejor energía de unión con la proteína Mpro en este servidor.
En RdRp, la energía de unión con CDS fue de -10.00 kcal/mol; en DT fue -6.511 kcal/mol para Luteolin 7-glucoside; en el caso de Remdesivir los valores obtenidos fueron -9.20 kcal/mol en CDS y -7.330 kcal/mol en DT (tabla 1). En CDS, la puntuación de Luteolin 7-glucoside es superior al control, por lo que hay una mejor energía de unión. En DT, la puntuación de Luteolin 7-glucoside es inferior a la del control.
La Luteolin y sus derivados poseen diferentes actividades biológicas, siendo las más destacadas la antioxidante, antimicrobiana, antiinflamatoria y anticancerígena (Žemlička et al., 2014). La Luteolin mostró una potente actividad antiviral contra el SARS-CoV (Yi et al., 2004), el virus de la encefalitis japonesa (Fan et al., 2016), la proteasa del VIH-1 (Mehla et al., 2011), Epstein-Barr virus, rotavirus Rhesus y virus Chikungunya (Zakaryan et al., 2017).
En cuanto a la inhibición del SARS-CoV-2, se ha informado que Luteolin tiene una excelente afinidad de unión a los residuos de aminoácidos del sitio activo de la proteína espiga del SARS-CoV-2 (Sen et al., 2020). Un estudio por acoplamiento molecular mostró que luteolin-7-glucoside puede potencialmente inhibir a Mpro en SARS-CoV-2 (Khaerunnisa et al., 2020), reportando un valor de -8.7 kcal/mol, utilizando Autodock 4.2, este valor es cercano al obtenido en el presente estudio con CDS para Mpro (-8.30 kcal/mol).
Otro estudio computacional informó que luteolin-7-glucoside tiene una energía de unión óptima para la inhibición de Mpro (-10.66 kcal/mol con Autodock4, -8.4 kcal/mol con Autodock Vina y -9.73 kcal/mol con Smine) (Giguet-Valard et al., 2020). Estos valores son cercanos a los obtenidos con CDS (-8.30 kcal/mol) y DockThor (-9.040 kcal/mol); se espera que esta molécula tenga potencial como inhibidor del SARS-CoV-2; además, se destaca el uso de flavonoides para la inhibición del SARS-CoV-2.
La literatura respalda el estudio de la Luteolin al demostrar actividad antiviral en varios virus y mostrar actividad contra el SARS-CoV-2. Sin embargo, son necesarios estudios in vitro o in vivo.
Syringetin glucopyranoside
Syringetin glucopyranoside con Mpro en CDS y DT mostró energías de unión de -8.20 kcal/mol y -8.191 kcal/mol, respectivamente, y Remdesivir fue de -8.30 kcal/mol y -7.919 kcal/mol. En CDS, la puntuación es más baja que Remdesivir. Sin embargo, este valor no está lejos del control. En el caso de DT, los resultados son superiores a Remdesivir, indicando mejor energía de unión que el control con la proteína Mpro en este servidor. RdRp muestra una tendencia similar a la Luteolin 7-glucoside, con valores de CDS superiores a Remdesivir. Mientras que con DT se reportaron valores menores (Cuadro 1).
La información sobre Syringetin glucopyranoside es mínima; Syringetin es un flavonoide, específicamente un flavonol. Se ha descrito gran variedad de actividades biológicas en los flavonoides, como antiinflamatorio, antioxidante, antibacteriano, antiviral, anticancerígeno y neuroprotector (Brodowska, 2017; Zakaryan et al., 2017). También se ha informado actividad antiviral en flavonoides contra ciertos virus de ARN, como el virus respiratorio sincitial (RSV) o poliovirus, y virus de ADN como el virus del herpes simple (HSV-1) (Naithani et al., 2010). Los compuestos de Damiana (Turnera diffusa) como la Luteolin y el Syringetin glucopyranoside poseen actividad antiinflamatoria, antibacteriana, antioxidante y antiviral (Govea-Salas et al., 2017).
Se ha evaluado la actividad antiviral in vitro de algunos flavonoides, incluida la Syringetin, contra el virus respiratorio sincitial (RSV). Los resultados mostraron actividad antiviral de los flavonoides (Xu et al., 2020). Por lo tanto, es prometedor utilizar e investigar flavonoides y flavonoles como agentes antivirales. Este trabajo es el primero en investigar esta molécula, por lo que recomendamos realizar un estudio más extenso para comprobar su utilidad en ensayos posteriores.
Las propiedades de estos compuestos bioactivos tienden a influir en los resultados de las infecciones virales, ya que tienen efectos antioxidantes que pueden ayudar a reducir los niveles de estrés oxidativo. Sus efectos antiinflamatorios e inmunomoduladores pueden reducir en gran medida el daño causado por la infección viral.
Se ha demostrado que los polifenoles tienen una acción inhibitoria contra el SARS-CoV-2, el SARS-CoV y el MERS-CoV. Las interacciones de los polifenoles con las proteínas virales y los receptores de las células huésped pueden interferir con la entrada y la replicación del virus. Los estudios in vitro han demostrado que los polifenoles pueden interrumpir el ciclo viral al unirse a proteínas. Estas interacciones podrían inhibir proteínas, como Mpro, RdRp, o alterar la unión de proteínas estructurales, como la proteína S.Por ejemplo, la curcumina es una de las moléculas más estudiadas in silico e in vitro como potente inhibidor de Mpro de SARS-CoV-2. Concentraciones de curcumina superiores a 30 µg/mL pueden reducir la actividad del SARS-CoV-2 Mpro en más del 50%, mientras que la concentración más alta de 75 µg/mL produjo una actividad residual del 28.1% (Gligorijevic et al., 2021). También se ha demostrado que los flavonoides tienen el potencial de inhibir la actividad de las proteínas virales del SARS-CoV-2 (Benarba & Pandiella, 2020).
Se estima que la ingesta de polifenoles en la población general es de 0.9 g al día; después de la ingestión, solo el 5-10% de la ingesta total de polifenoles se absorbe en el intestino delgado, mientras que el 90-95% restante puede acumularse en la luz del intestino grueso hasta el rango milimolar (Gligorijevic et al., 2021).
Los polifenoles por administrados por vía oral deberían tener efectos beneficiosos en la prevención y el tratamiento de la COVID-19, al menos en el tracto gastrointestinal. Después de la ingestión, los polifenoles interactúan con las proteínas de la cavidad bucal. Por lo tanto, los polifenoles podrían inhibir la entrada y la replicación del SARS-CoV-2, lo que reduce el riesgo de infección por SARS-CoV-2. Además, se encontró una alta expresión del receptor ACE2 para SARS-CoV-2 en células epiteliales de la mucosa oral y en las células epiteliales de la lengua, por lo que se considera que la cavidad oral es de alto riesgo potencial para el SARS-CoV-2 (Gligorijevic et al., 2021).
Los estudios futuros sobre los efectos benéficos de las moléculas bioactivas como polifenoles, taninos y flavonoides en COVID-19 también deben considerar la biodisponibilidad de los polifenoles, sus metabolitos y sus concentraciones efectivas para inducir un efecto.
Interacciones complejo proteína-ligando
La proteína Mpro consiste en un homodímero con cada polipéptido compuesto por tres dominios: I (residuos 8-101), II (residuos 102-184) y III (residuos 201-303). El sitio de unión se encuentra en una hendidura entre los dominios I y II. Su centro de reacción es Cys145-His164, siguiendo un mecanismo similar al de otros coronavirus (Pavlova et al., 2021).
Nsp12 consta de un dominio RdRp (residuos Ser367 a Phe920) y un dominio de extensión N-terminal específico de nidovirus (residuos Asp60 a Arg249). Se pliega en tres subdominios, a saber, pulgar, palma y dedos. Además, Nsp12 de SARS-CoV-2 posee un dominio de horquilla β recientemente identificado en su extremo N (Guedes et al., 2021).
La unión de Remdesivir y RdRp se estabiliza en el sitio activo con cuatro tipos de enlaces, tres enlaces de hidrógeno con Arg475, Tyr539 y Asn611, un enlace C-H (carbono-hidrógeno) con Asp543, un enlace de catión pi con Lys471 y un enlace de alquilo con Ala608 (Figura 2a). Remdesivir interactúa con los residuos del sitio activo de Mpro a través de enlaces H (hidrógeno) con Ser1 y Thr280, enlaces C-H con residuos Gly2 y Leu282, un enlace alquilo Pi con Phe291 e interacciones de Van der Waals con nueve residuos como se observa en la Fig. 1a. Un enlace doble de catión Pi con el residuo Lys471, un enlace alquilo con Ala608, un enlace C-H con Asp543 y enlaces H con residuos Arg475, Tyr539 y Asn611 (Fig. 1a).
Además, la Luteolin 7-glucoside presenta enlaces H con los residuos Ser1, Phe3, Lys5, Arg4 y Asn214 cuando interactúa en el sitio activo de Mpro (Fig. 1b). En RdRp, la Luteolin 7-glucoside se une a través de enlaces H con los residuos Arg473, Arg475, Asp543, Ser602, Ser679, Asp680 y un doble enlace C-H con el residuo Asp543 (Fig. 2b).
Syringetin glucopyranoside interactúa con Mpro y RdRp en su sitio activo solo con enlaces H y C-H. Las interacciones con Mpro son enlaces H con residuos Lys5 y Asp216, enlaces C-H con Leu282 y Gly283, y residuos Phe3 formando ambos tipos de enlaces (Fig. 1c). El acoplamiento con RdRp muestra interacciones de enlaces C-H con residuos Asp543 y Asp680, y enlaces H con Ile468, Arg473, Arg475 (doble enlace), Cys542, Asp543, Asp681 y Ser734 (Fig. 2c).
Cypellocarpin B se une al sitio activo de la proteína Mpro a través de enlaces H con Arg4, Phe3 y Asp216, las interacciones de Van der Waals con Gly2 Asn214, Leu282, residuos Thr280 y enlaces C-H con Gly283 (Fig. 1d). Cypellocarpin B con RdRp a través de cuatro enlaces de hidrógeno convencionales con residuos Lys471, Arg475, Asp680 y Ser734, fuerzas de Van der Waals en residuos Arg473, Asn611, Ala608, Leu678, Tyr539, Trp537, Asp538 y enlaces catiónicos C-H y Pi con residuos Ser 679 y Asp681, respectivamente (Fig. 2d).
En Cypellocarpin C, las interacciones con la proteína Mpro fueron formadas por residuos Asp216 a través de enlaces H, el residuo Gly283 a través de enlaces C-H y Arg4, Thr280 y Leu282 formando interacciones de Van der Waals (Fig. 1e). La cipelocarpina C se estabiliza en el sitio activo de la proteína RdRp mediante enlaces H (Ser469, Arg475, Cys 542, Asp543), interacciones de Van der Waals (Arg756, Ala467, Ile 468, Ala470, His359, Ser734, Arg473, Lys541, Arg 544, Asp680) y un doble enlace (catión Pi y alquilo Pi) en el residuo Lys471 (Fig. 2e).
Aunque la mayoría de las interacciones muestran variaciones en los tipos de enlaces y residuos, RdRp cuando interactúa con Remdesivir, Luteolin 7-glucoside, Syringetin glucopyranoside y Cypellocarpin B y C muestra una interacción típica del tipo de enlace de hidrógeno con el residuo Arg475.
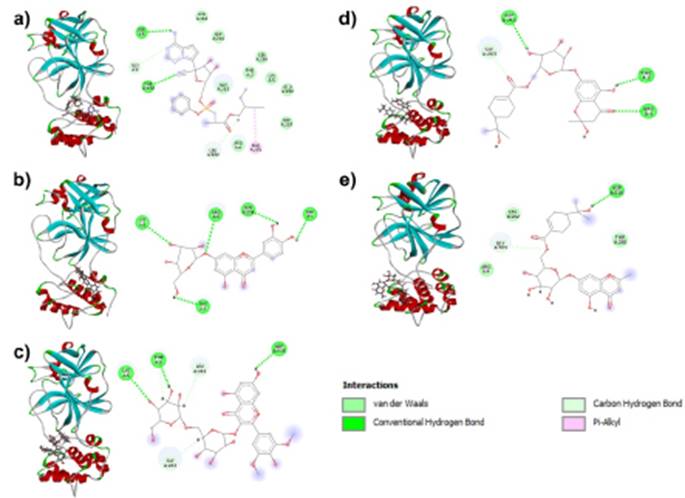
Fuente: Elaboración propia a partir de Discovery Studio Visualizer. Remdesivir, b) Luteolin 7-glucoside, c) Syringetin glucopyranoside, d) Cypellocarpin B, e) Cypellocarpin C. Se observan las interacciones de las moléculas con los residuos de aminoácidos de las proteínas.
Figura 1 Estructura tridimensional e interacciones 2D de SARS-CoV-2 Mpro.
Análisis de servidores de acoplamiento
Siete moléculas analizadas para Mpro por CDS (Remdesivir, Neochlorogenic acid, Syringetin glucopyranoside, Nordihydroguayretic acid, Cypellocarpin B, Cypellocarpin C y vitexin) mostraron un mayor valor energético, en comparación con DT. Al realizar el acoplamiento entre las moléculas y Mpro, DT mostró que cinco de los compuestos bioactivos tenían mayor energía de unión (-9.040, -8.191, -8.209, -8.100, -8.910 y -8.886 kcal/mol) que Remdesivir (-7.919 kcal/mol), mientras que en el servidor CDS, solo dos compuestos mostraron una energía de enlace superior (-9.00 y -8.60 kcal/mol) que el control (-8.30 kcal/mol). En cuanto a RdRp, todas las puntuaciones obtenidas por CDS fueron superiores a DT.
En el acoplamiento con RdRp, los resultados del servidor CDS mostraron que ocho de las moléculas tenían puntajes más altos (-9.30, -10.00, -10.60, -9.50, -9.30, -10.10, -9.80 y -9.60 kcal/mol) que Remdesivir (-9.20 kcal/mol). Por el contrario, el servidor DT presentó solo tres (-7.478, -7.542 y -7.427 kcal/mol) que fueron superiores a Remdesivir (-7.330 kcal/mol). Las diferencias en los valores de energía de enlace entre servidores pueden deberse principalmente a los diferentes algoritmos o motores de acoplamiento utilizados en cada servidor y las diferencias en las estructuras de proteínas virales predeterminadas en cada servidor.
Para el acoplamiento de moléculas pequeñas, CDS utiliza Autodock Vina como motor de acoplamiento (Kong et al., 2020). Se ha demostrado que existe una excelente concordancia entre las puntuaciones de acoplamiento obtenidas de CDS y el acoplamiento manual utilizando AutoDock Vina (Sen Gupta et al., 2020), lo que convierte a este servidor en una herramienta valiosa para analizar moléculas de interés contra el SARS-CoV-2. Sin embargo, se recomiendan estudios de confirmación in vitro o in vivo.
El servidor DT utiliza el programa DT como motor de acoplamiento. El servidor web utiliza las instalaciones informáticas de la plataforma brasileña de alto rendimiento (SINAPAD) y la supercomputadora SDumont (da Silveira et al., 2019; Guedes et al., 2021). DT ha sido evaluado con otros programas de acoplamiento molecular para la eficiencia de las funciones de puntuación (Rerank > MolDock > PLANTS > AutoDock Vina > DT) (González-Paz et al., 2020), concluyendo que DT es inferior en este aspecto a AutoDock Vina (el motor utilizado por CDS).
Además, existen diferencias a la hora de realizar el análisis; por ejemplo, el tamaño de la caja en CDS es de 30 x 30 x 30, y en DT es de 20 x 20 x 20. Además, en CDS se hacen 10 corridas, y la mejor corrida es la que se selecciona; en DT se realizan 24 corridas, y se toma el valor final.
Algunos estudios han obtenido resultados prometedores de DT al ser comparado con otros programas de acoplamiento como Glide, GOLD y AutoDock Vina (considerando varios objetivos moleculares y clases químicas de ligandos). Esto convierte a DT en un servidor prometedor para probar moléculas contra el SARS-CoV-2; sin embargo, al igual que con CDS, se recomiendan pruebas de confirmación in vitro o in vivo. Este servidor tiene un gran potencial para ser ampliamente utilizado en estudios de receptor-ligando (Santos et al., 2020).
En referencia a las proteínas de cada servidor, la proteína Mpro predeterminada en DT es un dímero de dos cadenas de aminoácidos homólogas (A y B). CDS usa una estructura monomérica para hacer el acoplamiento. Ambos servidores han usado la estructura reportada por Jin et al. (2020) (PDB 6LU7). Con RdRp; ambos servidores utilizan la estructura RdRp informada por Yin et al. (2020) (PDB 7BV2).
Se requiere un análisis completo de los servidores CDS y DT, así como de las proteínas que cada uno tiene para realizar el acoplamiento molecular. Sin embargo, pueden considerarse herramientas actuales para evaluar rápidamente moléculas con actividad potencial contra SARS-CoV-2.
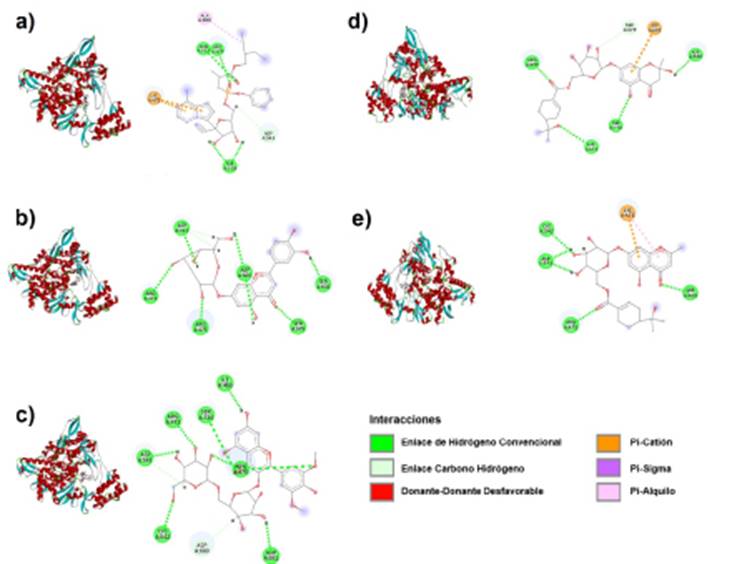
Fuente: Elaboración propia a partir de Discovery Studio Visualizer. a) Remdesivir, b) Luteolin 7-glucoside, c) Syringetin glucopyranoside, d) Cypellocarpin B, e) Cypellocarpin C. Se observan las interacciones de las moléculas con los residuos de aminoácidos de las proteínas.
Figura 2 Estructura tridimensional e interacciones 2D de la proteína RdRp del SARS-CoV-2.
Limitaciones del estudio
Los estudios in silico indican la interacción de una molécula o la energía de unión con la proteína de interés. Sin embargo, datos como la concentración que genera la interacción no pueden determinarse por este método, por lo que deben complementarse con experimentos in vitro e in vivo para verificar las actividades biológicas.
Las posibles variaciones genómicas en la región del sitio de unión de los objetivos moleculares pueden afectar drásticamente el modo de unión y la afinidad de los ligandos y, cambiar la identificación de compuestos prometedores. Es necesario demostrar la utilidad clínica de estas moléculas ya que los datos actuales aún son prematuros. Además, la eficacia y la seguridad deben estudiarse más a fondo in vivo y validarse en pacientes con COVID-19. También se debe considerar la biodisponibilidad, modos de administración, dosis seguras, tiempo de exposición y perfil farmacocinético de las moléculas.
Si bien los enfoques in silico no necesariamente garantizan un comportamiento antiviral, son el primer paso para futuros estudios in vitro e in vivo de moléculas que contienen actividades antivirales contra el SARS-CoV-2 y otros tipos de virus.
Conclusiones
Los resultados revelaron que cypellocarpin B, cypellocarpin C, luteolin 7-glucoside y syringetin glucopyranoside tienen las mejores afinidades con las proteínas del SARS-CoV-2, Mpro y RdRp, comparado con remdesivir. Los resultados del acoplamiento molecular confirman que las moléculas presentes en las plantas medicinales de México son opciones para el tratamiento contra el COVID-19. Sin embargo, se recomiendan estudios in vitro o in vivo para demostrar la seguridad y eficacia de estos compuestos. CDS y DT son herramientas útiles para evaluar moléculas con actividad potencial contra el SARS-CoV-2.
Abarca-Vargas, R. & Petricevich, V. L. (2018). Bougainvillea Genus: A Review on Phytochemistry, Pharmacology, and Toxicology. Evidence-Based Complementary and Alternative Medicine, 2018, 1-17. https://doi.org/10.1155/2018/9070927 [ Links ]
Aftab, S. O., Ghouri, M. Z., Masood, M. U., Haider, Z., Khan, Z., Ahmad, A. & Munawar, N. (2020). Analysis of SARS-CoV-2 RNA-dependent RNA polymerase as a potential therapeutic drug target using a computational approach. Journal of Translational Medicine, 18, 275. https://doi.org/10.1186/s12967-020-02439-0 [ Links ]
Beigel, J. H., Tomashek, K. M., Dodd, L. E., Mehta, A. K., Zingman, B. S., Kalil, A. C. ,Hohmann, E., Chu, H. Y., Luetkemeyer, A., Kline, S., Lopez de Castilla, D., Finberg, R. W., Dierberg, K., Tapson, V., Hsieh, L., Patterson, T. F., Paredes, R., Sweeney, D. A., Short, W. R., . . . Lane, H. C. (2020). Remdesivir for the Treatment of Covid-19 - Final Report. New England Journal of Medicine, 383(19), 1813-1826. https://doi.org/10.1056/nejmoa2007764 [ Links ]
Benarba, B. & Pandiella, A. (2020). Medicinal Plants as Sources of Active Molecules Against COVID-19. Frontiers in Pharmacology, 11,1189. https://doi.org/10.3389/fphar.2020.01189 [ Links ]
Benfenati, E., Gini, G., Hoffmann, S. & Luttik, R. (2010). Comparing In Vivo, In Vitro and In Silico Methods and Integrated Strategies for Chemical Assessment: Problems and Prospects. Alternatives to Laboratory Animals, 38(2), 153-166. https://doi.org/10.1177/026119291003800201 [ Links ]
Ben-Shabat, S., Yarmolinsky, L., Porat, D. & Dahan, A. (2019). Antiviral effect of phytochemicals from medicinal plants: Applications and drug delivery strategies. Drug Delivery and Translational Research, 10(2), 354-367. https://doi.org/10.1007/s13346-019-00691-6 [ Links ]
Brezáni, V. & Karel, Š. (2013) Secondary metabolites isolated from the genus Eucalyptus. Current Trends in Medicinal Chemistry, 7, 65-95. http://www.researchtrends.net/tia/article_pdf.asp?aid=5301&in=0&tid=61&vn=7 [ Links ]
Bright, K. R. & Gilling, D. H. (2016). Natural Virucidal Compounds in Foods. Viruses in Foods, 449-469. https://doi.org/10.1007/978-3-319-30723-7_16 [ Links ]
Brodowska, K. (2017). Natural flavonoids: classification, potential role, and application of flavonoid analogues. European Journal of Biological Research, 7(2), 108-123. http://dx.doi.org/10.5281/zenodo.545778 [ Links ]
Da Silva, F. M. A., da Silva, K. P. A., de Oliveira, L. P. M., Costa, E. V., Koolen, H. H., Pinheiro, M. L. B., de Souza, A. Q. L. & de Souza, A. D. L. (2020). Flavonoid glycosides and their putative human metabolites as potential inhibitors of the SARS-CoV-2 main protease (Mpro) and RNA-dependent RNA polymerase (RdRp). Memórias do Instituto Oswaldo Cruz, 115, e200207. https://doi.org/10.1590/0074-02760200207 [ Links ]
Da Silveira, N. J. F., Pereira, F. S. S., Elias, T. C. & Henrique, T. (2019). Web Services for Molecular Docking Simulations. Methods in Molecular Biology, 2053, 221-229. https://doi.org/10.1007/978-1-4939-9752-7_14 [ Links ]
Díaz, K., Espinoza, L., Madrid, A., Pizarro, L. & Chamy, R. (2018). Isolation and Identification of Compounds from Bioactive Extracts of Taraxacum officinale Weber ex F. H. Wigg. (Dandelion) as a Potential Source of Antibacterial Agents. Evidence-Based Complementary and Alternative Medicine , 2018, 2706417. https://doi.org/10.1155/2018/2706417 [ Links ]
Eastman, R. T., Roth, J. S., Brimacombe, K. R., Simeonov, A., Shen, M., Patnaik, S. & Hall, M. D. (2020). Remdesivir: A Review of Its Discovery and Development Leading to Emergency Use Authorization for Treatment of COVID-19. ACS Central Science, 6(5), 672-683. https://doi.org/10.1021/acscentsci.0c00489 [ Links ]
Fan, W., Qian, S., Qian, P. & Li, X. (2016). Antiviral activity of luteolin against Japanese encephalitis virus. Virus Research, 220, 112-116. https://doi.org/10.1016/j.virusres.2016.04.021 [ Links ]
Food and Drug Administration [FDA]. (2020) FDA Approves First Treatment for COVID-19, FDA Approves First Treatment for COVID-19. Available at: https://www.fda.gov/news-events/press-announcements/fda-approves-first-treatment-covid-19 [ Links ]
Fitriani, I.N.,Utami, W., Zikri, A.T., & Santoso, P. (2020). In Silico Approach of Potential Phytochemical Inhibitor from Moringa oleifera, Cocos nucifera, Allium cepa, Psidium guajava, and Eucalyptus globulus for the Treatment of COVID-19 by Molecular Docking. Res. Sq. https://doi.org/10.21203/rs.3.rs-42747/v1 [ Links ]
Giguet-Valard, A. G., Raguette, K., Morin, S., Bellance, R. & Ravin, J. S. (2020). Gossypetin Derivatives are also Putative Inhibitors of SARS-COV 2: Results of a Computational Study. Journal of Biomedical Research & Environmental Sciences, 1(6), 201-212. https://doi.org/10.37871/jbres1144 [ Links ]
Gligorijevic, N., Radomirovic, M., Nedic, O., Stojadinovic, M., Khulal, U., Stanic-Vucinic, D. & Cirkovic Velickovic, T. (2021). Molecular Mechanisms of Possible Action of Phenolic Compounds in COVID-19 Protection and Prevention. International Journal of Molecular Sciences, 22(22), 12385. https://doi.org/10.3390/ijms222212385 [ Links ]
González-Paz, L., Paz, J. L., Vera-Villalobos, J. & Alvarado, Y. J. (2020). Compuestos Fitoquímicos Dirigidos al Bloqueo de la Polimerasa Viral del SARS-CoV-2 Causante del COVID-19: un Análisis Comparativo de Funciones de Puntuación para Acoplamientos con Interés Biomédico. Revista Politécnica, 46(1), 7-20. https://doi.org/10.33333/rp.vol46n1.01 [ Links ]
Goodger, J. Q. & Woodrow, I. E. (2013). Oleuropeic and Menthiafolic Acid Glucose Esters from Plants. Studies in Natural Products Chemistry, 40, 427-452. https://doi.org/10.1016/b978-0-444-59603-1.00012-6 [ Links ]
Govea-Salas, M., Morlett-Chávez, J., Rodriguez-Herrera, R. & Ascacio-Valdés, J. (2017). Some Mexican Plants Used in Traditional Medicine. Aromatic and Medicinal Plants - Back to Nature. https://doi.org/10.5772/66637 [ Links ]
Guedes, I. A., Costa, L. S. C., dos Santos, K. B., Karl, A. L. M., Rocha, G. K., Teixeira, I. M., Galheigo, M. M., Medeiros, V., Krempser, E., Custódio, F. L., Barbosa, H. J. C., Nicolás, M. F. & Dardenne, L. E. (2021). Drug design and repurposing with DockThor-VS web server focusing on SARS-CoV-2 therapeutic targets and their non-synonym variants. Scientific Reports, 11(1). https://doi.org/10.1038/s41598-021-84700-0 [ Links ]
Hakki, Z., Cao, B., Heskes, A. M., Goodger, J. Q., Woodrow, I. E. & Williams, S. J. (2010). Synthesis of the monoterpenoid esters cypellocarpin C and cuniloside B and evidence for their widespread occurrence in Eucalyptus. Carbohydrate Research, 345(14), 2079-2084. https://doi.org/10.1016/j.carres.2010.07.029 [ Links ]
Jin, Z., Du, X., Xu, Y., Deng, Y., Liu, M., Zhao, Y., Zhang, B., Li, X. , Zhang, L., Peng, C., Duan, Y., Yu, J., Wang, L., Yang, K., Liu, F., Jiang, R., Yang, X., You, T., Liu, X., . . . Yang, H. (2020). Structure of Mpro from SARS-CoV-2 and discovery of its inhibitors. Nature, 582(7811), 289-293. https://doi.org/10.1038/s41586-020-2223-y [ Links ]
Khaerunnisa, S., Kurniawan, H., Awaluddin, R., Suhartati, S., Soetjipto, S. (2020). Potential Inhibitor of COVID-19 Main Protease (Mpro) From Several Medicinal Plant Compounds by Molecular Docking Study. Preprints, 2020030226. http://dx.doi.org/10.20944/preprints202003.0226.v1 [ Links ]
Khalifa, I., Zhu, W., Mohammed, H. H. H., Dutta, K. & Li, C. (2020). Tannins inhibit SARS-CoV-2 through binding with catalytic dyad residues of 3CLpro: An in silico approach with 19 structural different hydrolysable tannins. Journal of Food Biochemistry, 44(10), e13432. https://doi.org/10.1111/jfbc.13432 [ Links ]
Ko, W. C., Rolain, J. M., Lee, N. Y., Chen, P. L., Huang, C. T., Lee, P. I. & Hsueh, P. R. (2020). Arguments in favour of remdesivir for treating SARS-CoV-2 infections. International Journal of Antimicrobial Agents, 55(4), 105933. https://doi.org/10.1016/j.ijantimicag.2020.105933 [ Links ]
Kong, R., Yang, G., Xue, R., Liu, M. , Wang, F., Hu, J., Guo, X. & Chang, S. (2020). COVID-19 Docking Server: a meta server for docking small molecules, peptides, and antibodies against potential targets of COVID-19. Bioinformatics, 36(20), 5109-5111. https://doi.org/10.1093/bioinformatics/btaa645 [ Links ]
Ma, Y. L., Sun, P., Feng, J., Yuan, J., Wang, Y., Shang, Y. F., Niu, X. L., Yang, S. H. & Wei, Z. J. (2021). Solvent effect on phenolics and antioxidant activity of Huangshan Gongju (Dendranthema morifolium (Ramat) Tzvel. cv. Gongju) extract. Food and Chemical Toxicology, 147, 111875. https://doi.org/10.1016/j.fct.2020.111875 [ Links ]
Malin, J. J., Suárez, I., Priesner, V., Fätkenheuer, G. & Rybniker, J. (2020). Remdesivir against COVID-19 and Other Viral Diseases. Clinical Microbiology Reviews, 34(1). https://doi.org/10.1128/cmr.00162-20 [ Links ]
Mehla, R., Bivalkar-Mehla, S. & Chauhan, A. (2011). A Flavonoid, Luteolin, Cripples HIV-1 by Abrogation of Tat Function. PLoS ONE, 6(11), e27915. https://doi.org/10.1371/journal.pone.0027915 [ Links ]
Mukhtar, M., Arshad, M., Ahmad, M., Pomerantz, R. J., Wigdahl, B. & Parveen, Z. (2008). Antiviral potentials of medicinal plants. Virus Research , 131(2), 111-120. https://doi.org/10.1016/j.virusres.2007.09.008 [ Links ]
Moneriz, C., & Castro-Salguedo, C. (2020). Promising and potential drugs for the treatment of COVID-19. Revista chilena de infectología, 37(3), 205-215. https://doi.org/10.4067/s0716-10182020000300205 [ Links ]
Naithani, R., Mehta, R. G., Shukla, D., Chandersekera, S. N. & Moriarty, R. M. (2010). Antiviral Activity of Phytochemicals: A Current Perspective. Dietary Components and Immune Function, 421-468. https://doi.org/10.1007/978-1-60761-061-8_24 [ Links ]
Nguyen, H. L., Thai, N. Q., Truong, D. T. & Li, M. S. (2020). Remdesivir Strongly Binds to Both RNA-Dependent RNA Polymerase and Main Protease of SARS-CoV-2: Evidence from Molecular Simulations. The Journal of Physical Chemistry B, 124(50), 11337-11348. https://doi.org/10.1021/acs.jpcb.0c07312 [ Links ]
Paraiso, I. L., Revel, J. S. & Stevens, J. F. (2020). Potential use of polyphenols in the battle against COVID-19. Current Opinion in Food Science, 32, 149-155. https://doi.org/10.1016/j.cofs.2020.08.004 [ Links ]
Pavlova, A., Lynch, D. L., Daidone, I., Zanetti-Polzi, L., Smith, M. D., Chipot, C., Kneller, D. W., Kovalevsky, A., Coates, L., Golosov, A. A., Dickson, C. J., Velez-Vega, C., Duca, J. S., Vermaas, J. V., Pang, Y. T., Acharya, A., Parks, J. M., Smith, J. C. & Gumbart, J. C. (2021). Inhibitor binding influences the protonation states of histidines in SARS-CoV-2 main protease. Chemical Science, 12(4), 1513-1527. https://doi.org/10.1039/d0sc04942e [ Links ]
Piluzza, G., Campesi, G., Molinu, M. G., Re, G. A. & Sulas, L. (2020). Bioactive Compounds from Leaves and Twigs of Guayule Grown in a Mediterranean Environment. Plants, 9(4), 442. hts://doi.org/10.3390/plants9040442 [ Links ]
Santos, K. B., Guedes, I. A. , Karl, A. L. M. & Dardenne, L. E. (2020). Highly Flexible Ligand Docking: Benchmarking of the DockThor Program on the LEADS-PEP Protein-Peptide Data Set. Journal of Chemical Information and Modeling, 60(2), 667-683. https://doi.org/10.1021/acs.jcim.9b00905 [ Links ]
Saucedo-Pompa, S., Torres-Castillo, J., Castro-López, C., Rojas, R., Sánchez-Alejo, E., Ngangyo-Heya, M. & Martínez-Ávila, G. (2018). Moringa plants: Bioactive compounds and promising applications in food products. Food Research International, 111, 438-450. https://doi.org/10.1016/j.foodres.2018.05.062 [ Links ]
Sen, D., Debnath, P., Debnath, B., Bhaumik, S. & Debnath, S. (2020). Identification of potential inhibitors of SARS-CoV-2 main protease and spike receptor from 10 important spices through structure-based virtual screening and molecular dynamic study. Journal of Biomolecular Structure and Dynamics, 40(2), 941-962. https://doi.org/10.1080/07391102.2020.1819883 [ Links ]
Sen Gupta, P. S., Biswal, S., Panda, S. K., Ray, A. K. & Rana, M. K. (2020). Binding mechanism and structural insights into the identified protein target of COVID-19 and importin-α with in-vitro effective drug ivermectin. Journal of Biomolecular Structure and Dynamics , 40(5), 2217-2226. https://doi.org/10.1080/07391102.2020.1839564 [ Links ]
Singh, S., Sk, M. F., Sonawane, A., Kar, P. & Sadhukhan, S. (2020). Plant-derived natural polyphenols as potential antiviral drugs against SARS-CoV-2 via RNA-dependent RNA polymerase (RdRp) inhibition: an in-silico analysis. Journal of Biomolecular Structure and Dynamics , 39(16), 6249-6264. https://doi.org/10.1080/07391102.2020.1796810 [ Links ]
Tallei, T. E., Tumilaar, S. G., Niode, N. J., Fatimawali, Kepel, B. J., Idroes, R., Effendi, Y., Sakib, S. A. & Emran, T. B. (2020).Potential of Plant Bioactive Compounds as SARS-CoV-2 Main Protease (Mpro) and Spike (S) Glycoprotein Inhibitors: A Molecular Docking Study. Scientifica, 2020, 6307457. https://doi.org/10.1155/2020/6307457 [ Links ]
Torres-León, C., Aguirre-Joya, J. A., Czaja, A., & Aguillón-Gutiérrez, D. R. (2020). In silico Screening bioaktiver Verbindungen aus mexikanischen Wüstenpflanzen zur Vorhersage potenzieller Inhibitoren von SARSCoronavirus 2 (SARS-CoV-2). Z Arznei-Gewurzpfla, 2(4), 153-156. https://www.researchgate.net/publication/348977122_In_silico_Screening_bioaktiver_Verbindungen_aus_mexikanischen_Wustenpflanzen_zur_Vorhersage_potenzieller_Inhibitoren_von_SARSCoronavirus_2_SARS-CoV-2 [ Links ]
Torres-León, C., Ventura-Sobrevilla, J., Serna-Cock, L., Ascacio-Valdés, J. A., Contreras-Esquivel, J. & Aguilar, C. N. (2017). Pentagalloylglucose (PGG): A valuable phenolic compound with functional properties. Journal of Functional Foods, 37, 176-189. https://doi.org/10.1016/j.jff.2017.07.045 [ Links ]
Treml, J., Gazdová, M., Šmejkal, K., Šudomová, M., Kubatka, P. & Hassan, S. T. S. (2020). Natural Products-Derived Chemicals: Breaking Barriers to Novel Anti-HSV Drug Development. Viruses, 12(2), 154. https://doi.org/10.3390/v12020154 [ Links ]
Wang, Y. , Zhang, D., Du, G., Du, R., Zhao, J., Jin, Y., Fu, S., Gao, L., Cheng, Z., Lu, Q., Hu, Y., Luo, G., Wang, K., Lu, Y., Li, H., Wang, S., Ruan, S., Yang, C., Mei, C., . . . Wang, C. (2020). Remdesivir in adults with severe COVID-19: a randomised, double-blind, placebo-controlled, multicentre trial. The Lancet, 395(10236), 1569-1578. https://doi.org/10.1016/s0140-6736(20)31022-9 [ Links ]
Xu, F., Gao, M., Li, H. , Han, X., Zhang, X., Li, Y., Guo, D. A. & Liu, B. (2020). Three new bisflavonols from the seeds of Hovenia dulcis Thunb. and their anti-RSV activities. Fitoterapia, 143, 104587. https://doi.org/10.1016/j.fitote.2020.104587 [ Links ]
Yang, Y., Xiao, Z., Ye, K., He, X., Sun, B., Qin, Z., Yu, J. , Yao, J., Wu, Q., Bao, Z. & Zhao, W. (2020). SARS-CoV-2: characteristics and current advances in research. Virology Journal, 17(1). https://doi.org/10.1186/s12985-020-01369-z [ Links ]
Yi, L., Li, Z., Yuan, K., Qu, X., Chen, J., Wang, G., Zhang, H., Luo, H., Zhu, L., Jiang, P., Chen, L., Shen, Y., Luo, M., Zuo, G., Hu, J., Duan, D., Nie, Y., Shi, X., Wang, W., . . . Xu, X. (2004). Small Molecules Blocking the Entry of Severe Acute Respiratory Syndrome Coronavirus into Host Cells. Journal of Virology, 78(20), 11334-11339. https://doi.org/10.1128/jvi.78.20.11334-11339.2004 [ Links ]
Yin, W., Mao, C., Luan, X., Shen, D. D., Shen, Q., Su, H., Wang, X., Zhou, F., Zhao, W., Gao, M., Chang, S., Xie, Y. C., Tian, G., Jiang, H. W., Tao, S. C., Shen, J., Jiang, Y., Jiang, H., Xu, Y. , . . . Xu, H. E. (2020). Structural basis for inhibition of the RNA-dependent RNA polymerase from SARS-CoV-2 by remdesivir. Science,368(6498), 1499-1504. https://doi.org/10.1126/science.abc1560 [ Links ]
Yoshimoto, F. K. (2020). The Proteins of Severe Acute Respiratory Syndrome Coronavirus-2 (SARS CoV-2 or n-COV19), the Cause of COVID-19. The Protein Journal, 39, 198-216. https://doi.org/10.1007/s10930-020-09901-4 [ Links ]
Zakaryan, H., Arabyan, E., Oo, A. & Zandi, K. (2017). Flavonoids: promising natural compounds against viral infections. Archives of Virology, 162(9), 2539-2551. https://doi.org/10.1007/s00705-017-3417-y [ Links ]
Žemlička, L., Fodran, P., Lukeš, V., Vagánek, A., Slováková, M., Staško, A., Dubaj, T., Liptaj, T., Karabín, M., Birošová, L. & Rapta, P. (2014). Physicochemical and biological properties of luteolin-7-O-β-d-glucoside (cynaroside) isolated from Anthriscus sylvestris (L.) Hoffm. Monatshefte für Chemie - Chemical Monthly, 145, 1307-1318. https://doi.org/10.1007/s00706-014-1228-3 [ Links ]
Recibido: 13 de Julio de 2022; Aprobado: 29 de Diciembre de 2022; Publicado: 10 de Enero de 2023











 text in
text in 



