Los cultivos agrícolas están expuestos a factores abióticos y bióticos que pueden incidir negativamente en su productividad, i.e. inundaciones, sequías, suelos degradados y fitopatógenos. Estos últimos causan una disminución del rendimiento agrícola hasta del 40% (Valenzuela-Ruiz et al., 2020). Aunado a esto, el aumento poblacional y -en consecuencia- la creciente demanda de alimentos condujeron a la modificación de las prácticas agrícolas para mitigar las pérdidas ocasionadas por la incidencia de fitopatógenos, sin disminuir la calidad nutricional y la inocuidad de los alimentos producidos (FAO, 2018). La estrategia ampliamente utilizada para atender estos aspectos ha sido la aplicación de plaguicidas sintéticos, la cual aumentó mundialmente un 97.7% de 1992 (1.30 kg ha-1) a 2016 (2.57 kg ha-1) (FAO, 2018); mientras que en México el incremento de fungicidas sintéticos fue 76.9% (de 1.08 a 1.91 kg ha-1) en el mismo periodo (Roser, 2019). Se ha reportado que sólo el 0.1% de los plaguicidas sintéticos aplicados llega al cultivo agrícola de interés (Gill y Garg, 2014), lo cual genera graves afectaciones económicas y ambientales, como la contaminación de suelos y mantos acuíferos con residuos recalcitrantes, la degradación química y microbiana del suelo y la pérdida de su biodiversidad (de los Santos-Villalobos et al., 2018).
En este contexto, el manejo de fitopatógenos en los sistemas de producción agrícola debe ser atendido amplia e integralmente en congruencia con el objetivo dos, poner fin al hambre, de la Agenda 2030 para el Desarrollo Sostenible para América Latina y el Caribe. En éste se establece la necesidad de centrar esfuerzos para lograr la seguridad alimentaria, mejorar la nutrición y promover una agricultura sostenible (ONU, 2018). México ha coadyuvado a estos objetivos colateralmente ya que desde 1940 se iniciaron los primeros estudios y aplicaciones del control biológico y se crearon programas académicos con el fin de promover el manejo integrado fitosanitario (Gutiérrez-Samperio, J. Comunicación Personal. 2019). El manejo biológico de fitopatógenos es definido como “el uso de organismos benéficos para reducir los efectos negativos de patógenos de las plantas, a través de sus acciones antagónicas, ya sea por acción directa de reconocimiento génico, o indirectamente mediante metabolitos o productos derivados de éstos” (Valenzuela-Ruiz et al., 2020). En una concepción amplia, el manejo biológico se incentivó inicialmente con campañas implementadas por instancias gubernamentales, mediante el uso de insectos depredadores, parasitoides o insectos estériles; y posteriormente, con la inclusión de hongos entomopatógenos (Beauveria bassiana y Metarhizium), hongos filamentosos y bacterias, para el control de plagas de importancia nacional (Arredondo-Bernal y Rodríguez-del Bosque, 2015).
El uso de agentes de control biológico de origen microbiano (ACB-M) en México es una alternativa que ha sido utilizada para complementar el control convencional de fitopatógenos, mediante plaguicidas sintéticos (Arredondo-Bernal y Rodríguez-del Bosque, 2015). Por ejemplo, en 2017 en México, el área de siembra fue aproximadamente de 32.4 millones de hectáreas, cuyos principales cultivos fueron: maíz, sorgo, frijol, café, caña de azúcar y trigo (INEGI, 2017); en dichos cultivos los principales géneros de fitopatógenos reportados fueron Fusarium, Colletotrichum, Leveillula, Botrytis, Rhizoctonia, Pythium, Phytophthora, Pseudomonas, Candidatus Liberibacter asiaticus, Leifsonia y Xanthomonas, y los ACB-M más utilizados para controlar a estos fitopatógenos fueron las especies fúngicas Trichoderma harzianum, Paecilomyces lilacinus, P. fumosoroseus y Beauveria bassiana, y las especies bacterianas Bacillus subtilis, B. pumilus, B. amyloliquefaciens, B. licheniformis y B. thuringiensis (García-Juárez et al., 2016; Villarreal-Delgado et al., 2017; Delgado-Ortiz et al., 2019). Aunque la investigación en ACB es significativa (Cuadro 1 y 2), en ocasiones, la información detallada sobre los resultados obtenidos por la aplicación en campo de estos ACB a cultivos agrícolas de importancia nacional no es publicada o se registra en informes técnicos con acceso restringido, y se limita a describir aumentos de rendimientos agrícolas y/o inhibición de fitopatógenos.
El descubrimiento de nuevos agentes y/o el entendimiento de las interacciones planta-patógeno-ACB de origen microbiano (bajo condiciones de campo), así como estudios robustos sobre la afiliación taxonómica polifacética (clasificación consenso basada en la integración de todos los datos fenotípicos y genotípicos disponibles), los mecanismos de acción y ecología de dichos ACB en los agroecosistemas, mediante el empleo de los avances recientes de las ciencias ómicas, es determinante para el registro, comercialización, innovación y éxito de bioplaguicidas formulados. Por lo anterior, el objetivo de la presente revisión es describir el estado del conocimiento y analizar, en el marco de la celebración internacional de la Sanidad Vegetal, aspectos que limitan la bioprospección y uso extensivo de ACB-M en México, así como estrategias genómicas de vanguardia inherentes a las ciencias ómicas enfocadas a la identificación, selección y estudio de los mecanismos de acción de dichos agentes, así como la difusión y socialización del conocimiento científico generado para potenciar la innovación agro-biotecnológica y el éxito de bioplaguicidas.
Agentes de control biológico de origen microbiano (ACB-M) y bioplaguicidas en México. La búsqueda e introducción de enemigos naturales a fitopatógenos de importancia económica en México tomó relevancia a partir de la década de 1940, debido a la necesidad de generar estrategias de manejo eficientes y sostenibles. En más de 80 años del empleo de ACB-M en nuestro país (Bernal y Quezada, 1999), en el mercado nacional se encuentran disponibles 230 bioproductos de 40 empresas con registro ante COFEPRIS (https://www.gob.mx/cofepris). Sin embargo, entre estos productos existe un número limitado de ingredientes activos a nivel de grupo taxonómico (hongos, bacterias y/o virus), género o especie microbiana para el control de agentes causales de enfermedades de plantas cultivadas en México (Cuadro 1). Lo anterior refleja una limitada disponibilidad de bioproductos para su uso en el campo mexicano a pesar de los esfuerzos e investigaciones realizadas en nuestro país. Esto podría estar asociado al limitado apoyo otorgado al sector científico para la investigación sistémica y bioprospección de nuevos ACB-M; para el registro de patentes en el área biotecnológica, así como a la escasa estructura organizacional a nivel institucional para establecer departamentos especializados en el proceso de patentes y licenciamiento. Los escasos recursos financieros muchas veces han implicado el desarrollo de investigaciones parciales, principalmente in vitro, a partir de colecciones con limitada representatividad biológica y regional. Por otra parte, no se cuenta con acceso a la información documental que soporte científicamente su efectividad, y se tiene una deficiencia de datos estadísticos relativos a su producción, consumo y éxito a nivel nacional (De León y Mier, 2010); a excepción de productos como Fungifree®, el cual es un biofungicida efectivo para el manejo de enfermedades foliares como la antracnosis (C. gloeosporioides, C. acutatum y C. fragariae), cenicilla polvorienta (L. taurica, E. chichoracearum y S. humili) y moho gris (B. cinerea) (Galindo et al., 2015), y los trabajos realizado por el Centro Nacional de Referencia de Control Biológico SAGARPA-DGSV para el manejo de Diaphorina citri y langosta con hongos entomopatógenos (Ayala-Zermeño et al., 2015; Gallou et al., 2016).
Cuadro 1. Productos de control biológico de origen microbiano en México con registro ante COFEPRIS (https://www.gob.mx/cofepris).
| Ingrediente Activo | Producto comercial | Uso agrícola |
|---|---|---|
| Bacterias | ||
| Acetobacter lovaniensis | Q-Bacter | Bio-bactericida |
| Bacillus amyloliquefaciens | Serifel/Serenade Optimum / Stargus | Bio-fungicida |
| B. subtilis | Fungifree-Ab/ Agrobacilo/ Serenade As/ Serenade Max/ Bacillios G / Baktillis / Serenade/ Rador/ Vici Active/ Fungi-Q/ Fungisei/ Protec-Root/ Cx-9030 | Bio-fungicida |
| B. stearothermophilus | Bacillus 1537 / Labsa Bst / Bacillus Thermo | Bio-fungicida |
| B. thurigiensis | Dipel 2x /Xentari Grd/ Delta Bt/ Bt-K/ Dipel-2x/ Phc Beretta/ Larvix/ Thuricide Ph/ Newbt-2x Wp/ Mvp/ Xentari/ Lepinox 15 Wdg/ Crymax Gda/ Phc Condor Wp/ Dipel 2x/ Turinsil | Bio-insecticida |
| B. pumilus | Sonata As | Bio-fungicida |
| Chromobacterium subtsugae | Grandevo | Bio-insecticida |
| Streptomyces spp. | Blite Free | Bio-fungicida |
| Hongos | ||
| Beauveria bassiana | Bea-Sin/ Atento/ Bioin Dalife/Cercon Es/ Naturalis L | Bio-insecticida |
| Metarhizium anisopliae | Meta-Sin/ Biomett/ Meta-Noc/ Spectrum Meta | Bio-insecticida |
| Myrothecium verrucaria | Ditera Es/ Ditera Df | Bio-nematicida |
| Paecilomyces fumosoroseus | Pae-Sin/ Bioamin-Insect-1/ Pfr-97 20% Wdg/ Nofly Wp | Bio-insecticida |
| P. lilacinus | Lila-Sin / Chimal/ Bioact Wg/ Biostat/ Nemafin/ Spectrum Pae | Bio-nematicida |
| P. variotii | Nemaquim / Nemakill / Pha.Da / Nem P.B. / Sinnemakill / Nemphada / O.B Golf | Bio-nematicida |
| Pochonia chlamydosporia | Genexis Ph | Bio-nematicida |
| Trichoderma harzianum | Tricho-Sin/ Labrador/ Trianum P/ Phc T22/ Spectrum Trico-Bio/ Bioamin-Fung-2/ Rootshield Plus Wp | Bio-fungicida |
| T. lignorum | Mycobac | Bio-fungicida |
| T. viride | Funqui | Bio-fungicida |
| Verticillium lecanii | Verti-Sin/ Eday | Bio-insecticida |
| Mezclas | ||
| Bacillus subtilis, Rhodotorula minuta | Fungifree-Ab Plus | Bio-fungicida |
| B. subtilis, Bacillus spp. Torulopsis inconspicua | Obiettivo | Bio-fungicida |
| B. licheniformis, T. harzianum | Biowall | Bio-fungicida |
| Bacillus spp., aceite de clavo y neem | Roya Out | Bio-fungicida |
| B. subtilis, Trichoderma spp., P. lilacinus, quitosano, bicarbonato de potasio | Nemaxxion Biol | Bio-fungicida |
| B. bassiana, Nomurea rileyi , aceitedeneem, B. thuringiensis var. Kurstaki y var. Israelensis | Larbiol 2X | Bio-insecticida |
| B. bassiana, Nomuraea rileyi, Metarhizium anisopliae, Verticillium lecanii, P. fumosoroseus, concentrado oleico | Biomaxx Duo | Bio-insecticida |
| T. harzianum, T. viride, T. fasciculatum | Juq | Bio-fungicida |
| P. fumosoroseus, M. anisopliae, B. bassiana | Tri-Sin | Bio-insecticida |
| M. anisopliae, B. bassiana | Biomabb/ QUIM | Bio-insecticida |
| P. lilacinus, B. firmus | Arrecife | Bio-nematicida |
| P. lilacinus, B. subtilis, Pseudomonas fluorescens, extracto de ruezno de nuez, Yucca schidigera, guiche de lechuguilla, cáscara de nuez, higuerilla, gel de nopal, chitosan | Nemmax | Bio-nematicida |
Además, una de las principales limitantes para la transferencia de tecnología y uso extensivo de bioproductos a base de ACB-M es la insuficiente publicación y difusión de las investigaciones científicas desarrolladas por diversas universidades y centros de investigación del país, aún posterior al registro de la patente en cuestión (Bernal y Quezada, 1999). Al respecto, es de reconocer el importante papel de la Revista Mexicana de Fitopatología (https://www.rmf.smf.org.mx/), ya que en los últimos ocho años diversos investigadores han publicado alrededor de 50 contribuciones (23.7% de su publicación total) sobre aspectos relacionados al uso de ACB-M, i.e. artículos científicos, artículos de revisión, notas fitopatológicas y reportes fitopatológicos (Cuadro 2). Así mismo, se ha registrado por Scielo Analytics (https://analytics.scielo.org/) que de los 100 artículos de la RMF con más consultas en línea, el 24% corresponden a temas relacionados con ACB-M, entre los que destacan: El género Bacillus como agente de control biológico y sus implicaciones en la bioseguridad agrícola (Villarreal-Delgado et al., 2017), el modo de acción de Candida oleophila para el manejo de Penicillium expansum y Botrytis cinerea (Guerrero et al., 2011), y el biocontrol de la “escoba de bruja” con Trichoderma spp. bajo condiciones de campo en mango (Michel-Aceves et al., 2013), por mencionar algunos ejemplos.
En este sentido, con la finalidad de potenciar la investigación científica y enriquecer el espectro funcional de bioproductos registrados y comercializados en México, es determinante la incorporación de herramientas de vanguardia y robustas en estudios científicos sobre ACB-M, especialmente su afiliación taxonómica polifacética, fisiología, modo de acción in vitro, invernadero y campo, e interacciones con los factores bióticos y abióticos. Lo anterior conduciría al desarrollo de sistemas de producción, formulación, escalamiento e innovación costo-efectivos (Galindo et al., 2013; Serrano-Carreón et al., 2010). Esto permitiría a dependencias gubernamentales y profesionistas involucrados en la producción agrícola una transferencia tecnológica eficiente, segura y rigurosa de estos bioproductos a los agricultores.
Cuadro 2 Investigaciones publicadas en la Revista Mexicana de Fitopatología (https://www.rmf.smf.org.mx/) enfocadas al control biológico. Periodo: 2012 al 2020.
| Agente de control biológico | Análisis empleado para identificación | Cultivo | Agente fitopatógeno | Condiciones de evaluación | Mecanismo de acción propuesto | Herramienta empleada para detección de mecanismo de acción | Referencias |
|---|---|---|---|---|---|---|---|
| Bacillus y Burkholderia | Secuenciación del gen 16S ARNr | - | Colletotrichum gloeosporioides | In vitro | Se propone la excreción de metabolitos secundarios | Confrontación dual, estimación cualitativa con escala de 4 niveles de antagonismo y cuantitativo mediante análisis de imagen | Macedo-Castillo et al., 2012 |
| Paecilomyces lilacinus | Claves taxonómicas | Solanum tuberosum | Nematodos de vida libre | In vitro | Parasitismo | Potencial parasítico | Carrión y Desgarennes, 2012 |
| Pochonia hlamydosporia | Claves taxonómicas | Phaseolus vulgaris | Nacobbus aberrans | In vitro einvernadero | Parasitismo | Potencial parasitismo, colonización de raíces y prueba de efectividad | Franco-Navarro et al., 2013 |
| Trichoderma | Claves taxonómicas | Mangifera indica | Fusarium oxysporum y F. subglutinans | Campo | Parasitismo | Severidad de la enfermedad | Michel-Aceves et al., 2013 |
| Bacillus, Pseudomonas, Trichoderma y Penicillium | Claves taxonómicas, medios selectivos y pruebas bioquímicas | - | Rhizoctonia solani y F. oxysporum f sp. lycopersi | In vitro | Antibiosis, parasitismo | Confrontación en placa y suelo estéril | Rodríguez- Millán et al., 2013 |
| Trichoderma y Bacillus | Claves taxonómicas | Agave tequilana weber var. Azul | Fusarium | Vivero | Parasitismo, resistencia sistémica, competencia, producción de enzimas líticas | Potencial parasitismo, colonización de raíces y supresión del agente causal de la enfermedad | Tlapal-Bolaños et al.,2014 |
| B. subtilis y T. harzianum | Uso de microorganismos de productos comerciales | Capsicum annuum | Phytophthora capsici | Invernadero | Supresión de la enfermedad | Antagonismo, colonización y supresión del agente causal de la enfermedad | Lozano-Alejo et al., 2015 |
| C. musae, C. loeosporioides e Idriella lunata | Claves taxonómicas | - | Staphylococcus aureus, Streptococcus pneumoniae, S. epidermis, Escherichia coli y Pseudomonas aeruginosa | - | Antibiosis de metabolitos secundarios | Evaluación mediante sensidiscos con los extractos fúngicos (halos de inhibición) | Lagunes-Castro et al., 2015 |
| Actinomicetos | Claves taxonómicas | Solanum tuberosum | Ralstonia solanacearum, Pectobacterium carotovorum, P. infestans, Fusarium y R. solani | In vitro | Antibiosis, enzimas líticas, | Evaluación de extractos de actinomicetos mediante difusión en agar, halos de inhibición, confrontación dual, concentración mínima inhibitoria | Pérez-Rojas et al., 2015 |
| T. viride y B. subtillis | Uso de microorganismos de productos comerciales | - | Sclerotinia minor, S. sclerotiorum, y S. cepivorum | In vitro | Competencia, micoparasitismo y antibiosis | Confrontación dual | Pérez-Moreno et al., 2015 |
| Bacillus y Trichoderma | Claves taxonómicas y secuenciación del gen 18S ARNr y 16S ARNr | - | F. oxysporum, Botrytis cinerea, Penicillium crustosum, Aspergillus nidulans y Alternaria alternata | In vitro | Parasitismo, antibiosis, producción de enzimas líticas | Confrontación dual | Rios-Velasco et al., 2016 |
| Bacillus spp. | Claves taxonómicas | Capsicum chinense | F. equiseti y F. solani | In vitro e invernadero | Competencia, producción de metabolitos y enzimas líticas, promoción del crecimiento vegetal, inducción de resistencia sistémica | Confrontación dual, inducción de resistencia, severidad de la enfermedad | Mejía-Bautista et al., 2016 |
| Trichoderma | Claves taxonómicas y secuenciación del gen ITS1, ITS4 y tef1α | - | P. infestans | In vitro | Parasitismo | Confrontación dual | García-Núñez et al., 2017 |
| Hongosmicorrícicosarbusculares | Uso de microorganismos de productos comerciales | Agave cupreata | F. oxysporum | Invernadero | Competencia por sitio de colonización, cambios en la composición de exudados radiculares, inducción de resistencia, promoción del crecimiento vegetal | Colonización de raíces y severidad de la enfermedad | Trinidad-Cruz et al., 2017 |
| Saccharicola | Microorganismos provenientes de trabajos previos | Capsicum annuum | P. capsici, F. oxysporum y R. solani | In vitro e invernadero | Parasitismo, competencia, producción de enzimas, antibiosis | Confrontación dual, prueba de parasitismo de Riddell, microscopia de barrido, incidencia y severidad de la enfermedad | Uc-Arguelles et al., 2017 |
| Trichoderma y Bacillus | Microorganismos provenientes de trabajos previos | M. domestica | P. cactorum | Invernadero | Promoción del crecimiento vegetal, producción de fitohormonas, inducción de resistencia | Confrontación, severidad de la enfermedad | Ruiz-Cisneros et al., 2017 |
| Lecanicillium, alcarisporium, Sporothrix y Simplicillium | Claves taxonómicas | - | Hemileia vastatrix | In vitro | Micoparasitismo | Confrontación dual, parasitismo | Gómez-De la Cruz et al., 2017 |
| B. amyloliquefaciens y B. thuringiensis | Claves taxonómicas y secuenciación del gen 16S ARNr | - | P. capsici | In vitro e invernadero | Antibiosis, producción de lipopéptidos, promoción del crecimiento vegetal | Inhibición germinativa de zoosporas con inoculo bacteriano y filtrados, eficacia de biocontrol | Ley-López et al., 2018 |
| B. subtilis | AFLP (EcoR y MseI) | - | R. solani | In vitro | Antibiosis, producción de enzimas líticas , | Sobrenadante difusión en placa (metabolitos antagonistas), placas microtituladoras BIOLOG (diversidad metabólica) | Jiménez- Delgadillo et al., 2018 |
| Trichoderma y Bacillus | Claves taxonómicas y secuenciación del gen 18S ARNr, ITS1 e ITS4). Bacillus proviene de trabajos previos | S. lycoper- sicum | A. solani, F. oxysporum y P. infestans | In vitro | Producción de enzimas líticas, metabolitos promotores de crecimiento vegetal, antibiosis, inducción de resistencia, producción de lipopéptidos | Confrontación dual, tipo de antagonismo (escala de Bell) | Ruiz-Cisneros et al., 2018 |
| P. fluorescens | Claves taxonómicas y secuenciación del gen 16S ARNr | S. lycoper- sicum | A. alternata | In vitro e invernadero | Producción de compuestos extracelulares inhibidores enzimáticos, antibiosis | Difusión en agar de extractos, germinación de conidios y crecimiento micelial, incidencia y severidad de la enfermedad | Rodríguez- Romero et al., 2019 |
| Simplicillium y Lecanicillium | Secuenciación del gen 18S ARNr, ITS1 e ITS4 | - | Hemileia vastatrix | In vitro | Parasitismo | Confrontación dual, parasitismo | García-Neváreze Hidalgo- Jaminson, 2019 |
| T. harzianum | No se menciona | Carya illinoensis | Phymatotrichopsis omnivora | vivero | Antagonismo | Desinfestación reductiva | Samaniego- Gaxiola et al., 2019 |
| T. harzianum y B. subtilis | Microorganismos provenientes de trabajos previos | Arachis hypogaea | Aspergillus flavus, Fusarium sp., Sclerotinia minor y Thecaphora frezzi | In vitro y campo | Producción de enzimas líticas, promoción del crecimiento vegetal | Inoculación de sustrato estéril, incidencia de enfermedad, crecimiento de plantas, peso seco. | Illa et al., 2020 |
| B. amyloli- quefaciens y B. subtilis | Claves taxonómicas, pruebas bioquímicas, y metabolismo (tarjeta VITEX 2) | - | S. cepivorum | In vitro | Producción de metabolitos, sideróforos, enzimas líticas, promoción del crecimiento vegetal, tolerancia a estrés, antibiosis | Confrontación dual, actividad del ACC desaminasa, tolerancia a NaCl y prueba colorimétrica para producción de sideróforos y AIA | Ocegueda- Reyes et al., 2020 |
Afiliación taxonómica de ACB-M. El estudio taxonómico molecular de ACB-M se ha realizado convencionalmente con el gen 16S ARNr para la afiliación taxonómica de procariotas debido a que: i) está presente en dicho grupo de organismos y a menudo existe como una familia multigénica; ii) su función a lo largo del tiempo no ha cambiado, sugiriendo que su secuencia cambia lentamente en el curso evolutivo, y iii) su longitud (1500 pb) es lo suficientemente grande para propósitos técnicos y bioinformáticos, aunque limita la identificación de especies bacterianas debido a su restringida diversidad genética (Aguilar-Marcelino et al., 2020). De forma similar, en los eucariotas, la región ITS (internal transcribed spacer, por sus siglas en inglés), conformada por ITS1, el gen 5.8S ARNr e ITS2, es muy conservada a nivel de género, cuya longitud (650-1,100 pb) y limitada diversidad genética entre especies dificulta su uso para la diferenciación y afiliación taxonómica correcta. Además, la afiliación taxonómica de los ACB-M también se basa en el empleo de técnicas moleculares (RAPDs, ISSR, RFLP, AFLP) y/o técnicas de PCR de detección rápida mediante el uso de genes marcadores, tales como: 16S ARNr, 28S ARNr, ITS, β-tubulina, RPB2, factor de elongación y SCAR (Mazrou et al., 2020; Hassan et al., 2019). La resolución taxonómica a nivel de especie o subespecie (i.e. haplotipos) dependerá de la técnica molecular seleccionada, enzimas de restricción y genes o regiones seleccionadas.
La afiliación taxonómica correcta es determinante para la bioprospección de ACB-M bioseguros, ya que diversos géneros tienen especies con actividad antagónica contra fitopatógenos, pero pueden poseer otras especies causantes de enfermedades en plantas, animales y/o humanos. Por ejemplo, Bacillus es uno de los géneros bacterianos más reportados y estudiados a nivel mundial por su capacidad de control biológico. El grupo de B. cereus ha sido ampliamente estudiado y comprende, al menos, ocho especies estrechamente relacionadas: B. anthracis, B. cereus, B. thuringiensis, B. mycoides, B. pseudomycoides, B. weihenstephanensis, B. cytotoxicus y B. toyonensis. Sin embargo, los miembros de este grupo tienen un impacto significativo en la salud humana, la agricultura y la industria alimentaria; i.e. B. anthracis es el agente etiológico del ántrax, un patógeno obligado que representa una amenaza para la salud humana y de los herbívoros, mientras que B. thuringiensis es un agente de manejo biológico ampliamente utilizado y reconocido como un bioplaguicida seguro a nivel mundial (Liu et al., 2015). Por otro lado, B. cereus es un patógeno oportunista que a menudo causa intoxicación alimentaria y ha sido recientemente reportado como un fitopatógeno emergente. A nivel intraespecífico, miembros del grupo de B. cereus poseen secuencias del gen 16S ARNr muy similares y genomas altamente conservados (Ehling-Schulz et al., 2019), lo que dificulta su correcta afiliación taxonómica. Otro grupo que evidencia la importancia taxonómica con un enfoque molecular debido a restricciones de caracterización bioquímica, biológica y morfológica es el género Burkholderia (Espinosa-Victoria et al., 2020; Serret-López et al., 2021; de los Santos-Villalobos et al., 2018).
En los últimos años, la introducción de diversas estrategias y plataformas de secuenciación de siguiente generación (NGS, por sus siglas en inglés), herramientas genómicas, filogenómicas y bioinformáticas ha contribuido significativamente a una mejor comprensión de la evolución e interacción entre microorganismos y su ambiente (Jagadeesan et al., 2019). Además, estas herramientas han permitido el estudio de cepas microbianas a nivel genómico, lo que ha robustecido su correcta afiliación taxonómica y la identificación de genes involucrados en la colonización y adaptación al hospedero, sus mecanismos de acción, entre otros aspectos (Figura 1).
Estudios genómicos para la afiliación taxonómica de ACB de origen microbiano (ACB-M). En la actualidad, la afiliación taxonómica robusta de microorganismos se basa en la extracción, secuenciación y análisis de su genoma, parcial o total, apoyado de rasgos morfológicos, fisiológicos y metabólicos (Figura 2). Para esto, es determinante la secuenciación del ADN de alta calidad del microorganismo de interés y validar los datos obtenidos por un proceso de control de calidad, lo cual permite la exclusión de lecturas de ADN sin la calidad necesaria para análisis posteriores (Xi et al., 2019). FastQC es una herramienta digital que permite realizar numerosas comprobaciones de control de calidad de las secuencias obtenidas (Andrews, 2010). Posteriormente, se debe eliminar los adaptadores y remover datos de baja calidad de la información genómica obtenida, mediante el uso de los programas como Trimmomatic v0.36 (Bolger et al., 2014) y Cutadapt v1.18 (Martin, 2011). Posteriormente, el genoma es ensamblado, con programas como SPAdes v3.14.1, el cual ensambla los péptidos no ribosomales asociados a mecanismos de control biológico (Bankevich et al., 2012), hybird SPAdes (Antipov et al., 2016), Velvet v1.2.10 (Zerbino, 2008), IDBA-UD v1.1.1 (Peng et al., 2012), Canu (Koren et al., 2017) y SOAPdenovo2 (Luo et al., 2012). Una vez que el genoma ha sido ensamblado, se deben reordenar los contigs obtenidos basados en un genoma de referencia, mediante el programa Mauve contig Mover 2.4.0 (Darling et al., 2004). Finalmente, la anotación del genoma se lleva a cabo para identificar las secuencias codificantes y asociarles una función, o identificar proteínas hipotéticas; para esto el Centro Nacional de Información Biotecnológica (NCBI, por sus siglas en inglés) proporciona una herramienta de anotación de genoma procariótico automático. Similarmente, RAST y xBASE2 son servidores web empleados para anotar genomas de bacterias y arqueas (Aziz et al., 2008; Chaudhuri et al., 2008). Por último, la contaminación del genoma se puede cuantificar con diversos softwares como checkM y Quast, para procariotas y eucariotas (Gurevich et al., 2013; Parks et al., 2015).
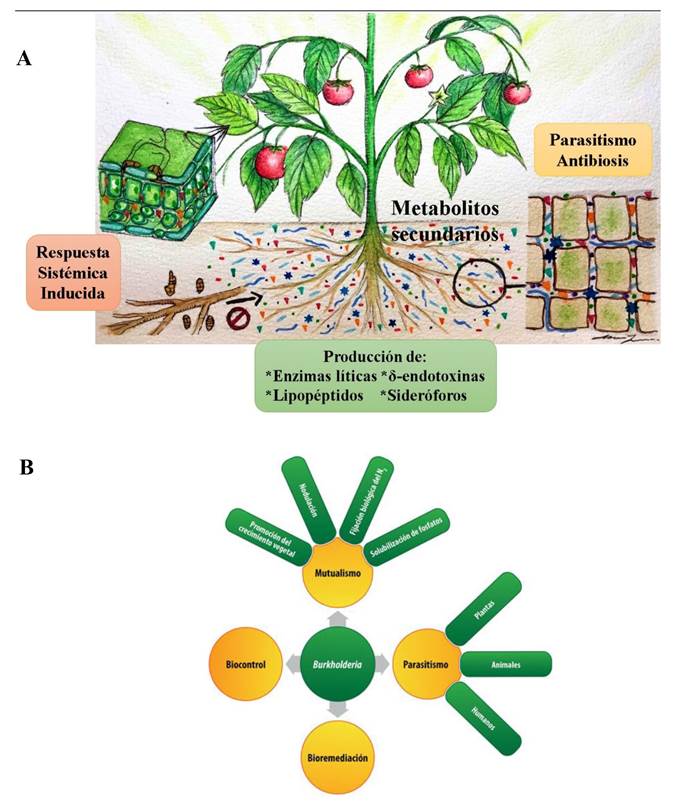
Figura 1 A. Mecanismos de acción más estudiados y reportados en agentes de control biológico de origen microbiano (ACB-M) (Fuente: Autores). B. Multiplicidad de funciones bióticas-abióticas que pueden estar asociados con un ACB-M. Caso Burkholderia (Fuente: Espinosa-Victoria et al., 2020).
Recientemente, se han desarrollado herramientas bioinformáticas in silico para la afiliación taxonómica de especies bacterianas (Figura 2), entre los cuales destacan los índices generales de relación del genoma (OGRIs, por sus siglas en inglés), tales como: la identidad de nucleótidos promedio (ANI, por sus siglas en inglés) y la Calculadora de distancia de genoma a genoma (GGDC, por sus siglas en inglés), para reemplazar el estándar de hibridación ADN-ADN (DDH, por sus siglas en inglés) (Richter et al., 2016). Lo anterior sólo se ha utilizado para el estudio en procariotas, ya que para cepas fúngicas no existen equivalentes de OGRIs (Raja et al., 2017).
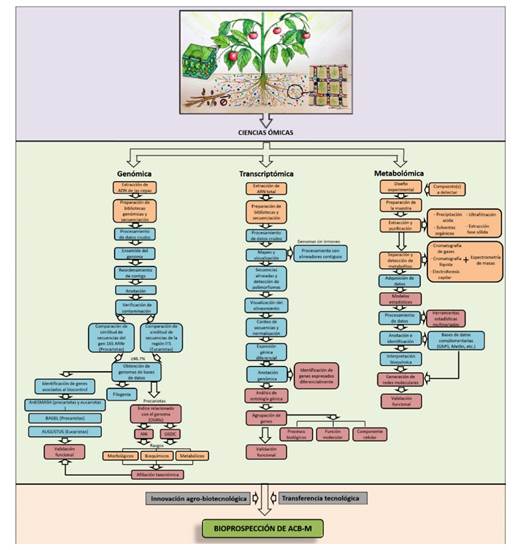
Figura 2 Flujo de procesos metodológicos genómicos y metabólicos empleados en investigación taxonómica y para establecer mecanismos de acción de agentes de control biológico de origen microbiano (ACB-M) aplicados en la supresión de fitopatógenos.
De esta manera, ANI es ampliamente utilizada para establecer la similitud -a nivel genómico- entre dos especies procariotas (Yoon et al., 2017), y es definida como el porcentaje promedio de identidad de secuencias de nucleótidos de genes ortólogos compartidos entre dos genomas (Sentausa y Fournier, 2013). El valor ANI del genoma en estudio corresponde al promedio de los valores de identidad entre todos los pares de fragmentos ortólogos entre dos genomas. El valor de ANI empleado para afiliar una cepa a una especie bacteriana previamente descrita es ≥ 95-96% (Varghese et al., 2015; Moriuchi et al., 2019).
Por otra parte, a diferencia de ANI (índice de tipo similitud), GGDC (http://ggdc.dsmz.de/) es un OGRI basado en la relación de distancia, que permite que se alinee el genoma de interés contra uno de referencia; posteriormente, se obtiene un conjunto de pares de segmentos de alta puntuación (HSP), titulados “coincidencias intergenómicas”, a partir de los cuales se calculan fórmulas de distancia específica para transformar los datos de HSP en el valor de GGDC (Meier-Kolthoff et al., 2013), utilizando un valor de GGDC >70% para afiliar taxonómicamente una cepa a una especie bacteriana previamente reportada.
En la actualidad, el uso de estas herramientas y estrategias enfocadas a una afiliación taxonómica robusta e integral de ACB-M (basadas en la secuenciación de genomas y estudios polifacéticos) se encuentra en auge en México. Por ejemplo, uno de estos trabajos fue recientemente publicado por de los Santos-Villalobos et al. (2019), quienes reportaron una nueva especie bacteriana con capacidad de control biológico contra Bipolaris sorokiniana, agente emergente causante de la mancha borrosa en el trigo, en el Valle del Yaqui, México. Inicialmente, esta cepa bacteriana (TE3T) fue afiliada al género Bacillus, con base a la secuenciación parcial del gen 16S ARNr (Valenzuela-Aragón et al., 2018). Posteriormente, Villa-Rodriguez et al. (2019) propusieron su afiliación taxonómica a la especie B. subtilis, mediante la secuenciación completa del gen 16S ARNr. Finalmente, la secuenciación y estudio completo del genoma, el análisis de OGRIs (ANI y GGDC), y la caracterización polifacética de la cepa TE3T permitieron una afiliación taxonómica robusta de este ACB a una nueva especie, nombrada B. cabrialesii (de los Santos-Villalobos et al., 2019). La inoculación de B. cabrialesii TE3T en plantas de trigo infectadas por B. sorokiniana resultó en la mitigación de la enfermedad reduciendo la escala de severidad de infección en hoja de 3-5 y la densidad de lesiones a 3.06 ± 0.6 cm-2 hoja, en comparación con plantas inoculadas con el fitopatógeno (8 - 9 y 6.46 ± 1.46 lesiones cm-2).
Estudio de los mecanismos de acción de ACB-M. Durante las últimas décadas se ha estudiado el efecto de ACB-M en el manejo de fitopatógenos. Lo anterior, ha permitido dilucidar los diferentes mecanismos empleados por dichos agentes para reducir el crecimiento, desarrollo e infección de fitopatógenos en plantas cultivadas. Entre los mecanismos de interacción con efectos supresivos más estudiados y reportados destacan i) micoparasitismo, ii) antibiosis, iii) actividad de enzimas líticas, iv) biosíntesis de sideróforos, v) producción de δ-endotoxinas, vi) biosíntesis de lipopéptidos, y vii) respuesta sistémica inducida (Figura 1A). Sin embargo, la acción de los ACB-M implican fases no parasíticas determinantes en su adaptación al medio edáfico o endofítico para garantizar su actividad supresiva, por lo que deben estudiarse de manera sistémica para diseñar estrategias de bioremediación de suelos, sustratos u órganos vegetales (Figura 1B).
En México, con base en las contribuciones publicadas en la Revista Mexicana de Fitopatología y revistas nacionales e internacionales en el área, existe un gran número de trabajos científicos desarrollados para la selección y evaluación in vitro de ACB-M promisorios. Aunque en menor proporción, también se han explorado mecanismos de acción específicos descritos anteriormente (Cuadro 2 y Figura 1). Sin embargo, la innovación de bio-plaguicidas desarrollados en México y otros países demanda la identificación de nuevos ACB y los mecanismos responsables de la actividad observada, tanto en nuevas cepas como en aquellas ya reportadas, debido a que no necesariamente los mecanismos o metabolitos que emplean dichos ACB in vitro son los responsables del efecto observado o están relacionados de forma directa con su bio-actividad en campo. En consecuencia, es determinante el empleo de estrategias de vanguardia e integradoras que permitan un mejor entendimiento de los mecanismos de acción de los ACB-M bajo condiciones de campo. Lo anterior incrementaría las probabilidades de éxito de estos agentes en su implementación agrícola y permitiría la optimización de procesos de producción y escalamiento de nuevos bioproductos, y la innovación de bioproductos ya registrados y disponibles en el mercado.
Estudios genómicos de ACB-M para la identificación de potenciales mecanismos de acción. En la actualidad, se han desarrollado diversas estrategias y plataformas enfocadas a la identificación de clusters de genes en el genoma asociados al control biológico, principalmente aquellos relacionados a las sintetasas de péptido no ribosomales (NRPS, por sus siglas en inglés) y las policétido sintasas (PKS, por sus siglas en inglés) (Figura 2). Estos metabolitos secundarios exhiben una significativa diversidad de actividades biológicas, y muchos de ellos son compuestos antimicrobianos, antifúngicos, antiparasitarios, antitumorales e inmunosupresores clínicamente valiosos (Ansari et al., 2004). Entre las herramientas empleadas para la identificación de dichos clusters de genes en el genoma de ACB de origen bacteriano se encuentran PRISM (Skinnider et al., 2017), CLUSEAN (Weber et al., 2009), RIPPMiner (Agrawal et al., 2017), mientras que para genomas fúngicos destacan SNAP (Jhonson et al., 2008), Augustus (Keller et al., 2011) y GeneMark-ES (Borodovsky y Lomsadze, 2011). Sin embargo, las herramientas AntiSMASH 5.0 y BAGEL4 han sido ampliamente reportadas y utilizadas para estos fines; no obstante, la identificación de genes asociados a la actividad de los ACB-M mediante estas herramientas es predictiva, por lo que, su expresión y función in vivo debe ser ampliamente estudiada y complementadas con otras estrategias que se describirán posteriormente.
AntiSMASH 5.0 utiliza la herramienta HMMer3 (http://hmmer.janelia.org/), y las traducciones de la secuencia de aminoácidos de todos los genes codificantes, mismas que se analizan con modelos de perfil oculto de Markov (pHMM, por sus siglas en inglés), basados en alineamientos de secuencias múltiples asociadas a proteínas o proteínas/dominios experimentalmente caracterizados (Blin et al., 2019). La etapa de detección utiliza una lógica de filtrado de pHMM negativos y positivos, y sus puntos de corte, donde se basa en el conocimiento de los componentes básicos mínimos de cada tipo clusters de genes reportados en la literatura científica.
Por otra parte, BAGEL4 traduce en seis proteínas [una para cada marco de lectura abierta (ORF, por sus siglas en inglés)], cada uno de los genes codificantes del genoma del ACB estudiado (Van Heel et al., 2018). Estas proteínas se examinan para detectar la ocurrencia de ciertos motivos proteicos (elementos conservados en una secuencia de aminoácidos que habitualmente se asocian con una función específica), y se compara con la base de datos de péptidos. Finalmente, las áreas de interés (AOI, por sus siglas en inglés) son identificadas y analizadas a detalle. Los ORF en el AOI se analizan de primera instancia usando Glimmer3 (Delcher et al., 2007). El programa está configurado para que Glimmer3 realice un modelo para cada AOI definida. Posteriormente, ORF pequeños se denominan en las regiones intergénicas. La configuración predeterminada para estos ORF es una longitud mínima de 72 bp (24 residuos de aminoácidos) y se permite una superposición de 10 pb con los ORF de Glimmer3.
Actualmente, el empleo de estas herramientas para el acercamiento integrativo de los potenciales mecanismos de acción de ACB-M se están desarrollando de forma exponencial en México. Ejemplo de ello, es el trabajo de Valenzuela-Ruiz et al. (2019) quienes emplearon esta estrategia para la identificación de un ACB-M afiliado taxonómicamente, a través del estudio de su genoma y OGRIs, como Bacillus paralicheniformis TRQ65. El estudio de los clusters de genes asociados a NRPS y PKS del genoma de B. paralicheniformis TRQ65 permitió identificar nueve genes asociados con la biosíntesis de lipopéptidos: lichenicidina, haloduracina, bacteriocina, enterocina y sonorensina (por BAGEL4), y ocho genes asociados con la biosíntesis de lipopéptidos: bacillibactina, butirocina, liquenicina, bacitracina y haloduracina (antiSMASH). La funcionalidad de dichos genes se validó mediante ensayos de confrontación dual contra Bipolaris sorokiniana, observando una zona de inhibición de 1.6 ± 0.4 cm, lo que confirmó la funcionalidad de los genes putativos encontrados en el genoma de la cepa TRQ65, asociados al control biológico de fitopatógenos (Villa-Rodríguez et al., 2019; Valenzuela-Ruiz et al., 2019).
Estudios transcriptómicos para la identificación de potenciales mecanismos de acción de ACB-M. La modulación en la expresión génica de un organismo, asociadas a sus interacciones con factores bióticos y/o abióticos, son reveladas por estudios transcriptómicos; los cuales se centran en el estudio de la expresión génica, patrones de co-expresión, cascadas de señalización y mecanismos moleculares regulados durante una condición biológica y tiempo específico (Aguilar-Marcelino et al., 2020).
Los estudios transcriptómicos son altamente dependientes del ARN total de alta calidad extraído bajo las condiciones específicas de estudio, seguido de la preparación y secuenciación de las muestras (Figura 2). Las lecturas obtenidas son filtradas mediante un análisis de calidad por FastQC (Andrews, 2010), se eliminan los adaptadores y lecturas de baja calidad por Trimmomatic v0.36 (Bolger et al., 2014) o Prinseq (Schmieder y Edwards, 2011). Posteriormente, se procede con al alineamiento y mapeo de las lecturas con un genoma (o transcriptoma) de referencia para identificar y cuantificar la regulación de los genes, así como conocer sus variantes alélicas (Rollano-Peñaloza y Mollinedo-Portugal 2017). Los organismos cuyos genomas no presenta intrones son procesados con alineadores contiguos, como Bowtie2 (Langmead y Salzberg 2012) o BWA (Li y Durbin 2009), diseñados para alinear ADN. Caso contrario, cuando se tienen genomas que poseen intrones es mejor utilizar alineadores como Tophat2 (Kim et al., 2013), Hisat2 (Kim et al., 2013), STAR (Dobin et al., 2013), GSNAP (Wu y Nacu 2010), SOAP2 (Li et al., 2009) o Kallisto (Bray et al., 2016). El ensamble de transcritos y la estimación de sus abundancias es realizado por lo general con Cufflinks y Cuffmerge (Trapnellet al., 2009); aunque también se han reportado estudios transcriptómicos utilizando DESeq (Love et al., 2014) o HTSeq-count (Anders y Huber, 2015).
Las secuencias alineadas pueden ser ordenadas con el uso de Samtools (Li, 2011) y Picard (http://broadinstitute.github.io/picard/). Esta información puede ser utilizada por programas como Bedtools (Quinland y Hall, 2010) para detectar polimorfismos de un solo nucleótido (SNPs, por sus siglas en inglés) (Rollano-Peñaloza y Mollinedo-Portugal, 2017). El análisis apropiado de expresión génica se basa en una normalización debido a los diversos tamaños de los genes y a la variación de la profundidad en la secuenciación de cada muestra. De esta manera, en el estudio de transcriptomas de novo es recomendado el software RSEM (Li y Dewey, 2011), que también proporciona valores de normalización. Posteriormente, para la identificación de genes expresados diferencialmente se debe realizar una anotación genómica, mediante un alineamiento de las secuencias de los genes que se fueron identificados como significativamente expresadas (Camacho et al., 2009). En seguida, se procede a identificar las familias de los genes y/o sus productos mediante softwares como HMMER (Potter et al., 2018), InterProScan (Mitchell et al., 2015) y SignalP (Petersen et al., 2011). Por último, se realiza un análisis de ontología génica (GO, por sus siglas en inglés), donde permite agrupar genes por procesos biológicos, función molecular, componente celular y el análisis de rutas metabólicas (KEGG) (Kanehisa et al., 2004) y otras agrupaciones de mayor complejidad (PANTHER) (Mi et al., 2013), y así herramientas como AgriGO (Tian et al., 2017) permiten visualizar -mediante arboles jerárquicos- los términos GO significativamente expresados. Otro enfoque puede ser el empleo de primers o iniciadores de genes específicos previamente identificados con mecanismos de acción de interés para su empleo en qPCR o ddPCR para cuantificar la expresión génica. Esta técnica evita el estudio transcriptómico de un organismo.
Diversas investigaciones han sido publicadas a nivel mundial sobre el uso de la transcriptómica para el entendimiento de mecanismos de control biológico durante la interacción planta-patógeno-ACB mediante el estudio de los perfiles de expresión génica en el ACB-M (en presencia del fitopatógeno in vitro o en campo); regulación de la expresión de genes en el fitopatógeno o en la planta hospedera (en presencia del ACB-M o sus metabolitos); impacto del fitopatógeno sobre los perfiles de expresión génica de su hospedero; y la modulación de estos perfiles por la presencia o ausencia del ACB-M. Por ejemplo, Duke et al. (2017) reportaron que el cultivo de Brassica napus (canola) es fuertemente afectado por Sclerotinia sclerotiorum, agente causal de la pudrición del tallo de canola; sin embargo, la inoculación de Pseudomonas chlororaphis PA23 a la canola en presencia de S. sclerotiorum, redujo la tasa de infección en 91%. Lo anterior, se asoció a la reprogramación transcripcional en la canola, i.e. las plantas infectadas con S. sclerotiorum regularon positivamente aproximadamente 8000 genes, mientras que el tratamiento preventivo con P. chlororaphis PA23 disminuyó 16 veces la expresión de los genes previamente regulados por el fitopatógeno. Además, en ausencia del patógeno, se observó que P. chlororaphis PA23 causó la regulación de genes de defensa en la canola, los cuales están involucrados en la respuesta sistémica inducida y la producción de especies reactivas de oxígeno.
Estudios metabolómicos para la identificación de potenciales mecanismos de acción de ACB-M. La metabolómica, como un enfoque científico basado en datos para detectar y cuantificar cientos de metabolitos por análisis diferencial (Lloyd et al., 2015), es ideal para el análisis de interacciones complejas, a nivel de metabolitos, durante las interacciones planta-microorganismos-ambiente.
Los estudios metabolómicos se basan primeramente en establecer la condición experimental para el estudio (Figura 2), con lo cual se definirá la estrategia de preparación de la muestra (enfocada el tipo de compuestos a detectar, incluyendo extracciones con solventes orgánicos o extracción de fase sólida), seguido por la preparación de la muestra ya sea concentrándola o purificándola (Mhlongo et al., 2018). Posteriormente, se realiza una separación y detección de metabolitos de interés a través de cromatografía de gases (GC, por sus siglas en inglés), cromatografía líquida (LC, por sus siglas en inglés), o la electroforesis capilar (CE, por sus siglas en inglés) acopladas a la espectrometría de masas (MS, por sus siglas en inglés) (Naz, 2014). La CE separa los compuestos según la carga y el tamaño, y ofrece un alto poder de resolución. La CE-MS se utiliza principalmente para vías metabólicas primarias-intermedias, y por lo general se acopla a un analizador de masas TOF (Ramautar et al., 2016). En los últimos años, la imagenología de MS (MSI, por sus siglas en inglés) ha avanzado y se ha aplicado en diferentes estudios metabólicos que proporciona la distribución de compuestos en superficie tisular (células, tejido o secciones específicas) (Mhlongo et al., 2018). En comparación con otras técnicas tradicionales de imagenología molecular, la MSI permite obtener mayor información al proporcionar una distribución de características con alta resolución para una amplia gama de metabolitos (Schwamborn, 2012).
La metabolómica, como otras herramientas ómicas, genera grandes cantidades de datos complejos que requieren instrumentos de almacenamiento y procesamiento. Lo anterior se puede procesar mediante herramientas estadísticas gratuitas como MarVis1, Mzine, XCMS, MAVEN, Metaboanalyst y MetAlign (Benton et al., 2008); así como programas comerciales: Markerlynx (Waters), soluciones de creación de perfiles (Shimadzu), Mass Profiler.pro (Agilent) y perfilador metabólico (Bruker) (Mhlongo et al., 2018). Además, se utilizan herramientas estadísticas multivariadas para reducir la dimensionalidad de los datos, la discriminación de variables, y agrupar mediante características compartidas entre muestras (i.e. n-cepas); dentro de los cuales se encuentran los análisis de componentes principales y el análisis discriminante de proyección ortogonal a estructuras latentes (OPLS-DA, por sus siglas en inglés) supervisado (Worley y Powers, 2013). Por último, se anotan e identifican los metabolitos mediante el uso de bases de datos complementarias que incorporan espectros de masas, nombres y estructuras de compuestos, modelos estadísticos y rutas metabólicas (Fukushima y Kusano, 2013). Recientemente, se han desarrollado varias bases de datos que incorporan herramientas estadísticas y metabolómicas basadas en la MS o resonancia magnética nuclear, i.e. MeRy-B, MeltDB y SetupX (Fukushima y Kusano, 2013).
La incorporación de enfoques metabolómicos para entender los mecanismos de acción de los ACB-M son de gran valor para el sector agrícola, debido a la amplia diversidad de metabolitos producidos por los ACB-M con alto potencial agro-biotecnológico, así como el conocimiento de los metabolitos implicados en el control biológico de fitopatógenos durante la interacción ACB-hospedero-fitopatógeno-ambiente. Por ejemplo, Vinale y colaboradores (2014), a través de enfoques metabolómicos, identificaron la producción de un nuevo metabolito producido por Trichoderma harzianum, nombrado como ácido isoharziánico, el cual inhibe el crecimiento de fitopatógenos como: Sclerotinia sclerotiorum y Rhizoctonia solani. Además, el ácido isoharziánico mejora la germinación de semillas de jitomate e induce resistencia sistémica en plantas. De forma similar, otros metabolitos asociados al control biológico fueron reportados para Trichoderma, tales como: ácido hetelídico, sorbicilinol, tricodermanona C, ácido giocládico y bisorbicilinol (Kang et al., 2011).
Es primordial que en México un importante productor agrícola, se fortalezcan los apoyos a la investigación científica de vanguardia enfocada a la afiliación taxonómica polifacética de ACB-M, y el estudio integrativo del conocimiento de los mecanismos de acción de dichos agentes durante la interacción con la planta hospedera, el fitopatógeno, y ambiente. Lo anterior, permitirá potenciar el desarrollo, innovación, y el éxito del uso de bioproductos formulados y registrados con el fin de proporcionar una alternativa ecológica sustentable que beneficien la sanidad de los cultivos.
Perspectivas agroecológicas de ACB-M con sustento en ciencias ómicas. El uso de ACB-M es una alternativa funcional y económicamente viable para el control de fitopatógenos. Sin embargo, es fundamental realizar y fortalecer inventarios regionales para identificar potenciales ACB-M y profundizar sobre sus mecanismos de acción bajo condiciones de campo, lo que permitirá identificar con mayor certeza diversos aspectos que impactan en su establecimiento y funcionalidad, tales como acciones específicas de supresión, así como densidad de umbrales de acción, dinámicas poblacionales, competencia inter-específica y mecanismos de adaptación físico-química al entorno edáfico y planta. Este conocimiento permitiría establecer dosis, frecuencia y sustratos de multiplicación - aplicación.
Entre los aspectos identificados como determinantes para incrementar la eficiencia de acción de los ACB-M en campo destaca la activación de la respuesta sistémica de la planta (ISR, por sus siglas en inglés), mediante el reconocimiento de los patrones moleculares asociados a microorganismos (MAMPs), a patógenos (PAMPs), o por daño (DAMPs) (Figura 2). Esta señalización en la planta permite detectar al patógeno y reducir su colonización en el sitio de infección, incluso activar mecanismos sistémicos en tejidos distales de la planta (Burketova et al., 2015). Así, la IRS es un estado de la planta que, mediante factores bióticos (i.e. ACB-M) o químicos, le provee protección contra sostenidos y futuros ataques de fitopatógenos (Ku´c, 1982). Este proceso fue primeramente reportado cuando el fitopatógeno estaba en contacto con la raíz; sin embargo, estudios sobre la colonización de inductores de resistencia mediante aplicaciones foliares han propuesto los mecanismos iniciales de dicha resistencia. Por ejemplo, Lamdan y colaboradores (2015) reportaron que los ACB pertenecientes a la especie Gliocladium virens y G. atroviride interactúan con la planta y patógeno, promoviendo el crecimiento de las plantas e induciendo respuesta sistémica, mediante proteínas secretadas por dichos hongos, lo cual reduce la enfermedad en las partes aéreas de la planta. Así, el empleo de elicitores microbianos permite inducir la respuesta sistémica y funcionan como activadores endógenos o exógenos mediante la reprogramación de los genes de resistencia (Gupta y Bar, 2020). El ergosterol, un componente principal de la membrana fúngica y sustrato de acción de muchos fungicidas químicos, ha funcionado como inductor de la resistencia en jitomate y tabaco (Granado et al., 1995; Vatsa et al., 2011). Por otra parte, en plantas hospederas y no hospederas de Cladosporium fulvum (patógeno del jitomate), se encontró que el gen CfHNNI1 es capaz de inducir la respuesta hipersensible en jitomate y tabaco (Xu et al., 2012), similar a lo reportado por Zhang y colaboradores (2013), quienes mencionaron que la proteína elicitora SCFE1 de Sclerotinia sclerotiorum indujo la inmunidad por PAMP en Arabidopsis thaliana. No obstante, en comparación con elicitores de bacterias y oomicetos, los elicitores fúngicos continúan sin ser comprendidos en su totalidad, y aquellos detectados hasta ahora se desconoce su función, por lo que prevalece su importancia para ser investigados (Liu et al., 2013).
De esta manera, las plantas sanas pueden ser tratadas con inductores de respuesta sistémica; sin embargo, éstos no resultan ser universales, por lo cual se necesitan más procesos de investigación. Actualmente, la inducción de respuesta preventiva en plantas es abordada con ACB-M con la finalidad de sensibilizar al hospedero para responder de forma rápida, fuerte, duradera, y que el gasto de energía requerido sea menor ante un fitopatógeno (Mauch-Mani et al., 2017). Lo anterior, debido a que las plantas en crecimiento en condiciones de campo están constantemente expuestas a diferentes estímulos de tipo abióticos y bióticos (bacterias, hongos, herbívoros, oomicetos, virus, artrópodos, entre otros) que podrían activar defensas en las plantas, e influir para mejorar su capacidad de respuesta manteniendo niveles basales de resistencia o una memoria bioquímica (Mauch-Mani et al., 2017). Así, cuando la planta percibe el estímulo, se producen cambios a nivel fisiológico, transcripcional, metabólico y epigenético, conocido como fase de inducción de respuesta preventiva, para que la planta desarrolle una respuesta más rápida y fuerte al potencial ataque, dado que ya estaba previamente preparada. Este tipo de resistencia previa puede ser duradera y mantenerse durante el ciclo de vida de la planta, incluso esta inducción de respuesta preventiva puede considerarse como transgeneracional (Mauch-Mani et al., 2017). No obstante, para el entendimiento de los mecanismos moleculares de la inducción de respuesta preventiva, se sugieren dos mecanismos potenciales: i) cambios epigenéticos en la metilación del ADN y las modificaciones de histonas que pueden ser portadoras de memoria de estrés y desencadenantes de respuestas inmunes, y ii) la acumulación de proteína quinasas activadas por mitógenos (MPK) (Espinas et al., 2016).
Por otra parte, otro de los enfoques en el estudio de los ACB-M es el empleo de sus metabolitos secundarios (MS), considerado como una solución verde en la agricultura ya que la mayoría de dichos ACB poseen la capacidad de secretar sus metabolitos (Köhl et al., 2019). De esta manera, los metabolitos bio-activos (poliquétidos, péptidos no ribosomales, metabolitos híbridos de péptido-péptido, terpenos, sideróforos, enzimas líticas, entre otros) son aislados y purificados (Figura 1 y 2). Estos metabolitos resultan de gran interés no sólo para la comunidad científica sino también para la industria agro-biotecnológica, debido a las actividades biológicas que poseen contra fitopatógenos, como hormonas o transportadores de elementos químicos durante la producción de bioplaguicidas y biofertilizantes (Woo et al., 2014). Los MS son altamente diversos en estructura química, pero las vías de biosíntesis se relacionan con el metabolismo primario, como la ruta del ácido shikimato, acetil Co-A, los derivados de azucares o aminoácidos (Pott et al., 2019). El entendimiento de estas rutas metabólicas es de gran importancia para la innovación de bioproductos enfocados al control de fitopatógenos, a través de técnicas tradicionales o aquellas catalogadas como ómicas.
Un enfoque clásico holístico, pero recientemente revalorado con el advenimiento de nuevas tecnologías como la metagenómica es el funcionamiento de suelos supresivos, cuya composición de la comunidad microbiana impide que los fitopatógenos presentes infecten o se multipliquen en la planta de interés. Dichos suelos pueden dividirse en dos categorías: i) suelos supresivos generales, los cuales tienen un alto contenido de biomasa microbiana, pero resulta en bajos niveles de supresión, y ii) los suelos supresivos específicos, que tiene una alta concentración de uno o más especies microbianas y resulta en altos niveles de protección de la planta contra patógenos (Mousa y Raizada, 2016). Shen y colaboradores (2015), reportaron que la composición de la comunidad microbiana de la rizósfera de plantas de chile inoculada con B. amyloliquefaciens (bacteria benéfica) indujo la supresión de Fusarium y aumentó la diversidad bacteriana potencialmente estimulantes (Acidobacterias, Firmicutes, Leptosphaeria y Phaeosphaeriopsis). Sin embargo, hace falta mayor número de estudios actuales sobre suelos supresivos, en especial donde se realizan aplicaciones constantes de ACB-M para el manejo de fitopatógenos, por lo que este tipo de estudios representa un reto para el sector agro-biotecnológico enfocado en garantizar la sanidad vegetal de forma sostenible e integradora, utilizando enfoques altamente precisos como las ciencias ómicas.
El desarrollo e innovación del sector agro-biotecnológico es determinante para la implementación extensiva y exitosa de ACB-M en México, pero representa un gran reto científico. Existen múltiples estudios de ACB-M para el manejo de fitopatógenos, pero aún requieren estudios integradores y creativos enfocados a la generación o innovación de alternativas rentables, efectivas y ecológicas para potenciar el uso de bioproductos bioplaguicidas con la calidad y seguridad que demanda la sanidad vegetal a nivel mundial.
CONCLUSIONES
El empleo masivo de ACB-M en nuestro país y en el mundo demanda mayor diversidad de bioplaguicidas registrados y comercializados. Sin embargo, es necesario abandonar la concepción reduccionista de la implementación mediante el uso de especímenes de colecciones microbianas y su escalabilidad regional irrestricta del entorno agroecológico. En necesario generar y disponer de un amplio acceso a información técnica-científica que soporte la efectividad relativa a las complejas interacciones con factores bióticos y/o abióticos del agroecosistema. Esto permitirá detonar procesos de generación, innovación y adopción más eficiente de estas estrategias agrobiotecnológicas. En México y a nivel internacional, la investigación sistémica y holística de ACB-M debe fortalecerse con la integración de enfoques vanguardistas a partir del surgimiento y desarrollo de las ciencias ómicas, las cual solo podrán ser potenciadas con capital humano altamente capacitado e infraestructura científica especializada. Esto permitiría desarrollar la nueva generación de ACB-M eficientes y costo-efectivos mediante la bioprospección de nuevos ACB-M y el estudio detallado de su afiliación taxonómica y sus mecanismos de acción a nivel de los agroecosistemas. Lo anterior basado en ejes temáticos transdisciplinarios, tales como microbiomas, dinámicas metapoblacionales, relaciones funcionales nivel comunidad, e interacción génica-metabólica en el sistema planta-microorganismos-ambiente en un contexto adaptativo y evolutivo. El entendimiento sistémico e integral de ACB-M en los agroecosistemas, la generación de redes de investigadores y empresas biotecnológicas, y la divulgación del conocimiento permitirá potenciar la adopción de ACB-M en el campo mexicano, contribuir a la sanidad vegetal y seguridad alimentaria de forma sostenible, económicamente viable y biosegura.











 text in
text in 


