INTRODUCCIÓN
Los mamíferos clasificados como rumiantes se caracterizan por la adaptación morfofisiológica de su sistema digestivo (Resende Jr et al., 2019; Rotta et al., 2014), fraccionado en cuatro cámaras: I) retículo, II) rumen, III) omaso y IV) abomaso (Qiyu et al., 2019). El abomaso secreta hidrolasas digestivas y su función es similar al estómago de los monogástricos (Agarwal et al., 2015). Los rumiantes se especializan por su capacidad para alimentarse de pastos y forrajes (Puppel y Kuczyńska, 2016), ya que pueden degradar polisacáridos estructurales p. ej., celulosa, hemicelulosa y pectina (DePeters y George, 2014), muy poco digestibles para las especies no rumiantes (Kittelmann et al., 2013; Zeng et al., 2017). La degradación del alimento se realiza principalmente por digestión fermentativa, llevada a cabo por microorganismos presentes en el rumen (Ginane et al., 2015; Wallace et al., 2017). Las moléculas resultantes de la fermentación ruminal son aprovechadas para satisfacer los procesos fisiológicos del animal (Kittelmann et al., 2013; Li et al., 2019a). La cuantificación de los analitos bioquímicos en el plasma y/o suero, otorgan una aproximación al metabolismo nutricional (García et al., 2015); además confieren información sobre alteraciones y ajustes homeostáticos (Moyano et al., 2018). Por tal motivo es importante comprender los procesos de catabolismo y anabolismo que se llevan a cabo en el rumiante para entender los niveles de analitos presentes (Puppel y Kuczyńska, 2016). Debido a ello, es necesario incrementar nuestra comprensión sobre el metabolismo de los monosacáridos, aminoácidos (aa) y ácidos grasos. Por lo tanto, se realizó una revisión bibliográfica sobre su metabolismo en los rumiantes y su asociación con diferentes analitos bioquímicos.
Abreviaturas
aa |
aminoácidos |
AcAc |
acetoacetato |
AGNE |
ácidos grasos no esterificados |
AGV |
ácidos grasos volátiles |
ALB |
albumina |
Arg |
arginina |
C=O |
grupo carbonilo |
C16:0 |
palmítico |
C3H3O3 |
piruvato |
C6H12O6 |
glucosa |
CO2 |
dióxido de carbono |
COL |
colesterol |
COOH |
grupo carboxilo |
CH4 |
metano |
FAD |
dinucleótido de flavina-adenina |
Glu |
glutámico |
H2CO3 |
carbónico |
HCl |
clorhídrico |
HCO3 |
anión hidrógenocarbonato |
His |
histidina |
Ile |
isoleucina |
K+ |
ion potasio |
Leu |
leucina |
Lys |
lisina |
Met |
metionina |
Na+ |
ion sodio |
NH3 |
amoníaco |
NNP |
nitrógeno no proteico |
pH |
potencial de hidrógeno |
Phe |
fenilalanina |
PLP |
cofactor piridoxal fosfato |
TAG |
triacilgliceroles |
Thr |
treonina |
Trp |
triptófano |
Val |
valina |
VLDL |
very low density lipoproteins |
β-HBA |
β-hidroxibutirato |
El Rumen
El rumen es una cámara de fermentación anaerobia (Armato et al., 2016), con un potencial de hidrógeno (pH) entre ácido y neutro de 5.5 a 7.0 (Jiang et al., 2017); siendo éste el principal determinante del tipo y número de microorganismos (Resende Jr et al., 2019), y una temperatura que oscila entre 38 a 42 ºC (Pourazad et al., 2016; Yazdi et al., 2016). El ecosistema ruminal está formado por tres grupos: I) bacterias, su concentración es de 1 x 1010 y 1 x 1011/mL de líquido ruminal (Valente et al., 2016), y está relacionada con el contenido energético de la dieta (Krause et al., 2013); además el nitrógeno no proteico (NNP), como la urea, debe ser convertido en amoniaco (NH 3) para que pueda ser utilizado por las bacterias (DePeters y George, 2014; Wallace et al., 2017), transformando proteína de mala calidad en proteína de alta calidad (Puppel y Kuczyńska, 2016; Jin et al., 2018); grupo II) protozoarios ciliados, su concentración oscila entre 1 x 104 y 1 x 106/mL de líquido ruminal, su función es controlar el número de bacterias en el rumen (Francisco et al., 2019), envuelven almidón que pasa al intestino, siendo una fuente de glucosa (C 6 H 12 O 6) para el rumiante (Wallace et al., 2017), no sintetizan proteína a partir de NNP (Jin et al., 2018); la mayoría son del género Isotricha o Entodinium (Gebreegziabher, 2016), y grupo III) hongos, se encuentran en una concentración de 1 x 103 a 1 x 105/mL de líquido ruminal, poseen actividad celulolítica principalmente en forrajes maduros (Valente et al., 2016); algunas especies son Neocallimastix frontalis, Caecomyces communis y Piromyces communis (Krause et al., 2013).
La Microbiota Ruminal Amilolítica-Celulolítica y la Fermentación Anaeróbica
La degradación de polisacáridos presente en los forrajes es llevada a cabo por bacterias celulolíticas (Bacteriodes succinogenes, Ruminococcus albus), amilolíticas (Bacteroides amylophylus, Streptococcus bovis), hemicelulolíticas (Butyrivibrio fibrisolvens, Bacteroides ruminicola) y pectinolíticas (Lachnospira multiparus, Succinivibrio dextrinosolvens (Valente et al., 2016), que obtienen C6H12O6 y otros monosacáridos como xilosa y fructosa-6-fosfato, a partir de celulosa y hemicelulosa (Krause et al., 2013). Los monómeros son absorbidos por microorganismos y vía glucolítica forman nicotinamida adenina dinucleótido en su forma reducida (NADH+H +), piruvato (C 3 H 3 O 3) y adenosina trifosfato (ATP) para su crecimiento y mantenimiento (Wallace et al., 2017; Francisco et al., 2019). La digestión fermentativa es anaeróbica (Kittelmann et al., 2013; Yazdi et al., 2016), por lo que el C3H3O3 funciona como captador de electrones, para generar NAD+ y ATP, retirando NADH+H+ (Górka et al., 2017).
Los ácidos grasos volátiles (AGV): acético (CH 3 -COOH), propiónico (CH 3 -CH 2 -COOH) y butírico (CH 3 -CH 2 -CH 2 -COOH) son los principales productos terminales de la digestión fermentativa (Aydin et al., 2017; Li et al., 2019a); son absorbidos a través de la pared del rumen e incorporados a la circulación mediante la vena porta (Resende Jr et al., 2019). Representan entre el 70-80% del combustible energético del rumiante (Mikołajczyk et al., 2019).
La flora ruminal sintetiza CH3-COOH a partir de la descarboxilación de C3H3O3 en acetil-coenzima A, liberando un carbono (Gebreegziabher, 2016; Chishti et al., 2020). Para la formación de CH3-CH2-CH2-COOH se requieren dos acetil-coenzima A (Górka et al., 2017; Resende Jr et al., 2019). Hay dos vías para la formación de CH3-CH2-COOH: I) vía reductiva directa, el C3H3O3 pasa a lactato, y éste a acrilil-coenzima A (Aydin et al., 2017), y II) vía aleatoria, se añade un carbono al C3H3O3 y el oxaloacetato formado se transforma en succinato; posteriormente se sintetiza CH3-CH2-COOH, perdiendo un carbono y formando dioxígeno molecular (Krehbiel, 2014; Gebreegziabher, 2016). Además, se forma dióxido de carbono (CO 2) y metano (CH 4) que se eliminan por eructo (Teklebrhan et al., 2020; Toral et al., 2017). La síntesis de CH4 es necesaria para la producción de cofactores oxidados en las rutas para la formación de CH3-COOH y CH3-CH2-CH2-COOH (Kozłowska et al., 2019). Las bacterias encargadas de esta función son Methanobrevibacter ruminantium, Methanobacterium formicicum y Methanomicrobium mobile (Baruah et al., 2019).
En la figura 1, se muestra la síntesis de AGV. La concentración ruminal de CH3-COOH, CH3-CH2-COOH y CH3-CH2-CH2-COOH en animales alimentados con forrajes; oscila entre 70:20:10% respectivamente, y en animales alimentados principalmente con cereales fluctúa entre 60:30:10% (Gebreegziabher, 2016).
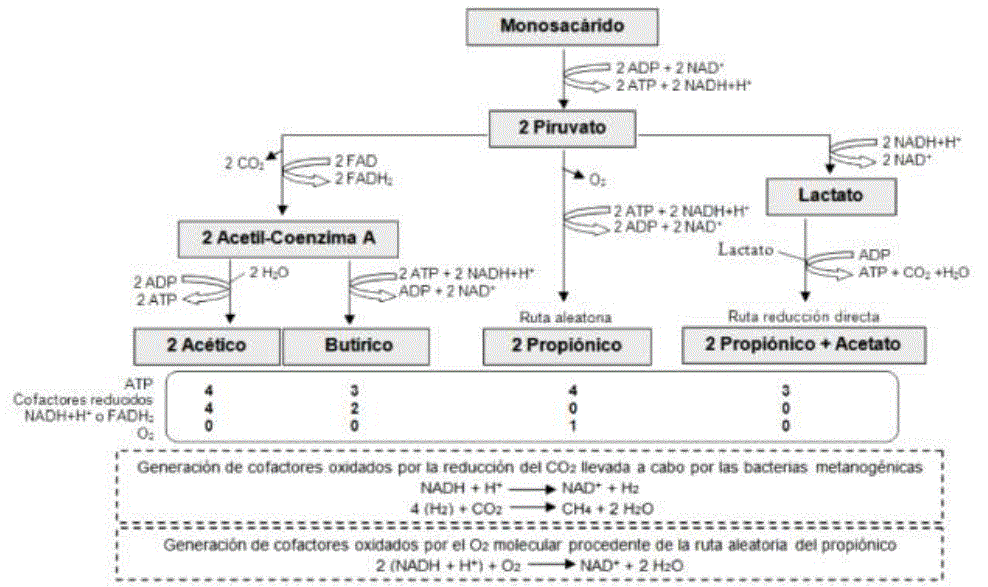
Fuente: información sintetizada de (Gebreegziabher, 2016)
Figura 1 Síntesis de ácidos grasos volátiles a partir de monosacáridos en el rumen
La Microbiota Ruminal Proteolítica y la Fermentación Anaeróbica
Los componentes proteicos suministrados en la dieta son fermentados por bacterias proteolíticas Bacteroides amylophylus, Bacteroides ruminicola, y algunas cepas de Butyrivibrio fibrisolvens (García et al., 2014), mediante sus proteasas microbianas, liberando péptidos (Alves et al., 2014; Rostom y Shine, 2018). Estos son absorbidos por el microorganismo, donde las peptidasas hidrolizan los enlaces peptídicos, liberando aa, utilizados para traducir proteínas propias o catabolizarlos para liberar energía (Li et al., 2019b; Silva et al., 2016). El producto final es el NH3 (Khezri et al., 2016; Carvalho et al., 2019), que sirve como sustrato de nitrógeno para las bacterias (Valente et al., 2016). El NH3 es absorbido mediante difusión pasiva a través de los canales de ion potasio (K +), ubicados en la membrana del rumen (García et al., 2014), por circulación portal llega al hígado donde es sintetizado en urea (Rostom y Shine, 2018).
La síntesis de urea comienza en la matriz mitocondrial (Shi et al., 2019) con la unión del anión hidrogenocarbonato (HCO 3 -) y el NH3, por medio de carbamoil fosfato sintetasa. El fosfato de carbamoil se une a la ornitina, por medio de ornitina transcarbamoilasa, generando citrulina. Esta se transporta al citoplasma donde reacciona con aspartato por medio de argininosuccinato sintasa, formando argininosuccinato; posteriormente argininosuccinato liasa lo divide, formando arginina (Arg) y fumarato (Hristov et al., 2019). Por último, la Arg cataliza la hidrólisis para sintetizar ornitina, agua (H 2 O) y urea (Gebreegziabher, 2016) (figura 2 ).
La urea pasa nuevamente a la circulación sanguínea, donde tiene tres rutas metabólicas: 1.) regresa al rumen vía saliva o a través de las capas epiteliales del rumen, con ayuda de las proteínas de transporte UT-B para ser reconvertida en NH3 (García et al., 2014; Carvalho et al., 2019), 2.) excretada en la orina o heces fecales (Schuba et al., 2017; Li et al., 2019b) o, 3.) formar parte del NNP de la leche (Alves et al., 2014; Jin et al., 2018) (figura 3).
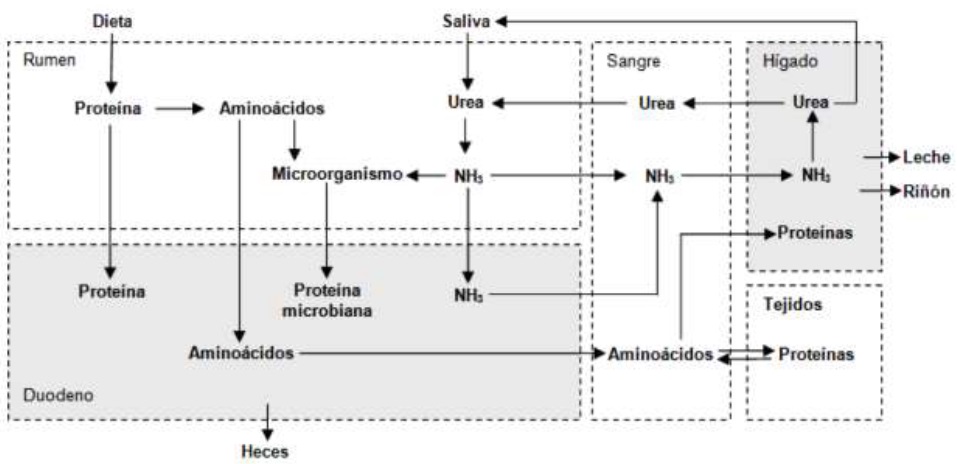
Fuente: información sintetizada de (Li et al., 2019b)
Figura 3 Metabolismo general de las proteínas en el rumiante
La Microbiota Ruminal Lipolítica y la Fermentación Anaeróbica
Los microorganismos encargados de catabolizar los componentes lipídicos de la dieta, son: Anaerovibrio lipolytica, Butyrivibrio fibrisolvens, Treponema bryantii, Eubacterium spp., Fusocillus spp y Micrococcus spp. (Valente et al., 2016). Las lipasas bacterianas por hidrólisis liberan ácidos grasos no esterificados (AGNE) y glicerol (Prieto et al., 2016); además alcoholes aminados (derivados de fosfolípidos) y galactosa (procedente de galactolípidos) (Toral et al., 2018). El glicerol, los alcoholes aminados y la galactosa son metabolizados a AGV (Silva et al., 2014; van Cleef et al., 2018). Los AGNE que se encuentran libres en el rumen, llevan un proceso de hidrogenación microbiana (Tran et al., 2017; Toral et al., 2017), resultado de la adición de hidrógeno a los ácidos grasos saturados, para formar ácidos grasos insaturados con dobles enlaces (Francisco et al., 2019). Este mecanismo es otra forma de eliminar los hidrógenos que resultan del catabolismo de los polisacáridos (Osorio et al., 2015; Prieto et al., 2016).
La absorción de los AGV se realiza en la pared del rumen (80%), en omaso (10%), y el resto pasa al abomaso para ser absorbidos en el duodeno (Yazdi et al., 2016). Los AGV se difunden pasivamente hacia el interior del epitelio ruminal (Agarwal et al., 2015; Yohe et al., 2019). El hidrógeno necesario para que los AGV se disocien en el epitelio, es donado por el carbónico (H 2 CO 3), formando CO2 y H2O, de la disociación se obtiene un hidrógeno para unirse a los AGV y se forma una molécula de HCO3- en la luz del rumen. Por lo tanto, este proceso ayuda a amortiguar el pH ruminal (Wang et al., 2016).
La absorción de los AGV se realiza de la misma forma para todos, aunque en el interior de las células epiteliales del rumen cambia su conformación (Qumar et al., 2016). Una parte del CH3-COOH se oxida por completo dentro de las células, como fuente de energía; mientras el resto es absorbido sin ser alterado, pasando al hígado por la vena porta (Loncke et al., 2015). El 80% del CH3-COOH que llega al hígado escapa de la oxidación, pasando a la circulación general para ser aprovechado por otros tejidos (Qumar et al., 2016).
En el citoplasma la conversión del CH3-COOH a acetil-Coenzima A es catalizado por acetil-Coenzima A sintetasa (Chishti et al., 2020). La mayor parte se oxida en el ciclo de Krebs o es utilizado para síntesis de ácidos grasos en los hepatocitos (Yohe et al., 2019). Una fracción del CH3-CH2-COOH es degradado y convertido en lactato (2-5%) antes o durante la absorción; el resto pasa en la circulación portal hacia el hígado, donde los hepatocitos lo sintetizan en C6H12O6, vía glucogénesis (Loncke et al., 2015). Para entrar al ciclo de Krebs el propionil-Coenzima A mediante propionil-Coenzima A carboxilasa, forma metilmalonil-Coenzima A, y posteriormente se forma succinil-Coenzima A (Gebreegziabher, 2016). El CH3-CH2-CH2-COOH es convertido casi en su totalidad a β- hidroxibutirato (β-HBA) en la mucosa ruminal (Agarwal et al., 2015). Este cuerpo cetónico, representa el 80% de las cetonas formadas (Górka et al., 2017). El CH3-COOH y el β-HBA se utiliza para la síntesis de ácidos grasos en el tejido adiposo y la glándula mamaria (García et al., 2015; Song et al., 2018).
Digestión y Absorción Posruminal
Aunque el rumiante se caracteriza por la fermentación microbiana en el rumen (Hristov et al., 2019), la digestión posruminal es vital, ya que dispone de lípidos, proteínas y algunos polisacáridos no estructurales que escapan de la fermentación (Agarwal et al., 2015). El alimento no fermentado junto con proteína microbiana, pasa al omaso por el orificio retículo-omasal, donde se absorben AGV, NH3, H2O, ion sodio (Na +) y K+ (Hussain et al., 2013; Freitas Jr et al., 2019). Posteriormente pasan al abomaso que contiene ácido clorhídrico (HCl) y pepsina (Rotta et al., 2014). El alimento es mezclado, pasando al duodeno (Hristov et al., 2019). El almidón y disacáridos que escapan de la digestión ruminal son hidrolizados por amilasas pancreáticas obteniéndose monosacáridos (Rotta et al., 2014).
La absorción se lleva a cabo en las vellosidades de los enterocitos (Harmon, 2009). Los monosacáridos se transportan en contra de su gradiente de concentración por medio del cotransportador de Na+ (Harmon y Swanson, 2020). La bomba ATPasa - Na+- K+ crea el gradiente de concentración del Na+ que aporta la energía (Bergman et al., 2019).
Otra forma de transporte para C6H12O6 es el transportador GLUT2 (Harmon, 2009). La proteína que llega al intestino delgado procede de la dieta que escapa de la fermentación, proteína endógena (García et al., 2015) y la contenida en los microorganismos que están unidos al alimento (Batista et al., 2016; Golshan et al., 2019). El catabolismo inicia en el abomaso por la pepsina e hidrólisis acida; posteriormente en el duodeno por enzimas pancreáticas y duodenales (tripsinasa, quimiotripsinasa y carboxipeptidasa), que rompen enlaces peptídicos para liberar aa y pequeños péptidos para su absorción en yeyuno e íleon (Emery, 2015; Hristov et al., 2019). La absorción consiste en un transporte a través de Na+ dependiente, el consumo de energía se asocia con el flujo continuo de Na+ hacia el exterior, como resultado de la actividad de la bomba ATPasa - Na+ - K+ (Silva et al., 2016). El Na+ que entra a la célula a favor de un gradiente de concentración, va unido a una molécula de aa a través de la membrana celular (Emery, 2012; Rostom y Shine, 2018).
Los lípidos que llegan al abomaso en forma de AGNE representan entre el 70 y 80%, el resto son fosfolípidos de origen microbiano (Aibibula et al., 2015; Toral et al., 2018). Estos últimos son emulsionados por sales biliares e hidrolizados por lipasas pancreáticas para liberar AGNE (Dawson y Karpen, 2015; Kohan et al., 2015). La micela se forma de sales biliares, AGNE saturados, triacilgliceroles (TAG) y lecitina (Cao et al., 2018), transportándose hasta las vellosidades de los enterocitos (Park et al., 2019). Los AGNE de menos de 12 carbonos, se absorben y son transportados por vena porta al hígado unidos por enlaces no covalentes en la albumina (ALB) (Dawson y Karpen, 2015). En cambio, los AGNE de 12 o más carbonos, son esterificados para formar TAG y fosfolípidos (Vargas, 2019). Los TAG, cantidades pequeñas de mono y diacilgliceroles, fosfolípidos y colesterol (COL) son unidos a apoproteínas para formar quilomicrones y lipoproteínas de muy baja densidad ( very low density lipoproteins VLDL ), que salen al sistema linfático, para ser incorporados al torrente sanguíneo (Kohan et al., 2015; Prieto et al., 2016). Los lípidos se absorben por difusión o pinocitosis (Walther y Farese Jr, 2012).
Metabolismo de Monosacáridos en Rumiantes
El torrente sanguíneo es el medio por el cual los nutrientes absorbidos se dirigen al hígado y a otros órganos para su catabolismo o anabolismo, según la necesidad celular (Goyal y Longo, 2015). Las enzimas juegan un papel muy importante en el metabolismo, ya que son proteínas catalizadoras de reacciones específicas (Jindal y Warshel 2017); sin ellas las reacciones biológicas serían muy lentas para la vida celular (Ramsay et al., 2019). Su función es unirse temporalmente a una molécula, parta aplicar cambios atómicos (Menger y Nome, 2019). El metabolismo de los monosacáridos gira en torno al suministro y destino de C6H12O6, siendo este monómero la principal fuente de energía para las células (Hooijberg et al., 2017). La vía catabólica de C6H12O6 es la glucólisis, llevada a cabo en el citoplasma celular (Dashty, 2013). Este proceso consta de ocho reacciones: 1) la glucosa (C 6 H 12 O 6) ingresa al citoplasma para ser fosforilada (adición de un grupo fosfato), a partir de ATP. Esta reacción es catalizada por la hexoquinasa. La glucosa- 6-fosfato (C 6 H 11 O 9 P) (aldohexosa) resultante abunda en todas las células, ya que la gran mayoría de C6H12O6 que ingresa al citoplasma termina siendo fosforilada, con el fin de impedir que pueda atravesar de regreso la membrana citoplasmática y difundirse al medio extracelular (Donnelly y Finlay, 2015); 2) la C6H11O9P presenta isomerización [una molécula es transformada en otra que posee los mismos átomos, pero dispuestos de forma distinta -cambia de lugar el grupo carbonilo (C=O)-] y es transformada en fructosa- 6-fosfato (cetohexosa). Reacción catalizada por glucosa-6-fosfato isomerasa (Dashty, 2013); 3) la fructosa-6-fosfato, es fosforilada a partir de ATP, en los carbonos 1 y 6 para dar lugar a la fructosa-1,6-bisfosfato. Reacción catalizada por fosfofructoquinasa (Ashrafi y Ryan, 2017); 4) la fructosa-1,6-bisfosfato es dividida en dos: gliceraldehido-3-fosfato y dihidroxiacetona fosfato. Reacción catalizada por aldosa (Watts y Ristow, 2017); 5) triosa fosfato isomerasa cataliza la conversión de dihidroxiacetona fosfato para obtener más gliceraldehido-3-fosfato (Bommer et al., 2020); 6) el gliceraldehido-3-fosfato es oxidado y fosforilado, en los carbonos 1 y 6 formando 1,3-bisfosfoglicerato por gliceraldehido- fosfato deshidrogenasa (Poher et al., 2018). Posteriormente, transfiere su grupo fosfato, para sintetizar ATP y se transforma en 3-fosfoglicerato. Reacción catalizada por fosfoglicerato quinasa (Dashty, 2013); 7) el 3-fosfoglicerato presenta isomerización del C3 al C2 y es transformado en 2-fosfoglicerato por la fosfoglicerato mutasa (Donnelly y Finlay, 2015). Posteriormente la enolasa propicia la formación de un enlace doble, eliminando una molécula de H2O y formando fosfoenolpiruvato (Bommer et al., 2020) y 8) el fosfoenolpiruvato transfiere su grupo fosfato, para sintetizar ATP y se transforma en C3H3O3, reacción catalizada por piruvato quinasa (figura 4).
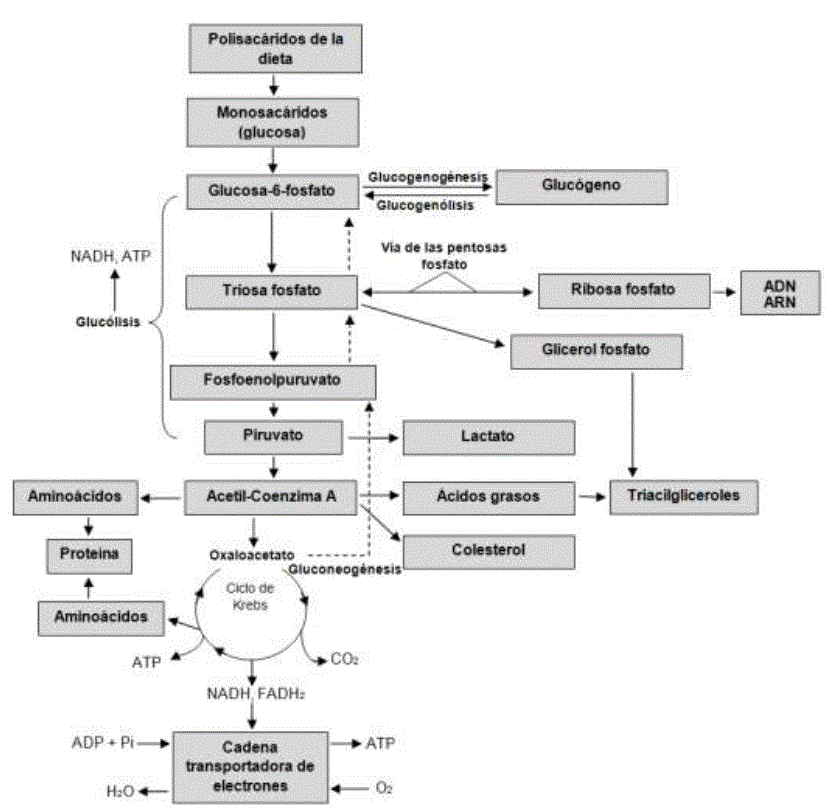
Fuente: información sintetizada de (Dashty, 2013)
Figura 4 . Metabolismo general de los monosacáridos
El C3H3O3 sale del citoplasma e ingresa a la matriz mitocondrial, utilizando la fuerza protón- motriz generada por la cadena respiratoria (Poher et al., 2018). Por cada C6H12O6 se generan dos C3H3O3, dos ATP, dos NADH+H+, dos hidrogeniones y dos moléculas de H2O (Dashty, 2013; Watts y Ristow, 2017). Las células aerobias metabolizan el C3H3O3 a acetil-Coenzima A, por medio de piruvato deshidrogenasa (Edinburgh et al., 2017), permitiendo su ingreso al ciclo de Krebs para su participación en la fosforilación oxidativa (Bergman et al., 2019).
Por cada acetil-Coenzima A que ingrese en el ciclo de Krebs se producen 12 ATP. Este proceso es fuente esencial de intermediarios para otras rutas metabólicas, p. ej., glucogenogénesis en el hígado y músculo estriado (Dashty, 2013; Edinburgh et al., 2017), ruta de las pentosas fosfato (figura 4) y síntesis de lípidos y aa. La ruta de las pentosas fosfato, es una vía metabólica alterna que no produce ATP (Kohan et al., 2015), sintetiza equivalentes reductores como nicotinamida adenina dinucleótido (NADPH), para la síntesis de novo de ácidos grasos, esteroides, el mantenimiento de glutatión para la actividad antioxidante (Chen et al., 2016) y fuentes de ribosa para la síntesis de ácidos nucleicos y nucleótidos (Norris et al., 2016).
El intermediario triosa fosfato de la glucólisis forma la porción de glicerol en los TAG (Edinburgh et al., 2017). Por otro lado, el C3H3O3 y los intermediarios del ciclo de Krebs suministran los esqueletos carbonados para la síntesis de aa (Valdebenito et al., 2016) y la acetil-Coenzima A es el precursor de AGNE, COL y hormonas esteroideas (Edinburgh et al., 2017). La gluconeogénesis sintetiza C6H12O6 a partir de lactato, aa y glicerol (Cantalapiedra et al., 2015; Campos et al., 2018), en el citoplasma y la mitocondria de los hepatocitos (Chen et al., 2016; Qaid y Abdelrahman, 2016). En esta ruta se consumen seis ATP por cada C6H12O6 producida (Gebreegziabher, 2016) y el propionato CH3-CH2- COOH es el único AGV glucogénico (Wallace et al., 2017).
La importancia de la glucogénesis en rumiantes (figura 4), se debe a que su organismo absorbe cantidades pequeñas de C6H12O6 por el tracto digestivo y su capacidad de almacenar glucógeno en el hígado es limitada (Qaid y Abdelrahman, 2016).
Metabolismo de Ácidos Grasos en Rumiantes
El metabolismo de los lípidos depende principalmente de ácidos grasos y COL (Watts y Ristow, 2017). La fuente de AGNE de cadena larga es proporcionada por la dieta o por síntesis de novo a partir de acetil-Coenzima A, que se deriva de monosacáridos o esqueletos carbonados de aa (Walther y Farese Jr, 2012). La síntesis de ácidos grasos inicia en la mitocondria con la formación de acetil-Coenzima A, a partir de la oxidación de CH3-COOH y CH3-CH2-CH2-COOH (Vargas, 2019). Dentro de la mitocondria, se produce acetil-Coenzima A; sin embargo, la membrana mitocondrial es impermeable a su paso. Por lo tanto, se requiere del sistema tricarboxilato y de la acción de citrato sintetasa para convertir acetil-Coenzima A en citrato y permitir su paso al citoplasma celular (Civeira et al., 2013; Nunes-Nesi et al., 2013).
Una vez en el citoplasma, el citrato es trasformado nuevamente en acetil-Coenzima A por medio de ATP-citrato liasa, obteniéndose además oxaloacetato y adenosina difosfato (ADP) (Walther y Farese Jr, 2012). Como el proceso para la síntesis de ácidos grasos es endergónico (acumula energía a partir de carbonos), el acetil-Coenzima A presenta carboxilación [se estructura un grupo carboxilo (COOH) en la molécula], a través de su unión con HCO - en una reacción catalizada por acetil-Coenzima A carboxilasa (García et al., 2014).
El oxaloacetato es reducido por malato deshidrogenasa a malato, y este a su vez, es convertido en C3H3O3 por malato deshidrogenasa, dando a la donadora de electrones nicotinamida adenina dinucleótido fosfato en su forma reducida (NADPH+H +) (Watts y Ristow, 2017; Vargas, 2019). A partir de malonil-Coenzima A, la síntesis de ácidos grasos se realiza por elongación, mediante ácido graso sintasa (Du et al., 2018). Este complejo proteico efectúa síntesis, reducción, deshidratación, y nuevamente reducción, condensando los grupos de malonil-Coenzima A con acetil-Coenzima A (Civeira et al., 2013; Norris et al., 2016). En la elongación se van añadiendo grupos de dos carbonos al ácido graso, obteniendo palmítico (C16:0) como ácido graso final (Shi et al., 2018).
Los ácidos grasos (figura 5), se pueden oxidar a acetil-Coenzima A mediante β-oxidación mitocondrial, o esterificarse con glicerol para formar TAG y funcionar como la principal reserva energética del organismo (Osorio et al., 2015). La síntesis de TAG inicia con la formación de glicerol-3-fosfato (Fong et al., 2016), posteriormente acil-Coenzima A graso sintasa activa ácidos grasos y tres de ellos se esterifican a la molécula (Civeira et al., 2013).
En el catabolismo de TAG se hidrolizan los enlaces éster en C1 o en C3, obteniendo AGNE. Reacción catalizada por lipasa sensible a hormona (McFadden, 2020). Los AGNE se transportan en el torrente sanguíneo, mediante unión no covalente con ALB, donde son captados y oxidados por miocitos o hepatocitos, o almacenados por adipocitos (Edinburgh et al., 2017). La β-oxidación se realiza en la matriz mitocondrial (Morita et al, 2016), llevándose a cabo mediante, la activación de ácidos grasos por medio de tiosinasa en acil-Coenzima A (Walther y Farese Jr, 2012); este proceso requiere ATP para formar adenilil (Fukao et al., 2014). El acil-Coenzima A activado entra a la matriz mitocondrial por medio de la carnitina palmitoiltransferasa (Nunes-Nesi et al., 2013; Morita et al, 2016), y se oxida por medio de acil-Coenzima A graso deshidrogenasa (Houten y Wanders, 2010). Los átomos de hidrógeno son aceptados por el dinucleótido de flavina-adenina (FAD) que se reduce a FADH2 (Norris et al., 2016). Posteriormente, enoil-Coenzima A hidratasa introduce H2O en el doble enlace recién formado entre C2 y C3 (Kong et al., 2017) y β-hidroxiacil Coenzima A deshidrogenasa forma al 3-cetoacil-Coenzima A (Walther y Farese Jr, 2012; Martines et al., 2017). Los dos átomos eliminados se transfieren a NAD+ generando NADH+H+ (Kohan et al., 2015).
Por último tiolasa divide el C1 y C2 del 3-cetoacil-Coenzima A, liberando acetil-Coenzima A (Martines et al., 2017), esto acorta la cadena de acil-Coenzima A de dos carbonos, necesitándose otra Coenzima A, para finalizar la molécula recién acortada (Kong et al., 2017). Estos pasos se repiten hasta dejar un acil-Coenzima A de cuatro carbonos,donde se repiten los cuatro pasos, sólo que en vez de liberarse un acetil-Coenzima A se liberan dos (Civeira et al., 2013).
Cuando se trata de un ácido graso impar la penúltima repetición deja un acil-Coenzima A graso de cinco carbonos y éste se somete a los cuatro pasos anteriores, pero los dos pasos finales dan una molécula de acetil-Coenzima A y una molécula de propionil- Coenzima A de tres carbonos (Houten y Wanders, 2010). La acetil-Coenzima A como producto de la β-oxidación de los ácidos grasos, puede tener tres destinos: a) entrar al ciclo de Krebs para oxidarse hasta CO2 y H2O para la liberación de energía (Fukao et al., 2014; Panov et al., 2014); b) fungir como precursor para la síntesis de COL y otros esteroides (Walther y Farese Jr, 2012) y c) participar en la cetogénesis (Watts y Ristow, 2017). Los cuerpos cetónicos acetoacetato (AcAc), β-HBA y acetona (Garzón y Espinosa, 2018), sirven como sustrato para la producción de ATP (McFadden, 2020). Se sintetizan en el hígado, en concentraciones bajas, pero cuando disminuye la C6H12O6 intracelular su síntesis se eleva (Norris et al., 2016).
La cetogénesis tiene lugar en la matriz mitocondrial (Fukao et al., 2014). Cuando las reservas hepáticas de glucógeno disminuyen, se estimula la actividad de la carnitina palmitoiltransferasa, provocando el transporte de AGNE hacia el interior de la mitocondria hepática (Walther y Farese Jr, 2012), donde se realiza una serie de sucesivas β- oxidaciones que conducen a la formación de acetil-Coenzima A (McFadden, 2020). Esta molécula se combina con oxaloacetato para su ingreso al ciclo de Krebs (García et al., 2015). Si esta oxidación es completa se liberará CO2 y átomos de hidrógeno, que donarán sus electrones para efectuar reacciones óxido reducción, que culminarán con la formación de H2O y ATP (McFadden, 2020).
Si el oxaloacetato se reduce el acetil-Coenzima A, se acumula dentro de la mitocondria hepática (Walther y Farese Jr, 2012); por lo que dos moléculas de acetil-Coenzima A reaccionan para formar acetoacetil-Coenzima A, catalizada por tiolasa (Fukao et al., 2014). El acetoacetil-Coenzima A se une con otra molécula de acetil-Coenzima A para formar β-hidroxi-β-metilglutaril-CoA, catalizada por 3-hidroxi-3-metilglutaril-CoA sintasa (Norris et al., 2016). Por último, la molécula, se metaboliza en AcAc (figura 5) y sale de la mitocondria al citoplasma, donde puede reducirse en β-HBA o descarboxilarse, hasta acetona (García et al., 2015).
Metabolismo de Aminoácidos en Rumiantes
El metabolismo de los aa involucra la transaminación y desaminación (Dong et al., 2016), reacciones necesarias para el anabolismo y catabolismo de las proteínas (Golshan et al., 2019). Los aa Arg, histidina (His), isoleucina (Ile), leucina (Leu), lisina (Lys), metionina (Met), fenilalanina (Phe), treonina (Thr), triptófano (Trp) y valina (Val), son producidos en su mayoría por fermentación ruminal (Zhou et al., 2019). Los aa están compuestos por un grupo amino (-NH 2) y un grupo COOH; además de una cadena lateral R, que les da propiedades hidrofílicas, hidrofóbicas, ácidas, básicas y aromáticas (Rostom y Shine, 2018). La transaminación se lleva a cabo por aminotransferasas, el grupo -NH2 se transfiere de un aa ácido a un aa cetoácido (Zhou et al., 2019; Batista et al., 2016). Las aminotransferasas se localizan en el citoplasma y mitocondrias, teniendo dos tipos de especificidad: I) el tipo de aa que dona el -NH2 (Emery, 2015) y II) el cetoácido que acepta el -NH2 (Dong et al., 2016). Aunque las enzimas varían dependiendo del tipo de aa que unen, la mayoría usan glutámico (Glu) como donador de -NH2 (Rostom y Shine, 2018).
Estas reacciones requieren del cofactor piridoxal fosfato (PLP) (Witus et al., 2013). En la desaminación oxidativa los aa pierden el -NH2, reacción catalizada por glutamato deshidrogenasa (Dong et al., 2016). Los esqueletos carbonados resultantes se degradan para obtener uno de los siete productos metabólicos posibles: acetil-Coenzima A, acetoacetil-Coenzima A, C3H3O3, cetoglutarato, succinil-Coenzima A, fumarato u oxaloacetato (Rostom y Shine, 2018). Los aa que se degradan de acetil-Coenzima A, a acetoacetil-Coenzima A se conocen como cetogénicos (Lys y Leu) (Batista et al., 2016). Los esqueletos carbonados de los aa glucogénicos se degradan a C3H3O3 o un intermediario del ciclo de Krebs, pero también pueden convertirse en C6H12O6 mediante glucogénesis (Emery, 2012). El NH3 resultante de la desaminación de los aa (figura 6) se transporta a los hepatocitos periportales para participar en la ureagénesis (García et al., 2014).
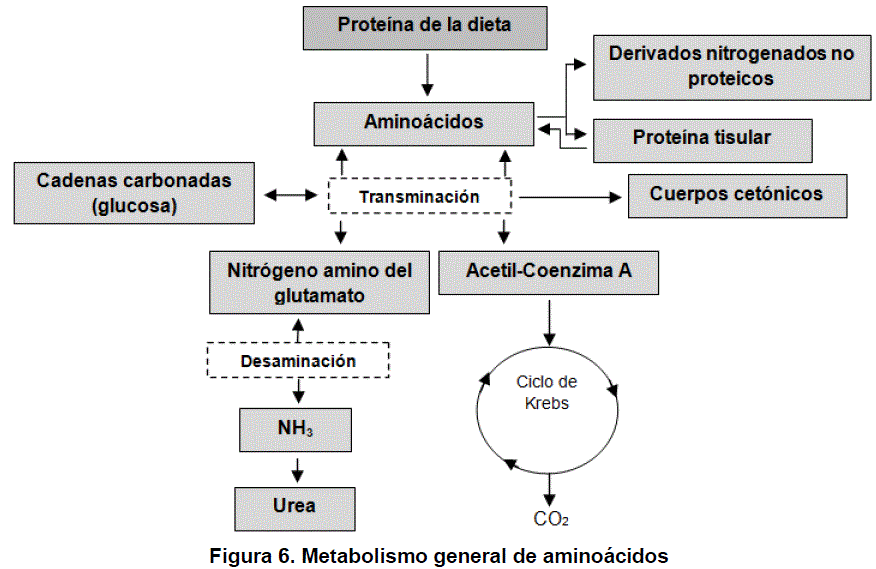
Fuente: información sintetizada de (Golshan et al., 2019)
Figura 6 . Metabolismo general de aminoácidos
CONCLUSIÓN
Los elementos científicos presentados sobre el anabolismo y el catabolismo de los nutrientes, manifiestan que la absorción intestinal de glucosa en los rumiantes es limitada. Por lo tanto, la microbiota ruminal juega un papel importante en la transformación, asimilación y síntesis de cada uno de los monómeros bioquímicos; elementos de vital importancia en la glucogénesis, proteogénesis, ureagénesis, lipogénesis y cetogénesis; procesos metabólicos que confieren información sobre las alteraciones y los ajustes homeostáticos en los rumiantes.











 text in
text in 




