Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista bio ciencias
On-line version ISSN 2007-3380
Revista bio ciencias vol.6 Tepic Jan. 2019 Epub Mar 18, 2020
https://doi.org/10.15741/revbio.06.e537
Artículos Originales
Análisis de caracteres morfológicos y fragmentos RAPDs de doce especies de la Familia Crassulaceae
1Universidad Autónoma del Estado de Morelos, Facultad de Ciencias Agropecuarias. Av. Universidad 1001, Cuernavaca, Morelos, C. P. 62210, México.
2Universidad Autónoma del Estado de Morelos, Escuela de Estudios Superiores de Xalostoc. Av. Nicolás Bravo s/n, Parque Industrial Cuautla, C.P. 62717 Xalostoc, Ayala, Morelos. México.
La familia Crassulaceae es la más representativa de México debido al alto endemismo de la mayoría de sus especies y por su significado histórico-cultural; son cotizadas y valoradas en el mercado internacional, esto ha provocado una intensa recolecta y saqueo de plantas, frutos y semillas de manera ilegal, con fines de venta y registro en otros países. Las características morfológicas se utilizan para estudiar la diversidad genética, identificar plantas cultivadas y el método de la caracterización molecular mide la variabilidad genética y la diversidad genética con la finalidad de conservar y proteger los recursos genéticos. Con base en lo anterior, el objetivo de la investigación fue la caracterización morfológica y molecular mediante RAPD (Amplificación Aleatoria de DNA Polimórfico) de doce especies de la familia Crassulaceae. Se estudiaron doce especies de la familia Crassulaceae, Echeveria runyonii, E. perle, E. agavoides, E. pumila var. glauca x E. Walther., E. elegans, E. pulvinata, E. derembergii, E. setosa, Pachyphytum oviferum, Sedum clavatum, S. nussbaumerianum, y S. palmeri, las cuales son especies endémicas. Para la caracterización morfológica se usaron algunas características utilizadas para la descripción de plantas de algunas especies de la familia Crassulaceae. Se realizó la caracterización molecular mediante RAPD. Los datos se procesaron utilizando el Sistema de Análisis Multivariado y Taxonómico Numérico (NTSYSpc 2.1). Los caracteres cuantitativos separaron a las doce especies en cinco grupos según el análisis de conglomerados. Se obtuvieron seis grupos con el análisis RAPDs. Las especies con mayor similitud genética fueron: E. runyonii y E. derembergii, lo que indica mayor posibilidad genética de cruzamiento para hibridación interespecífica.
Palabras clave: Especies endémicas; Análisis morfológico y molecular; agrupamiento; y análisis multivariado
The Crassulaceae family is the most representative of Mexico due to the high endemism of most of its species and to its historical-cultural significance. They are prized and valued on the international market, causing an intense and illegal harvest and sacking of plants, fruits and seeds, with the purpose of selling and recording them in other countries. Morphological characteristics are used to study genetic diversity, to identify cultivated plants and the method of molecular characterization measures genetic variability and diversity with the purpose of conservation and protection of genetic resources. On that basis, the objective of this research work was the morphological and molecular characterization by RAPD (Random Amplified Polymorphic DNA) of twelve species of the Crassulaceae family. Twelve species of the family Crassulaceae were studied: Echeveria runyonii, E. perle, E. agavoides, E. pumila var. glauca x E. Walther., E. elegans, E. pulvinata, E. derembergii, E. setosa, Pachyphytum oviferum, Sedum clavatum, S. nussbaumerianum, and S. palmeri, which are endemic species. For morphological characterization, some characteristics used for plant description of some species from the Crassulaceae family were employed. Molecular characterization was performed by RAPD. Data were processed using the Numerical Taxonomic and Multivariate Analysis System (NTSYSpc 2.1). Quantitative characters separated the twelve species into five groups according to the cluster analysis. Six groups were obtained with the RAPD analysis. The most genetically similar species were: E. runyonii and E. derembergii, indicating more genetic crossing possibilities for interspecific hybridization.
Key words: Endemic species; Morphological and molecular analysis; clustering; multivariate analysis
Introducción
La familia Crassulaceae es la más representativa de México, debido al alto endemismo de la mayoría de sus especies, por su significado histórico-cultural y por el papel que desempeñan en la estructura y función de los ecosistemas áridos del país (Toledo, 1988); tiene gran diversidad genética, la cual se manifiesta con variación en el porte de la planta, tamaño, forma, espesor y color de las hojas, tamaño y forma de la inflorescencia (Borys et al., 2005).
México es considerado el segundo centro de diversidad, con representación de aproximadamente 300 (89.8 %) especies endémicas (t’ Hart, 1997), siendo este el porcentaje de endemismo más alto dentro de las familias y géneros en este país (Thiede, 1995). México es un importante lugar de origen de gran diversidad de especies vegetales (Colinas, 2003).
Debido a su forma inigualable y la belleza de sus flores, las Crassulaceae son cotizadas y valoradas en el mercado internacional; lo anterior ha provocado una intensa recolecta y saqueo de plantas, frutos y semillas de manera ilegal, con fines de venta y registro en otros países (Bravo-Hollis, 1978). La conservación y utilización de los recursos genéticos es de importancia estratégica para la humanidad; las diferentes especies vegetales han estado sometidas a una activa interacción con el ambiente, lo cual ha generado un gran número de genotipos adaptados a diferentes condiciones locales, ampliando la diversidad genética (Becerra & Paredes, 2000).
En el estado de Morelos están presentes el 70.5 % de las familias de plantas vasculares de México, el 36.4 % de los géneros y el 14.2 % de las especies. Estas cifras revelan que el estado es un reservorio importante de diversidad de todos los grupos vegetales y a distintos niveles de jerarquía taxonómica (Rendón & Fernández, 2007).
Las características morfológicas se han venido utilizando para estudiar la diversidad genética, identificar plantas cultivadas, conservar los recursos genéticos (Yasmin et al., 2006), así como también el método de la caracterización molecular que mide la variabilidad genética directamente a nivel de la molécula de DNA (Watson & Eyzaguirre, 2002). Los marcadores moleculares se han convertido en herramientas importantes en estudios de diversidad genética, como los RAPDs (Bered et al., 2005 y Yasmin et al., 2006).
Las asociaciones de marcadores genéticos por medio del análisis de agrupamiento se consideran muy útiles ya que incluyen un gran número de caracteres y porque es una clasificación jerárquica que puede ser usada para determinar los niveles umbrales de similitud entre especies (Singh, 1999). También, indican la formación de agrupaciones muy distintas a las consideradas a partir del planteamiento clásico basado en el análisis morfológico y anatómico (García, 2003).
Eggli (2003) y Acevedo-Rosas et al. (2004) se basaron en caracteres morfológicos para describir y caracterizar a las Crassulaceae. Van Ham et al. (1994) usaron iniciadores universales para hacer amplificación de DNA mediante PCR de tres regiones no codificantes del DNA del cloroplasto con el propósito de estudiar la variación en la longitud de la secuencia de DNA en las crasuláceas y especies relacionadas. De igual manera, Van Ham & t’ Hart (1998), analizaron una zona de restricción del DNA del cloroplasto de 44 especies de seis subfamilias Crassulaceae utilizando 12 endonucleasas de restricción a fin de establecer las relaciones filogenéticas entre ellas.
El análisis morfológico y molecular en las Crassulaceae es de gran importancia para conocer la diversidad genética y la relación entre genotipos, ya que esto permitirá una clasificación correcta, la conservación de genotipos y la utilización de recursos genéticos. Actualmente existe un gran número de colecciones de germoplasma con genotipos de un alto valor agronómico que podrían ser usados como progenitores en los programas de mejoramiento genético; sin embargo, en muchas ocasiones se desconoce su grado de diversidad y la relación existente entre materiales, lo que dificulta su utilización. Los recursos genéticos en México, no se han aprovechado adecuadamente debido a la limitada información disponible sobre las características y la diversidad genética de muchas especies (Becerra & Paredes, 2000).
Con base en lo anterior, el objetivo de la investigación fue evaluar la diversidad y relaciones genéticas realizando la caracterización morfológica y molecular mediante RAPDs de doce especies de la familia Crassulaceae. Esta caracterización permitirá generar información complementaria para los fitomejoradores, y para una futura utilización del material genético para realizar cruzas interespecíficas.
Materiales y Métodos
El material genético estudiado fueron doce especies de la familia Crassulaceae cultivadas en maceta, Echeveria runyonii topsy-turvy (Haworthia x E. Shaviana) (México) (Maatz, 2012), E. perle von Nuremberg (E, gibbiflora var. metalica x E. potosina) (Mexico y San Luis potosi), E. agavoides Lem. (San Luis Potosi), E. pumila var. glauca x E. Walther (México), Echeveria elegans Rose (México), E. pulvinata (Puebla), E. derembergii (México) E. setosa Rose & J.A. Purpus (Puebla), Pachyphytum oviferum J.A. Purpus (San Luis Potosi), Sedum clavatum (México), S. nussbaumerianum Bitter (México), S. palmeri S. Watson (México) (Sajeva & Constanzo, 1997) (Figura 1).

Figura 1 Especies de la familia Crassulaceae estudiadas. a: Echeveria runyonii topsy-turvy, b: E. perle von nuremberg, c: E. agavoides Lem., d: E. pumila var. glauca x E. Walther., e: E. elegans Rose, f: E. pulvinata, g: E. derembergii, h: E. setosa Rose & J.A. Purpus, i: Pachyphytum oviferum J.A. Purpus, j: Sedum clavatum, k: S. nussbaumerianum Bitter, l: S. palmeri S. Watson.
Las plantas se obtuvieron de viveros comerciales, fueron seleccionadas por su porte, color y forma de las hojas, ya que son especies que se distinguen por su valor decorativo, así como por la capacidad que tienen de producir hijuelos, además, de ser algunas de las especies que más se comercializan. Se evaluaron 30 plantas para el análisis morfológico, de cada una de las especies señaladas.
Caracterización morfológica
La evaluación se realizó al momento de la floración de las 30 plantas. Las unidades de medición en cm se midieron con regla de 30 cm y con vernier digital para obtener mm; y el color de los órganos se evaluó con una carta de colores (RHS color chart®). Las flores se observaron al microscopio para evaluar el número de estambres y carpelos (Tabla 1). Se usaron algunas características señaladas por Martínez-Ávalos & Mora-Olivo (2000), García-Ruíz & Pérez-Calix (2007) utilizados para la descripción de algunas especies de la familia Crassulaceae.
Tabla 1 Caracteres morfológicos cualitativos y cuantitativos evaluados en doce especies de la familia Crassulaceae.
| Traits | Quantitative | Qualitative |
|---|---|---|
| Form of the plant | Height (cm) and Diameter (mm) | Rosette and Branch |
| Stem | Number of sheets | Phylotaxy |
| Leaves | Length (cm) and width (cm), thickness at the base and middle part (mm) | Apex shape, Petiole or pseudopeciolus, Color |
| Inflorescence | Number per plant, length (cm), flowers per inflorescence, peduncle length (cm), peduncle diameter (mm). | Origin: basal, middle, apical, ramifications: primary, secondary. Type of inflorescence. |
| Bracts | Number, length (mm), width (mm), base thickness and middle part (mm). | Phylotaxy, bract shape, apex shape and base shape |
| Flower | Length (mm), basal diameter (mm) and pedicel length (mm). | |
| Chalice | Form, Free or merged | |
| Separations | Number of sepals, length (mm) and width (mm). | |
| Corolla | Diameter (mm) | Shape |
| Petals | Number | Color |
| Stamens | Number | |
| Carpels | Number, and length of carpels (mm) | Free or merged |
| Time of flowering | Date |
Caracterización molecular
Se utilizó una planta por especie para obtener el DNA. Se pesaron 400 mg de tejido foliar de cada una de las doce especies, para extraer el DNA de acuerdo a la metodología reportada por Andrade-Rodríguez et al. (2004); en el último paso, el DNA obtenido se disolvió en 25 µL de 0.1 TE (1 mM Tris-HCl pH 8.0, 0.1 mM EDTA) con 20 ng∙µL-1 de RNAse, calentado a 37 °C por 40 min. y almacenado a -20 °C hasta su utilización.
La integridad del DNA de todas las plantas se estimó mediante electroforesis en gel de agarosa (ultrapura GIBCO®) al 1 %, el gel se tiño con bromuro de etidio (1 µg mL-1 ). En los pozos del gel se cargaron 10 µL constituidos por 1 µL de muestra de DNA, 7 µL de agua destilada deionizada autoclaveada y 1 µL de buffer de carga (40 % glicerol, 0.1 % azul de bromofenol, TE pH 8.0). La electroforesis se realizó a temperatura ambiente con buffer TAE (0.04 M Tris-base, ácido acético glacial, 1 mM EDTA) durante dos horas, tiempo en que la banda de referencia migró 7.0 cm aproximadamente. Transcurrido este tiempo, las muestras de DNA se visualizaron y se foto documentaron en un analizador de geles (Syngene® GVM20).
La concentración del DNA y su pureza fueron cuantificadas a partir de la solución de DNA obtenida, para cada muestra se preparó una dilución de 1:150 [5 µL de DNA de la muestra más 995 µL de TE 10x (10 mM Tris HCl pH 8.0, 1 mM EDTA pH 8.0)], para cuantificar la concentración y pureza del DNA por medio de un espectrofotómetro Genesys 6® de luz ultravioleta. La concentración se estimó mediante la fórmula:
Donde:
OD260 = Densidad óptica de la solución de DNA leída a la longitud de onda de 260 nm.
DF = Factor de dilución.
50 µg µL-1 = Concentración de DNA determinada para un valor de 1.0 a 260 nm.
La pureza se evaluó como la proporción de las lecturas a longitudes de onda de 260 y 280 nm (260/280). Los valores entre 1.8 y 2.0 de densidad óptica (OD) indican alto grado de pureza. Valores menores a 1.8 indican contaminación de la muestra de DNA con proteínas y/u otros elementos absorbentes de luz UV; mientras que valores mayores indican contaminación por cloroformo, fenol u otra sustancia orgánica. Una vez obtenida la lectura para cada muestra de DNA se prepararon las soluciones de trabajo a 20 ng µL-1.
Análisis RAPDs
Se realizó amplificación (TECHNE TC-412®) de DNA con 68 iniciadores (Operon Technologies Inc.) de secuencia aleatoria de diez nucleótidos del kit OPA, OPB, OPC, más ocho iniciadores: SAP, de estos, se eligieron 20 iniciadores (Tabla 2) con base en la cantidad y brillo de los fragmentos RAPDs amplificados, mismos que fueron utilizados para efectuar el estudio de similitud genética.
Tabla 2 Secuencia de los veinte iniciadores seleccionados para la caracterización molecular de doce especies de la familia Crasulaceae.
| Primers | Key | Sequence of bases |
|---|---|---|
| 2 | OPA02 | 5´ TGC CGA GCT G 3´ |
| 4 | OPA04 | 5´ AAT CGG GCT G 3´ |
| 7 | OPA07 | 5´ GAA ACG GGT G 3´ |
| 8 | OPA08 | 5´ GTG ACG TAG G 3´ |
| 9 | OPA09 | 5’ GGG TAA CGC C 3’ |
| 10 | OPA10 | 5´ GTG ATC GCA G 3´ |
| 1 | OPB-01 | 5´ GTT TCG CTC C 3´ |
| 6 | OPB-06 | 5´ TGC TCT GCC C 3´ |
| 7 | OPB-07 | 5´ GGT GAC GCA G 3´ |
| 8 | OPB-08 | 5´´GTC CAC ACG G 3´ |
| 10 | OPB-10 | 5´ CTG CTG GGA C 3´ |
| 12 | OPB-12 | 5´ CCT TGA CGC A 3´ |
| 18 | OPB-18 | 5´ CCA CAG CAG T 3´ |
| 19 | OPB-19 | 5´ ACC CCC GAA G 3´ |
| 8 | OPC08 | 5´ TGG ACC GGT G 3´ |
| 9 | OPC09 | 5´ CTC ACC GTC C 3´ |
| 13 | OPC13 | 5´ AAG CCT CGT C 3´ |
| 16 | OPC16 | 5´ CAC ACT CCA G 3´ |
| 25 | SAP-03 | 5´ TGG GAC CTC C 3´ |
| 26 | SAP-04 | 5´ GGA GCT ACC T 3´ |
La mezcla de reacción consistió de 10 μL de dNTPs (5 µM de cada dNTP, GIBCO BRL), 2.5 µL de amortiguador PCR (10x), 1.5 µL MgCl2 (3 mM), 2.0 µL iniciador (10 pmol µL−1), 0.3 µL (1.5 U) de taq DNA polimerasa nativa (Invitrogen), 4 µL DNA (20 ng µL−1) ajustando a 25 µL con 4.7 µL agua desionizada esterilizada.
El programa que se usó para la amplificación mediante la reacción en cadena de la polimerasa (PCR) consistió en un ciclo inicial de predesnaturalización de DNA a 94° C por 4 min y 35 ciclos integrados por las etapas siguientes: 94° C por 1 min para separar bandas de DNA, 36 °C por 1 min. para alineación de iniciador, 72 °C por 2 min. para extensión o polimerización y un ciclo de extensión final a 72 °C por 10 min.
La separación de fragmentos amplificados se hizo mediante electroforesis en gel de agarosa (Invitrogen) 1.5 % (p/v) con bromuro de etidio (10 μg L−1). La electroforesis se realizó a 75 V por 4 h. Los geles con los fragmentos de DNA amplificados se foto documentaron en un Sistema de Documentación y Análisis de Electroforesis (Syngene GVM20®, Kodak®).
Análisis estadístico
El tamaño de los fragmentos de DNA producidos por los iniciadores RAPDs se obtuvo utilizando el programa Gene tools Labworks 4.0®. La presencia de marcadores RAPDs se determinó mediante el análisis de los perfiles electroforéticos generados por cada una de las especies de Crassulaceae. Para la construcción del dendrograma, los datos se analizaron con base en la presencia (indicada por 1) o la ausencia (indicada por 0) de fragmentos amplificados con los iniciadores decámeros seleccionados.
Los datos morfológicos y moleculares se procesaron utilizando el Sistema de Análisis Multivariado y Taxonómico Numérico (NTSYSpc 2.1). El coeficiente de distancia empleado para el agrupamiento fue la distancia Euclidiana. Los análisis de agrupamiento se realizaron sobre la relación de matrices con el método de ligamiento promedio aritmético de grupos de pares no ponderados (siglas en inglés, UPGMA) (Avise, 1994). El programa NTSYS propone el punto de corte en el dendograma de acuerdo con una evaluación de congruencia comparándolo con una gráfica tridimensional de proyección para observar el número de grupos formados.
Resultados y Discusión
Caracterización morfológica
Los caracteres cualitativos permitieron observar dos tipos de características en las especies estudiadas, el primero son las características discriminativas con las cuales se caracteriza a cada una de las especies y las hace distintivas, lo cual manifiesta la diversidad morfológica de las Crassulaceae; en estas características se incluye hábito de crecimiento, forma de la hoja, color de la hoja, origen de la inflorescencia, tipo de inflorescencia, forma de las brácteas y color; el segundo tipo de características son las de similitud que comparten entre estas especies, como forma del ápice, forma de la base, ramificación de la inflorescencia, filotaxia de las brácteas, forma del cáliz, forma de la corola y pétalos, cárpelos y época de floración, debido a que son caracteres cualitativos no se corrió un análisis multivariado.
Los cuatro primeros componentes principales explicaron el 81 % de la variabilidad morfológica observada. El componente principal 1 (PC1) con un valor propio de 9.795 explicó el 40.81 % de variación, donde las variables de mayor contribución fueron largo de hoja, grosor en la base de la hoja, grosor en la parte media de la hoja, diámetro del pedúnculo, longitud de bráctea, longitud de flor, diámetro basal, longitud de sépalos, ancho de la corola y longitud de carpelos. El componente principal 2 (PC2), explica 21.07 % y las variables que describen esta variación dentro de este componente son longitud de inflorescencia, longitud del pedúnculo, ancho de la bráctea, grosor de la base de la bráctea, grosor de la parte media de la bráctea. En el componente principal 3 (PC3), las variables altura, número de hojas y número de flores por ramificación son las que explican 13.37 % de la variabilidad total. El componente principal 4 (PC4) la variable longitud del pedicelo explica el 6.13 % de la varianza (Tabla 3).
Tabla 3 Proporcion de la varianza global, vectores y valores propios del análisis de componentes principales (PC) de los 24 caracteres morfológicos que describen la variacion en 12 especies de Crassulacea.
| Trait | PC1 | PC2 | PC3 | PC4 |
|---|---|---|---|---|
| Height | 0.481 | 0.289 | 0.699 | 0.216 |
| Diameter | -0.659 | -0.463 | -0.304 | -0.286 |
| Number of sheets | -0.180 | -0.325 | -0.728 | 0.255 |
| Leaf length | -0.679 | -0.528 | 0.062 | -0.263 |
| Sheet width | -0.613 | -0.563 | 0.208 | -0.358 |
| Thickness at the base of the leaf | -0.848 | -0.404 | 0.141 | 0.022 |
| Thickness in the middle part of the leaf | -0.844 | -0.116 | 0.004 | 0.189 |
| Number of inflorescences | 0.068 | -0.372 | -0.337 | -0.167 |
| Inflorescence length | -0.506 | -0.819 | -0.054 | 0.112 |
| Number of flowers per branch | -0.336 | -0.411 | 0.768 | -0.179 |
| Pedicle length | -0.396 | -0.780 | 0.058 | 0.255 |
| Pedicle diameter | -0.771 | 0.351 | -0.131 | -0.200 |
| Number of bract | -0.379 | -0.566 | 0.634 | -0.172 |
| Bract length | -0.836 | 0.126 | 0.261 | 0.295 |
| Bract width | -0.655 | 0.594 | 0.326 | -0.221 |
| Thickness of the base of the bract | -0.523 | 0.666 | 0.341 | -0.084 |
| Thickness of the middle part of the bract | -0.593 | 0.662 | 0.227 | 0.034 |
| Flower length | -0.863 | 0.036 | -0.089 | 0.400 |
| Basal diameter | -0.834 | 0.220 | -0.383 | -0.017 |
| Pedicel length | -0.485 | -0.125 | 0.249 | 0.719 |
| Sepals length | -0.744 | 0.450 | -0.238 | -0.041 |
| Sepals width | -0.636 | 0.390 | -0.386 | -0.074 |
| Corolla width | -0.847 | 0.276 | -0.291 | -0.092 |
| Carpels length | -0.679 | 0.325 | 0.236 | -0.038 |
| Own values | 9.795 | 5.058 | 3.210 | 1.471 |
| Explained variance (%) | 40.814 | 21.077 | 13.378 | 6.132 |
| Accumulated variance (%) | 40.814 | 61.891 | 75.269 | 81.401 |
López-Gutiérrez et al., (2015) realizaron la comparación morfológica entre poblaciones silvestres y manejadas de Opuntia atropes (Cactaceae) en Michoacán, México; las variables morfológicas evaluadas permitieron que los componentes principales separaran a los individuos cultivados de los no cultivados.
En otra investigación realizada por Martínez-Moreno et al., (2006) reportan que los tres componentes principales muestran el 84.16 % de la variabilidad total existente en los individuos, misma que se obtiene con la medición de las 18 variables. En otra investigación realizada por Morales et al., (2016) reportan que los primeros dos PC explicaron el 91 % de la varianza con nueve variables. Ambas investigaciones son congruentes al porcentaje reportado en este estudio, con el 81 % de varianza acumulada con 24 variables morfológicas evaluadas.
Los caracteres cuantitativos separaron a las doce especies en cinco grupos según el análisis de conglomerados, con un coeficiente de distancia taxonómica media de 0.28. En el primer grupo se ubicaron siete especies: E. runyonii, P. oviferum, E. elegans, E. perle, E. pumila, E. pulvinata, y E. agavoides, siendo donde se agruparon mayor número de especies. Las características generales compartidas por las especies del primer grupo son las hojas arrosetadas, la forma sagitada-elíptica de sus hojas, y sobre todo una característica distintiva entre ellas es la inflorescencia racimo de racimos y con disposición de cima unípara escorpoidea. El segundo grupo se integró por E. derembergii y E. setosa, estas especies son similares en la forma de la roseta; sin embargo, E. setosa presenta tricomas en las hojas. El tercer grupo correspondió a S. clavatum, separandose de las Echeverias y Pachyphytum, que aunque tiene similitud con ellas por compartir la morfología de hojas arrosetadas forma de la flor, se distinguió por sus flores pequeñas (Figura 2).

Figura 2 Fenograma de doce especies de la familia Crassulaceae, construido por el método de UPGMA (ligamiento promedio) a partir de 35 caracteres cuantitativos de la planta. El coeficiente de distancia se refiere a la Distancia Taxonómica Media. Línea punteada: referencia para formación de grupos.
El cuarto grupo se conformó por S. nussbaumerianum, esta especie se agrupa con S. palmeri (del quinto grupo), a una distancia taxonómica de 0.39; éstas son similares en hábito de crecimiento e inflorescencias; estas dos especies de Sedum a su vez se unen con las otras diez especies de crasuláceas con un coeficiente de distancia taxonómica media de 0.51 siendo estas las más lejanas de las otras especies de crasuláceas del dendograma (Figura 2).
Como se observa, P. oviferum se agrupó con seis especies de Echeveria, esto fue debido a que comparten características similares en cuanto a forma de las hojas, color, tamaño e inflorescencia, pero cabe mencionar que su crecimiento no es en roseta, sino que presenta ramificación ascendente.
Las especies más similares de acuerdo con el coeficiente de distancia taxonómica media fueron E. runyonii con P. oviferum, en contraste, las especies más distintas al resto fueron S. nussbaumerianum y S. palmeri, que entre sí tienen un coeficiente de distancia taxonómica media de 0.39.
Conforme a los resultados, se puede decir que las Crassulaceae son una familia muy compleja, debido a que es fácil su hibridación y puede haber cruzamientos no solo entre especies del mismo género sino de diferentes géneros, siendo la posible causa de que compartan similitud morfológica entre géneros (Jones et al., 2010, Jimeno-Sevilla et al., 2013, Jimeno-Sevilla et al., 2014 y Rodríguez-Rojas et al., 2015). También es probable que la clasificación taxonómica específica actual no sea la correcta. En los análisis filogenéticos de Crassulaceae repetidamente se han encontrado cladogramas plagados de politomías carentes de resolución (Acevedo-Rosas et al., 2004).
Caracterización molecular
Se seleccionaron 20 iniciadores para el análisis molecular, por presentar el mayor número de productos amplificados, bandas más brillantes y mejor definidas. Los iniciadores generaron fragmentos que variaron de 231 a 4,774 pb.
Los iniciadores seleccionados para las doce especies de la familia Crassulaceae produjeron 594 fragmentos RAPDs, el 99 % de los cuales fueron polimórficos y permitieron diferenciar a las doce especies con 76 marcadores. Los iniciadores OPA-09, OPB-18, y SAP-03 no generaron fragmentos marcadores (Tabla 4).
Tabla 4 Iniciadores usados para estudiar doce especies de la familia Crassulaceae.
| Primers | Amplified fragments | Polymorphism (%) | Markers fragments (pb) |
|---|---|---|---|
| OPA02 3′TGCGCCCTTC 5’ | 28 | 100 | 4171,4244, 1050 |
| OPA04 3′AATCGGGCTG 5’ | 38 | 100 | 3478, 3058, 1453, 641,585, 334, 240 |
| OPA07 3′GAAACGGGTG 5’ | 32 | 100 | 3211, 2634, 525 |
| OPA08 3′GTGACGTAGG 5’ | 32 | 100 | 1919, 1259, 1013, 801, 745, 395 |
| OPA09 3′ GGGTAACGCC 5’ | 22 | 100 | 0 |
| OPA10 3′GTGATCGCAG 5’ | 26 | 100 | 1858,1732,1618 |
| OPB01 5’ GTTTCGCTCC 3’ | 21 | 96 | 3095, 1229, 1066, 308 |
| OPB06 3′ TGCTCTGCCC 5’ | 25 | 96 | 2528,1956, 2526, 887, 1581, 373 |
| OPB07 3′GGTGACGCAG 5’ | 45 | 100 | 3135, 796, 231 |
| OPB08 5’ GTCCACACGG 3’ | 25 | 98 | 734, 694,595 |
| OPB10 3′CTGCTGGGAC 5’ | 39 | 100 | 2723, 2306, 2244,1441, 847, 1344, 267, 1303, 712, 441, 294 |
| OPB12 3′ CCTTGACGCA 5’ | 29 | 96 | 3275, 2506, 312 |
| OPB18 3′ CCACAGCAGT 5’ | 24 | 100 | 0 |
| OPB19 3′ ACCCCCGAAG 5’ | 32 | 100 | 4774,3172,1604, 4587, 1580, 1469, 636, 625, 395 |
| OPC08 3′ TGGACCGGTG 5’ | 30 | 100 | 2968, 1206, 1430 |
| OPC09 3′ CTCACCGTCC 5’ | 28 | 100 | 309 |
| OPC13 3′ AAGCCTCGTC 5’ | 24 | 96 | 2791 |
| OPC16 3′ TTTGCCCGGA 5’ | 26 | 100 | 788,353, 306, 282, 241 |
| SAP03 3′ TGGGACCTCC 5’ | 26 | 92 | 0 |
| SAP04 3′ GGAGCTACCT 5’ | 24 | 100 | 2935, 2682, 618, 333, 249 |
| Total | 594 | 99 | 76 |
Los iniciadores que generaron mayor número de marcadores fueron el OPB-10 y el OPB-19, estos permitieron caracterizar mayor número de especies. Los iniciadores que mostraron tres marcadores fueron OPA-02, OPA07, OPA-10, OPB-07, OPB-08, OPB-12, OPC-08, estos marcadores se presentaron en al menos dos especies; el OPC-13 y OPC-09 produjeron solo un fragmento marcador cada uno, y fueron útiles para caracterizar a una planta. Ningún iniciador por sí solo permitió caracterizar a todas las especies. E. agavoides y E. pulvinata fueron las que mostraron mayor presencia de marcadores (Tabla 4).
Los fragmentos RAPDs totales y polimórficos obtenidos en la caracterización de las doce especies de la familia Crassulaceae, fueron mayores a los obtenidos por Montalvo et al. (2010) quien obtuvo el total de 97 bandas de las cuales el 62.8 % fueron polimórficas y en cambio en la presente investigación se obtuvieron 594 bandas, el 99 % de las cuales fueron polimórficas. El análisis de agrupamiento obtenido por Montalvo et al. (2010), les permitió la diferenciación de todos los genotipos, pudieron definir la presencia de un grupo formado por los individuos 1, 2 y 3, mientras que el genotipo 4 estaba más alejado genéticamente, en nuestro estudio se separaron 6 grupos diferentes en donde los grupos no se comportaron de acuerdo a su clasificación taxonómica, cabe mencionar que el estudio de Montalvo et al., (2010) consto de 4 semillas de una misma especie en comparación al estudio realizado donde se compararon especies de diferente género.
La similitud de las doce especies de la familia Crassulaceae determinada con los 594 fragmentos varió de 0.576 a 0.692; de las 66 similitudes genéticas obtenidas, seis fueron menores a 0.6 y correspondieron a E. perle con E. pumilla, E. agavoides con E. elegans, E. agavoides con P. oviferum, E. pumilla con E. setosa, y E. pumila con S. palmeri; en contraste, la mayor similitud genética se obtuvo para E. runyonii con E. derembergii (0.692), E. setosa con S. palmeri (0.685), E. perle con E. agavoides (0.668), E. runyonii con E. perle (0.667), E. runyonii con S. palmeri (0.663), E. derembergii con P. oviferum (0.663), P. oviferum con S. nussbaumerianum (0.660). Se esperaba que la mayor similitud genética se tuviera para especies del mismo género, pero no fue así, dentro de los pares con mayor similitud se encuentran varios de diferentes géneros como son E. setosa con S. palmeri y P. oviferum con S. nussbaumerianum.
Las asociaciones de marcadores genéticos por medio del análisis de agrupamiento se consideran muy útiles ya que incluyen un gran número de caracteres y porque es una clasificación jerárquica que puede ser usada para decir los niveles umbrales de similtud entre especies (Singh, 1999).
El análisis del conglomerado obtenido, muestra que a un nivel de 0.65 de distancia genética se formaron seis agrupaciones: 1) E. runyonii, E. derembergii, P. oviferum, S. nussbaumerianum, siendo esta la agrupación que incluye más especies; 2) E. setosa, S. palmeri, 3) E. elegans, E. pulvinata, 4) S. clavatum, 5) E. perle, E. agavoides, y 6) E. pumila, este último grupo es el más distinto al resto, ya que es la última que se incorpora al grupo con una distancia de similitud de 0.61 con las demás especies (Figura 3).
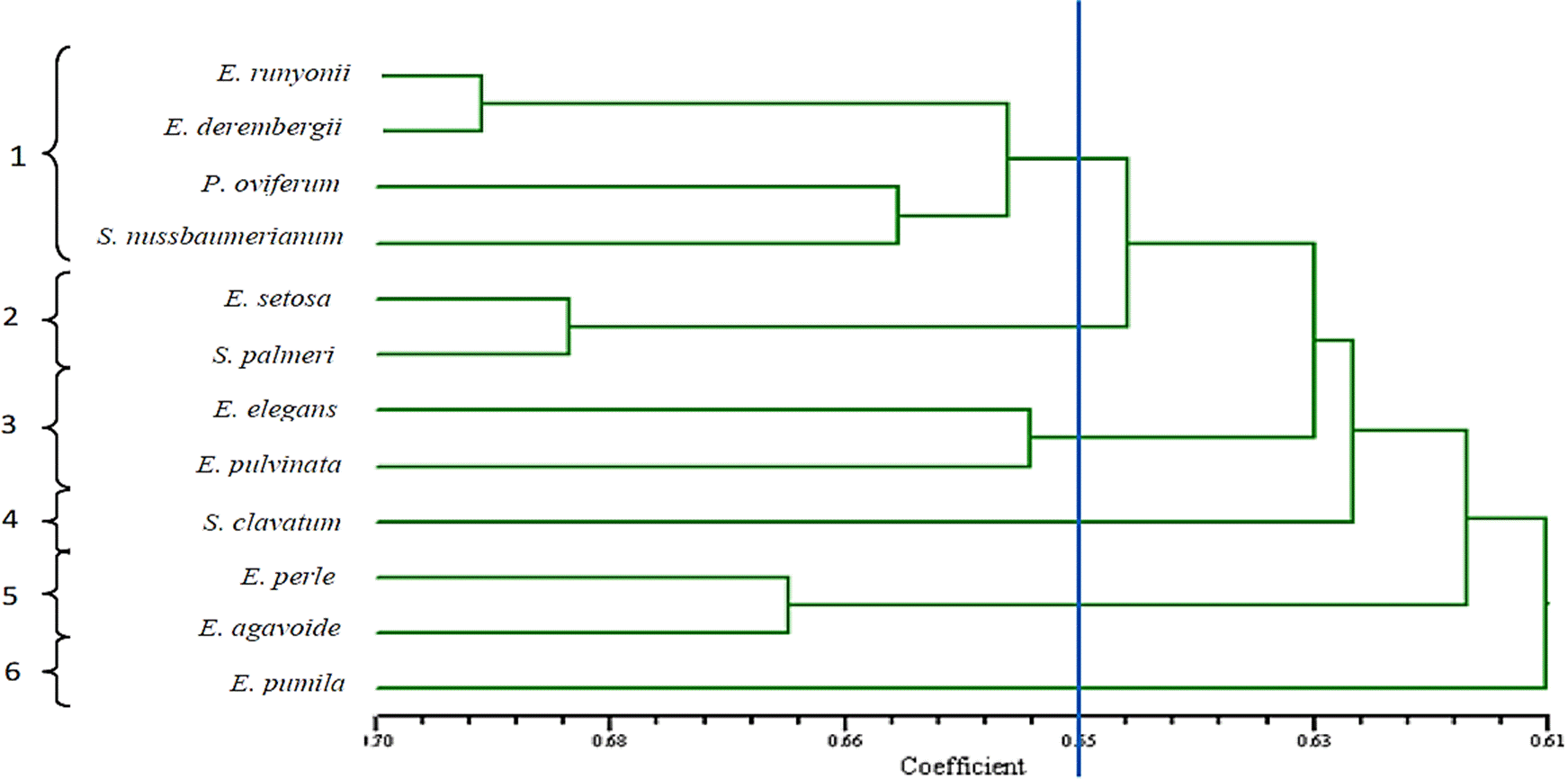
Figura 3 Dendograma generado a partir de datos de marcadores moleculares con de 12 especies de la familia Crassulaceae.
Las especies más similares entre sí (con un coeficiente de 0.62) fueron E. runyonii con E. derembergii, estas fueron las que menor distancia genética presentaron. En contraste, la especie más distinta al resto es claramente E. pumilla ya que es la que se incorpora al grupo con mayor coeficiente de distancia (0.6) (Figura 3).
La agrupación obtenida con base en caracteres morfológicos tuvo tres similitudes con la agrupación generada con base en fragmentos RAPDs, E. elegans estuvo agrupada con E. pulvinata al igual que E. perle con E. agavoides, la tercera similitud fue que Sedum clavatum no se agrupó con ninguna de las once especies del estudio.
Carrillo-Reyes et al. (2008) investigaron a Thompsonella y a un grupo de Echeverias (Crassulaceae), su relación filogenética basada en caracteres moleculares y morfológicos, realizaron un dendograma con el análisis molecular, observando que se agruparon especies de un género con las del otro género, similar a lo obtenido en la presente investigación en donde hay una incongruencia de agrupación genética referente a la clasificación taxonómica, en ambas investigaciones. Ellos concluyen que es necesario dilucidar las relaciones y los límites genéricos del grupo Echeveria y, además, en el clado de Acre como un todo.
Acevedo-Rosas (2004) estudió la filogenética molecular de Graptopetalum (Crassulaceae), concluyó diciendo “este estudio arroja nueva luz sobre las interpretaciones de las relaciones sistemáticas dentro de Crassulaceae y el papel que la geografía, el hábitat, los polinizadores y otros factores ecológicos pueden jugar en la conducción de la evolución de estas plantas suculentas”, ya que en su estudio se presentaron agrupaciones de distintos géneros así como en nuestras agrupaciones de este estudio. En diversos estudios se ha discutido la mala clasificación taxonómica de la familia Crassulaceae, esta investigación muestra que es de gran importancia realizar un estudio que englobe a todas las especies de esta familia para elucidar la correcta clasificación de los géneros, mediante un programa que permita obtener marcadores moleculares que las identifiquen y clasifiquen correctamente.
Los fragmentos RAPDs totales y polimórficos obtenidos en la caracterización de doce especies de la familia Crassulaceae, objetivo de esta investigación, fueron mayores a los obtenidos por García (2003) quien empleo marcadores genéticos AFLP en Crassulaceae, en donde concluye que obtuvo un total de 497 bandas distintas con cinco combinaciones usadas, siendo polimórficas 375 (92.4 %). En otro estudio realizado por Montalvo et al. (2010) en plantas de la familia Cactaceae (Pilosocereus sp), se reporta el empleo de marcadores ISTR, en donde se utilizaron seis combinaciones de oligonucleótidos ISTR, y se obtuvo el total de 97 bandas de las cuales el 62.8 % fueron polimórficas. En cambio, en la presente investigación se obtuvieron 594 bandas, de las cuales el 99 % fueron polimórficas. Carrillo-Reyes et al. (2008) realizaron un trabajo para identificar grupos monofiléticos para esclarecer si Altamiranoa y Villadia deben reconocerse como géneros independientes, argumentando que a Altamiranoa la dividieron en dos grupos, Subcampanulatae y Gamopetalae pero después vieron la variabilidad de la inflorescencia en otros géneros de Crassulaceae y propusieron la fusión de ambos taxones bajo el nombre de Villadia; posteriormente debido a una errónea interpretación de las láminas de los tipos nomenclaturales de Villadia y Thomsonella Britton & Rose, fue entonces que decidieron que el nombre legitimo para Thompsonella era Villadia y se transfirió a todas las especies de Villadia a Altamiranoa; pero en 1940 detectaron esta equivocación y se separó a Thompsonella de Villadia y Altamiranoa.
En 1996 se argumentó una superposición de caracteres apoyándose en la filogenia de `t Hart, que propuso la inclusión de Altamiranoa en Sedum y el reconocimiento de Villadia, pero aún algunos investigadores consideran a Altamiranoa como un género independiente. También Mort et al. (2001), mencionan que el “clado Acre” comprende un tercio de las especies de crasuláceas, pero está plagado de una serie de relaciones no resueltas.
Carrillo-Reyes et al. (2008) concluyen que su análisis combinado muestra igualmente numerosas relaciones no resueltas y escasos grupos con soporte. Entonces consideran que debido a que las especies de Sedum se entremezclan con las de todos estos géneros, además de que otras se posicionan en grupos independientes, es necesario primero definir el límite y la composición del resto de los taxones. Por lo tanto, se podría decir que las Crassulaceae son una familia muy compleja, debido a que es fácil su hibridación y pudo haber cruzamientos no solo entre especies del mismo género sino de diferentes géneros, siendo esta la respuesta a que compartan similitud genética con otros géneros.
La agrupación del dendograma morfológico de las doce especies evaluadas no fue congruente con la agrupación molecular, por lo cual esto indica que no hubo correspondencia entre los marcadores morfológicos y moleculares evaluados, lo cual es coherente ya que son diferentes métodos de evaluación y probablemente exista una errónea clasificación de especies, para confirmar lo anterior se tendría que establecer un banco de germoplasma para evaluar y clasificar correctamente las especies, realizar la evaluación por lo menos en dos ciclos, utilizando los componentes principales morfológicos que arrojo la presente investigación y correr más iniciadores que puedan discriminar correctamente a las especies y permitan ubicarlas correctamente.
Conclusiones
Los marcadores morfológicos y moleculares detectaron diversidad genética entre especies. La agrupación obtenida de acuerdo con las características morfológicas fue congruente con la clasificación taxonómica dado que en general agrupo a especies del mismo género. En contraste, en el estudio molecular se agruparon especies de diferentes géneros. Lo cual indica que no hubo correspondencia entre los marcadores morfológicos y moleculares evaluados. El análisis molecular detecto que las especies con mayor similitud genética fueron E. runyonii y E. derembergii, lo que indica mayor posibilidad genética de cruzamiento para hibridaciones interespecíficas futuras.
REFERENCIAS
Acevedo-Rosas, R., Cameron, K., Sosa, V. and Pell, S. (2004). A molecular phylogenetic study of Graptopetalum (Crassulaceae) based on ETS, ITS, RPL16, and TRNL-F nucleotide sequences. American Journal of Botany, 91: 1099-1104. https://doi.org/10.3732/ajb.91.7.1099 [ Links ]
Andrade-Rodríguez, M., Villegas-Monter, A., Carrillo-Castañeda, G. and García-Velázquez, A. (2004). Polyembryony and identification of Volkamerian lemon zygotic and nucellar seedlings using RAPD. Pesquisa Agropecuária Brasileira, 39: 551-559. http://dx.doi.org/10.1590/S0100-204X2004000600006 [ Links ]
Avise, J. C. (1994). Molecular markers, natural history and evolution. Chapman and Hall. New York, N Y. 7(6): 766-767. https://doi.org/10.1046/j.1420-9101.1994.7060766.x [ Links ]
Becerra, V. V. & Paredes, C. M. (2000). Uso de marcadores bioquímicos y moleculares en estudio de diversidad genética. Agricultura técnica (Chile), 60: 216-223. http://dx.doi.org/10.4067/S0365-28072000000300007 [ Links ]
Bered, F., Terra, T. F., Spellmeier, M. and Neto, J. F. B. (2005). Genetic variation among and within sweet corn populations detected by RAPD and SSR markers. Crop Breeding and Applied Biotechnology 5: 418-425. https://pdfs.semanticscholar.org/293d/0a884480b6e7087da53f06d143ea82618126.pdf [ Links ]
Borys, M. W., Leszczyñska-Borys, H. and Galván, J. L. (2005). Echeveria gibbiflora D. C. -a new ornamental plant from México. I. vase life. Acta Horticulturae, 683: 271-277. http://wwwlib.teiep.gr/images/stories/acta/Acta%20 683/683_31.pdf [ Links ]
Bravo-Hollis, H. (1978). Las cactáceas de México. Vol. 1. Universidad Nacional Autónoma de México. México. 755 p. [ Links ]
Carrillo-Reyes, P., Sosa, V. and Mort, M. E. (2008). Thompsonella and the “Echeveria group” (Crassulaceae): phylogenetic relationships based on molecular and morphological characters. Taxon. 57:863-874. https://www.researchgate.net/publication/233690220_Thompsonella_and_the_Echeveria_group_Crassulaceae_Phylogenetic_relationships_based_on_molecular_and_morphological_characters [ Links ]
Colinas, L. M. T. (2003). ‘‘Importancia de los Estudios Post-Cosecha de Plantas Ornamentales Nativas de México’’. In: Mejía, M.J.M. y F.A. Espinosa (comps.). Plantas Nativas de México con Potencial Ornamental: Análisis y Perspectivas. Universidad Autónoma Chapingo. pp. 175-179. [ Links ]
Eggli, U. (2003). Illustrated Handbook of succulent Plants: Crassulaceae. Springer. Zürich Switzerland. 458 p. [ Links ]
García, R. I. & Pérez-Calix, E. (2007). Una nueva especie de Echeveria (Crassulaceae) originaria del estado de Jalisco, México. Acta Botánica Mexicana 78: 125-132. http://www.scielo.org.mx/scielo.php?script=sci_arttext&pid=S0187-71512007000100009 [ Links ]
García, R. (2003). Relaciones interespecíficas del género Pachyphytum (Crassulaceae), empleando marcadores genéticos AFLP. Universidad de Colima, Biotecnología. Tecomán, Colima. 143 p. [ Links ]
García-Ruiz, I. & Pérez-Calix, E. (2007). Una especie nueva de Echeveria (Crassulaceae) originaria del estado de Jalisco, México. Acta Botánica Mexicana , 78: 125-132. http://www.redalyc.org/pdf/574/57407809.pdf [ Links ]
Jimeno-Sevilla, H. D., Hernández-Ramírez, A. M. and Krömer, T. (2013). Echeveria rosea Lindley (Crassulaceae): a hummingbird-dependent succulent epiphyte. Cactus and Succulent Journal. 85: 128-131. https://doi.org/10.2985/0007-9367-85.4.128 [ Links ]
Jimeno-Sevilla, H., Hérnandez-Ramírez, A. M., Ornelas, J. F. and Marten-Rodríguez, S. (2014). Morphological and néctar traits in Echeveria rosea Lindley (Crassulaceae) linked to humminbird pollination in Central Veracruz, Mexico. Haseltonia 19:17-25. https://doi.org/10.2985/026.019.0104 [ Links ]
Jones, C. E., Shropshire, F. M., Allen, R. L. and Atallah, Y. C. (2010). Pollination and reproduction in natural and mitigation populations of the many-stemmed Dudleya, Dudleya multicaulis (Crassulaceae). California Botanical Society 57: 42-53pp https://www.jstor.org/stable/pdf/41425844.pdf?seq=1#page_scan_tab_contents [ Links ]
López-Gutiérrez, D. M., Reyes-Agüero, J. A., Muñoz, A., Robles, J. and Cuevas, E. (2015). Comparación morfológica entre poblaciones silvestres y manejadas de Opuntia atropes (Cactaceae) en Michoacán, México. Revista Mexicana de Biodiversidad 86: 1072-1077. https://doi.org/10.1016/j.rmb.2015.08.006 [ Links ]
Maatz, Tiky. (2012) Nichi”, an interesting Echeveria hybrid made in Argentina. Cactus and Succulents Journal. 84 (1): 30-32. https://doi.org/10.2985/0007-9367-84.1.30 [ Links ]
Martínez-Ávalos, J. G. & Mora-Olivo, A. (2000). Una nueva especie de Echeveria (Crassulaceae) del estado de Tamaulipas, México. Acta Botánica Mexicana 52: 43-48. https://www.redalyc.org/pdf/574/57405203.pdf [ Links ]
Martínez-Moreno, E., Corona-Torres, T., Avitia-García, E., Castillo-González, A. M., Terrazas-Salgado, T. and Colinas-León, M. T. (2006) Caracterización morfométrica de frutos y semillas de nanche (Byrsonima crassifolia (L.) H.B.K.) Revista Chapingo Serie Horticultura 12(1): 11-17. http://www.redalyc.org/articulo.oa?id=60912103 [ Links ]
Montalvo, G. F., Ortiz, G.M., Quiala, M. E., Keb-Llanes, M., Rojas, L. E., Bautista, A. M., Alonso, E. M., Quiroz, M. A., Robde, W. and Sánchez, T. L. F. (2010). Primer reporte del empleo de Marcadores ISTR en Cactaceae (Pilosocereus sp). Revista Colombiana de Biotecnología 2: 223-229. https://revistas.unal.edu.co/index.php/biotecnologia/article/view/18772 [ Links ]
Morales, N. C. R., Avendaño, A. C., Melgoza, C. A., Gil, V. K. C; Quero, C. A., Jurado, G. P. and Martínez, S. M. (2016). Caracterización morfológica y molecular de poblaciones de pasto banderita (Bouteloua curtipendula) en Chihuahua, México. Revista Mexicana de Ciencias Pecuarias 7(4): 455-469. http://www.redalyc.org/articulo.oa?id=265648207005 [ Links ]
Mort, M. E., Soltis, D. E., Soltis, P. S., Francisco-Ortega, J. and Santos-Guerra, A. (2001). Phylogenetic relationships and evolution of Crassulaceae inferred frommatK sequence data. American Journal of Botany , 88: 76-91. https://doi.org/10.2307/2657129 [ Links ]
Rendón C. A & Fernández N. R. (2007) Plantas con potencial uso ornamental del estado de Morelos, México. Polibotánica. 23: 121-165. http://www.redalyc.org/pdf/621/62102308.pdf [ Links ]
Rodríguez-Rojas, T. J., Andrade-Rodríguez, M., Canul-Ku, J., Castillo-Gutiérrez, A., Martínez-Fernández, E. and Guillén-Sánchez, D. (2015). Viabilidad de polen, receptividad del estigma y tipo de polinización en cinco especies Echeveria en condiciones de invernadero. Revista Mexicana de Ciencias Agrícolas 6: 111-123. http://www.scielo.org.mx/scielo.php?pid=S2007-09342015000100010&script=sci_arttext&tlng=en [ Links ]
Sajeva, M., & Constanzo, M. (1997). Succulents The Illustrated Dictionary. Ed. Timber Press. Portland, Oregon. 239p. [ Links ]
Singh, G. (1999). Plant Systematics. Enfield, NH: Science Publishers Inc.756 p. [ Links ]
´t Hart H. (1997). Taxonomic groups. Crassulaceae. In: Olfield S. (comp.) Status survey and conservation action plan. Cactus and Succulent Plants. IUCN. Cambridge, U.K. pp. 20-23 [ Links ]
Thiede, J. (1995). Quantitative phytogeography, species richness and evolution of American Crassulaceae. In: Evolution and systematics of the crassulaceae. (Eds.): t’ Hart & Eggli. Backhuys Publishers. Leiden. Netherland. 89-123. http://www.academia.edu/29809697/Thiede_J._1995_Quantitative_phytogeography_species_richness_and_evolution_of_American_Crassulaceae._In_t_Hart_H._Eggli_U._Eds._Evolution_and_systematics_of_the_ Crassulaceae._Backhuys_Publishers_Leiden_pp._89-123 [ Links ]
Toledo, V. M. (1988). La diversidad biológica de México. Ciencia y Desarrollo, 81:17-30. http://www.ejournal.unam.mx/cns/no34/CNS03407.pdf [ Links ]
Van Ham, R. C. H. J. & t’ Hart, H. (1998). Phylogenetic relationships in the Crassulaceae inferred from chloroplast DNA restriction site variation. American Journal of Botany, 85: 123-134. [ Links ]
Van Ham, R. C. H. J., t’ Hart, H., Mes, T. H. M. and Sandbrink, J. M. (1994). Molecular evolution of noncoding regions of the chloroplast genome in the Crassulaceae and related species. Current genetics, 25: 558-566. https://link.springer.com/content/pdf/10.1007/BF00351678.pdf [ Links ]
Watson, J. W. & Eyzaguirre, P. B. (2002). Proceedings of the Second International Home Gardens Workshop: Contribution of home gardens to in situ conservation of plant genetic resources in farming systems. Winzenhausen, federal Republic of Germany. International Plant Genetic Resources Institute. Rome. 192 p. https://www.bioversityinternational.org/fileadmin/_migrated/uploads/tx_news/Home_gardens_and_in_situ_conservation_ of_plant_genetic_resources_in_farming_systems_753.pdf [ Links ]
Yasmin S, M. S., Islam, M., Kondoker, M., Nasiruddin, S. and Alam. (2006). Molecular characterization of potato germplasm by Random Ampliefied Polymorphic DNA markers. Biotechnology. 5(1): 27-31. http://docsdrive.com/pdfs/ansinet/biotech/2006/27-31.pdf [ Links ]
Como citar este artículo: Andrade-Rodríguez, M., Rodríguez-Rojas, T.J., Castillo-Gutiérrez, A., Villegas-Torres, O.G., Guillén-Sánchez, D. (2019). Analysis of morphological characters and Rapd fragments of twelve species of the Crassulaceae Family. Revista Bio Ciencias 6, e537. doi: https://doi.org/10.15741/revbio.06.01.09
Recibido: 25 de Junio de 2018; Aprobado: 18 de Octubre de 2018











 text in
text in 


