Introducción
La tuberculosis (TB) causada por Mycobacterium tuberculosis (Mtb) representa un riesgo para la salud pública y una amenaza a la seguridad sanitaria; es una de las principales causas de morbilidad y mortalidad a nivel mundial1. Es una enfermedad infecto-contagiosa que afecta principalmente a los pulmones2. La organización mundial de la salud (OMS), reportó que en el año 2021 murieron 1.4 millones de personas, y considera que es la enfermedad más mortífera después de la COVID-19; se estima que 10.6 millones de personas se enfermaron de TB en el año 20213. Es evidente que durante la pandemia causada por COVID-19, el sistema de atención, diagnóstico y tratamiento de TB fue deficiente; esto aumenta la posibilidad de un remonte de la enfermedad, con un posible impacto en la salud mundial4. En la mayoría de los casos el virus SARS-CoV-2 ocasiona una infección respiratoria en el individuo infectado, la cual se puede complicar en diferentes grados que van de leves a graves. El uso de medicamentos inmunosupresores en COVID-19 puede conducir a un aumento en la expresión génica de Mtb. Algunos reportes, describen casos de co-infección entre Mtb y SARS-CoV-2, con una prevalencia de TB entre los pacientes con COVID-19 de entre 0.47 y 4.47 %, y se reporta que la tasa de mortalidad en pacientes con TB/COVID-19 fue más alta que la de los pacientes afectados solo por uno de estos patógenos4-6.
En la práctica clínica cotidiana, el diagnóstico certero de la TB es por medio del cultivo del microorganismo, y se requieren bacterias en crecimiento para realizar pruebas de susceptibilidad a los medicamentos, lo que resulta un desafío médico importante y vuelve lento el procedimiento7. Aunado a esto, existe el problema del desarrollo progresivo de la TB farmacoresistente, lo que refuerza la necesidad urgente de investigar nuevas moléculas para diagnóstico y control de la TB8. El diagnóstico oportuno; tanto como el tratamiento eficaz de los individuos infectados, podría ayudar a reducir la TB. Por esta razón, es indispensable realizar investigación de biomarcadores novedosos para el diseño de métodos de control. En los últimos años, se ha investigado a los microRNAs como moléculas prometedoras para tales efectos, debido a su alta estabilidad, sensibilidad y especificidad9. Los miRNA son pequeños RNA reguladores no codificantes que actúan reprimiendo la expresión de proteínas a nivel postranscripcional, y tienen funciones importantes en muchos procesos fisiológicos y fisiopatológicos10. Los mecanismos de regulación de los miRNAs, se basan en la complementariedad de secuencias entre el miRNA y el RNAm blanco; si la unión es perfecta resulta en la degradación del RNAm, si la unión es parcial, se reprime la traducción11. La deadenilación del RNAm conduce a la inestabilidad y por ende la degradación del RNAm10. Después de cualquiera de estos mecanismos, se activa la respuesta inmune innata del huésped, con la producción de citocinas y quimiocinas11.
El desarrollo en las ciencias omicas ha permitido la rápida identificación y caracterización de pequeños RNA no codificantes, los cuales forman parte de un complejo sistema de regulación génica, y se ha encontrado una expresión diferencial de estos en individuos infectados con TB. Durante la infección con Mtb, se activa la respuesta inmunitaria del huésped, en esta interacción huésped-Mtb, se manipula el perfil de miRNAs; esto implica la regulación de varios procesos biológicos mediados por esas moléculas11. Algunos miRNAs que se modifican durante una infección por Mtb; también se producen en células inmunitarias contenidas en el granuloma y conducen a la respuesta inmunitaria adaptativa; también se pueden secretar en el medio extracelular, a través de procesos como la apoptosis o necrosis, la encapsulación dentro de microvesículas o exosomas y mediante la unión a lipoproteínas de alta densidad (HDL), entre otras. Esto permite patrones estables de expresión de miRNAs asociados a la infección por TB12.
Se sabe poco sobre la patogénesis molecular de la enfermedad, pero existen reportes recientes que demuestran la importancia de los miRNA en la TB pulmonar y que pueden ser detectados en sangre de pacientes infectados; por lo que actualmente son señalados como candidatos para el diagnóstico. Los miRNA, que son modulados en respuesta a la infección con Mtb, son miR-125b, miR-155, miR-144, miR-3179, miR-147, miR-146a/b, miR-886-5p, let-7e, let-7i13,14. La presencia y regulación de estos miRNA en humanos infectados por TB indica su importancia en la patogénesis y sobrevivencia del bacilo, por lo que es indispensable su investigación durante la infección.
El miRNA 146b ha sido asociado a la regulación de diversas vías de señalización y algunos de sus genes blanco descritos son AKT3, IL6, IRAK1, NFKB1, TLR415; y se ha reportado que se sobre expresa en el suero de pacientes con TB activa16. El miR-155 modula la producción de mediadores inflamatorios en respuesta a estímulos microbianos regulando negativamente la expresión de TAK1 y la proteína de unión a TRAF6. También se ha observado que, en la infección por TB, miR-155 se sobre expresa e inhibe la autofagia inducida por IFN-γ; algunos de los genes blanco a los que ha sido asociado son AKT1, APAF1, ATP6V1H, CASP315. Interesantemente, miR-155 tiene doble función durante la infección con TB; por un lado, mantiene la supervivencia de los macrófagos infectados con Mtb y, por otro lado, promueve la supervivencia y la función de las células T específicas de Mtb17. Considerando la importancia de los miRNA en la patogénesis de la TB humana, donde se han detectado en sangre de pacientes enfermos; en este trabajo, se realizó la predicción de los genes a los que se unen los miR-146a, miR-146b y miR-155 y se realizó el análisis de las rutas metabólicas en las que estos intervienen.
Material y métodos
Selección de los miRNA de estudio
Los tres miRNA usados para el análisis, fueron seleccionados mediante una búsqueda bibliográfica en la base de datos del NCBI, a través, de la colección de revistas biomédicas del PubMed Central (https://www.ncbi.nlm.nih.gov/pmc/)18, se seleccionaron los miRNAs estudiados en humanos, que se reportaron de manera consistente en al menos cinco revistas científicas y encontrados como sobre expresados en individuos infectados en ensayos experimentales, de estos se seleccionaron tres, al azar. Se usó la base de datos miRBase19 para obtener los datos de los miRNA de interés, como el número de acceso y secuencia madura de cada miRNA, mismos que fueron los siguientes: hsa-mir-146a-3p: MIMAT0004608 (CCUCUGAAAUUCAGUUCUUCAG), hsa-miR-146b-3p: MIMAT0004766 (GCCCUGUGGACUCAGUUCUGGU), hsa-miR-155-3p: MI0000681 MIMAT0004658 (CUCCUACAUAUUAGCAUUAACA).
Predicción de las rutas y genes blanco de los miRNA
Para determinar los genes diana de los miRNA analizados, se usaron tres modelos ontológicos, lo que permitió obtener la metainformación más acertada a la realidad y describir la semántica de los datos más objetivos. Se usaron los siguientes programas: TargetScan Human20, miRDB21,22 particularmente analizados con la herramienta target ontology23 y miRTarBase23,24. Los criterios de inclusión de los genes blanco a estudiar fueron los siguientes. Para el software TargetScan Human20, se incluyeron para el análisis, solo los genes con puntaje context, arriba de -0.20. En el caso de Target ontology23, solo los genes blancos que cumplieron un target score arriba de 77, fueron seleccionados. Para el software miRTarBase23,24, se seleccionaron los genes blanco de cada miRNA de acuerdo con la evidencia experimental validada al menos por dos métodos y que estuvieran reportados en artículos relacionados al tema de estudio. Adicionalmente, los genes que se encontraron de manera consistente en al menos dos de los programas utilizados fueron los que se tomaron en cuenta para su revisión. Finalmente, de estos se seleccionaron al azar al menos dos genes blanco para revisar su relevancia en la infección con TB. A cada gen se le asignó una vía metabólica o regulatoria, usando la información de la librería de genes y genomas de Kyoto (KEGG)25.
Resultados
Predicción de genes diana y análisis de las rutas metabólicas de los miRNAs
Los análisis bioinformáticos, permitieron predecir genes diana para los miR-146a, miR-146b y miR-155 (Cuadro 1); algunos de estos genes tienen grandes implicaciones durante la infección de TB. El miR-146a, puede regular genes involucrados en adhesión celular y procesos de formación del fagosoma (CLDN16, ATP6V1C2) (Figura 1). El miR-146b está involucrado en las rutas metabólicas de degradación de valina, leucina e isoleucina y la ruta de señalización de la hormona tiroidea entre otras (Figura 2). El miR-155 interviene en las rutas del sistema de intercambio de azufre y el metabolismo de triptófano; de acuerdo con las predicciones del programa KEGG (enero, 2022) (Figura 3).
Cuadro 1 Predicción de las principales rutas metabólicas y genes a los que se unen e interceptan los miRNA miR-146a, miR-146b y miR-155
| miRNA | Ruta metabólica KEGG | Genes blanco-destacados | Valor P |
|---|---|---|---|
| miR-146a | Moléculas de adhesión celular | CLDN16 (claudin 16) | 0.02 |
| Fagosoma | ATP6V1C2 (ATPase H+ transporting V1 subunit C2) | 0.03 | |
| miR-146b | Degradación de valina, leucina e isoleucina | BCKDHB (branched chain keto acid dehydrogenase) ABAT (4-aminobutyrate aminotransferase) | 0.006 |
| Ruta de señalización de la hormona tiroidea | THRA (thyroid hormone receptor alpha) RXRB (retinoid X receptor beta) | 0.01 | |
| miR-155 | Sistema de intercambio de azufre | MOCS2 (molybdenum cofactor synthesis 2) | 0.006 |
| Metabolismo de triptófano | IDO-1 (indoleamine 2,3-dioxygenase 1) | 0.01 |
El análisis se realizó a partir de la secuencia madura -3p de cada miRNA.
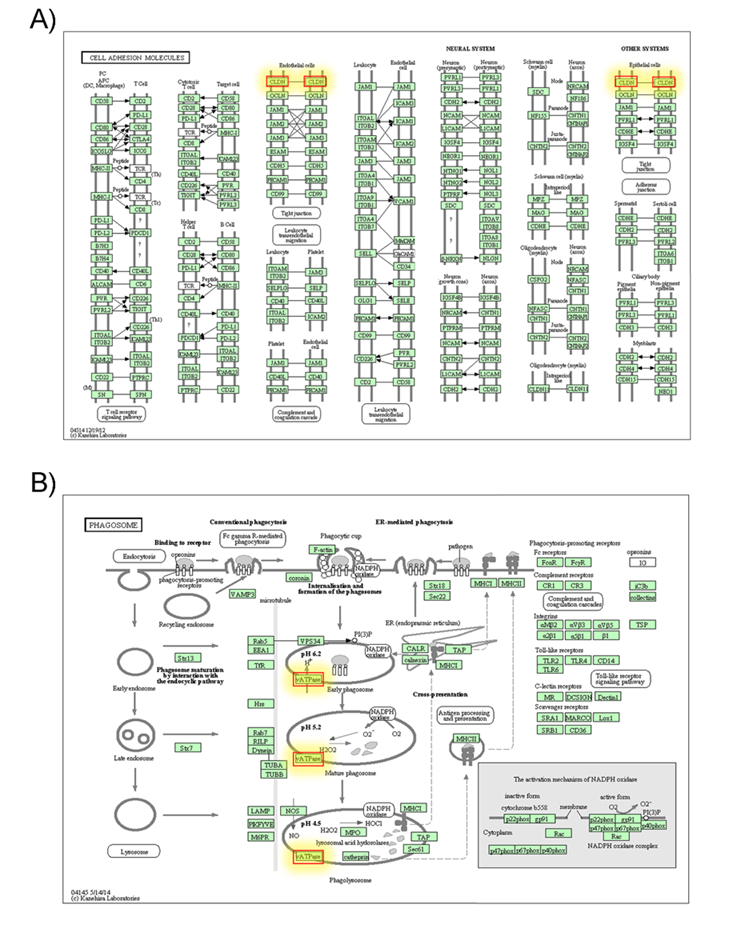
Figura 1 Rutas metabólicas predichas KEGG para el miRNA 146a de acuerdo con los genes a los que se une. Panel A) Moléculas de adhesión celular, Panel B) Fagosoma. Se resaltan (amarillo) los genes destacados en esta investigación
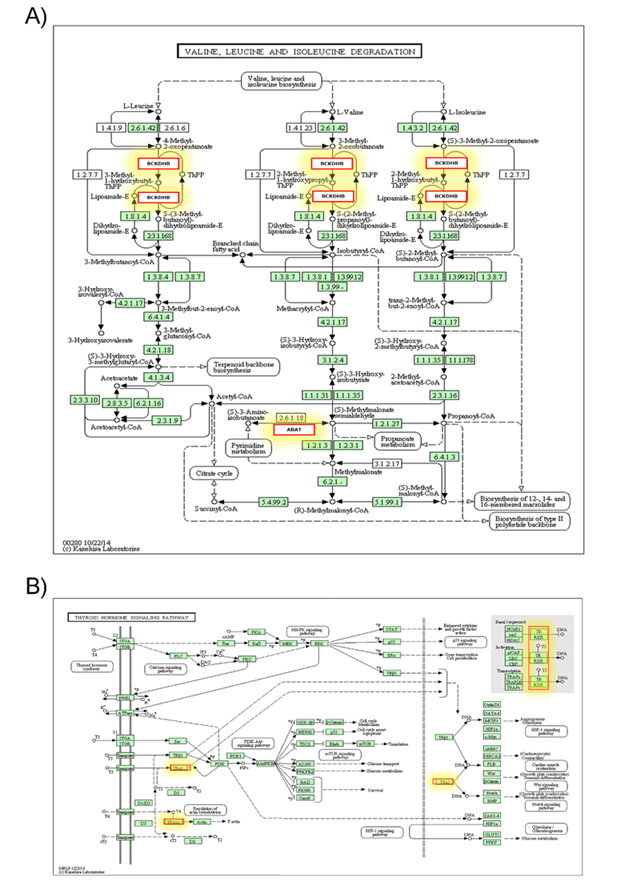
Figura 2 Rutas metabólicas predichas KEGG para el miRNA 146b de acuerdo con los genes a los que se une. Panel A) Degradación de valina, leucina e isoleucina, Panel B) Ruta de señalización de la hormona tiroidea. Se resaltan (amarillo) los genes destacados en esta investigación
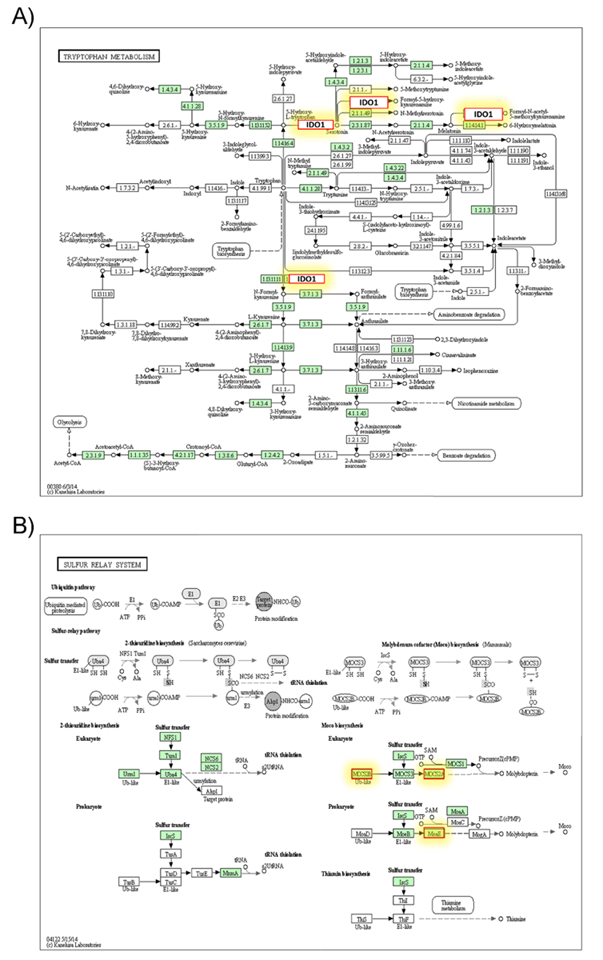
Figura 3 Rutas metabólicas predichas KEGG para el miRNA 155 de acuerdo con los genes a los que se une. Panel A) Metabolismo del triptófano, Panel B) Sistema relay de azufre. Se resaltan (amarillo) los genes destacados en esta investigación
Los tres miRNA tienen genes diana validados en la base de datos. En el caso específico del miR-146a, sus principales rutas metabólicas KEGG son moléculas de adhesión celular y fagosoma; pero también participa en la regulación del metabolismo del cáncer, uno de los genes destacados en este proceso es ZEB2, que es un factor de transcripción que desempeña un papel en las vías de señalización del factor de crecimiento transformante β, que son esenciales durante el desarrollo fetal temprano y su desregulación ha sido caracterizada en diferentes tipos de cáncer26. El software miRTarBase no muestra intersección de los miRNA en las rutas metabólicas, ya que no comparten genes diana predichos.
Discusión
En el análisis del miR-146a se encontraron dos rutas metabólicas predichas importantes, donde hay dos genes con los que interacciona este miRNA en rutas independientes; uno de ellos es el gen CLDN16 que codifica a la claudina 16. Las claudinas son proteínas con dominios transmembrana que se encuentran en la zona de unión estrecha entre las células de los epitelios y endotelios; junto a otras proteínas forman poros y son componentes clave del canal paracelular. Los canales paracelulares en la unión estrecha, tienen propiedades de selectividad iónica, dependencia del pH y otros efectos27. La sobreexpresión de claudina 16 ha sido asociada a cáncer de ovario y otras enfermedades, y se ha determinado su importancia en la reabsorción de magnesio celular27.
En un estudio se determinó el patrón de expresión de algunas claudinas (2 y 4), y se analizaron los cambios estructurales en biopsias de colon en pacientes con TB. Los resultados demuestran que la claudina 2 se expresa en la zona de unión estrecha entre las células y no se observaron cambios estructurales en el tejido analizado28. Recientemente, se investigó el efecto de la infección por TB en la expresión de proteínas de unión celular en el sistema nervioso central (SNC). Estos resultados sugieren que Claudina-5 disminuye su expresión y cambia su localización dentro de la célula en respuesta a la infección con Mtb de la cepa N15, sugiriendo que Mtb afecta la expresión de proteínas cerebrales de las uniones celulares. Este daño, consistió en cambios celulares sugerentes de toxicidad, debido a que se observaron signos de necrosis29.
El gen ATP6V1C2 codifica a una enzima que es una ATPasa, algunos estudios han sugerido la importancia de las ATPasas tipo P en la fisiología y la supervivencia intracelular de las micobacterias30. Un perfil transcripcional en humanos, de las ATPasas en condiciones de hipoxia, estrés oxidativo, inanición e intoxicación por agentes químicos y procesos de infección in vitro e in vivo, evidenció la expresión diferencial de estos transportadores frente a estas condiciones. La ATPasa es una bomba de protones altamente conservada que se expresa en las células31. Recientemente, se realizó un estudio donde se investigaron dos compuestos que inhiben el crecimiento de cepas de TB sensibles y resistentes a los medicamentos, en este estudio, a través de ensayos transcriptómicos mostraron cambios en la expresión de ciertos genes en respuesta a la infección con TB; uno de esos genes fue ATP6V1C2 el cual se encontró sobre expresado en respuesta a la infección con TB32.
El miR-146b interacciona con genes destacados que participan en la producción de energía en las células, tales como la deshidrogenasa (BCKDHB); y con aminotransferasa (ABAT) involucrada en la degradación de valina, leucina e isoleucina. Algunos reportes indican que el aumento en la actividad del lactato deshidrogenasa sérica es un indicador de diagnóstico presuntivo de neumonía y otras infecciones como la TB33.
El miR-155 demostró interacción con los genes MOCS2 e IDO-1, los cuales provienen del sistema de intercambio de azufre y del metabolismo de triptófano respectivamente. El gen MOCS2 codifica para dos proteínas diferentes MOCS2A y MOCS2B, estas dos juntas forman la enzima molibdopterina sintasa que participa en la biosíntesis del cofactor de molibdeno (MoCo), que es un grupo prostético. Las enzimas dependientes de MoCo intervienen en muchos procesos biológicos; interesantemente, el MoCo funciona directamente en las enzimas etilbenceno deshidrogenasas y otras34. El molibdeno (Mo) es necesario para que varias enzimas como el sulfito oxidasa, el aldehído oxidasa, entre otras puedan tener su función. La función de esas enzimas es la descomposición de sustancias en el cuerpo, algunas de las cuales son tóxicas si no se metabolizan. Algunas Micobacterias tienen genes que codifican para MoCo. Mtb posee múltiples homólogos que codifican sintasa en la biosíntesis de MoCo; esto sugiere que su expansión puede cumplir diferentes funciones celulares35.
Las enzimas de Mo son catalizadores en la generación de energía y reacciones de desintoxicación, entre otras funciones. Se sabe que los sustratos convertidos por las enzimas Mo bacterianas, que son importantes para la virulencia son del grupo que se generan en el huésped durante la inflamación o red de señalización. Esto sugiere que podrían ser objetivos importantes de fármacos36. Las enzimas de Mo catalizan importantes reacciones redox. Las micobacterias tienen varias enzimas que contienen Mo; estas ayudan a regular la latencia de Mtb. El cofactor MoCo, es el cofactor común de las enzimas Mo en las micobacterias37. En algunos experimentos se ha identificado una vía novedosa que usa Mtb para la resistencia al estrés (hipoxia) impuesto por el huésped; esta capacidad de Mtb de persistir en condiciones de hipoxia, contribuye a la TB latente en el huésped. Mtb adquirió a través de transferencia horizontal el gen moaA1-D1, el cual está involucrado en la biosíntesis del MoCo; a saber; estos genes tienen homólogos presentes en todo el género Mycobacterium; interesantemente los genes moaA1-D1 se inducen bajo condiciones de hipoxia38. Evolutivamente, el complejo Mtb ha desarrollado mecanismos para tener éxito, en parte por la adquisición de genes involucrados en la patogénesis. Descifrar y conocer los mecanismos a través de los cuales Mtb causa enfermedad, es relevante para identificar objetivos desconocidos y de interés para el desarrollo de nuevo métodos de control, diagnóstico y terapia de la enfermedad. El estudio amplio de la biosíntesis de las enzimas MoCo de Mtb, ayudará a identificar objetivos farmacológicos prometedores para controlar la TB, especialmente la TB latente.
El gen IDO-1 codifica a una enzima que se encuentra principalmente en macrófagos, la enzima participa en la degradación del triptófano que genera quinurenina; esta ruta metabólica constituye un mecanismo de modulación de la respuesta inmune. La indolamina 2,3-dioxigenasa (IDO-1) es un enzima que se encuentra en numerosas células39. La IDO-1 ayuda a la degradación del triptófano en quinurenina, en el interior de las células, y por tanto regula la disponibilidad del triptófano. Esto tiene amplias implicaciones en la respuesta inmune del organismo. Se ha reportado que la proteína IDO-1 se sobreexpresa en respuesta a la infección con Mtb en macrófagos humanos y murinos in vitro. La sobreexpresión de IDO-1 se ha correlacionado también, con la expresión de otros marcadores inflamatorios, como la proteína C reactiva y el mal pronóstico de pacientes con TB40,41.
Estudios in vitro demuestran que la actividad de IDO-1 en las células presentadoras de antígeno, inhibe la proliferación de células T específicas de antígeno micobacteriano. La actividad de IDO-1 podría tener una función importante en la inhibición de la respuesta inmunitaria adaptativa específica para Mtb, y podría ayudar a que el patógeno sobreviva en el huésped infectado. El metabolismo del triptófano es un medio para regular las funciones de las células T, como en la evasión del sistema inmunitario inducido por tumores, la tolerancia periférica y la inflamación durante la infección40,42. La activación del metabolismo del triptófano es un mecanismo antimicrobiano que se presenta contra algunas bacterias patógenas. La activación de IDO-1 y el metabolismo de triptófano en los macrófagos dentro del SNC, se relaciona con demencia asociada a el SIDA y otras enfermedades cerebrales inflamatorias43. Intervenir la actividad de la enzima IDO-1, es una estrategia prometedora para desarrollar tratamientos en los trastornos neurológicos asociados con el VIH. En relación con la alta prevalencia de individuos infectados con VIH y Mtb. Así como una terapia (dirigida al huésped) efectiva contra la TB.
Si bien, este análisis se realizó en los miRNA que se han descrito en humanos, es importante destacar que el genoma de M. bovis tiene similitud a nivel de nucleótidos de más del 99.95 % con M. tuberculosis; no obstante, M. bovis ha perdido parte de su genoma debido a mutaciones genéticas, por mecanismos de deleción; lo que lo hace más pequeño (M. bovis AF2122/97: 4’345,492 pb) comparado con M. tuberculosis (CDC1551: 4’403,836 pb)44,45. Interesantemente, se ha sugerido que M. tuberculosis surgió de M. bovis durante el periodo en que el ganado fue domesticado por el hombre, aproximadamente hace 10-15,000 años, al infectar al humano46. Esta aseveración está fundada en la observación de la infección (causada por varias cepas de M. bovis) en diferentes hospederos animales incluyendo al humano; mientras que la infección natural de M. tuberculosis, aparentemente hasta la fecha de esta publicación, se encuentra restringida al humano47. Esta similitud tan estrecha entre estas dos especies hace que el estudio de productos génicos, proteicos, así como miRNA que pueden ser análogos o equivalentes entre M. tuberculosis y M. bovis, sea viable para tratar de comprender un poco más de la patogénesis de la tuberculosis causada por estas dos especies, y quizá para sentar las bases para el diseño de nuevos biomarcadores o posibles blancos terapéuticos.
Conclusiones e implicaciones
Los hallazgos mostrados aquí, sugieren que los miR-146a, miR-146b y miR-155 están asociados a la activación de mecanismos moleculares específicos en respuesta a la TB. Los genes con los cuales los miR-146a y miR-155 interaccionan o se unen, están involucrados en la respuesta inmune y en procesos celulares imprescindibles durante una infección por TB.











 text in
text in 


