Introducción
Oaxaca cuenta con una gran variedad de climas y microclimas debido principalmente a su compleja orografía lo cual permite la existencia de un mosaico de vegetación privilegiado. Particularmente, la selva baja caducifolia (SBC) es un tipo de bosque tropical que se caracteriza por la pérdida total de las hojas durante una marcada temporada de secas. Florísticamente, predominan especies de las familias Anacardiaceae, Asteraceae, Burseraceae, Cactaceae, Fabaceae, Malpighiaceae y Rubiaceae, entre otras (Trejo, 2005). Este tipo de vegetación ocupa 3.38% (6,649,200 ha) del territorio nacional (INEGI, 2005) y en Oaxaca, exclusivamente en la región de la costa, representa 1.26% (79,120 ha) de la cobertura forestal (Semarnat-Conafor, 2014). Poco se conoce sobre la diversidad de macromicetos asociados a ese tipo de vegetación y el escaso conocimiento que se tiene de los hongos tropicales, en general, hace evidente la necesidad de elaborar inventarios de especies que puedan ayudar a establecer patrones de diversidad y comprender mejor su dinámica ecológica (Díaz-Moreno et al., 2005).
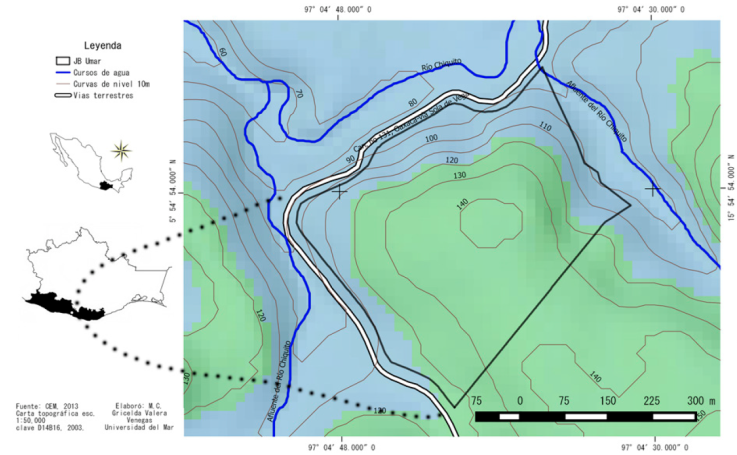
El estado de Oaxaca se divide en 8 regiones socioculturales, de las cuales la región de la costa está conformada por los distritos de Jamiltepec, Juquila y Pochutla (Fig. 1). En esta región son escasos los estudios sobre la diversidad de hongos y los pocos trabajos que existen son contribuciones parciales, reportando a la fecha 163 especies de macromicetos (Álvarez et al., 2016; Barbosa-Reséndiz et al., 2020; Cobos-Villagrán et al., 2020; Guzmán y Pérez-Patraca, 1972; Ortega-López et al., 2019; Pérez-Silva, 1973, 1977; Ramírez-Cruz et al., 2006; Raymundo y Valenzuela, 2003; Raymundo et al., 2020; Singer, 1976; Villarruel-Ordaz et al., 2015), de las cuales solo 20 están registradas creciendo en SBC, y el resto, en otros tipos de vegetación como manglar, selva mediana subcaducifolia, bosque tropical perennifolio y subperennifolio, bosque mesófilo e incluso en bosques mixtos de pino-encino.
El continuo deterioro de las áreas naturales, la tasa de deforestación, el cambio constante del uso de suelo y todas las actividades antropogénicas en detrimento de la diversidad biológica, están ocasionando una perdida irreversible de los recursos naturales mucho antes de saber qué es lo que tenemos y cómo podemos utilizarlo. Es indiscutible entonces que la estrategia a seguir es buscar otras alternativas de estudio de la biodiversidad que nos permitan conocer de manera más rápida la identidad de las especies. Se han propuesto diferentes técnicas moleculares para tal fin, en donde destaca el código de barras genético como una alternativa de solución (Garibay-Orijel, 2014). Los códigos de barras genéticos son una herramienta de la biología molecular que ha revolucionado las tareas de identificación taxonómica, aportando un método confiable y relativamente económico. El principio de esta técnica se basa en identificar una región del ADN que permita discernir entre la variación inter e intraespecífica (Paz et al., 2011). Para el caso de los hongos, el marcador aceptado como código de barras genético universal es la región de los interespaciadores transcritos ribosomales (ITS) (Badotti et al., 2017; Schoch et al., 2012).
Con la finalidad de contribuir al conocimiento de la funga de la región de la costa, a la de Oaxaca, y a su vez, a la de México, en este trabajo se presenta un listado de macromicetos de una zona con selva baja caducifolia, se ilustran los nuevos registros y las recolectas recientes se acompañan de códigos de barras genéticos.
Materiales y métodos
El trabajo de campo se llevó a cabo en la Unidad de Manejo para la Conservación de Vida Silvestre “Jardín Botánico Puerto Escondido” (UMA-JBPE) de la Universidad del Mar, Oaxaca, México; el mismo está conformado por 15 ha destinadas a la restauración y conservación y 2 ha destinadas a mantener una serie de colecciones de plantas vivas. Éste se encuentra ubicado en el Km 239 de la carretera federal 131 Sola de Vega-Puerto Escondido, en el municipio de San Pedro Mixtepec, Distrito de Juquila. Geográficamente, se sitúa entre las coordenadas 15°54’14.75” - 15°55’1.06” N, 97°04’52.93” - 97°04’29.21” O, y en un rango altitudinal de 80 a 150 m snm (Fig. 1). La zona presenta lomeríos con pendientes de suaves a pronunciadas y con predominio de áreas planas a muy onduladas (INEGI, 2013). El clima es cálido subhúmedo con lluvias en verano de cuatro meses y sequía por el resto del año con temperatura promedio de 27 °C. Estas condiciones permiten el establecimiento de selva baja caducifolia, cuyos elementos arbóreos predominantes en nuestra zona de estudio son: Ceiba aesculifolia (Kunth) Britten et Baker fil., Cochlospermum vitifolium (Willd.) Spreng., Bursera simaruba L. Sarg., Amphipterygium adstringens (Schltdl.) Schiede ex Standl. y Enterolobium cyclocarpum (Jacq.) Griseb.
Usualmente los hongos macroscópicos desarrollan sus fructificaciones durante el periodo del año que presenta una mayor precipitación pluvial y temperatura apropiada, lo cual normalmente ocurre en nuestra zona de estudio entre los meses de julio a octubre. Durante dicho periodo de lluvias de los años 2007 al 2013, 2018 y 2019, se llevaron a cabo 27 visitas a la UMA-JBPE, donde se realizaron recorridos aleatorios de aproximadamente 4 horas en cada visita con la finalidad de recolectar y describir esporomas de macromicetos de acuerdo a las técnicas convencionales propuestas por Cifuentes et al. (1986). Todos los especímenes recolectados fueron fotografiados digitalmente y se secaron con una deshidratadora NESCO como método de preservación.
La identificación de las especies se realizó siguiendo las técnicas de microscopía de rutina (Largent et al., 1986) y con la ayuda de claves de identificación taxonómica y guías de campo como las de Cappello (2006), Cappello et al. (2013), Guzmán (2003), Guzmán y Piepenbring (2011) y Wartchow et al. (2008), entre otras. Para la validez de los nombres científicos, se consideró la base de datos en línea del catálogo de la vida (Roskov et al., 2020) y el Index Fungorum (2020). Los especímenes recolectados durante la temporada de lluvias del 2018 y 2019 fueron identificados tradicionalmente, usando caracteres morfológicos y se complementó con identificación molecular mediante códigos de barras genéticos, para lo cual se empleó la región de los ITS (Schoch et al., 2012). El ADN genómico de los esporomas se extrajo con el kit DNeasy Plant (Qiagen) siguiendo el protocolo del fabricante. Se amplificó la región ITS utilizando una mezcla de oligonucleótidos ITS1F e ITS4 (Gardes y Bruns, 1993; White et al., 1990). Para la reacción en cadena de la polimerasa (PCR) se utilizó Taq-&LOADTM MasterMix (MP Biomedicals) con las siguientes condiciones: 5 min a 94 ºC de desnaturalización seguidos de 35 ciclos de 30 s a 94 ºC, 30 s a 55 ºC y 60 s a 72 ºC, finalizando con 5 min a 72 °C (Pérez-Pazos et al., 2019). La amplificación de los productos de PCR se corroboró mediante electroforesis en gel de agarosa al 1% teñido con GelGreen® Nucleic Acid Gel Stain (Biotium). Los productos de PCR fueron limpiados con Exo-SAP-IT™ PCR Product Cleanup Reagent (Applied Biosystems) según las instrucciones del fabricante. La secuenciación de los productos de PCR se realizó en el Laboratorio de Secuenciación Genómica de la Biodiversidad y de la Salud, del Instituto de Biología, UNAM, con los cebadores empleados en la PCR en un secuenciador ABI 3100 (Applied Biosystems). La identificación molecular se determinó comparando las secuencias obtenidas, con la base de datos del Centro Nacional de Información Biotecnológica (NCBI por sus siglas en inglés) por medio del algoritmo BLAST y con la base de datos de UNITE (Abarenkov et al., 2010).
Así mismo, se hizo un análisis de acumulación de especies a partir de un modelo de regresión gaussiana y aplicando el método de mínimos cuadrados para determinar el número de especies estimado en la zona de estudio (Chapra y Canale, 2011).
Todo el material estudiado cuenta con el permiso expedido por la Subsecretaría de Gestión para la Protección Ambiental (con número de registro MEX/JB-065-OAX/12) y se encuentra depositado en el laboratorio de Colecciones Biológicas de la Universidad del Mar, campus Puerto Escondido, Oaxaca.
Resultados
Durante las 27 visitas a la UMA-JBPE, se recolectaron 225 ejemplares de macromicetos pertenecientes a 160 morfoespecies, de las cuales 35.6% (57 spp.) fueron determinadas taxonómicamente a nivel de especie y 5 más con la notación de “affinis” (Tabla 1). De las 160 morfoespecies reconocidas, 12 pertenecen al filo Ascomycota y 148 a Basidiomycota, mientras que los órdenes mejor representados son los Agaricales, con 38 géneros y 96 morfoespecies; le siguen los Polyporales con 18 géneros y 25 morfoespecies y los Xylariales con 4 géneros y 10 morfoespecies. Sobresalen los géneros Lepiota y Marasmius, con 14 y 9 morfoespecies respectivamente, seguidos de Psatyrella y Xylaria, cada uno con 7 morfoespecies.

Tabla 1 Listado de especies presentes en la UMA “Jardín Botánico Puerto Escondido”. El arreglo taxonómico está basado en Roskov et al. (2020) e Index Fungorum (2020). *** Nuevo registro para México, ** Nuevo registro para el estado de Oaxaca, * Nuevo registro para la región de la costa.
De los 40 especímenes recolectados entre 2018 y 2019, se obtuvieron 33 secuencias de ADN que corresponden a 30 especies identificadas molecularmente, algunas de ellas confirmadas con datos morfológicos (Tabla 2).
Tabla 2 Similitud nucleotídica de los ejemplares de macromicetos con código de barras de la UMA-Jardín Botánico Puerto Escondido, con los registros de la base de datos del Genbank. ID-COL: Número de recolecta; CGB: código de GenBank; ID-TAX: identificación taxonómica; BC: resultado de Blast más cercano; SN: porcentaje de similitud nucleotídica; VE: valor de significado estadístico (e-value); CGBC: código del GenBank del Blast más cercano.
| ID-COL | CGB | ID-TAX | BC | SN (%) | VE | CGBC |
|---|---|---|---|---|---|---|
| VO 1750 | MT939261 | Leucoagaricus sp. | Leucoagaricus sp. | 82.12 | 8X10-177 | KU647726 |
| VO 1754 | MT939262 | Trametes aff. variegata | Hexagonia variegata | 97.59 | 0 | MH856864 |
| VO 1757 | MT939263 | Podoschypha sp. | Podoschypha sp. | 86.00 | 0 | UDB033817 |
| VO 1764 | MT939264 | Trichaptum sp. | Trichaptum byssogenum | 94.61 | 1X10-137 | KR265130 |
| VO 1765 | MT939265 | Hexagonia sp. | Hexagonia apiaria | 96.20 | 0 | KC867362 |
| VO 1766 | MT939266 | Flavodon flavus | Flavodon flavus | 100 | 0 | JN710543 |
| VO 1767 | MT939267 | Crinipellis sp. | Crinipellis aff. iopus | 92.60 | 0 | FJ167636 |
| VO 1768 | MT939268 | Auricularia nigricans | Auricularia nigricans | 99.30 | 0 | JX065172 |
| VO 1769 | MT939269 | Crassisporus microsporus | Crassisporus microsporus | 99.50 | 0 | MK116496 |
| VO 1770 | MT939270 | Leucocoprinus sp. | Leucocoprinus sp. | 98.60 | 0 | KR154960 |
| VO 1771 | MT939271 | Volvariella sp. | Volvariella taylorii | 89.30 | 1X10-158 | MK412340 |
| VO 1772 | MT939272 | Crassisporus microsporus | Crassisporus microsporus | 99.50 | 0 | MK116496 |
| VO 1773 | MT939273 | Lepiota sp. | Lepiota sp. | 99.80 | 0 | MH211803 |
| VO 1774 | MT939274 | Psathyrella aff. cacao | Psathyrella cacao | 97.40 | 0 | NR_148106 |
| VO 1794 | MT939275 | Psathyrella sp. | Psathyrella candolleana | 96.70 | 0 | KT273363 |
| VO 1795 | MT939276 | Ganoderma martinicense | Ganoderma martinicense | 100 | 0 | MG654184 |
| VO 1827 | MT939277 | Scytinopogon sp. | Scytinopogon aff. pallescens | 100 | 0 | MK328887 |
| VO 1828 | MT939278 | Leucoagaricus sp. | Leucoagaricus americanus | 86.90 | 5X-143 | MN808630 |
| VO 1829 | MT939279 | Marasmiellus sp. | Marasmiellus ramealis | 94.60 | 0 | KY404985 |
| VO 1830 | MT939280 | Gloeophyllum striatum | Gloeophyllum striatum | 99.50 | 0 | KJ141171 |
| VO 1831 | MT939281 | Hohenbuehelia portegna | Hohenbuehelia portegna | 100 | 0 | KY426798 |
| VO 1889 | MT939282 | Agaricus aff. porphyropos | Agaricus porphyropos | 98.20 | 0 | MF511109 |
| VO 1890 | MT939283 | Tricholosporum sp. | Tricholosporum sp. | 87.40 | 2X10-99 | KR002902 |
| VO 1891 | MT939284 | Phlebopus beniensis | Phlebopus beniensis | 99.40 | 0 | MG996746 |
| VO 1892 | MT939285 | Lepiota sp. | Lepiota sp. | 86.80 | 0 | MH211692 |
| VO 1893 | MT939286 | Russula sp. | Uncultured Russula | 98.00 | 0 | KU175680 |
| VO 1894 | MT939287 | Gymnopus gibbosus | Gymnopus gibbosus | 99.80 | 0 | KY061203 |
| VO 1895 | MT939288 | Geastrum aff. violaceum | Geastrum violaceum | 96.60 | 0 | KF988450 |
| VO 1896 | MT939289 | Clavulina sp. | Clavulina sp. | 94.40 | 0 | MK547190 |
| VO 1897 | MT939290 | Marasmius congregatus | Marasmius congregatus | 99.10 | 0 | KP635166 |
| VO 1898 | MT939291 | Scytinopogon sp. | Scytinopogon aff. pallescens | 100 | 0 | MK328887 |
| VO 1899 | MT939292 | Marasmius aff corrugatiformis | Marasmius corrugatiformis | 96.40 | 0 | KX953757 |
| VO 1900 | MT939293 | Scytinopogon sp. | Scytinopogon aff. pallescens | 100 | 0 | MK328887 |
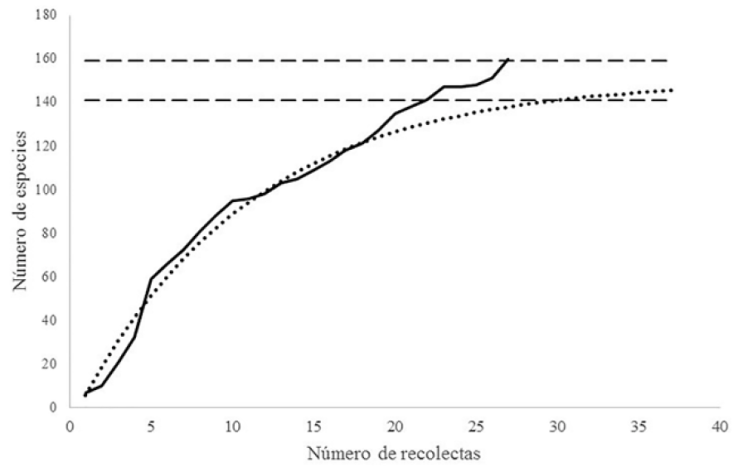
Con base en el modelo de acumulación de especies, se estima que en la zona de estudio pueden existir 150 taxones con un error estándar de ± 9 especies. La completitud del inventario, traducido como el estado de conocimiento, se determinó a partir del cociente del número de especies observado entre el número de especies estimado (160/150), que para nuestro caso es de 1.06, lo cual indica que conocemos alrededor de 6% más de lo esperado (Fig. 2).
Discusión
El bajo porcentaje en la determinación taxonómica a una categoría de especie (35.6%; menos de la mitad de las morfoespecies reconocidas), es reflejo de la carencia de estudios sistemáticos sobre macromicetos tropicales, los cuales pudieran servir como referencia para la identificación de especies; siendo ésto un factor importante que limita el conocimiento (Guzmán, 1998). Es necesario revertir esta situación, ya que los estudios sobre diversidad biológica del suelo forestal, indican que los trópicos son el hábitat de una mayor diversidad de hongos (O’Brien et al., 2005; Tedersoo et al., 2014). No sería de extrañarse que algunas de las morfoespecies que no pudieron ser identificadas fueran registros nuevos para la ciencia. Aun así, en términos de la contribución al conocimiento de la funga local, se registró un aumento de 22% (35 spp.) de registros nuevos para la región de la costa, de los cuales 28 especies se citan por primera vez para el estado de Oaxaca. Actualmente, se registran para todo el estado 716 especies de macromicetos, por lo que los resultados obtenidos generaron un aumento en el conocimiento de aproximadamente 4%. El aporte al conocimiento de la diversidad de macromicetos no solo fue a nivel regional, ya que también se citan 8 nuevos registros para el país (Calocybe carnea, C. gambosa, Marasmius congregatus, Gymnopus gibbosus, Phlebopus beniensis, Foraminispora rugosa, Ganoderma martinicense y Crassisporus microsporus), contribuyendo con ésto al conocimiento de la micobiota mexicana (Tabla 1; Fig. 3). En cuanto al conocimiento sobre los macromicetos que crecen en selva baja caducifolia de la región de la costa, hubo un avance bastante significativo, ya que aumentó 270% de lo previamente registrado, dando un total de 74 especies ahora conocidas.

Figura 3 1. Ophiocordyceps sobolifera; 2. Annulohypoxylon truncatum; 3. Hypoxylon rubiginosum; 4. Xylaria filiformis; 5. Calvatia cyathiformis; 6. Calvatia rugosa; 7. Leucoagaricus lilaceus; 8. Leucocoprinus birnbaumii; 9. Leucocoprinus fragilissimus; 10. Macrolepiota excoriata; 11. Hygrocybe cantharellus; 12. Calocybe carnea; 13. Calocybe gambosa; 14. Marasmius congregates; 15. Gymnopus gibbosus; 16. Hohenbuehelia portegna; 17. Pleurotus djamor; 18. Volvariella bombycina; 19. Volvopluteus gloiocephalus; 29. Parasola plicatilis; 21. Auricularia fuscosuccinea; 22. Phlebopus beniensis; 23. Geastrum lageniforme; 24. Geatrum minimum; 25. Gloeophyllum striatum; 26. Phylloporia spathulata; 27. Foraminispora rugosa; 28. Ganoderma martinicense; 29. Crassisporus microsporus; 30. Flavodon flavus; 31. Lentinus crinitus; 32. Lentinus velutinus; 33. Panus neostrigosus; 34. Thelephora versatilis; 35. Tremella wrightii. Escala: barra = 1 cm.
La proporción de especies del filo Ascomycota en relación al total de macromicetos en la zona de estudio es de 7.5%. Esto concuerda con lo reportado por varios autores quienes citan un valor que oscila entre 10 y 20% (Mori-del Águila et al., 2011; Villarruel-Ordaz y Cifuentes, 2007; entre otros). A pesar de ser un patrón observado principalmente en bosques templados y que existe una marcada carencia de estudios sobre macromicetos, particularmente los de afinidad tropical, los datos hasta ahora analizados señalan que el patrón antes mencionado es constante en la naturaleza. Por otro lado, el orden de los Agaricales es uno de los grupos más diversos dentro del filo Basidiomycota. La proporción de especies de este grupo en relación con el total de la funga macroscópica, puede oscilar entre 30 y 50% (Cifuentes et al., 1996; Villarruel-Ordaz et al., 2015); sin embargo, al parecer este valor tiende a aumentar en los bosques tropicales, tal como lo permiten suponer nuestros resultados, ya que en la zona de estudio, las 96 especies que pertenecen a los Agaricales representan alrededor de 60%.
La riqueza de especies concuerda con lo registrado por autores como Cannon y Kirk (2007), Cifuentes y Guzmán (1981), Guzmán y Piepenbring (2011), Lodge (2001) y Sunum et al. (2015), quienes señalan que géneros como Xylaria, Lepiota y Marasmius, tienden a estar muy bien representados en las zonas tropicales. En el caso del género Xylaria, se registra una riqueza de 28 especies para todo Oaxaca (Chacón y Guzmán, 1983; Medel, 2007; Medel et al., 2010; Pérez-Silva, 1975; San Martín y Rogers, 1989, 1995; San Martín et al., 1997; Villarruel-Ordaz et al., 2015; Welden y Guzmán, 1978), mientras que en la zona de estudio se identificaron 7 morfoespecies, lo cual representa 25% de lo que se conoce para todo el estado. Situación similar ocurre en el caso de los géneros Lepiota, Marasmius y Psathyrella.
El uso de códigos de barras genéticos se ha constituido como una herramienta poderosa en la identificación de especies, particularmente cuando éstas presentan una divergencia genética grande y una variación morfológica pequeña, como ocurre en las especies crípticas (Bickford et al., 2006; Kipling y Rubinoff, 2006; Paz et al., 2011). En muchas ocasiones, la comparación de secuencias de ITS ha revelado que una morfoespecie, cuya identificación está basada solo en la morfología, está constituida por más de una especie genética (Ramírez-López et al., 2015; Seifert, 2008; Yahr et al., 2016). Los criterios para separar especies por medio de códigos de barras suelen ser subjetivos y varían dependiendo del clado (De Salle et al., 2005; Garnica et al., 2016); sin embargo, existe un consenso que establece conservadoramente un valor estándar de 97% de similitud nucleotídica (3% de divergencia genética) entre las secuencias para ser consideradas conespecíficas (Anslam et al., 2018; Begerow et al., 2010; Kõljalg et al., 2013). Las secuencias obtenidas en nuestro estudio, al ser comparadas con secuencias de referencia depositadas en las bases de datos genéticos públicas, como UNITE y la del NCBI, muestran un alto grado de certidumbre en la identificación molecular, ya que 18 de las 33 secuencias analizadas presentan valores por encima del estándar (Tabla 2). Si consideramos ese valor estándar de similitud nucleotídica como parámetro para establecer un código de barras genético confiable, entonces nuestros resultados contribuyen con 18 códigos de barras de especies tropicales en las bases de datos públicas.
Los ejemplares identificados como Trametes aff. variegata (ID-COL VO-1754) y Psathyrella aff. cacao (ID-COL VO 1774); aun cuando presentan valores de similitud nucleotídica de 97.6% y 97.4%, respectivamente, fueron considerados con la connotación “affinis” debido a la enorme plasticidad fenotípica que se presenta en esas especies, por lo cual es necesario realizar una revisión más detallada de la morfología. En el caso del espécimen identificado como Agaricus aff. porphyropos (ID COL VO 1889), éste presenta un valor de 1.8 de divergencia genética. Optamos por considerarlo “affinis”, ya que autores como Drewinski et al. (2017) y Parra et al. (2018) han empleado dentro del género Agaricus valores de 0.1% hasta 1.2% de divergencia genética para considerar a 2 especies conespecíficas y/o separar especies hermanas. Aun cuando los datos moleculares respaldan la idea de que se trata de A. porphyropos, es necesario revisar con mayor detalle la morfología de nuestro espécimen. Por otro lado, Geastrum aff. violaceum (ID-COL VO 1895), con un valor de 96.6% de similitud nucleotídica, corresponde morfológicamente con G. violaceum, citada para México por Bautista-Hernández et al. (2015), sin embargo, debido a su alto valor de variación genética (3.4%), es probable que se trate de una especie críptica, la cual podría ser nueva para la ciencia.
Es notable señalar que, durante las recolectas de 2013 (a partir del número 15 en la Fig. 2), se observó un aumento en el número de especies acumuladas. Se sabe que a medida que se van realizando más muestreos, el número de especies que se registran va disminuyendo, no obstante, en nuestro caso no ocurrió de esa manera (Jiménez-Valverde y Hortal, 2003). Este aumento en la riqueza de especies podría ser resultado del huracán Carlota, que azotó las costas de Oaxaca en junio del 2012. Los huracanes son un factor que modifica la estructura y la composición de los ecosistemas (Navarro-Martínez et al., 2012). Durante un huracán, se abren claros en los bosques por el derribo de árboles (Pat-Aké et al., 2018), generando áreas de acceso a nuevas especies adaptadas a esos microclimas particulares. Una perturbación grave como la generada por los huracanes induce modificaciones en el ambiente y en la abundancia de sustratos, dando como resultado una variación en las comunidades fúngicas (Bergues-Pupo y Almarales, 2019; Lodge y Cantrell, 1995; Penttilä et al., 2013). El aumento de diversidad fúngica es un fenómeno que se ha observado en estudios hechos sobre el impacto de huracanes en el estado de Jalisco (Álvarez-Manjarrez y Garibay-Orijel, com. pers.). Consideramos que el incremento de la riqueza observada en la curva de acumulación, así como la tendencia a seguir aumentando el número de nuevos registros, es una respuesta conjunta entre el esfuerzo de muestreo, el efecto de un fenómeno natural como lo fue el huracán Carlota, la propia resiliencia ecológica y la implementación de técnicas moleculares para la identificación de especies.
Por todo lo anterior, concluimos que, a pesar de la escasez de bibliografía taxonómica útil para la identificación de hongos en bosques tropicales, el aporte al conocimiento de la diversidad de macromicetos para la región de la costa y para Oaxaca es bueno, con un incremento de 22 y 4% de nuevos registros, respectivamente. El valor casi triplicado (54 spp.) del número de especies previamente conocidas, creciendo en selva baja caducifolia de la región de la costa, es un reflejo de lo poco estudiado que está este tipo de vegetación, en cuanto a la funga se refiere. Del mismo modo, el bajo porcentaje de similitud nucleotídica que presentan 15 de nuestros 33 códigos de barras, no indica la necesidad de aumentar el muestreo de hongos en los bosques tropicales.
La mala identificación taxonómica de los especímenes, la carencia de datos descriptivos de las especies, así como la ausencia de secuencias de referencia disponibles en las bibliotecas públicas de datos genéticos actúan como limitantes para el uso de los códigos de barras en el proceso de la determinación taxonómica. Es necesario revertir este fenómeno fomentando la formación de micólogos que realicen una taxonomía integrativa, considerando las características de identificación morfológica, uso de técnicas moleculares y de datos ecológicos, entre otros.
Es factible pensar que nuestros resultados de la completitud del inventario no son incorrectos y que la diversidad de macromicetos de esa zona aún no se conoce por completo, por lo cual se sugiere continuar con el estudio de los hongos de la región.











 text new page (beta)
text new page (beta)