Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista mexicana de ingeniería química
Print version ISSN 1665-2738
Rev. Mex. Ing. Quím vol.10 n.2 Ciudad de México Aug. 2011
Biotecnología
Producción de proteínas recombinantes en Escherichia coli
Recombinant protein production in Escherichia coli
Álvaro R. Lara*
Departamento de Procesos y Tecnología Universidad Autónoma Metropolitana–Cuajimalpa Artificios 40, Col. Hidalgo, Delegación Álvaro Obregón, México, D. F, C.P. 01120. *Autor para la correspondencia. E–mail: alara@correo.cua.uam.mx
Recibido 11 de Febrero 2011.
Aceptado 11 de Abril 2011.
Resumen
La producción de proteínas recombinantes (PR) es una de las aportaciones más importantes de la biotecnología moderna. El hospedero bacteriano más importante para la producción de PR es Escherichia coli, aunque otros como Bacillus subtilis y Bacillus megaterium están cobrando cada vez más importancia. Debido a su impacto económico y en el sector de la salud humana, el presente trabajo se enfocará principalmente a la producción de PR terapéuticas en E. coli. Se abordarán en general los aspectos de diseño del vector, la cepa, el cultivo y la purificación de la PR, con énfasis en las oportunidades que la ingeniería metabólica ofrece para mejorar la producción de PR y las estrategias de cultivo.
Palabras clave: proteína recombinante, Escherichia coli, ingeniería metabólica, biorreactores.
Abstract
Recombinant proteins production (R P) is one of the most important contributions of modern biotechnology. The most important bacterial host for RP production is Escherichia coli, although B acillus subtilis and Bacillus megaterium are also gaining importance. Due to its economical relevance and impact on human health, the present review is foc used on the production of RP in E. coli. General aspects of vector design, strains election as well as cultivation and downstream processing are covered. Particularly, the opportunities to improve cultivation schemes and downstream through metabolic engineering are discussed.
Keywords: recombinant proteins, Escherichia coli, metabolic engineering, bioreactors.
1 Introducción
La producción de PR en bacterias es un a tecnología que surgió hace cerca de 30 años y respondió a una necesidad de proveer proteína de uso terapéutico con un abasto asegurado (que no dependiera de fuentes animales) y calidad constante. La primera PR aprobada para su uso en humanos fue la insulina humana producida en Escherichia coli por la empresa Genentech. Desde entonces, la tecnología de producción de PR ha tenido un avance muy importante. Hasta el año 2010, el número de productos biofarmacéuticos aprobados por la Administración de Alimentos y drogas de los EUA (FDA) sumaba más de 200, de los cuales la gran mayoría son PR (Walsh, 2010). La importancia económica de las PR de uso terapéutico es innegable: las 10 PR de mayor venta durante 2009 sumaron un total de 50,000 millones de dólares en ventas (Walsh, 2010), lo que representa casi la mitad de las reservas internacionales de México al 15 de septiembre de 2010.
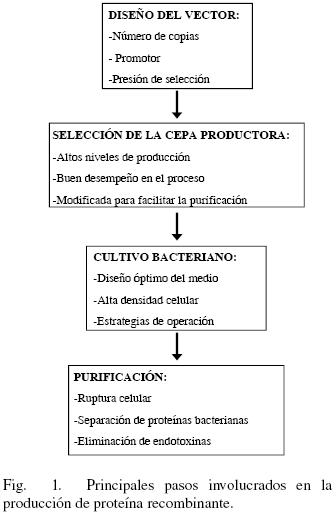
Muchas de las PR terapéuticas requieren modificaciones post–traduccionales que no pueden ser llevadas a cabo por cepas bacterianas silvestres, por lo que son preferentemente producidas por células eucariontes superiores. A pesar de ello, la bacteria E. coli sigue siendo una plataforma ampliamente usada para la producción de PR, ya que presenta una serie de ventajas importantes (Demain y Vaishnav, 2009): i) su genoma es conocido desde hace varios años, lo que amplía considerablemente las posibilidades de su manipulación genética, ii) existe una gran cantidad de conocimiento acumulado sobre su fisiología y metabolismo, iii) se tienen varios vectores bien establecidos para la producción de PR, iv) puede crecer rápido en medios muy simples, con altos niveles de producción de PR. Actualmente, cerca del 30 % de las PR de uso terapéutico son producidas empleando E. coli (Ferrer–Miralles y col, 2009), las más importantes pueden consultarse en la tabla 1. De un total de 58 productos aprobados por la FDA en el periodo 2006–2010, 17 son producidos usando E. coli y 32 empleando células de mamífero (Walsh, 2010). Una visión global de los aspectos involucrados en la producción de proteína recombinante por bacterias se muestra en la Fig. 1. Dichos aspectos se abordarán a lo largo del presente trabajo.
2 Vectores de expresión
La información genética de la proteína a producir usualmente se inserta en un vector (plásmido) de expresión. Los elementos esenciales de un vector de expresión para bacterias se muestran en la Fig. 2. El tamaño del plásmido es muy variable. En general, se prefieren plásmidos de alto número de copias (por arriba de 500 copias por célula). El número de copias es, en principio, función del origen de replicación del plásmido (aunque las condiciones ambientales del cultivo y el estado fisiológico de la bacteria tienen una fuerte influencia sobre el número de copias –Sayadi y col, 1989–). Generalmente se asume que un alto número de copias del plásmido resulta en un alto número de copias del gen de interés, y por lo tanto, la cantidad de PR producida será mayor cuanto mayor sea el número de copias. Modificaciones al origen de replicación de pMBl (por ejemplo, la familia de plásmidos pUC) son muy empleadas en la producción de PR. Sin embargo, el empleo de plásmidos con alto número de copia también induce una alta carga metabólica para la célula (observada como una reducción en la velocidad de crecimiento y en el rendimiento de biomasa en sustrato). También se ha observado que una alta concentración de ARN mensajero pude conducir a destrucción de los ribosomas y muerte celular (Baneyx, 1999). Un número de copias menor al de los plásmidos pUC puede favorecer una mayor producción de PR (Jones y col, 2000). En este punto, también influyen factores como la estabilidad del ARN mensajero (Carriery col., 1998).
Generalmentese asume que un alto número de copias del plásmido resulta en un alto número de copias del gen de interés y por lo tanto, la cantidadde PR producida será mayor cuanto mayor sea el número de copias. Modificaciones al origen de replicación de pMB1 (por ejemplo, la familia de plásmnidos pUC) son muy empleadas en la producción de PR. Sin embargo, el empleo de plásmidos con alto número de copia también induce una alta carga metabólica para la célula (observada como una reducción en la velocidad de crecimiento y en el rendimiento de biomasa en sustrato). También se ha observado qwue una alta concentración de ARN mensajero puede conducir a destrucción de ribosomas y muerte celular (Baneyx, 1999). Un número de copias menor al de los plásmidos pUC puede favorecer una mayor producción de PR (Jones y col., 2000). En este punto, también influyen factores como la estabilidad del ARN mensajero (Carrier y col., 1998).
En todo caso, deberá encontrarse el nivel de expresión óptimo para la PR producida en una cepa de E. coli en particular, lo cual también dependerá del promotor empleado. El promotor a emplear debe ser cuidadosamente seleccionado con el fin de poder regular la intensidad de inducción. Una expresión muy alta del gene heterólogo puede conducir a la formación de cuerpos de inclusión (agregados intercelulares de proteína), que pueden ser una ventaja para la purificación de PR, pero que también requieren pasos adicionales en la purificación, además de requerir recuperar el plegamiento adecuado de la PR antes de la formulación del producto final.
Existen varios promotores que han sido usados en experimentos de laboratorio, muchos de los cuales son poco prácticos para la producción industrial de PR. La Tabla 2 muestra algunos ejemplos de los promotores más comunes. Un problema encontrado con varios de estos promotores es la expresión basal (expresión del gene heterólogo en condiciones no inducidas). Idealmente el promotor a usar debería ser completamente regulable, muy fuerte, con una expresión basal mínima, a la vez que permitiera diferentes grados de inducción, preferentemente mediante un cambio en las condiciones de cultivo, para minimizar efectos tóxicos y maximizar la formación de PR. Mientras que los promotores lac y trp se han usado para experimentos de laboratorio durante mucho tiempo, son poco habituales a escala industrial. El promotor lac requiere la adición de un inductor que no es metabolizado (IPTG) y que es altamente tóxico. Esto representa una gran desventaja para procesos industriales, ya que se debe garantizar que dicho inductor sea completamente eliminado del producto, y además no debe ser enviado a aguas residuales municipales. En el caso del promotor trp (al igual que el promotor PhoA), la inducción ocurre cuando se agota un nutriente (triptófano en el caso del primero, fosfatos en el segundo caso). Esto dificulta el control de la fisiología bacteriana y determinar el momento preciso de la inducción. También impone una restricción en la formulación de los medios de cultivo. En el caso de que se aplique un inductor metabolizable, como la arabinosa, el cultivo está sujeto a adaptar su metabolismo a dicha fuente de carbono, que puede no ser la óptima para la producción de una PR (Sorensen y Mortensen 2005).
Un promotor más atractivo para aplicaciones industriales es el promotor pL/pR del fago lambda. Este promotor está regulado por la proteína cI857, que impide efectivamente la transcripción de los genes bajo control de pL/pR a temperaturas por debajo de los 37 °C. Dada la naturaleza termolábil de cI857, cuando la temperatura se incrementa (normalmente a 42 °C), la expresión es muy potente (Valdéz–Cruz y col., 2009).
El empleo del promotor pL/pR permite obtener altas densidades celulares (concentraciones de biomasa mayores a 40 g/L, medidas como peso seco), manteniendo el nivel de expresión del gene heterólogo a un nivel despreciable (normalmente cuando las temperaturas del cultivo están entre 28 y 32 °C). El incremento de temperatura es una operación sencilla a varias escalas de producción y permite manipular con exactitud el momento de la inducción (por ejemplo, una vez alcanzada una cierta densidad celular) sin agregar elementos secundarios al medio de cultivo, con la consecuente reducción de costos. Aunque el incremento de temperatura es más lento a medida que aumenta la escala del biorreactor, un calentamiento más lento puede de hecho favorecer la producción de PR (Caspeta y col, 2009). Una de las desventajas de la inducción por temperatura es que las células necesariamente experimentan un estrés fisiológico conocido como choque térmico, que conduce a respuestas generalizadas en el fluxoma, metaboloma y transcriptoma de E. coli (Wittmann y col., 2007) y que limitan el crecimiento y las funciones celulares una vez que se ha iniciado la inducción.
Existen además promotores inducibles a temperaturas bajas (máxima actividad a 20 °C) derivados de pL (Lim y col., 2000). Estos promotores, aunque mucho menos estudiados, pueden resultar atractivos para su aplicación industrial, debido a que se acoplarían los siguientes beneficios: el cultivo puede operarse a una temperatura óptima para el crecimiento de E. coli (37°C), y una vez alcanzada la densidad celular deseada, la temperatura puede reducirse conduciendo a la expresión del gen heterólogo. La solubilidad del oxígeno es 33 % mayor a 20 °C que a 37 °C, lo que resulta ampliamente benéfico para aumentar la transferencia de oxígeno al biorreactor, siendo ésta una de las principales limitaciones en los cultivos de alta densidad celular, como se explicará más adelante. Por otra parte, ha sido propuesto que aunque la velocidad de transcripción y traducción es menor a bajas temperaturas, la velocidad de plegamiento de la proteína es prácticamente la misma a 20 °C que a 37 °C, lo que sería benéfico para producir proteínas correctamente plegadas y evitar la formación de cuerpos de inclusión.
El gene heterólogo debe ser optimizado para su expresión en la bacteria, principalmente en el uso de codones y sobre–expresando ARN's de transferencia poco comunes. El ARN mensajero es una molécula de vida media muy corta, especialmente en E. coli, donde puede degradarse en menos de 20 s (Wang y col., 2004). Es posible incrementar la estabilidad del ARN mensajero alterando su conformación y eliminando la expresión de nucleasas como la RNAsa III (Sorensen y Mortensen, 2005).
Normalmente el plásmido de expresión contiene un gene que proporciona resistencia a antibiótico (en el ejemplo de la Fig. 2, resistencia a ampicilina). Esto permite emplear una presión de selección para asegurar que se cultivan únicamente las células que contienen plásmidos, ya que debido a efectos de segregación, el plásmido podría perderse y las células libres de plásmido crecerían más rápido (al carecer de la carga metabólica impuesta por el plásmido), desplazando a las células portadoras del mismo, con la consecuente pérdida de productividad. Sin embargo, la expresión del gene de resistencia al antibiótico es probablemente el origen principal de carga metabólica (Rozkov y col., 2004). Con la finalidad de reducir la carga metabólica, se han desarrollado sistemas que permiten seleccionar durante el cultivo a las células portadoras de información genética, contenida en el plásmido, que complemente alguna auxotrofía, por ejemplo, a algún aminoácido (Cranenburgh y col., 2001; Vidal y col., 2008). Dichas estrategias han mostrado ser muy eficientes, y también resultan en una reducción de costos en los cultivos industriales.
3 Selección de la cepa de producción
Un paso importante para alcanzar altas productividades consiste en la selección de la cepa de producción adecuada. Para ello deben considerarse aspectos cinéticos, el genotipo y el fenotipo de las cepas. En general se buscan cepas con baja tasa de mutación o de inserción de secuencias provenientes del plásmido. Las cepas derivadas de E. coli K–12 y E. coli B son las más empleadas. Los avances en el conocimiento de la fisiología de E. coli y en el desarrollo de herramientas moleculares han permitido la obtención de cepas diseñadas específicamente para la producción de proteína recombinante. En particular, la cepa BL21 es preferida en el ámbito industrial. Además de carecer de las proteasas Lon y Omp–t (lo cual reduce considerablemente la degradación de la PR dentro de la bacteria), produce bajas cantidades de acetato (un subproducto metabólico altamente indeseable del que se hablará más adelante), lo que resulta ampliamente atractivo (Phue y col., 2005). Otras cepas derivadas de BL21 incluyen versiones carentes de la RNAsa E (BL21 Star–pLysS), versiones diseñadas para incrementar la producción de PR que requieren el uso de codones raros en E. coli (cepa denominada Rosetta) y versiones que contienen copias extra de los genes argU, ileY y leuW que codifican para ARN's de transferencia y que reconocen codones poco usuales en E. coli (cepa denominada BL21–CodonPlus). La cepa AD494 es una mutante de K–12 en la tioredoxina reductasa (trxB) que permite la formación de enlaces disulfuro en citoplasma, que usualmente son reducidos en E. coli, lo que es un cuello de botella para el plegamiento de las PR. La cepa Origami (también derivada de K–12) contiene además una mutación en la glutationa reductasa (gor), para facilitar más la formación de enlaces disulfuro (Sorensen y Mortensen 2005; Graumann y Premstaller, 2006).
Todas las cepas modificadas que se han mencionado están disponibles comercialmente. La elección de una cepa en particular dependerá de varios factores, incluyendo el tamaño de la PR a producir, la presencia de enlaces (o puentes) disulfuro, la posibilidad de optimizar el uso de codones, entre otros. Es importante señalar que en los últimos años ha existido una intensa investigación para mejorar las capacidades de producción de PR en cepas de E. coli mediante ingeniería metabólica. Ese punto será tratado más adelante.
Las bacterias Gram–positivas como Bacillus subtilis y Bacillus megaterium también se han usado para la producción de PR. De hecho, B. subtilis es muy importante en la producción industrial de proteasas y de algunas vitaminas. Al carecer de membrana externa, estas bacterias pueden secretar una gran cantidad de protéınas plegadas al medio extracelular y contienen una baja cantidad de pirógenos (Fu y col., 2007). Sin embargo, los plásmidos recombinantes son poco estables en B. subtilis, y los rendimientos de PR son menores que en bacterias Gram–negativas, principalmente debido a la alta actividad de proteasas, cuya producción aumenta cuando B. subtilis encuentra condiciones de estrés (Huang y col., 2003), que también pueden conducir su esporulación. A pesar de las ventajas que dichas bacterias pueden tener en la producción de PR, es poco probable que en el corto plazo sean relevantes para el mercado farmacéutico. Por el contrario, su importancia en la industria de enzimas y química fina probablemente crezca.
4 Cultivo bacteriano
Para maximizar la productividad de PR, se prefiere alcanzar altas densidades celulares en el biorreactor. En un biorreactor de escala de laboratorio, se pueden alcanzar rutinariamente densidades mayores a los 100 g/L de biomasa empleando las condiciones de cultivo adecuadas. Existen, sin embargo, limitaciones en la escala industrial que hacen que valores tan altos de biomasa no sean alcanzables. Se ha acumulado una considerable experiencia en el cultivo de E. coli, lo que ha permitido mejorar notoriamente las productividades de los cultivos. La Tabla 3 resume algunos de los valores más altos de PR producida en cultivos de E. coli. A pesar de los considerables avances, hay todavía muchas dificultades técnicas que superar. Enseguida se detallan algunos de los aspectos más importantes para el diseño y operación de cultivos de alta densidad celular de E. coli.
4.1 Medio de cultivo
El medio de cultivo debe estar balanceado para la producción de biomasa y PR. Para ello, debe tomarse en cuenta la composición de la biomasa. Los principales componentes incluyen la fuente de carbono, nitrógeno, fósforo, azufre, potasio, y magnesio. Es necesario también incluir cofactores enzimáticos como Mb, Co, Ni, Fe, Ca, Cu, Mn, entre otros. Los factores de crecimiento como la tiamina son necesarios para un adecuado crecimiento. Aunque en cultivos en matraz agitado se agregan mono y di fosfato de potasio para formar un amortiguador de pH, esto no es necesario en cultivos en biorreactor, en los que el pH es controlado mediante la adición de ácido o base. También debe observarse que varios de los componentes del medio pueden resultar tóxicos a ciertas concentraciones; por ejemplo, el amonio es tóxico a partir de 3 g/L (Shiloach y Fass, 2005). Esta cantidad de amonio puede ser limitante para cultivos de alta densidad celular si se agrega en su totalidad al inicio del cultivo. Sin embargo, el pH puede controlarse con hidróxido de amonio, que además de servir como base es una fuente de amonio fácilmente asimilable, reduciendo con ello la acumulación de iones amonio. El medio de cultivo puede contener fuentes complejas de nitrógeno, como extracto de levadura o casaminoácidos. No obstante, existe una clara tendencia a eliminar el uso de componentes complejos, ya que no permiten un control adecuado de la fisiología, y reducen la reproducibilidad del cultivo.
Tomando en cuenta un rendimiento de biomasa típico de 0.5 g biomasa/g glucosa, se requerirían al menos 200 g/L de glucosa para llegar a una concentración celular de 100 g/L. La glucosa es tóxica a concentraciones superiores a los 50 g/L, por lo que no puede emplearse una concentración de glucosa inicial tan alta (Shiloach y Fass, 2005; Lara y col., 2008). Por otro lado, la producción de acetato debida a altas velocidades de consumo de glucosa impediría llegar a altas densidades celulares. Para evitar esto, se han desarrollado modos de operación del cultivo más apropiados.
4.2 Transferencia de oxígeno al biorreactor
Los cultivos de E. coli para la producción de PR son llevados a cabo bajo condiciones aerobias. De esta manera se obtienen mayores rendimientos de biomasa y producto, debido a que la generación de energía es mayor que bajo condiciones anaerobias. Además, bajo condiciones anaerobias la glucosa o fuente de carbono es oxidada sólo parcialmente por E. coli, lo que conlleva a la acumulación de subproductos de fermentación ácido–mixta, que resultan tóxicos. La demanda de oxígeno para la oxidación completa de glucosa a CO2 es una función directa de la velocidad de crecimiento de la bacteria conocida como velocidad específica de consumo de oxígeno (qo2). La velocidad de consumo de oxígeno (VCO) en un cultivo se obtiene multiplicando la velocidad específica de consumo por la concentración de biomasa:

Para evitar que la bacteria esté limitada por oxígeno, se requiere suministrar oxígeno al biorreactor a una velocidad que sea al menos igual que la velocidad de consumo. La velocidad de transferencia de oxígeno (VTO) al biorreactor es una función de muchas variables de operación del biorreactor, como la velocidad de agitación, potencia volumétrica suministrada, constante de transferencia de masa (kLa), temperatura, presión, composición del gas de entrada al biorreactor, entre otros.
Los parámetros operacionales como la velocidad de agitación no se pueden incrementar más allá de un límite económicamente permisible, por lo que la VTO en biorreactores normalmente no excede los 0.15 mmolCh/g h (Knoll y col, 2007). Esta VTO permitiría mantener condiciones aerobias a un cultivo de E. coli de no más de 40 g/L, creciendo a una velocidad de 0.4 h–1. Evidentemente, la VTO de los biorreactores convencionales no permitiría el desarrollo de altas densidades celulares en cultivos de E. coli. Para mantener condiciones aerobias en un biorreactor se puede recurrir a otras estrategias. Por ejemplo, emplear aire enriquecido con oxígeno u oxígeno puro para aumentar la solubilidad del oxígeno y con ello la VTO. También se puede manipular parámetros operacionales del biorreactor para incrementar la solubilidad del oxígeno. Por ejemplo, se ha reportado una VTO de casi 1 mol O2/L h en un biorreactor operado a una presión de 10 bar (Knoll y col., 2007). Se ha demostrado que el uso de presiones elevadas en los biorreactores no afectan la fisiología ni la productividad de cultivos de E. coli recombinante (Lara y col, en prensa). La VTO máxima del biorreactor puede llegar a ser el parámetro que determine la biomasa alcanzable. También puede manipularse la fisiología del cultivo para reducir la VCO, como se explica más adelante.
4.3 Modo de operación y sobreflujo metabólico
Como se mencionó anteriormente, la producción de acetato bajo condiciones aerobias es un problema constante en los cultivos de E. coli. La producción de acetato es indeseable ya que representa un derroche de esqueletos carbonados, afecta el pH y el gradiente de protones transmembranal, entre otros efectos. Por ejemplo, en la Fig. 3, se muestra la drástica disminución en el contenido de plásmido de células de E. coli como resultado de acumulación de acetato (Lara y Ramírez, en prensa).

La producción de acetato bajo condiciones aerobias se atribuye a un desbalance entre los flujos de carbono proveniente de glucosa (la cual es transportada a través del sistema de fosfotransferasa, PTS) en la glicólisis y el ciclo de los ácidos tricarboxílicos, lo que conduce a una acumulación de acetil coenzima A, la cual se transforma a acetato que se transporta hacia afuera de la célula (De Anda y col., 2006). En la Fig. 4 pueden verse detalles de estos procesos metabólicos. Este fenómeno de producción de acetato es conocido como sobreflujo metabólico. Una manera de evitar el sobreflujo metabólico es restringir la velocidad de consumo de glucosa mediante la adición controlada de una solución concentrada al biorreactor. Esto puede hacerse mediante esquemas de alimentación constante, alimentación con incremento lineal, con incremento exponencial, o empleando algoritmos de control más sofisticados para controlar el consumo mediante efectos indirectos del metabolismo, como la variación de la tensión de oxígeno disuelto (TOD) o el pH.

Un enfoque diferente ha permitido eliminar el acetato producido por E. coli mediante el uso de membranas semi–permeables (enfoque denominado como biorreactor con diálisis). Con ésta técnica se ha logrado la producción de más de 190 g/L de biomasa (Fuchs y col., 2002). Este enfoque, sin embargo, no impide el desperdicio de carbono ni contempla el control de la fisiología en el biorreactor.
El método de cultivo más empleado para alcanzar densidades celulares altas y evitar la producción de acetato es el cultivo alimentado exponencialmente. En este cultivo se utiliza una fase lote corta, para acumular biomasa a una velocidad de crecimiento máxima. Una vez agotada la fuente de carbono del lote (usualmente menos de 15 g/L de glucosa o glicerol), se inicia la alimentación de una solución concentrada de glucosa (de hasta 700 g/L). La velocidad de adición de glucosa se incrementa exponencialmente para mantener una velocidad de crecimiento constante (y con ello condiciones fisiológicas bien definidas). Para evitar el sobreflujo metabólico, debe mantenerse una tasa de consumo de glucosa menor al valor umbral que dispara el sobreflujo metabólico (Eiteman y Altman, 2006). Este valor dependerá de la cepa, el medio de cultivo y la temperatura del mismo, pero es común que corresponda a valores de velocidad específica de crecimiento menores a 0.15 h–1.
Al reducir la tasa de consumo de sustrato se reducirá por consecuencia la demanda de oxígeno para oxidarlo, lo que permite alcanzar densidades celulares mayores a las que se alcanzarían en un cultivo por lote, pero sin limitación por oxígeno. Ya que la variable objetivo en este tipo de sistemas es la velocidad de crecimiento (una variable macroscópica que se puede medir con mayor facilidad que la velocidad específica de consumo glucosa), los cultivos por lote alimentado se operan utilizando una bomba programable de acuerdo al algoritmo de control descrito por la siguiente ecuación (Lara y col., 2008):

Donde F es el flujo de alimentación, μset es la velocidad de crecimiento deseada, ΥX/S es el rendimiento de biomasa por sustrato, ms es el coeficiente de mantenimiento (fracción del sustrato empleada para funciones de mantenimiento celular), ρF es la densidad de la solución de alimentación, CXF y VLF son la concentración de biomasa y el volumen de líquido en el biorreactor al inicio de la alimentación, CSF es la concentración de glucosa en la solución de alimentación, t y tF son el tiempo de proceso y el tiempo al inicio de la alimentación, respectivamente. Con este método, ha sido posible llegar a concentraciones de biomasa de más de 100 g/L.
La Tabla 3 muestra que las mayores concentraciones de proteína recombinante alcanzadas se han logrado con cultivos alimentados exponencialmente, o en modo pH–stat. Este último modo de operación consiste en agregar glucosa una vez que el pH se incrementa por encima de un valor determinado (debido a re–consumo de acetato). Una vez dosificada la glucosa, el pH vuelve a caer debido al consumo de sustratos y producción de acetato. Con este método se pueden mantener velocidades de crecimiento relativamente altas evitando una acumulación excesiva de acetato.
Los cultivos por lote normalmente no permiten la acumulación de altas densidades celulares, debido a la acumulación de acetato. Sin embargo, Lara y col (2008) reportaron la producción de más de 8 g/L de proteína recombinante en un cultivo por lote empleando una cepa modificada. Las modificaciones al metabolismo central de esta cepa se explicarán más adelante.
4.4 Heterogeneidades ambientales
Otro problema común en los cultivos industriales es la existencia de gradientes espaciales en parámetros relevantes como pH, TOD, concentración de sustrato, CO2 disuelto, entre otros. Esto se debe principalmente a que el escalamiento ascendente de los biorreactores generalmente conduce a un incremento en el tiempo de circulación (tc), definido como el tiempo que una partícula en el biorreactor tarda en dar un recorrido al mismo y regresar al punto de partida (Lara y col., 2006a). La Tabla 4 muestra que empleando los criterios más comunes de escalamiento, se obtienen incrementos sustanciales en el tiempo de circulación en el biorreactor. Los criterios mostrados consisten en incrementar la escala de operación del biorreactor manteniendo constante el parámetro mencionado. Con excepción de una velocidad de agitación constante (N), que mantendría el tc sin variaciones, los demás criterios conducen a incrementos de hasta 100 veces en el tc. Sin embargo, mantener N constante requiere de motores cada vez más potentes, incluso inexistentes, que restringen este criterio para su aplicación.

Como resultado, el incremento en el tc es prácticamente inevitable. Esto conlleva a que existan regiones con diferentes concentraciones. Por ejemplo, la adición del agente de control de pH (una solución concentrada de base) se adiciona normalmente la superficie del caldo de cultivo y tardaría hasta 100 s en mezclarse completamente en el biorreactor. Lo mismo ocurre con la solución de glucosa en un cultivo alimentado. Una manera de estudiar los efectos que las condiciones de gran escala pueden tener en los cultivos es la simulación de las condiciones esperadas, empleando biorreactores de laboratorio, enfoque denominado escalamiento descendente (Lara y col., 2006a).
En biorreactores industriales, las bacterias viajan cíclicamente entre regiones con alta y baja concentración de sustrato. Como resultado, su metabolismo sufre desviaciones con exposiciones a gradientes de sustrato de duración tan corta como 2 s (Lara y col., 2009). Otro parámetro importante que presenta gradientes es la TOD, cuya importancia ya ha sido explicada. La exposición cíclica de E. coli a gradientes de TOD desata procesos transcripcionales en tiempos tan cortos como 17 s (Lara y col., 2006b). Sandoval–Basurto y col. (2005) reportaron que gradientes de TOD con tc de 180 s tienen un fuerte impacto en la producción de proinsulina humana, provocando una disminución de hasta 94 % en su producción. Esto demuestra la importancia de las heterogeneidades en la producción de PR por E. coli, que debe ser tomada en cuenta durante el escalamiento de los procesos productivos, para un diseño más informado de las cepas y procesos.
5 Purificación de PR
La purificación de las PR producidas por E. coli puede seguir varias vertientes (Cisneros–Ruíz y Rito–Palomares, 2005), dependiendo de la localization de la PR, la cual también proporciona ciertas ventajas según cada caso, como puede verse en la Tabla 5. Si la PR fue acumulada en el citoplasma, el primer paso es la ruptura celular, la cual se puede llevar a cabo empleado medios mecánicos o químicos. La homogeneización a alta presión es una operación escalable para este fin. La acumulación de PR en el citoplasma con frecuencia lleva a la acumulación de cuerpos de inclusión, lo cual depende de factores como la secuencia de la proteína, la fuerza del promotor, la temperatura, velocidad de crecimiento, entre otros factores, pero hasta el momento no es fácil predecir su formación. En ocasiones es deseable la formación de cuerpos de inclusión (incluso se agregan proteínas de fusión para conducir a su formación), ya que protegen a la PR de la proteólisis, y pueden ser separados por centrifugación continua. Sin embargo, los cuerpos de inclusión pueden tener agregadas grandes cantidades de contaminantes como ADN, ARN y chaperonas, haciendo que la proporción de PR en el cuerpo de inclusión llegue a ser tan baja como el 60 % (Singh y col, 2006; Ferrer Miralles y col, 2009). En la industria se han establecido protocolos de separación de estos contaminantes por centrifugación (Wong y col, 1996; Heeboll–Nielsen y col, 2003). En el caso de productos solubles en el citoplasma, deben tomarse medidas para evitar la proteólisis del producto, como reducir la temperatura o agregar agentes acomplejantes. Si las proteínas son acumuladas en el espacio periplásmico, la aplicación de un choque osmótico permite la extracción de la proteína a la vez que causa muy poco daño a las células.
La recuperación de la proteína puede llevarse a cabo empleando filtros de diversos tamaños y/o con superficie cargada. La filtración también es escalable y está bien establecida en la escala industrial. Los ácidos nucleicos pueden ser removidos mediante extracciones o bien agregando agentes policatiónicos como la polietinilamina. La proteína puede ser capturada o precipitada selectivamente empleando una variedad de materiales que han sido desarrollados para este propósito, como algunas resinas de interacción hidrofóbica (Kato y col, 2004).
En el caso de obtener cuerpos de inclusión, la proteína debe replegarse. La eficiencia de este paso es la influencia más fuerte para la productividad del proceso. La proteína obtenida de los pasos anteriores tiene diferentes estados conformacionales. Para obtener la proteína correctamente plegada y en su caso, los puentes disulfuro, el método más empleado es disolver el agregado con agentes caotrópicos, detergentes, o alcalinizar. Manipulando correctamente el estado de oxidación–reducción de la solución, es posible promover tanto el plegamiento correcto como la formación de los puentes disulfuro (Singh y Panda, 2005).
La purificación final de la proteína puede hacerse mediante métodos cromatográficos como intercambio amónico y catiónico, interacción hidrofóbica y cromatografía de fase reversa. Una revisión completa de estos métodos está fuera del alcance de este trabajo. La PR purificada debe además tener un contenido mínimo de proteína bacteriana, particularmente de endotoxinas. Es común también la eliminación de uno o dos aminoácidos terminales (p. ej. Metionina) que no pertenecen a la secuencia original de la proteína. La adición de polietilénglicol (PEGilacióri) a la proteína permite reducir reacciones inmunogénicas adversas (Kumagai y col., 2010).
6 Ingeniería metabólica aplicada a la producción de PR
La ingeniería metabólica tiene como objetivo la modificación del metabolismo para incrementar las capacidades celulares hacia la producción o el fenotipo deseado. Las aplicaciones de la ingeniería metabólica a la producción de PR por E. coli son muy variadas, pero se resumirán sólo algunas de ellas. Por ejemplo, una de las principales desventajas de la producción de PR en E. coli es que la bacteria no es capaz de hacer modificaciones post–traduccionales. Se han hecho esfuerzos conducentes a introducir vías metabólicas de glicosilación en E. coli que, aunque no son como las de humanos, han dado buenos resultados para mejorar la función de la PR (Waker y col., 2002; Skretas y col., 2009). Otros desarrollos intentan modificar el estado de oxidación–reducción intracelular o en el periplasma para lograr la formación de enlaces disulfuro en E. coli (Masip y col., 2004, Nguyen y col., 2011). Para facilitar la purificación y el procesamiento post–traduccional, también se ha buscado establecer estrategias eficientes para que la PR se transporte al espacio periplasmático (DeLisa y col., 2003), lo que también ha sido un área de investigación muy activa.
Las herramientas modernas de análisis fisiológico global han permitido estudiar las respuestas de E. coli a condiciones de producción y elucidar estrategias de mejora del metabolismo celular. Por ejemplo, Choi y col. (2003) estudiaron el transcriptoma de cultivos de alta densidad celular de E. coli produciendo PR. Basados en sus resultados, seleccionaron y co–expresaron dos genes (glpF, codifica para un transportador de glicerol y prsA, codifica para una fosforibosil pirofosfato fosfotransferasa), logrando aumentar la producción de PR a más del doble. Estudios proteómicos y del fluxoma también han arrojado información valiosa sobre la fisiología bacteriana en cultivos de alta densidad celular (Lemuth y col, 2008).
El estudio de respuestas transcripcionales a gradientes ambientales empleando sistemas de escalamiento descendente ha permitido generar cepas modificadas con una mejor producción de PR. Lara y col. (2006b) estudiaron la expresión de 21 genes en respuesta a oscilaciones de TOD. Basados en sus resultados, bloquearon parcialmente la ruta de fermentación ácido–mixta, lo que resultó en una mayor velocidad de crecimiento incluso bajo gradientes de TOD y aumentando al doble la producción de PR bajo condiciones constantes u oscilantes de TOD (Lara y col., 2006c).
Con el objetivo de reducir el sobreflujo metabólico y mejorar el desempeño en cultivos de alta densidad celular, Lara y col. (2008) emplearon una cepa de E. coli modificada en el sistema de transporte de glucosa. En esta cepa, el sistema nativo PTS fue bloqueado, y la glucosa es transportada a través de la permeasa de galactosa, que está sobre–expresada a nivel cromosomal en la cepa modificada (De Anda y col, 2006). Como se observa en la Fig. 5, el transporte a través de la permeasa de galactosa no depende de PEP como en el PTS (ver Fig. 4). Esto permite tener más PEP disponible para funciones biosintéticas. Las cepas mutantes en el PTS también tienen un mayor flujo de carbono hacia la vía de las pentosas fosfato, que incrementaría la cantidad de precursores de ácidos nucleicos para mantener una mayor cantidad de plásmido.

Dado que la velocidad de consumo de glucosa en este sistema es 33 % menor que en una cepa nativa, el sobreflujo metabólico es fuertemente reducido. Esto ha permitido el cultivo de la cepa modificada en modo lote empleando hasta 100 g/L de glucosa inicial, lo que condujo a una producción de 52 g/L de biomasa y más de 8 g/L de PR y solo 2 g/L de acetato (que después fue consumido por la bacteria), en contraste con la cepa nativa, que produjo 34 g/L de biomasa, menos de 4 g/L de PR y cerca de 14 g/L de acetato. La concentración de PR alcanzada en este estudio es la más alta reportada para un cultivo por lote. Cabe hacer notar que ni el medio ni la cepa fueron optimizadas para la producción de la PR modelo (proteína verde fluorescente), por lo que la producción podría mejorar aún más.
El cultivo por lote de esta cepa modificada empleando altas concentraciones de glucosa es una opción atractiva ante los cultivos alimentados que, como se ha explicado, presentan desventajas como tiempos de cultivo prolongados y presencia de gradientes de sustrato. Sin embargo, uno de los principales retos es el suministro de oxígeno, ya que se tiene una densa población celular respirando activamente. Se ha demostrado que la cepa es cultivada satisfactoriamente en biorreactores presurizados empleando hasta 130 g/L de glucosa inicial, con una mínima producción de acetato, mientras que la sobre–presión del biorreactor llegó a casi 5 bar y la VTO a 0.45 mol O2/L h (Knabben y col., 2010). Esta VTO es la más alta reportada para un cultivo por lote.
Los ejemplos anteriores muestran el potencial de la ingeniería metabólica no solo para incrementar la producción de PR y su modificación, sino también para mejorar el desempeño de E. coli ante condiciones de proceso y facilitar las etapas de purificación.
Perspectivas
El mercado de PR producidas por bacterias es muy importante a nivel mundial. Las ventajas de la producción de PR sobre otros organismos alienta la investigación para superar las desventajas de sistemas microbianos, como lograr modificaciones post–traduccionales y producir protéınas de elevado peso molecular. Los avances recientes en este campo, mantienen vigente el interés en lograr sistemas de expresión y de cultivo bacteriano cada vez más eficientes. La ingeniería metabólica y un mejor entendimiento de la fisiología de la bacteria durante la expresión y bajo condiciones de producción industrial permitirán sin duda incrementar los rendimientos y abrirán nuevas opciones de procesamiento de PR.
Referencias
Baneyx, F. (1999). Recombinant protein expression in Escherichia coli. Current Opinion in Biotechnology 10, 411–421. [ Links ]
Carrier, T.A., Jones, K.L. y Keasling, J.D. (1998). mRNA stability and plasmid copy number effects on gene expression from an inducible promoter system. Biotechnology and Bioengineering 59, 666–672. [ Links ]
Caspeta, L., Flores, N., Pérez, N.O., Bolívar, F. y Ramírez, O.T. (2009). The effect of heating rate on Escherichia coli metabolism, physiological stress, transcriptional response, and production of temperature–induced recombinant protein: A scale–down study. Biotechnology and Bioengineering 102, 468–482. [ Links ]
Cisneros–Ruiz, M. y Rito–Palomares. M. (2005). Estrategias de bioingeniería para la recuperación primaria de productos biológicos. Revista Mexicana de Ingeniería Química 4,131–139. [ Links ]
Cranenburgh, R.M., Hanak, J.A., Williams, S.G. y Sherratt, D.J. (2001). Escherichia coli strains that allow antibiotic–free plasmid selection and maintenance by repressor titration. Nucleic Acids Research 29, E26. [ Links ]
Choi, J.H., Lee, S.J., Lee, S.J. y Lee, S.Y. (2003). Enhanced production of insulin–like growth factor I fusion protein in Escherichia coli by the coexpression of the down–regulated genes identified by transcriptome profiling. Applied and Environmental Microbiology 69, 4737–4742. [ Links ]
Choi, J.H., Keumb, K.C. y Lee, S.Y. (2006). Production of recombinant proteins by high cell density culture of Escherichia coli. Chemical Engineering Science 61, 876 – 885. [ Links ]
De Anda, R., Lara, A.R., Hernández, V., Hernández–Montalvo, V., Gosset, G., Bolívar, F. y Ramírez, O.T. (2006). Replacement of the glucose phosphotransferase transport system by galactose permease reduces acetate accumulation and improves process performance of Escherichia coli for recombinant protein production without impairment of growth rate. Metabolic Engineering 8, 281–290. [ Links ]
Delisa, M.P., Tullman, D. y Georgiou, G. (2003). Folding Quality Control in the Export of Proteins via the Bacterial Twin Arginine Translocation Pathway. Proceedings of the. National Academy of Sciences of the U.S.A. 100, 6115–6120. [ Links ]
Demain, A.L. y Vaishnav, P. (2009). Production of recombinant proteins by microbes and higher organisms. Biotechnology Advances 27, 297–306. [ Links ]
Eiteman, M.A. y Altman, E. (2006). Overcoming acetate in Escherichia coli recombinant protein fermentations. Trends in Biotechnology 24, 530–536. [ Links ]
Ferrer–Miralles, N., Domingo–Espín, J., Corchero, J.L., Vázquez, A. y Villaverde, A. (2009). Microbial factories for recombinant pharmaceuticals. Microbial Cell Factories 8, 17. [ Links ]
Fu, L.L., Xu, Z.R., Li, W.F., Shuai, J.B., Lu, P. y Hu, C.X. (2007). Protein secretion pathways in Bacillus subtilis: implication for optimization of heterologous protein secretion. Biotechnology Advances 25, 1–12. [ Links ]
Fuchs, C., Köstera, D., Wiebuscha, S., Mahrb, K., Eisbrennerb, G. y Märkla, H. (2002). Scale–up of dialysis fermentation for high cell density cultivation of Escherichia coli. Journal of Biotechnology 93, 243–251. [ Links ]
Graumann, K. y Premstaller, A. (2006). Manufacturing of recombinant therapeutic proteins in microbial systems. Biotechnology Journal 1,164–186. [ Links ]
Heebøll–Nielsen, A., Choe, W.S., Middelberg, A.P. y Thomas, O.R. (2003). Efficient inclusion body processing using chemical extraction and high gradient magnetic fishing. Biotechnology Progress 19, 887–898. [ Links ]
Hu, S.Y., Wu, J.L. y Huang, J.H. (2004). Production of tilapia insulin–like growth factor–2 in high cell density cultures of recombinant Escherichia coli. Journal of Biotechnology 107, 161–171. [ Links ]
Huang, H., Ridgway, D., Gu, T. y Moo–Young, M. (2003). A segregated model for heterologous amylase production by Bacillus subtilis. Enzyme and Microbial Technology 32, 407–413. [ Links ]
Jana, S. y Deb, J.K. (2005). Strategies for efficient production of heterologous proteins in Escherichia coli. Applied Microbiology and Biotechnology 67, 289–298. [ Links ]
Jones, K.L., Kim, S.W. y Keasling, J.D. (2000). Low–copy plasmids can perform as well as or better than high–copy plasmids for metabolic engineering of bacteria. Metabolic Engineering 2, 328–338. [ Links ]
Kato, Y., Nakamura, K., Kitamura, T., Hasegawa, M. y Sasaki, H. (2004). Hydrophobic interaction chromatography at low salt concentration for the capture of monoclonal antibodies. Journal of Chromatography A 1036, 45–50. [ Links ]
Knabben, I., Regestein, L., Marquering, F., Steinbusch, S., Lara, A.R. y Büchs, J. (2010). High cell–density processes in batch mode of a genetically engineered Escherichia coli strain with minimized overflow metabolism using a pressurized bioreactor. Journal of Biotechnology 150, 73–79. [ Links ]
Knoll, A., Bartsch, S., Husemann, B., Engel, P., Schroer, K., Ribeiro, B., Stöckmann, C., Seletzky, J. y Büchs, J. (2007). High cell density cultivation of recombinant yeasts and bacteria under non–pressurized and pressurized conditions in stirred tank bioreactors. Journal of Biotechnology 132, 167–179. [ Links ]
Kumagai, I., Asano, R., Nakanishi, T., Hashikami, K., Tanaka, S., Badran, A., Sanada, H. y Umetsu, M. (2010). Integration of PEGylation and refolding for renaturation of recombinant proteins from insoluble aggregates produced in bacteria–application to a single–chain Fv fragment. Journal of Bioscience and Bioengineering 109, 447–452. [ Links ]
Lara, A.R., Galindo, E., Palomares, A.L. y Ramírez, O.T. (2006a). Living with heterogeneous bioreactors: Understanding the effect of environmental gradients in cells. Molecular Biotechnology 34, 355–381. [ Links ]
Lara, A.R., Leal, L.I., Flores, N., Gosset, G., Bolívar, F. y Ramírez, O.T. (2006b). Transcriptional and metabolic response of recombinant Escherichia coli to spatial dissolved oxygen tension gradients simulated in a scale–down system. Biotechnology and Bioengineering 93, 373–385. [ Links ]
Lara, A.R., Vázquez–Limón, C., Gosset, G., Bolívar, F., López–Munguía, A. y Ramírez, O.T. (2006c). Engineering Escherichia coli to improve culture performance and reduce by–product formationduring recombinant protein production under transient intermittent anaerobic conditions. Biotechnology and Bioengineering 94, 1164–1175. [ Links ]
Lara, A.R., Caspeta, L., Gosset, G., Bolívar, F. y Ramírez, O.T. (2008). Utility of an Escherichia coli strain engineered in the substrate uptake system for improved culture performance at high glucose and cell concentrations: an alternative to fed–batch cultures. Biotechnology and Bioengineering 99, 893–901. [ Links ]
Lara, A.R., Taymaz–Nikerel, H., van Gulik, W., Heijnen, J.J., Ramírez, O.T. y van Winden, W. (2009). Fast dynamic response of Escherichia coli fermentation metabolism to aerobic and anaerobic glucose pulses. Biotechnology and Bioengineering 104, 1153–1161. [ Links ]
Lara, A.R. y Ramírez, O.T. (2011). Production of plasmid DNA for therapeutic applications. En: Methods in Molecular Biology 3rd ed., (A. Lorence, ed.). Humana Press. En prensa. [ Links ]
Lara, A.R., Knabben, I., Caspeta, L., Sassi, J., Ramírez, O.T. y Büchs, J. (2011). Comparison of oxygen enriched air vs pressurized cultivations to increase oxygen transfer and to scale–up plasmid DNA production fermentations. Engineering in Life Sciences. En prensa. [ Links ]
Lemuth, K., Hardiman, T., Winter, S., Pfeiffer, D., Keller, M.A., Lange, S., Reuss, M., Schmid, R.D. y Siemann–Herzberg, M. (2008). Global transcription and metabolic flux analysis of Escherichia coli in glucose–limited fed–batch cultivations. Applied and Environmental Microbiology 74, 7002–7015. [ Links ]
Lim, J., Thomas, T. y Cavicchioli, R. (2000). Low temperature regulated DEAD–box RNA helicase from the antartic archaeon, Methanococcoides burtonii. Journal of Molecular Biology 297, 553–567. [ Links ]
Masip, L., Pan, J., Haldar, S., Penner–Hahn, J.A., Delisa, M.P., Georgiou, G., Bardwell, J.C.A. y Collet, J.F. (2004). Engineering a de novo pathway for the formation of protein disulfide bonds in bacteria. Science 304, 1185–190. [ Links ]
Nguyen, V.D., Hatahet, F., Salo, K.E.H., Enlund, E., Zhang, C. y Ruddock L.W. (2011). Pre–expression of a sulfhydryl oxidase significantly increases the yields of eukaryotic disulfide bond containing proteins expressed in the cytoplasm of E.coli. Microbial Cell Factories 10, 1. [ Links ]
Palomares, L.A., Estrada–Mondaca, S. y Ramírez, O.T. (2004). Production of recombinant proteins: challenges and solutions. Methods in Molecular Biology 267, 15–52. [ Links ]
Palomares, L.A., Lara, A.R. y Ramírez, O.T. (2010). Bioreactor Scale–Down. En: Encyclopedia of Industrial Biotechnology: Bioprocess, bioseparation and cell technology. (M.C. Flickinger, ed.), Pp. 1–13. John Wiley and Sons, Nueva York. [ Links ]
Phue, J.N., Noronha, S.B., Hattacharyya, R., Wolfe, A.J. y Shiloach, J. (2005). Glucose metabolism at high density growth of E. coli B and E. coli K: Differences in metabolic pathways are responsible for efficient glucose utilization in E. coli B as determined by microarrays and Northern blot analyses. Biotechnology and Bioengineering 90, 805–820. [ Links ]
Rozkov, A., Avignone–Rossa, C., Ertl, P., Jones, P., O'Kennedy, R., Smith, J., Dale, J. y Bushell, M. (2004). Characterization of the metabolic burden on Escherichia coli DH1 cells imposed by the presence of a plasmid containing a gene therapy sequence. Biotechnology and Bioengineering 88, 909–915. [ Links ]
Sandoval–Basurto, E., Gosset, G., Bolívar, F. y Ramírez, O.T. (2005). Culture of Escherichia coli under dissolved oxygen gradients simulated in a two–compartment scale–down system: metabolic response and production of recombinant protein. Biotechnology and Bioengineering 89, 453–463. [ Links ]
Sayadi, S., Barbotin, J.N. y Thomas, D. (1989). Effect of environmental growth conditions on plasmid stability, plasmid copy number, and catechol 2,3–dioxygenase activity in free and immobilized Escherichia coli cells. Biotechnology and Bioengineering 33, 801–808. [ Links ]
Shiloach, J. y Fass, R. (2005). Growing E. coli to high cell density–A historical perspective on method development. Biotechnology Advances 23, 345–357. [ Links ]
Singh, S.M. y Panda, A.K. (2005). Solubilization and refolding of bacterial inclusion body proteins. Journal of Bioscience and Bioengineering 99, 303–310. [ Links ]
Skretas, G., Carroll, S., DeFrees, S., Schwartz, M.F., Johnson, K.F. y Georgiou, G. (2009). Expression of an active human glycosyltransferase (Sialyltransferase ST6GalNAcI) in Escherichia coli strains with oxidizing cytoplasm. Microbial Cell Factories 8, 50. [ Links ]
Sorensen, H.P. y Mortensen, K.K. (2005). Advanced genetic strategies for recombinant protein expression in Escherichia coli. Journal of Biotechnology 115, 113–128. [ Links ]
Vallejo, L.F., Brokelmann, M., Marten, S., Trappe, S., Cabrera–Crespo, J., Hoffmann, A., Gross, G., Weich, H.A. y Rinas, U. (2002). Renaturation and purification of bone morphogenetic protein–2 produced as inclusion bodies in high–cell–density cultures of recombinant Escherichia coli. Journal of Biotechnology 94, 185–194. [ Links ]
Valdéz–Cruz, N.A., Caspeta, L., Pérez, N.O., Ramírez, O.T. y Trujillo–Roldán, M.A. (2009). Production of recombinant proteins in E. coli by the heat inducible expression system based on the phage lambda pL and/or pR promoters. Microbial Cell Factories 9, 18. [ Links ]
Vidal, L., Pinsach, J., Striedner, G., Caminal, G. y Ferrer, P. (2008). Development of an antibiotic–free plasmid selection system based on glycine auxotrophy for recombinant protein overproduction in Escherichia coli. Journal of Biotechnology 134,127–136. [ Links ]
Wacker, M., Linton, D., Hitchen, P.G., Nita–Lazar, M, Haslam, S.M., North, S.J., Panico, M., Morris, H.R., Dell, A., Wren, B.W. y Aebi, M. (2002). N–linked glycolylation in Campilobacter jejuni and its functional transfer into E. coli. Science 298, 1790–1793. [ Links ]
Walsh, G. (2010). Biopharmaceuticals benchmarks 2010. Nature Biotechnology 28, 917–924. [ Links ]
Wang, Y., Liu, C.H., Storey, J.D., Tibshirani, R.J., Herschlag, D. y Brown, P.O. (2004). Precision and functional specificity in mRNA decay. Proceeding of the National Academy of Sciences of the U.S.A. 99, 5860–5865. [ Links ]
Wittmann, C., Weber, J., Betiku, E., Krömer, J., Böhm, D. y Rinas, U. (2007). Response of fluxome and metabolome to temperature–induced recombinant protein synthesis in Escherichia coli. Journal of Biotechnology 132, 375–384. [ Links ]
Wong, H.H., O'Neill, B.K. y Middelberg, A.P. (1996). Centrifugal processing of cell debris and inclusion bodies from recombinant Escherichia coli. Bioseparation 6, 361–372. [ Links ]
Xie, L., Hall, M.A., Eiteman, A. y Altman, E. (2003). Optimization of recombinant aminolevulinate synthase production in Escherichia coli using factorial design. Applied Microbiology and Biotechnology 63, 267–273. [ Links ]
Zhou, L., Zhang, K. y Wanner, B.L. (2004). Chromosomal expression of foreign and native genes from regulable promoters in Escherichia coli. Methods in Molecular Biology 267, 123–134. [ Links ]














