Introducción
La variabilidad intra e interespecífica en el género Trichoderma dificulta la identificación de las especies. Aún en la actualidad, esto provoca discrepancias entre autores, respecto a diferentes taxas de los clados clasificados (Druzhinina y Kubicek, 2005 6B; Martínez et al., 2015).
En las primeras caracterizaciones morfológicas y culturales de aislamientos del género Trichoderma se detectó polimorfismo. Este fue uno de los motivos para que se designaran nueve especies agregadas (Rifai, 1969). La variabilidad entre aislamientos de una misma especie se observó, además, en aspectos, fisiológicos y bioquímicos (Gakegne, 2018) y moleculares (Pandya et al., 2017). Esta, en algunos casos, motivó que ciertos aislados se identificaran como especies diferentes. En relación con esto, Lieckfeldt et al. (1999) demostraron que la especie Trichoderma viride Pers. ex S. F. Gray estaba constituida por dos tipos morfológicamente distintos (tipo I y II) y distinguieron al tipo I como el “verdadero” T. viride (el anamorfo de Hypocrea) y el tipo II como una nueva especie, identificada molecularmente y nombrada como Trichoderma asperellumSamuels Lieckfeldt & Nirenberg (Samuels et al. 1999) y en 2010 la de Trichoderma asperelloides Samuels sp. nov, como nuevas especies. Además, con la utilización de estas técnicas se reubicaron cepas de Trichoderma harzianum Rifai en especies como T. asperellum, Trichoderma atroviride Bissett y Trichoderma longibrachiatum Rifai (Lieckfeldt et al., 1999; Hermosa et al., 2000), y cepas de T. atroviride o T. viride como T. asperellum (Watanabe et al., 2005). Consecuentemente, el taxa en Trichoderma incrementó de nueve, a más de 100 (Druzhinina et al., 2006); 254 (Bissett et al., 2015) y actualmente, se notifican 453 especies (Index Fungorum, 2021).
La incompatibilidad o compatibilidad somática o vegetativa evidencia la existencia o no de variabilidad genética entre aislados de una misma especie de hongo, ya que cepas con cercanía genética entre ellas, muestran patrones similares de compatibilidad y viceversa (Galdames, 2001).
La compatibilidad entre aislados de Trichoderma se trató de demostrar sobre la base que las células vegetativas posiblemente basado en la cantidad de núcleos que poseen estas (Samuels et al., 1998), pueden formar heterocarión entre varias especies por medio de la anastomosis hifal, fusión de protoplastos o las transferencias nucleares (Barcellos et al., 2011). Precisamente por esto, la compatibilidad vegetativa permite evaluar las relaciones genéticas entre especies y/o cepas, y es un marcador útil para mostrar diversidad o variabilidad genética en diferentes especies de hongos (Ortuño et al., 2013; Moo koh et al., 2018).
En la actualidad, los marcadores moleculares son ampliamente utilizados para el estudio de relaciones taxonómicas y diversidad genética en disímiles organismos, incluyendo los hongos (El_Komy et al., 2015). Los RAPD (del inglés Random Amplified Polymorphic DNA, Polimorfismo del ADN Amplificado al Azar) son de gran utilidad en la detección de polimorfismo intra e interespecífico en hongos, debido a que la técnica produce perfiles específicos individuales de ADN molde, mediante la amplificación de fragmentos aleatorios que se distribuyen por todo el genoma (Khattak et al., 2018). Esto ha permitido su utilización en el mapeo, como la generación de huellas genéticas (Hassan et al., 2019).
En la Micoteca del Centro Nacional de Sanidad Agropecuaria (CENSA) se conservan aislados con elevadas potencialidades como Agentes de Control Biológico, sin embargo, entre los más promisorios se demostró la existencia de variabilidad patogénica (Cruz-Triana et al., 2018; Gakegne, 2018; Infante y Martínez, 2020; Duarte et al., 2021), que se destacan por su acción biorreguladora sobre una amplia gama de enfermedades en diversos cultivos de interés económico y fisiológica (Infante et al., 2015). Por ello, el presente trabajo tuvo como objetivo caracterizar aislados de Trichoderma tomando como base aspectos morfo-culturales, su compatibilidad vegetativa y variabilidad molecular.
Materiales y métodos
Procesamiento de muestras y aislamiento de Trichoderma
Para el análisis, las muestras vegetales, primeramente se lavaron cuidadosamente con abundante agua corriente; durante una hora y secaron con papel de filtro Whatman No. 4. Seguidamente, se desinfectaron con hipoclorito de sodio al 1 % durante 30 s y posteriormente con alcohol al 70 % durante 1 min; entre cada desinfección y al final las muestras se lavaron con tres cambios de agua destilada estéril por tres minutos cada uno y secaron en papel adsorbente estéril. A continuación, las muestras se cortaron en fragmentos de 1 cm y se mezclaron para homogenizar. Por último, se seleccionaron segmentos al azar y sembraron en placas Petri de 90 mm de diámetro que contenían medio Papa Dextrosa Agar (PDA + antibiótico; cloranfenicol 0,1g L-1) e incubaron a 28 ± 2°C a oscuridad. También, se realizó el montaje de los fragmentos en cámara húmeda; la incubación se realizó a temperatura ambiente.
Las evaluaciones se realizaron diarias hasta la aparición de estructuras reproductivas, durante 24 - 48 h.
Para la identificación, se realizaron preparaciones tomando fracciones de las estructuras fúngicas visibles (micelio y/o esporas); las cuales se visualizaron al microscopio óptico Zeiss (modelo Axiostar plus; fabricado en Alemania) con aumento de 400x. Después se realizaron varias siembras de la colonia obtenida como Trichoderma; hasta obtener un cultivo puro.
Los aislamientos y cepas de Trichoderma objeto de estudio tienen diferentes orígenes (Tabla 1).
Table 1: Geographic origin of Trichoderma isolates and strains.
| Aislamientos de Trichoderma | Origen geográfico |
| T.13 | Cuba1. Guantánamo |
| T.17 | Cuba1. Pinar del Río |
| T.75 | Cuba1. La Habana |
| T.78 | Cuba1. Mayabeque |
| T.90 | Cuba1. Cienfuegos |
| T. atroviride (IMI206040) | Suecia2 |
| T. asperellum (215D) | Holanda2 |
| T. harzianum (G108) | Guatemala2 |
| T. viride | Holanda2 |
1Aislamientos cubanos de Trichoderma, conservados en el Laboratorio de Micología Vegetal (LMV) del CENSA.
2Cepas de referencia conservadas en el cepario del Laboratorio Nacional de Genómica para la Biodiversidad (Langebio), Centro de Investigación y de Estudios Avanzados (Cinvestav) del Instituto Politécnico Nacional (IPN), México.
1Cuban Trichoderma isolates, preserved in the Vegetable Mycology Laboratory (VML) of CENSA.
2Reference strains conserved in the strain collection of the National Laboratory of Genomics for Biodiversity (Langebio), Center for Research and Advanced Studies (Cinvestav) from the National Polytechnic Institute (IPN), Mexico.
Caracterización morfo - cultural de aislamientos de Trichoderma
La caracterización micromorfológica de los aislamientos de Trichoderma (T.) (T.13, T.17, T.75, T.78 y T.90) se realizó tomando como base los aspectos morfológicos señalados en las claves taxonómicas referidas por Rifai (1969) y Gams y Bissett (1998): dimensiones y forma de los conidióforos, fiálides y conidios, y además la ornamentación de estos últimos; mientras que para la caracterización cultural (macroscópica) se tuvo en cuenta el color, forma y textura de las colonias.
Las colonias de Trichoderma se obtuvieron a partir de suspensiones de esporas (10 µL de una suspensión con una concentración de 107 conidios.mL-1), procedentes de cultivos puros y monospóricos de tres días de sembrados en el medio de cultivo Agar Malta (AM) (BioCen). La suspensión se depositó centralmente en placas Petri de 90 mm de diámetro, que contenían medio AM (BioCen, pH 5,5), incubadas a temperatura de 20±2°C y régimen de oscuridad constante. Las evaluaciones [características de las colonias: color, forma y textura] comenzaron a partir de las 24 h de la inoculación, hasta las 72 h; se determinaron por observación visual, documentadas mediante fotografías tomadas con una cámara digital marca Canon PowerShot ELPH 180 8x (20 MP) (Proveedor Costa Rica).
La caracterización micromorfológica de los aislamientos se realizó mediante observaciones microscópicas de las estructuras fúngicas desarrolladas en los microcultivos. Para ello, sobre portaobjetos estériles se vertió asépticamente una película fina de medio de cultivo PDA (BioCen; esterilizado a 121°C, durante 15 min y pH ajustado a 5,5) sobre la cual se sembró un fragmento pequeño de micelio de los aislamientos individualmente, proveniente de la zona de crecimiento activo en la periferia de las colonias, crecidas sobre PDA durante tres días, a 28 ± 2°C y régimen de oscuridad constante. Posteriormente, los portaobjetos sembrados se colocaron en placas Petri de 22 mm de diámetro esterilizadas y se incubaron a temperatura de 28 ± 2°C y régimen de oscuridad constante. A las 24 - 48 h de la siembra se visualizaron y midieron las estructuras fúngicas (30 conidióforos, 30 fiálides y 30 conidios), en un microscopio óptico marca Zeiss (modelo Axiostar plus; fabricado en Alemania) con aumento de 100x y 400x. Las estructuras, se documentaron como evidencia gráfica con una cámara digital marca Canon PowerShot ELPH 180 8x (20 MP) (Proveedor Costa Rica).
Variabilidad genética de aislamientos de Trichoderma
Compatibilidad vegetativa (Cv)
Para realizar el ensayo, se utilizaron cultivos de los aislados de Trichoderma del LMV del CENSA, y de las cepas de T. atroviride (IMI206040), T. viride (TV1), T. asperellum (215D) y T. harzianum (G108), conservadas en el Laboratorio de Genómica para la biodiversidad, Langebio (Guanajuato, México). Todos los aislados y cepas crecieron durante cinco días en medio PDA (Difco) e incubados a 28 ± 2°C y régimen de oscuridad constante.
Para estimar las relaciones genéticas, se realizaron confrontaciones entre los aislamientos cubanos y entre estos y las cepas de referencia de Trichoderma; (Tabla 1). Para ello, un disco de micelio de seis mm de diámetro de cada aislado y cepa de Trichoderma, se sembró frente a otro, a una distancia de 70 mm centralmente, en placas Petri de 90 mm de diámetro contentivas de medio PDA (Difco). Posteriormente, las placas se incubaron bajo oscuridad constante a 28 ± 2ºC y evaluaron a las 72 h. Las evaluaciones de las interacciones se realizaron macroscópicamente y la compatibilidad de los aislamientos se determinó, según el tipo de reacción entre ellos:
Incompatible: Formación de una línea sin crecimiento evidente en la zona de contacto entre las colonias, normalmente coloreada o pigmentada; o cuando un aislado sobrecrece al otro.
Compatible: no se observa formación de línea en la zona de interacción después del contacto entre las colonias, aparentando una sola colonia.
Se tomó evidencia gráfica de las interacciones, con una cámara digital marca Canon PowerShot ELPH 180 8x (20 MP) (Proveedor Costa Rica).
Variabilidad molecular de aislamientos de Trichoderma
Método de extracción del ADN
Sobre celofán colocado en el centro de placas Petri (Ø = 90 mm), contentivas de medio de cultivo PDA (Difco), se sembró centralmente un disco de cada aislamiento cubano (Tabla 1) por placa. Las placas se incubaron por 72 h a una temperatura de 28 ± 2°C y oscuridad constante. Posteriormente, el micelio se colectó con una espátula al retirar el celofán de la placa y se congeló a -20°C. Para la extracción del ADN total se usó el protocolo DNeasy Plant Mini Kit (Qiagen, MBH, Germany). El pellet se resuspendió en 40 - 60 μL solución amortiguadora TE 0,5X, incluido en el juego de reactivos. La calidad del ADN se verificó por electroforesis en geles de agarosa al 0,8 %, con solución amortiguadora de corrida TAE 1X (Tris base 40 mM, ácido acético 57,1 mL y EDTA 2 mM a pH 8), durante 45 min a 100V, teñido con bromuro de etidio (cinco mg.mL-1) y visualizado en un fotodocumentador (Biorad Gel Doc XR). La concentración del ADN se determinó en un espectrofotómetro (nanodrop ND 1000), a las longitudes de onda de 260 y 280 nm, tomándose como muestras con buena calidad aquellas cuyos valores estuvieron entre 1,8 a 2 (Sambrook et al., 1989). La suspensión de ADN se conservó a 4°C hasta su uso en la técnica para detectar polimorfismo entre los aislados.
Análisis RAPD
En el ensayo se usaron los aislados cubanos de Trichoderma (Tabla 1). Para la amplificación por PCR con cebadores arbitrarios, se utilizó una mezcla de reacción cuyo volumen final de 25 μL contenía: solución amortiguadora de PCR (Tris HCl 20 mM a pH 8,4, KCl 50 mM), MgCl2 2 mM, dNTP 0,2 mM, Taq ADN polimerasa 1U (Invitrogen), 100 ng de ADN genómico total y 0,2 μM de cada cebador: OPA02, OPA13, OPB07, OPB09, OPB10, OPE14, OPG02, OPG10, OPH03, OPH19 y OPJ20 (Firma Operon Technologies, Alameda).
Las amplificaciones se realizaron en un termociclador API Thermal Cycler; Applied Biosystems, E.U. con el siguiente programa: un ciclo de desnaturalización inicial a 94°C durante cinco min, seguido de 45 ciclos de: 94°C durante un min (desnaturalización), 30°C durante 40 s (alineamiento) y 72 °C durante dos min (extensión); con un ciclo de extensión final a 72 °C, durante 10 min.
Los fragmentos amplificados se separaron por electroforesis en geles de agarosa al 1,5 % en TAE 1X, a 100 V durante 1,5 h. La visualización y el registro de los datos se realizaron como se describió anteriormente.
Análisis de los datos
A partir de los datos se confeccionó una matriz binaria donde se le dio el valor 1 a la presencia de la banda y 0 a la ausencia, solo se tuvieron en cuenta las bandas bien definidas. Posteriormente, se calculó el índice de similitud de Jaccard, se realizó un análisis de conglomerados por el método de UPGMA y un remuestreo tipo “bootstrap” de 10000, para estimar la robustez del dendrograma original, utilizando el paquete de software FreeTree (Hampl et al., 2001).
Resultados y discusión
Caracterización morfo - cultural de aislamientos de Trichoderma
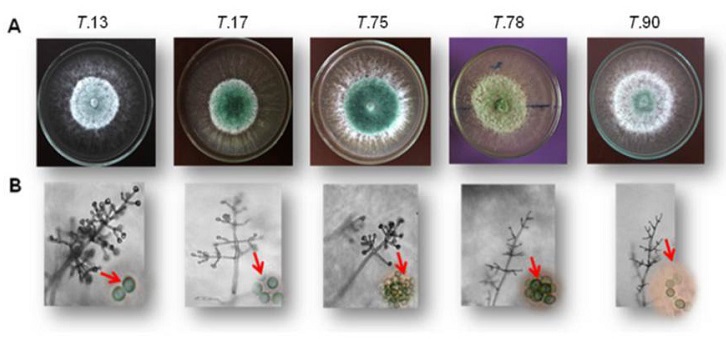
Los aislamientos de Trichoderma mostraron las siguientes características generales: rápido crecimiento, circular uniforme, colonias inicialmente blanquecinas, que se tornan de color verde o azulado a las 72 h; presencia de hifas hialinas septadas, conidióforos hialinos en penachos compactados en forma piramidal, fiálides alargadas y ensanchadas en el centro, y conidios ovalados de paredes ligeramente rugosas observadas con aumento de 1000X.
Estas características ubican los aislamientos fundamentalmente en la especie: T. viride, según aspectos descritos en las claves propuestas por Rifai (1969) y Gams y Bissett (1998).
Las características morfológicas observadas en los aislamientos coinciden con notificaciones realizadas por Samuels et al. (1999) y El-Sobky et al. (2019) en cuanto a: presencia de micelio hifal septado e hialino, conidióforo hialino, conidios generalmente ovalados, unicelulares, pequeños, formando racimos terminales, hialinos, de color verde al llegar a la maduración. Así como con las notificadas por Lieckfeldt et al. (1999), en cuanto a la ornamentación de los conidios.
No obstante, los aislamientos entre sí, presentaron diferencias en la coloración de las colonias y la morfometría de las estructuras; evidenciándose variabilidad (Tabla 2, Figura 1A y B).
Tabla 2 Características culturales y morfológicas de los aislamientos de Trichoderma, observadas a las 72 h.
Table 2: Morphological and cultural characteristics of Trichoderma isolates, observed at 72 h.
| Aislamientos | Parámetros | ||||
| Culturales (Fig. 1A) | Morfológicas (Fig.1B) | ||||
| Colonia | Conidióforos (µm) | Fiálides (µm) | Conidios (µm) | ||
| T. 13 | Verde brillante más oscuro después de las 72h. Presenta anillos que se unifican al pasar el tiempo. | (69) 71,4-88,1 (89,5) × (1,9) 2,3-4,6. | (4,7) 7,5-13,5 (15,9) × 2,3-3,7 (l/a 3,4). | Redondeados y/o elipsoidales 4,8-5,6 (6,4) × 3,2-4 (4,48) (l/a 1,4). | |
| T. 17 | Color verde azulado, con crecimiento uniforme, que llega a verde claro olivo durante la maduración de las esporas. | (40,1) 41-88,6 × 1,9-4,7 (5,1). | (5,6) 6,1-8,4 (9,8) × 2,3-3,3 (l/a 2,6). | Redondeados y/o elipsoidales 4,8-5,9 (6,4) × 3,2-4,8 (5,6) (l/a 1,3). | |
| T. 75 | Verde olivo oscuro, hasta la maduración de sus esporas. Presenta micelio aéreo blanquecino. | (50,4) 101,2-140 (198,7) × (2,8) 3,3-5,6 (6,5). | 5,1-10,3 (12,1) × 2,8-3,7 (l/a 2,5). | Redondeados y/o elipsoidales 4-5 (5,1) × 3,2-4,2 (l/a 1,24). | |
| T. 78 | Micelio aéreo de coloración verde olivo amarillento. | (38,2) 40,1-79,3 × (1,9) 2,8-5,1. | (6,5) 8,9-9,3 (10,3) × (2,3) 2,8-4,7 (l/a 2,4). | Redondeados y/o elipsoidales 3,2-4,8 × 2,4-3,8 (4) (l/a 1,4). | |
| T. 90 | Verde claro, micelio aéreo abundante. | (38,7) 46,6-93,3 × 3,7-4,7 (5,6) µm (l/a 1,5). | (3,7) 4,7-6,1 × (1,9) 2,3-3,3 µm (l/a 2,3). | Redondeados y/o elipsoidales 4-6,4 × (3,2) 3,5-4,5 (5,6) µm (l/a 1,2). | |
Si solo se tuviera en cuenta la variabilidad observada en los caracteres morfológicos y culturales (Tabla 2), los aislamientos se ubicarían en distintas especies, según lo descrito en la clave de Gams y Bissett (1998). Precisamente esto es uno de los motivos de identificaciones erróneas de especies (Druzhinina et al., 2006), depositadas en las bases de datos (Druzhinina y Kubicek, 2005 6B; Samuels et al., 2010).
Por esta razón, es necesario usar una taxonomía polifásica que aborde varios criterios de clasificación, que permitan identificar con mayor precisión y confiabilidad las especies del género (Samuels et al., 1999; Lieckfeldt et al., 1999; El-Sobky et al., 2019; Haouhach et al., 2020).
La variabilidad se observó, además, al nivel fisiológico, donde algunos de estos aislados pudieron esporular a 35ºC, T.13 y T.90 procedentes de la región oriental y central del país, donde la mayor parte del año las temperaturas medias son elevadas (Infante et al., 2015). También presentaron diferencias en la excreción de enzimas hidrolíticas, T.75 tuvo los mayores niveles de actividad quitinasa, mientras que T.13 y T.17 los mayores de glucanasa (González et al., 2012). Es posible que la variabilidad detectada intraespecífica esté relacionada con su proceso evolutivo, aspecto a seguir investigando.
Compatibilidad vegetativa (Cv)
Entre las cepas de referencia (Tabla 1), tomadas como patrones, se observaron reacciones de compatibilidad entre cepas de la misma especie (Figura 2A-E) e incompatibilidad entre especies diferentes (Figura 3A-C), excepto para la interacción F, G y H (Figura 2), las cuales fueron compatibles, aun siendo especies diferentes.
Figura 2 Reacciones de compatibilidad vegetativa entre las cepas de referencia. A) T. atroviride IMI206040 -T. atroviride IMI206040, B) T. virens - T. virens, C) T. asperellum 215D - T. asperellum 215D, D) T. harzianum 204 - T. harzianum 204, E) T. viride - T. viride, F) T. atroviride IMI206040 - T. asperellum 215D, G) T. asperellum 215D - T. viride, H) T. atroviride IMI206040 - T. viride. [1er hongo (izquierda) - 2do hongo (derecha)].
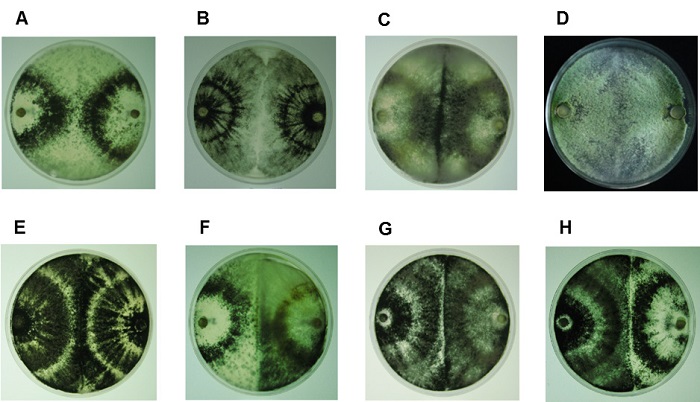
Figure 2: Vegetative compatibility reactions between reference strains. A) T. atroviride IMI206040 -T. atroviride IMI206040, B) T. virens - T. virens, C) T. asperellum 215D - T. asperellum 215D, D) T. harzianum 204 - T. harzianum 204, E) T. viride - T. viride, F) T. atroviride IMI206040 - T. asperellum 215D, G) T. asperellum 215D - T. viride, H) T. atroviride IMI206040 - T. viride. [1st fungus (left) - 2nd fungus (right)].
Esto indica, que estas especies deben estar genéticamente estrechamente relacionadas, pues la compatibilidad se logra cuando los alelos de todos los loci son idénticos (Galdames, 2001). Este aspecto se tomó como criterio en las observaciones del ensayo para los enfrentamientos de las cepas de referencia con los aislados cubanos.
En las pruebas de confrontación realizadas entre los cinco aislados de Trichoderma en estudio y las cepas de referencia antes mencionadas, se observaron también reacciones de compatibilidad e incompatibilidad.
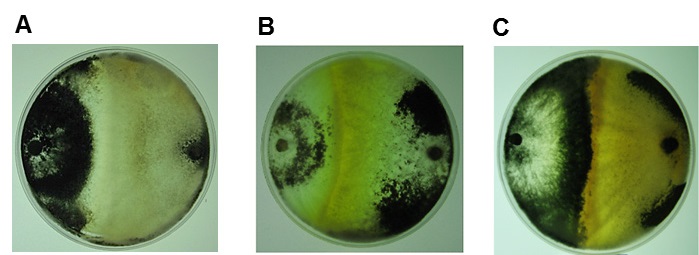
Las confrontaciones entre los aislados en estudio y las cepas de referencia T. harzianum (G108), mostraron una clara reacción de incompatibilidad (Figura 4A-E), determinada por la presencia de una barrera de inhibición [cambio de coloración en las colonias (excreción de metabolitos al medio, Figura 4A) o inhibición del crecimiento de una de las colonias, Figura 4E].
Figura 4 Reacciones de incompatibilidad vegetativa entre los aislamientos de Trichoderma y las cepas de referencia. A) T.13 - T. harzianum (G108), B) T.17 - T. harzianum (G108), C) T.75 - T. harzianum (G108), D) T.78 - T. harzianum (G108), E) T.90 - T. harzianum (G108). [1er hongo (izquierda)- 2do hongo (derecha)].
Barcellos et al. (2011) notificaron que la reacción de incompatibilidad se rige por un loci het, que limita la formación de heterocariones entre individuos genéticamente distintos. Es decir, si la diferencia es en uno o más loci het, las células que participan en la fusión son compartimentadas y se someten a un proceso lítico que conduce a la muerte celular. En estos casos los aislados son incompatibles; el fenómeno se identifica por la vacuolación, disolución de núcleos y generación de pigmentos, lo que se describió como barrera de reacción (Galdames, 2001).
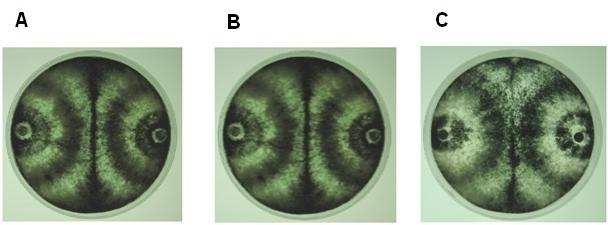
Por otra parte, en las interacciones de las cepas de T. asperellum (215D), T. atroviride (IMI206040) y T. viride (TV1) con los aislados en estudio se observaron reacciones de compatibilidad (Fig. 5A-Ñ), al igual que entre los propios aislados (Figura 6A, B y C).
Figura 5 Reacciones de compatibilidad vegetativa entre los aislamientos de Trichoderma y las cepas de referencia. A) T.13 - T. viride, B) T.17 - T. viride, C) T.75 - T. viride, D) T.78 - T. viride, E) T.90 - T. viride, F) T.13 - T. asperellum 215D, G) T.17 - T. asperellum 215D, H) T.75 - T. asperellum 215D, I) T.78 - T. asperellum 215D, J) T.90 - T. asperellum 215D, K) T.13 - T. atroviride IMI206040, L) T.17 - T. atroviride IMI206040, M) T.75 - T. atroviride IMI206040, N) T.78 - T. atroviride IMI206040, Ñ) T.90 - T. atroviride IMI206040. [1er hongo (izquierda) - 2do hongo (derecha)].

Figure 5: Vegetative compatibility reactions between Trichoderma isolates and the reference strains. A) T.13 - T. viride, B) T.17 - T. viride, C) T.75 - T. viride, D) T.78 - T. viride, E) T.90 - T. viride, F) T.13 - T. asperellum 215D, G) T.17 - T. asperellum 215D, H) T.75 - T. asperellum 215D, I) T.78 - T. asperellum 215D, J) T.90 - T. asperellum 215D, K) T.13 - T. atroviride IMI206040, L) T.17 - T. atroviride IMI206040, M) T.75 - T. atroviride IMI206040, N) T.78 - T. atroviride IMI206040, Ñ) T.90 - T. atroviride IMI206040. [1st fungus (left) - 2nd fungus (right)].
La compatibilidad observada entre estas especies pudiera estar relacionada con la existencia de un complejo entre T. viride - T. atroviride (ambas pertenecen a la sección Trichoderma, según análisis por ITS) (Lübeck et al., 2000). Consecuentemente, la identificación de T. asperellum por técnicas moleculares a partir de T. viride, justificó que T. atroviride y T. asperellum poseen características fenotípicas y talla del fragmento ITS muy similares (Hermosa et al., 2000). Este aspecto explica la compatibilidad existente entre los aislados de estas especies. Es por ello que, algunos aislados de T. atroviride o T. viride se reclasificaron como T. asperellum (Sánchez-García et al., 2017).
En relación con esto, Stocco (2014) demostró que entre las especies de la sección Trichoderma, que incluye a T. viride, más de una especie puede compartir la misma secuencia de SSU (gen de la pequeña subunidad 18S), lo que indica pocas diferencias entre estas. No se conoce si esta región está correlacionada con la Cv, y que, a ello, se deban los resultados obtenidos. Independientemente, la evaluación de la Cv tiene importancia para la identificación de aislamientos (Galdames, 2001).
Teniendo en cuenta la compatibilidad vegetativa que mostraron los aislamientos en estudio con las cepas de referencia T. asperellum 215D, T. atroviride IMI206040 y T. viride, se puede inferir, que estos tienen relación taxonómica con cualquiera de las tres especies antes mencionadas.
Cook y Baker (1983) con el uso de esta técnica (Cv) confirmaron la identificación taxonómica de aislados de Trichoderma realizada previamente por ellos, a partir de aspectos morfológicos. En relación con esto, Galdames (2001), opinó que este tipo de bioensayo (“prueba de compatibilidad”) puede ser considerado como un criterio de clasificación de las especies del género Trichoderma, debido a la consistencia de los resultados, al comparar el agrupamiento obtenido entre 21 cepas de Trichoderma spp., por técnicas moleculares (RAPD y AFLPs) y la Cv.
De forma general, esta prueba demuestra que cepas de una misma especie del hongo comparten patrones similares de compatibilidad, lo que se reduce entre especies más alejadas. Por tal motivo, puede ser considerada como una alternativa, simple y rápida para valorar la especie a que pertenece un nuevo aislado, siempre y cuando se cuente con cepas de diferentes especies correctamente identificadas molecularmente.
Desde el punto de vista práctico, es importante, ya que tiene implicación en la formulación de productos biológicos cuyo ingrediente activo es este antagonista y donde se utilicen consorcios de especies, debido a que algunos de ellos pueden inhibir al otro, sobre todo debido a liberación de compuestos no-volátiles (Lelay et al., 2007). Algunas investigaciones aclaran que cuando se aplica un consorcio de microorganismos de la misma especie (relación intraespecífica) o de especies diferentes (relación interespecífica) se podría obtener mayor inhibición de los patógenos, así como proporcionarle beneficios a la planta (Moo koh et al., 2018). En este sentido, Gallegos-Morales et al. (2022), evidenciaron compatibilidad in vitro entre T. asperellum, T. harzianum y Trichoderma lignorum; y al realizar co-aplicaciones tuvieron hasta el 86 % de rendimientos por planta, y disminución en la incidencia (71 %) y severidad por Fusarium sp., (59 %) en el cultivo de Chile (Capsicum annuum L.).
Análisis RAPD
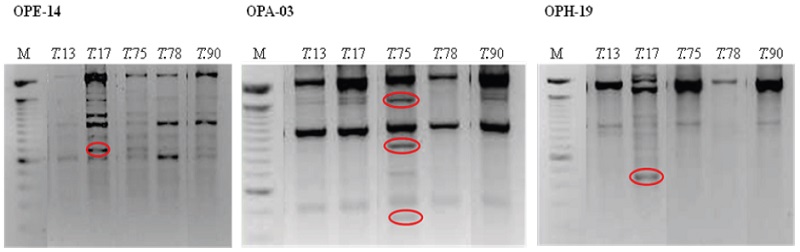
Los 11 marcadores generaron un patrón de amplificación claro y reproducible para cada aislamiento, con un total de 92 bandas reproducibles. De estas, 65 fueron polimórficas, para un 70,7 % de polimorfismo. Los iniciadores OPA-13, OPB-07, OPB-09, OPH-19 y OPJ-20 tuvieron 70 o más porciento de polimorfismo (Tabla 3), destacándose el OPH-19 con 100 % de polimorfismo.
Tabla 3 Total de bandas generadas a partir de los aislamientos cubanos de Trichoderma con el uso de 11 iniciadores RAPD.
Table 3: Total of bands generated from the Cuban Trichoderma isolates using eleven RAPD primers.
| Iniciador | No. Total de bandas | No. de bandas Polimórficas | % de Polimorfismo |
| OPA13 | 4 | 3 | 75 |
| OPB07 | 10 | 7 | 70 |
| OPB09 | 10 | 7 | 70 |
| OPB10 | 6 | 4 | 66,7 |
| OPE14 | 11 | 6 | 54,5 |
| OPG02 | 6 | 4 | 66,7 |
| OPH03 | 8 | 5 | 62,5 |
| OPH18 | 5 | 3 | 60 |
| OPH19 | 10 | 10 | 100 |
| OPG10 | 10 | 6 | 60 |
| OPJ20 | 12 | 10 | 83,3 |
| Total | 92 | 65 |
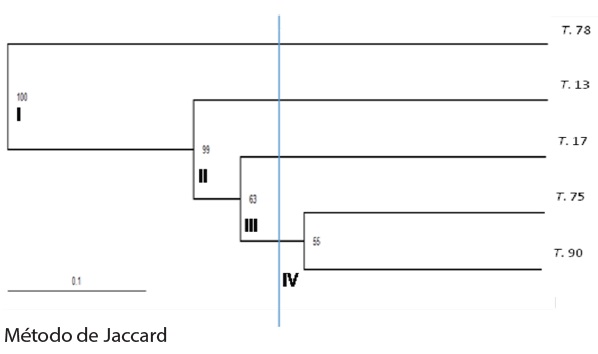
Los datos generados por los RAPD formaron cuatro grupos, una evidencia más de la variabilidad intraespecífica entre los aislados en estudio (Figura 7).
La variabilidad obtenida por RAPD entre los aislados puede ser usada para la detección de estos. El iniciador OPE-14 amplificó una banda en el aislado T.17 que lo diferencia de T.13, para T.75 con el iniciador OPH-03 se produjeron tres bandas que lo diferencian de T.78, y OPH-19, produjo una banda en T.17 que lo distingue del resto de los aislados (Figura 8). Diferencias que puntualizan dicha variabilidad. Desde otro punto de vista, estos resultados tienen importancia para el diseño de cebadores específicos, para su identificación, aspecto primordial para la autenticación, protección, y monitoreo de estos en campo.
Resultados similares obtuvieron Chandulal et al. (2016), quienes encontraron con cinco iniciadores RAPD variabilidad intraespecífica entre diez aislados de Trichoderma sp., obtenidos de suelo rizosferico de tomate (Solanum lycopersicum L.). También, Ranga et al. (2017) detectaron elevada variabilidad intraespecífica en nueve aislados de Trichoderma, con el uso de 15 iniciadores. Asimismo, coinciden con los obtenidos por Hernández et al. (2013), quienes con el uso de cuatro iniciadores RAPD evidenciaron variabilidad intraespecífica en diez aislados de Trichoderma spp. También, Hewedy et al. (2020) revelaron un elevado polimorfismo de 80 % de las bandas reproducibles en las cepas con siete iniciadores RAPD.
Por su parte, El_Komy et al. (2015), encontraron elevada variabilidad intraespecífica en 30 aislados de T. asperellum, con ocho cebadores RAPD, aunque esto no se relacionó con la actividad quitinasa y glucanasa de estas cepas. En sus investigaciones Kumar y Sharma (2011) declaran que, la variabilidad detectada con marcadores RAPD se relaciona con otros marcadores, fisiológicos o patogénicos. Los resultados del ensayo coinciden con los informado por estos autores, aunque parcialmente con los de El_Komy et al. (2015), ya que los aislados de Trichoderma cubanos, presentan marcada variabilidad enzimática (González et al., 2012).
Conclusión
Los resultados del presente estudio demuestran la variabilidad entre los aislados de Trichoderma sobre la base de caracteres morfológicos y culturales, así como genéticos, demostrada por la compatibilidad vegetativa y marcadores RAPD.
En general, estos resultados revisten gran importancia para la identificación, autenticación, protección y monitoreo de estos, en sistemas productivos. Para su uso como futuros controladores biológicos donde la variabilidad entre estos, sea la complementación, con vistas a la obtención de productos con mayor alcance, para su implementación dentro de un manejo integrado sustentable.











 text new page (beta)
text new page (beta)