Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
TIP. Revista especializada en ciencias químico-biológicas
Print version ISSN 1405-888X
TIP vol.13 n.1 Ciudad de México 2010
Artículo de revisión
Producción y características de cutinasas: Una alternativa interesante para biocatálisis a nivel industrial
Production and characteristics of cutinases: an interesting alternative for industrial level biocatalysis
Denise Castro-Ochoa, Carolina Peña-Montes y Amelia Farrés*
Departamento de Alimentos y Biotecnología, Facultad de Química, Universidad Nacional Autónoma de México, Circuito Exterior Universitario s/n, Coyoacán, C.P. 04510, México, D.F. *E-mail: farres@servidor.unam.mx
Artículo recibido el 29 de abril de 2010.
Aceptado el 27 de mayo de 2010.
Resumen
Las cutinasas (EC 3.1.1.74) son enzimas que catalizan la hidrólisis del polímero lipídico cutina, un componente estructural de la cutícula de las plantas. Estas enzimas muestran propiedades catalíticas de esterasas y lipasas, debido a que pueden hidrolizar ésteres solubles y triacilgliceroles, además de las reacciones inversas de síntesis en medios con baja actividad de agua. Esa versatilidad ha promovido su aplicación en áreas como la industria de alimentos, detergentes, producción de biodiesel, degradación enzimática de sustancias tóxicas y polímeros sintéticos. Las cutinasas han sido aisladas principalmente de hongos, siendo la más estudiada la cutinasa de Fusarium solani. Sin embargo, en los últimos años ha habido un mayor interés por la búsqueda de catalizadores con nuevas propiedades, por lo cual se han aislado y estudiado cutinasas en otras fuentes. En este trabajo se analizan la clasificación y estructura de estas enzimas, fuentes y medios de producción, y se profundiza en algunas áreas de aplicación con mayor auge en la actualidad.
Palabras Clave: Biocatálisis, biodiesel, cutina, cutinasa, esterasa, lipasa.
Abstract
Cutinases (EC 3.1.1.74) are enzymes that catalyze hydrolysis of lipidic polymer cutin, a structural component of plant cuticles. These enzymes display catalytic properties of esterases and lipases, as they can hydrolyze soluble esters and triacylglycerols, besides the reverse reactions of synthesis in a low-water environment. such versatility has promoted their application in areas such as the food industry, detergents, biodiesel production, enzymatic degradation of toxic substances and synthetic polymers. cutinases have been isolated mainly from fungi, being Fusarium solani cutinase the most studied. Nevertheless, there has been an increasing interest in the search of biocatalysts with new interesting properties in the last years, thus cutinases have been isolated and studied from other sources. In this review the classification, structures, sources and production of these enzymes are discussed, and applications in emergent areas are detailed.
Key Words: Biocatalysis, biodiesel, cutin, cutinase, esterase, lipase.
Introducción
Las cutinasas son enzimas muy versátiles que pueden catalizar, además de la ruptura del enlace éster de la cutina, reacciones de hidrólisis y síntesis in vitro de una amplia variedad de sustratos1. Estas propiedades catalíticas de las cutinasas tienen un uso potencial en diferentes industrias, por ejemplo, en alimentos, detergentes, producción de biodiesel, entre otras aplicaciones1-5. Asimismo, la elucidación de su estructura ha sido objeto de intensa investigación, nuevas estructuras tridimensionales y nuevas técnicas de biología molecular han provisto de herramientas para adaptar a las cutinasas a diferentes usos6-9.
La cutina es el componente principal de la cutícula, una estructura multicapa que cubre las paredes celulares externas de la epidermis en los órganos aéreos de las plantas, cuya función es brindar protección contra la pérdida de agua y el ataque de microorganismos10. La cutina es un polímero que consta de ácidos grasos de cadena larga que están unidos entre sí por enlaces tipo éster creando una red tridimensional rígida10. Está formada principalmente por ácidos grasos 16:0 y 18:1 con grupos hidroxilo o epóxidos situados en la parte media de la cadena o al final, en el lado opuesto al grupo carboxilo, aunque la composición precisa varía dependiendo de la especie11. El modo de penetración de esta barrera por microorganismos patógenos ha sido un tema polémico durante mucho tiempo. Mientras algunos reportes sugieren que la penetración puede ser por mera fuerza física debido al crecimiento del patógeno, otros autores presentan evidencias de que la degradación enzimática de la cutícula por cutinasas es esencial para la penetración en al menos algunas interacciones planta-hongo12.
Las cutinasas son enzimas inducibles producidas por microorganismos capaces de degradar la pared celular de las plantas. Catalizan la ruptura del enlace éster de la cutina originando la liberación de monómeros de cutina13. Las cutinasas han sido encontradas en diversas fuentes14-17, muchos hongos (principalmente patógenos) han sido reportados como productores de cutinasas, las cuales pueden asistir en la penetración de la cutícula de la planta. A partir de los trabajos pioneros de la actividad cutinasa del hongo fitopatógeno Fusarium solani f. sp. pisi14,18 y, de la demostración de la actividad cutinasa en otros hongos también patógenos, ha habido un especial interés en el papel de las cutinasas como determinantes de patogenicidad o virulencia en estos microorganismos. Evidencia del papel crítico de las cutinasas en la penetración de tejido sano del huésped ha sido demostrada en algunos hongos incluyendo Alternaria alternata19, Colletotrichum gloeosporioides20, Mycosphaerella sp.21 y Venturia inaequalis22, sin embargo, en estudios con otros hongos como Alternaria brassicicola23, Botrytis cinerea24 y Colletotrichum lagenarium25, en los cuales la patogenicidad se conservó intacta aun después de suprimir la actividad cutinasa por interrupción del gen de la enzima, han generado evidencia contradictoria, sugiriendo que las cutinasas no son requeridas para la patogenicidad del hongo.
Clasificación
Históricamente las enzimas suelen clasificarse con base en la especificidad de sustrato o reacción química que catalizan. De acuerdo con la nomenclatura de La Unión Internacional de Bioquímica y Biología Molecular26, las cutinasas reciben el número de clasificación EC 3.1.1.74, el cual las permite identificar como hidrolasas que actúan sobre enlaces ésteres de ácidos carboxílicos del polímero cutina.
El sistema de clasificación con base en la especificidad del sustrato en ocasiones presenta ciertos problemas. En el caso de las carboxilesterasas (3.1.1.x) la preferencia de sustratos frecuentemente se traslapa entre las clases definidas27, por lo que se considera a las cutinasas como intermedio entre lipasas y esterasas6. Asimismo, se ha observado que algunas enzimas que no están dentro de esta clasificación también pueden llevar a cabo la ruptura del enlace éster, como es el caso de algunas proteasas e incluso proteínas que no están clasificadas como enzimas (e.g. albúmina sérica) que también pueden llevar a cabo esta reacción28,29. Debido a la disponibilidad cada vez mayor de la información de secuencias en bases de datos públicas, una alternativa complementaria para la clasificación e identificación de enzimas ha sido la comparación de secuencias de aminoácidos, que además puede proporcionar un cuadro más claro sobre la relación evolutiva entre enzimas de diferente origen30.
Aplicaciones de cutinasas
En años recientes, la actividad esterolítica de las cutinasas ha sido ampliamente explorada. Carvalho et al.1 discuten a detalle las reacciones de biocatálisis que pueden llevar a cabo las cutinasas. In vitro las cutinasas despliegan actividad hidrolítica hacia una amplia variedad de ésteres, desde ésteres sintéticos solubles (e.g., p-nitrofenil ésteres) a triglicéridos de cadena larga insolubles31,32. Además en medios no acuosos o de baja actividad de agua, pueden catalizar reacciones de síntesis como esterificación y transesterificación de diferentes sustratos33. Estas reacciones de hidrólisis y síntesis tienen un uso potencial en la industria de alimentos, en la de lácteos para la hidrólisis de grasa de leche, en la industria de detergentes y oleoquímica, en la síntesis de triglicéridos estructurales, polímeros y surfactantes, en la síntesis de productos para el cuidado personal, degradación de insecticidas, sustancias tóxicas y polímeros sintéticos, y en la producción de biodiesel, entre otras aplicaciones1-5. A continuación se detallan algunas de las aplicaciones importantes.
Alimentos
Modificación de grasas y aceites
Las grasas y aceites son constituyentes importantes de los alimentos. El valor nutricional, sensorial y las propiedades físicas de un triglicérido son influenciados enormemente por factores tales como la posición estereoespecífica del ácido graso en el glicerol, el largo de cadena del ácido graso, y su grado de insaturación34. La estructura básica de grasas y aceites puede ser modificada de diferentes maneras: modificación química del ácido graso (hidrogenación), ruptura del enlace éster (hidrólisis), y reorganización de ácidos grasos en la cadena principal del triglicérido (interestificación)35. De esta manera, un lípido poco deseable y de bajo costo puede ser modificado en un producto con aplicaciones importantes con un alto valor.
La transesterificación de grasas y aceites, así como la esterificación selectiva de alcoholes, se puede obtener empleando cutinasa en bajas actividades de agua1.
La hidrólisis parcial de las grasas en la leche es una transformación típica mediada por enzimas endógenas de productos lácteos. Este proceso se puede realizar por enzimas de la microflora nativa en el producto o la adición intencional de enzimas exógenas36. Dependiendo del grado y especificidad de la hidrólisis, la grasa de leche modificada enzimáticamente exhibirá distintas notas de sabor, mantequilla, queso, crema, etc. Las grasas de leche hidrolizada por enzimas lipolíticas o grasas de leche lipolizada (LMF) ha llegado a ser por consiguiente un importante ingrediente en la industria alimentaria36. Algunos de sus usos actuales y potenciales son adición a los productos de panadería, cereales, dulces, productos lácteos, palomitas de maíz saborizadas, aderezos de ensaladas, botanas, etc. Regado et al.36 realizaron un análisis de la hidrólisis parcial de grasas de leche de vaca, ovejas y cabras empleando lipasas microbianas y la cutinasa de Fusarium solani pisi. Los ácidos grasos liberados fueron analizados por HPLC. El grado de hidrólisis y el perfil de los ácidos grasos de los productos finales fueron similares a los reportados en varios productos LMF.
Síntesis de aromas
Los ésteres de ácidos grasos son componentes importantes de aromas naturales usados en la industria de los alimentos. Por ejemplo, etil butirato e isoamil acetato son compuestos encontrados en el aroma de fresas y bananas, respectivamente37. Estos ésteres son obtenidos tradicionalmente por extracción de fuentes naturales, fermentación o síntesis química38. Sin embargo, estos métodos suelen ser costosos o de bajo rendimiento. Con el creciente interés por los productos "naturales", la industria del sabor está cada vez más interesada en el empleo de la biotecnología para la producción de estos compuestos, particularmente por métodos enzimáticos. La síntesis de ésteres mediante reacciones de síntesis directa en solventes orgánicos o transesterificación catalizadas por enzimas lipolíticas ha sido ampliamente reportada34. La cutinasa recombinante de F. solani pisi fue empleada para catalizar la reacción de transesterificación de butil acetato con hexanol para la obtención de hexil acetato, un éster de cadena corta con aroma frutal. Algunos parámetros relevantes para la actividad de la enzima como el contenido de agua, temperatura, molaridad del buffer, pH, surfactantes y concentraciones de hexanol y butil acetato fueron analizados mediante diseño factorial33. El empleo de cutinasas en reacciones de transesterificación de etanol y ácidos grasos de cadena corta (C2-C6) en medio orgánico también ha sido estudiada39.
Lipofilización de antioxidantes naturales
Los antioxidantes en alimentos retardan la rancidez causada por oxidación atmosférica protegiendo así a los lípidos y los compuestos liposolubles como vitaminas o carotenoides. Muchos compuestos han sido usados como antioxidantes en alimentos, incluyendo principalmente sustancias fenólicas artificiales como el butilhidroxianisol (BHA) y butilhidroxitolueno (BHT), los cuales se ha reportado pueden producir carcinogénesis40. Por ello ha habido un gran interés por el uso de antioxidantes naturales. Los ácidos fenólicos son antioxidantes naturales que están presentes en frutas, vegetales, hierbas aromáticas y especias. Son de interés particular debido a sus propiedades potenciales como antioxidantes, quelantes, antiinflamatorios, antialergénicos, antivirales y anticarcinógenos41. Los ácidos fenólicos (ferúlico, caféico, cumárico, etc.) generalmente son solubles en agua, por lo que su carácter hidrofílico reduce su efectividad en la estabilización de grasas y aceites40. Por lo tanto, la lipofilización o modificación de estos compuestos vía esterificación con moléculas alifáticas (ácidos grasos o alcoholes) se puede emplear como una herramienta para modificar su solubilidad en interfases agua-aceite, aire-aceite manteniendo sus propiedades funcionales originales41. La esterificación química y enzimática de compuestos fenólicos ha sido reportada por diversos autores42. Stamatis et al.43 estudiaron la esterificación enzimática de antioxidantes fenólicos naturales como los derivados del ácido benzoico y cinámico con alcoholes alifáticos, monosacáridos y alquilglucósidos empleando varias lipasas, esterasas y cutinasas en medios no acuosos.
Detergentes
La fabricación de detergentes biológicos es una tecnología en rápido crecimiento. Las lipasas, proteasas, amilasas y celulasas son enzimas que se añaden a las formulaciones de detergentes para la remoción de grasas, proteínas, carbohidratos, etc.34 El uso de cutinasas en formulaciones de detergentes para lavandería y lavaplatos ha sido evaluado44,45. La cutinasa presentó algunas ventajas en comparación con la lipasa comercial (LipolaseTM) en la remoción de triacilgliceroles en un solo proceso de lavado, así como en la hidrólisis de grasas en ausencia de calcio45.
Degradación de insecticidas, ftalatos y polímeros sintéticos
Insecticidas
El malatión es un insecticida y acaricida organofosforado sintético utilizado para el control de insectos en campos de cultivo, frutas, vegetales y ganado, y también ampliamente usado como sustituto del DTT2,46,. Éste, o sus residuos expuestos en el ambiente son absorbidos rápidamente por prácticamente todas las rutas incluyendo el aparato gastrointestinal, la piel, las membranas mucosas y los pulmones47. A partir de pruebas en animales y por la práctica en su uso se ha demostrado que afecta el sistema nervioso central de invertebrados, sistema inmune de cierta fauna vertebrada, y las glándulas suprarrenales, hígado y sangre de pescados48,49. El malatión también puede ocasionar mutaciones perceptibles en diversos tipos de células humanas cultivadas, incluyendo glóbulos blancos y células linfáticas2. El empleo de cutinasas y esterasas en la degradación de malatión ha sido evaluado. Kim et al.2 reportaron que la cutinasa de Fusarium oxysporum f. sp. pisi degradó 50 y 60% de la cantidad inicial de malatión en 15 y 30 minutos de reacción respectivamente.
Ftalatos
Los ftalatos son compuestos químicos usados principalmente en la fabricación de cloruro de polivinilo y a menudo en pinturas, lacas y cosméticos3. El dihexil ftalato (DHP), uno de los ftalatos más usados, también se utiliza en la elaboración de juguetes, guantes de vinilo, collares, zapatos y bandas transportadoras que se utilizan en operaciones del envasado de alimentos50,51. Los ftalatos han sido encontrados en sedimentos, agua y aire, y también en alimentos, ya que ellos pueden migrar del material de empaque51,52. El di-(2-etilhexil)-ftalato (DEHP), n-benzil butil ftalato (BBP) y DHP son ésteres ftálicos que están presentes principalmente en los materiales de empaquetado (papeles, cartulinas) para alimentos acuosos grasos y secos51,52. Durante los últimos años, los ftalatos de ocurrencia más común incluyendo el DHP, fueron clasificados como contaminantes y compuestos interruptores endocrinos (EDCs) conocidos por su efecto potencial sobre la salud humana al interferir con las hormonas endógenas del cuerpo53. Considerando la importancia de la búsqueda de biocatalizadores que sean efectivos en la degradación de ftalatos presentes en el ambiente y la estimación de la toxicidad de sus productos de degradación, se ha investigado la eficacia de la cutinasa de Fusarium oxysporum f. sp. pisi en la degradación de los ftalatos DEHP, DHP, BBP, DPeP y DPrP50-52,54,55.
Polímeros sintéticos
Los polímeros son de gran importancia y su producción ha aumentado en los últimos años. Sin embargo, el excesivo consumo de plásticos sintéticos derivados del petróleo ha tenido un impacto adverso en el medio ambiente debido a que la mayoría de estos plásticos sintéticos no se degrada en el ambiente56. Por esta razón, el desarrollo de polímeros o compuestos biodegradables, se presenta como una solución a la conservación del medio ambiente y al impulso de nuevas aplicaciones. A la fecha, se han desarrollado algunos tipos de poliésteres biodegradables como el poli-(ácido láctico) (PLA), poli (butileno-succinato) (PBS), poli-(butileno succinato-co-adipato) (PBSA) y poli-(caprolactona) (PCL), sin embargo, su costo de producción sigue siendo elevado comparado con los plásticos sintéticos no degradables4,57. Una comprensión de los mecanismos por los cuales los polímeros sintéticos naturales o sintéticos son degradados por las enzimas microbianas sería útil en el diseño de nuevos plásticos degradables. Las cutinasas son enzimas capaces de hidrolizar una amplia variedad de ésteres sintéticos; la degradación de plásticos biodegradables ha sido reportada para las cutinasas de los microorganismos F. solani f. sp. pisi, Pseudozyma jejuensis sp. nov., Aspergillus oryzae y Cryptococcus sp.4,16,57,58.
Producción de biodiesel
El biodiesel se ha convertido en un combustible alternativo en lo referente a los recursos energéticos tradicionales basados en combustibles fósiles, debido a la disminución de las reservas y aumento en el precio del petróleo, así como su contribución a la conservación del medio ambiente59. El biodiesel es un combustible renovable, biodegradable y no tóxico que se obtiene principalmente a partir de aceites vegetales o grasa animal mediante el proceso de transesterificación. Químicamente se describe como ésteres monoalquílicos de ácidos grasos de cadena corta o larga60. Se obtiene por transformación de triacilgliceroles en alquil ésteres de ácido graso en presencia de un alcohol59,60. A la fecha se han desarrollado varios procesos para la producción de biodiesel empleando ácidos, álcalis o enzimas como catalizadores de la reacción de transesterificación. Si bien es cierto que a nivel industrial se emplea la catálisis alcalina para la producción de biodiesel, hoy en día la catálisis enzimática ha ganado mucha atención59. Se han reportado diversos estudios empleando enzimas lipolíticas, principalmente lipasas en reacciones de transesterificación para la producción de biodiesel60. Sin embargo, las reacciones de transesterificación también se han evaluado usando esterasas y cutinasas empleando varios sistemas61. La cutinasa de F. solani pisi fue usada para catalizar la reacción de transesterificación entre una mezcla de triacilglicéridos y metanol en micelas reversas de bis(2-etilhexil) sulfosuccinato de sodio (AOT) en isooctanato con el propósito de producir biodiesel5. Los estudios se realizaron por lotes en pequeña escala, se estudió la influencia de la concentración inicial de enzima y alcohol, y la relación molar del sustrato. Conversiones de 75 % fueron obtenidas con tiempos de reacción de 24 horas, lo cual hace el proceso enzimático altamente competitivo cuando se compara con reacciones similares catalizadas por lipasas para la producción de biodiesel empleando metanol.
Producción
Microorganismos productores
Las cutinasas son enzimas hidrolíticas cuya producción se descubrió en hongos fitopatógenos que crecían en medios conteniendo cutina como única fuente de carbono18. Las primeras cutinasas que se aislaron fueron del hongo fitopatógeno F. solani f. pisi14. Posteriormente, se reportó la producción de cutinasas en polen de plantas17, bacterias15, levaduras16 e incluso en hongos no patógenos62,63, sin embargo, a la fecha los hongos fitopatógenos se siguen considerando como los principales productores de cutinasas. Algunos de los microorganismos productores de cutinasas son listados en la Tabla I.

Medio y condiciones de producción
La producción de enzimas es afectada por diversos factores como concentración y tipo de nutrientes, así como condiciones de crecimiento del microorganismo (temperatura, pH, velocidad de agitación, etc.). Las cutinasas microbianas son producidas en su mayoría en medios de cultivos sumergidos, sin embargo, también se ha empleado el método de fermentación en estado sólido64. El efecto de la fuente de carbono, nitrógeno, así como iones divalentes ha sido analizado en la producción de cutinasas65-67. En general, éstas suelen ser inducidas cuando se usa cutina o hidrolizado de ésta como fuente de carbono, e inhibidas por glucosa13. Con respecto a las condiciones de cultivo del microorganismo, se han evaluado temperatura68 y pH69, aunque existen muy pocos reportes en la literatura.
Efecto de nutrientes sobre la producción de enzimas
Las cutinasas son enzimas extracelulares generalmente inducidas por la presencia de cutina, monómeros de cutina o análogos de éstos en el medio de cultivo13,14 sin embargo, esto varía dependiendo del microorganismo analizado. F. solani f. sp. pisi produce una cutinasa extracelular cuando se cultiva empleando cutina o hidrolizado de cutina13. Cuando la cutinasa se indujo empleando el hidrolizado, se observó que los ω-hidroxi ácidos grasos fueron más eficientes en la inducción que los ácidos más polares de la cutina. Los alcoholes primarios n-alifáticos con 14 o más átomos de carbono también indujeron a la enzima13. Las cutinasas de Glomerella cingulata12 y Botrytis cinerea70 también fueron expresadas después del crecimiento del microorganismo en presencia de cutina o monómeros de cutina como inductores. Por el contrario, las cutinasas de Pseudomonas putida15, Thermonospora fusca ATCC 2773065 y Thermoactinomyces vulgaris NRLL B-1611771 sólo se producen en presencia de cutina debido a que, los hidrolizados de cutina o ácidos grasos individuales fueron inactivos como inductores de la producción de la enzima y en algunos casos inhibidores del crecimiento del microorganismo. En el hongo Alternaria brassicicola se encontraron dos clases de cutinasas23,72. La primera solamente fue expresada por contacto con cutina y no fue inducida por cera superficial o monómeros de cutina, en contraste, la segunda clase únicamente fue inducida por monómeros de cutina o por una prolongada exposición a la cutina intacta23,72. La cantidad de cutinasa producida también es afectada por la fuente y concentración de cutina. La cutina de las frutas Cucurbita maxima, Cucurbita moschata y manzana "Grannysmith" fueron usadas para inducir la actividad cutinolítica en dos cepas de Fusarium solani f. sp. cucurbitae73. La actividad fue inducida a diferentes niveles por las tres cutinas en cada cepa. La cepa que produjo mayor actividad fue la SAM410 con cutina de C. moschata, mientras que la cepa PGB153 alcanzó su máxima actividad con cutina de C. maxima. En ambos casos la menor actividad se obtuvo empleando la cutina de manzana como inductor73. Con respecto a la concentración de cutina, se analizaron concentraciones de 0.1, 0.5, 2.5 y 5 g/L, observando un incremento en la actividad producida con el aumento de concentraciones de cutina de hasta 2.5 g/L73.
Una de las principales desventajas del empleo de cutina como inductor de cutinasas es el alto consumo de tiempo y bajo rendimiento del proceso de producción, por lo que se han buscado alternativas a la cutina en la producción de cutinasas74. Se han explorado varias fuentes de carbono como glúcidos, ácidos grasos, aceites, así como fuentes de nitrógeno orgánicas e inorgánicas. Se ha observado que la adición de glucosa al medio de cultivo inhibe la producción de las cutinasas12,13,65, sin embargo, para la cutinasa de Colletotrichum lindemuthianum se obtuvieron concentraciones óptimas de glucosa para la máxima producción de la enzima66. La adición de glúcidos como fructosa, maltosa, galactosa, lactosa y sacarosa disminuyeron la producción de cutinasa de Fusarium oxysporum74, mientras que los ácidos y aceites analizados incrementaron la actividad de la cutinasa secretada por el hongo, el mejor resultado se obtuvo empleando aceite de linaza, cuyo valor fue incluso más alto que el obtenido con cutina. En el caso de la cutinasa de Thermonospora fusca, la adición de ácidos grasos o aceite provocó la inhibición del crecimiento del microorganismo65. Con respecto a la fuente de nitrógeno, se han obtenido buenos resultados empleando extracto de levadura en la producción de las cutinasas de F. oxysporum74 y C. lindemuthianum75.
Del mismo modo, se ha explorado con éxito el empleo de pulpa de manzana, cáscaras de tomate y papa en la producción de cutinasas65,71. Éstos se consideran subproductos agrícolas usados principalmente para alimentación animal, por lo que representan un potencial como inductores de bajo costo para la producción comercial de cutinasas microbianas vía fermentación.
Finalmente, además de las desventajas del uso de cutina como inductor antes mencionadas, en algunas ocasiones la producción de la enzima silvestre es muy baja; por lo que se ha estudiado y logrado la expresión exitosa de estas enzimas en sistemas heterólogos como Escherichia coli, Pichia pastoris, Saccharomyces cerevisiae, entre otros8,63,76,77.
Estructura
En 1992 se determinó experimentalmente la primera estructura de una cutinasa, fue la cutinasa del hongo Fusarium solani6. A la fecha existen 49 estructuras de cutinasas depositadas en la base de datos de proteínas (PDB)78, en su mayoría son variantes que incluyen uniones covalentes con inhibidores, así como mutantes construídas por diseño racional sobre la secuencia de la cutinasa de F. solani. Además de la de F. solani, recientemente se han elucidado las estructuras de las cutinasas de los microorganismos G. cingulata, Cryptococcus sp. y A. oryzae79-81.
La estructura de la cutinasa de F. solani ha sido la más estudiada y sobre ésta se han centrado las bases del estudio de la relación estructura-función de las cutinasas en general. No obstante los estudios de las estructuras de las cutinasas de G. cingulata y A. oryzae han permitido la determinación de nuevas propiedades estructurales y funcionales de estas enzimas.
La estructura tridimensional de la cutinasa de F. solani pisi es una molécula compacta, ~45 x 30 x 30 Â3 que consta de 197 aminoácidos con un peso molecular de alrededor de 22,000 daltons6,82. La enzima presenta un plegamiento conocido como α/β hidrolasa6, que es común en varias enzimas hidrolíticas de origen filogenético y función catalítica muy diferentes83. La estructura central de las α/β hidrolasas está formada principalmente por láminas β-plegadas paralelas conectadas por hélices-α83. Los residuos catalíticos constituyen siempre una triada altamente conservada: un nucleófilo (serina, cisteína o ácido aspártico), un residuo ácido y un residuo de histidina totalmente conservado84. La familia del plegamiento α/β hidrolasa incluye proteasas, lipasas, esterasas, deshalogenasas, peroxidasas y epóxido hidrolasas, haciéndolo uno de los plegamientos de proteínas más versátil y extenso84.
Las cutinasas son uno de los miembros más pequeños de las serín hidrolasas85. Su estructura está compuesta por una lámina-β central de cinco cadenas paralelas cubierta por cuatro hélices-α85. En la figura 1 se muestra un diagrama esquemático del plegamiento canónico de las cutinasas. El sitio activo consiste en la triada catalítica Ser 120, Asp175 e His1886. El péptido Gly-Tyr-Ser-Gln-Gly que contiene el aminoácido del sitio activo Ser 120, presenta una alta homología con la secuencia consenso Gly-(Tyr or His)-Ser-X-Gly comúnmente presente en lipasas. La serina 120 tiene una conformación ε (Φ = 59°, Ψ = -122°) típica de los residuos nucleofílicos de todos los miembros de las α/β hidrolasas86. Dos puentes disulfuro están presentes en la cutinasa de F. solani. El primero, Cys 31-Cys 109, uniendo el extremo del N-terminal a un giro-β y participa en la estabilización del plegamiento molecular global. El segundo puente disulfuro, Cys 171-Cys 178, se puede asumir que juega un papel importante en la estabilización de dos giros-β consecutivos, los cuales se localizan en el residuo catalítico Asp 17586.
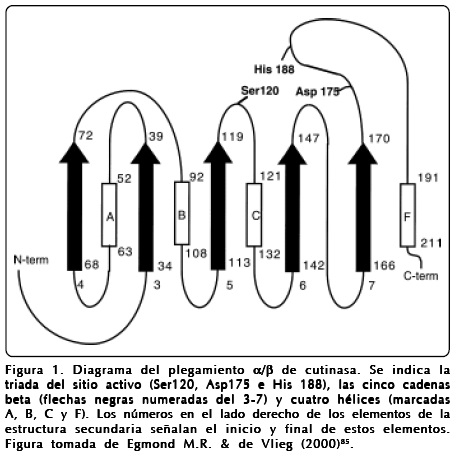
En la mayoría de las lipasas, el sitio activo se encuentra cubierto por una "tapa" anfipática. La apertura de esta tapa después de la interacción con una interfase hidrofóbica hace el sitio catalítico accesible al sustrato e incrementa la velocidad de hidrólisis87. En algunas lipasas, una segunda consecuencia del movimiento de la tapa es la formación de la cavidad oxianiónica88. Como en las serín proteasas, en lipasas la cavidad oxianiónica provee un ambiente electrofílico que estabiliza la carga negativa generada durante el ataque nucleofílico del enlace hidrolizable del sustrato89. A diferencia de las lipasas, el residuo catalítico Ser 120 de la cutinasa de F. solani no se encuentra cubierto bajo un lazo ("loop") o "tapa", sino que es accesible al solvente además, presenta la cavidad oxianiónica preformada. Esto podría explicar porqué las cutinasas no presentan activación interfacial6.
El estudio de la estructura de la cutinasa de G. cingulata por su parte reveló que la triada catalítica (Ser 136, Asp 191, e His 204) adopta una configuración inusual. La His 204 se gira hacia fuera del sitio activo en una posición donde es incapaz de participar en la catálisis, con el anillo de imidazol 11 Â lejos de su posición esperada79. Mediante experimentos de espectrometría de resonancia magnética nuclear, se confirmó la importancia de este residuo en el mecanismo de la enzima, por lo que se sugiere que durante el ciclo catalítico la cutinasa experimenta un cambio conformacional orientando a la His 204 de una conformación inactiva a una conformación activa, en la cual la triada asume una configuración clásica79.
La reactividad, estabilidad y estructura de la cutinasa de Aspergillus oryzae fue comparada con la de F. solani81. La cutinasa de A. oryzae es una proteína monomérica con un plegamiento α/β compuesto de una lámina-β de cinco cadenas paralelas rodeadas por 10 hélices-α. Esta enzima posee un sitio activo compuesto de los residuos Ser 126, Asp 181, e His 194 de la triada catalítica81. La comparación de las estructuras reveló que ambas cutinasas tienen un plegamiento similar. Sin embargo, la enzima de A. oryzaepresenta varias características estructurales que difieren significativamente de la de F. solani. La estructura de la cutinasa de A. oryzae contiene un único puente disulfuro entre Cys63 y Cys76 que enlaza la hélice 2 con la cadena 2 de la lámina-β central. Este puente disulfuro no había sido reportado previamente en ninguna estructura de cutinasas81. Los otros dos enlaces disulfuro que presenta la estructura de la cutinasa de A. oryzae entre Cys37-Cys115 y Cys177-Cys184 se conservan en las estructuras de las otras cutinasas reportadas6,79-81. Además se encontró que las enzimas de A. oryzae y F. solani presentan diferencias en la actividad y especificidad, lo cual se esclareció mediante el detalle molecular provisto por la estructura. La cutinasa de A. oryzae hidroliza preferentemente sustratos de cadena larga lo cual se debe a un surco profundo continuo que se extiende a través del sitio activo, mientras que la de F. solani que tiene mayor preferencia por sustratos de cadena corta presenta un surco bajo e interrumpido81. En la comparación de termorresistencia, los experimentos de termoactividad y termodinámica mostraron mayor termorresistencia de la cutinasa de A. oryzae, atribuida al puente disulfuro adicional que presenta la estructura de esta enzima comparada con la de F. solani81.
Conclusiones
Las cutinasas son enzimas producidas principalmente por hongos fitopatógenos, cuya relación con la patogenicidad del microorganismo y su efecto en la invasión al huésped sigue en controversia.
Las cutinasas tienen propiedades catalíticas muy versátiles con un gran potencial en aplicaciones biotecnológicas. El uso de estas enzimas en biocatálisis es un área emergente que ha generado resultados interesantes, por lo que resulta necesaria la búsqueda de nuevas enzimas, así como profundizar en el estudio de sus aplicaciones a nivel industrial y generar rutas alternativas para la producción de compuestos químicos interesantes con el uso de éstas. Por otra parte, es importante también estudiar la inmovilización de cutinasas con el fin de obtener biorreactores enzimáticos que puedan ser escalables a nivel industrial.
La aplicación industrial de las cutinasas requiere la sobreexpresión de la proteína, ya sea en la cepa silvestre o bien en un sistema heterólogo. Por lo que es necesario intensificar los estudios sobre desarrollo de procesos y sistemas de biorreactores para la producción de estas enzimas.
El conocimiento de las relaciones estructura-función de enzimas permite, a través de técnicas de mutagénesis o evolución dirigida, la generación de nuevos biocatalizadores adaptados a las condiciones utilizadas en los procesos industriales. Existen pocas estructuras elucidadas de cutinasas, por lo que la resolución de la estructura tridimensional de nuevas enzimas es una herramienta muy importante y necesaria para tal efecto.
Agradecimientos
PAPIIT-DGAPA-UNAM: Proyecto IN2148092 por los recursos financieros otorgados para la realización de investigación en esta área.
Referencias
1. Carvalho, C.M.L., Aires-Barros, M.R. & Cabral, J.M.S. Cutinase: from molecular level to bioprocess development. Biotechnol. Bioeng. 66(1), 17-34 (1999). [ Links ]
2. Kim, Y.h., Ahn, J.Y., Moon, S.H. & Lee, J. Biodegradation and detoxification of organophosphate insecticide, malathion by Fusarium oxysporum f. sp. pisi cutinase. Chemosphere 60, 1349-1355 (2005). [ Links ]
3. Kim, H.Y., Lee, J., Ahn, J.Y., Gu, M.B. & Moon, S.H. Enhanced degradation of an endocrine-disrupting chemical, butyl benzyl phthalate, by Fusarium oxysporum f. sp. pisi cutinase. Appl. Environ. Microbiol. 68(9), 4684-4688 (2002). [ Links ]
4. Murphy, C.A., Cameron, J.A., Huang, S.J. & Vinopal, R.T. Fusarium polycaprolactone depolymerase is cutinase. Appl. Environ. Microbiol. 62(2), 456-460 (1996). [ Links ]
5. Badenes, S.M., Lemos, F. & Cabral, J.M.S. Transesterification of oil mixtures catalyzed by microencapsulated cutinase in reversed micelles. Biotechnol. Lett. 32, 399-403 (2010). [ Links ]
6. Martínez, C., de Geus, P., Lauwereys, M., Matthyssens, G. & Cambillau, C. Fusarium solani cutinase is a lipolytic enzyme with a catalytic serine accessible to solvent Nature 356, 615, 618 (1992). [ Links ]
7. Araujo, R. et al. Tailoring cutinase activity towards polyethylene terephthalate and polyamide 6, 6 fibers. J. Biotechnol. 128, 849-857 (2007). [ Links ]
8. Koschorreck, K., Liu, D., Kazenwadel, C., Schmid, R.D. & Hauer, B. Heterologous expression, characterization and site-directed mutagenesis of cutinase CUTAB1 from Alternaría brassicicola. Appl. Microbiol. Biotechnol. 87, 991-997 (2010). [ Links ]
9. Brissos, V., Eggert, T., Cabral, J.M. & Jaeger, K.E. Improving activity and stability of cutinase towards the anionic detergent AOT by complete saturation mutagenesis. Protein Eng. Des. Sel. 21, 387-393 (2008). [ Links ]
10. Taiz, L. & Zeiger, E. Plant physiology (Sinauer Associates, Inc, Sunderland, 2002). [ Links ]
11. Walton, T.J. & Kolattukudy, P.E. Determination of the structure of cutin monomers by a novel depolymerization procedure and combined gas chromatography and mass spectrometry. Biochemistry 11(10), 1885-1897 (1972). [ Links ]
12. Farah Diba, A.B. etal. Induction and expression of cutinase activity during saprophytic growth of the fungal plant pathogen, Glomerella cingulata. Asia Pacific J. Biol. Biotechnol. 13(2), 63-69 (2005). [ Links ]
13. Lin, T.S. & Kolattukudy, P.E. Induction of a Biopolyester hydrolase (cutinase) by low levels of cutin monomers in Fusarium solani f. sp. pisi. J. Bacteriol. 133(2), 942-951 (1978). [ Links ]
14. Purdy, R.E. & Kolattukudy, P.E. Hydrolysis of plant cuticle by plant pathogens. Purification, aminoacid composition, and molecular weight of two isozymes of cutinase and a nonspecific esterase from Fusarium solani f. pisi. Biochemistry 14(13), 2824-2831 (1975). [ Links ]
15. Sebastian, J., Chandra, A.K., Kolattukudy, P.E. Discovery of a cutinase-producing Pseudomonas sp. cohabiting with an apparently nitrogen-fixing Corynebacterium sp. in the phyllosphere. J. Bacteriol. 169(1), 131-136 (1987). [ Links ]
16. Seo, H.S. et al. Pseudozyma jejuensis sp. nov., a novel cutinolytic ustilaginomycetous yeast species that is able to degrade plastic waste. FEMS Yeast Res. 7(6), 1035-1045 (2007). [ Links ]
17. Shayk, M. & Kolattukudy, P.E. Production of a novel extracellular cutinase by the pollen and the chemical composition and ultrastructure of the stigma cuticle of nasturtium ( Tropaeolum majus). Plant Physiol. 60, 907-915 (1977). [ Links ]
18. Purdy, R.E. & Kolattukudy, P.E. Depolymerization of a hydroxy fatty acid biopolymer, cutin, by an extracellular enzyme from Fusarium solani f. pisi isolation and some properties of the enzyme. Arch. Biochem. Biophys. 159, 61-69 (1973). [ Links ]
19. Tanabe, K., Nishimura, S. & Kohmoto, K. Pathogenicity of cutinase and pectic enzymes-deficient mutants of Alternaria alternata japanese pear pathotype. Ann. Phytopathol. Soc. Jpn. 54, 552-555 (1988). [ Links ]
20. Dickman, M.B. & Patil, S.S. Cutinase deficient mutants of Colletotrichum gloesporioides are nonpathogenic to papaya fruit. Physiol. Mol. Plant Pathol. 28, 235-142 (1986). [ Links ]
21. Dickman, M.B., Podila, G.K. & Kolattukudy, P.E. Insertion of cutinase gene into a wound pathogen enables it to infect intact host. Nature 342, 446-448 (1989). [ Links ]
22. Köller, W., Parker, D.M. & Becker, C.M. Role of cutinase in the penetration of apple leaves by Venturia inaequalis. Phytopathology 81, 1375-1379 (1995). [ Links ]
23. Yao, C. & Köller, W. Diversity of cutinase from plant pathogenic fungi different cutinases are expressed during saprophytic and pathogenic stages of Alternaria brassicicola. Mol. Plant-Microbe Interact. 8, 122-130 (1995). [ Links ]
24. van Kan, J., van 't Klooster, J., Wagemakers, C., Dees, D. & van der Vlugt-Bergmans, C. Cutinase A of Botrytis cinerea is expressed, but not essential, during penetration of gerbera and potato. Mol. Plant-Microbe Interact. 10(1), 30-38 (1997). [ Links ]
25. Bonnen, A.M. & Hammerschmidt, R.N. Role of cutinolytic enzymes in infection of cucumber by Colletotrichum lagenarium. Physiol. Mol. Plant Pathol. 35(6), 475-481 (1989). [ Links ]
26. http://www.chem.qmul.ac.uk/iubmb/enzyme/ [ Links ]
27. Fojan, P., Jonson, P.H., Petersen, M.T.N. & Petersen, S.B. What distinguishes an esterase from a lipase: a novel structural approach. Biochimie 82, 1033-1041 (2000). [ Links ]
28. Peña-Montes, C., González, A., Castro-Ochoa, D. & Farrés, A. Purification and biochemical characterization of a broad substrate specificity thermostable alkaline protease from Aspergillus nidulans. Appl. Microbiol. Biotechnol. 78(4), 603-612 (2008). [ Links ]
29. Córdova, J., Ryan, J.D., Boonyaratanakornkit, B.B. & Clark, D.S. Esterase activity of bovine serum albumin up to 160 °C: a new benchmark for biocatalysis. Enzyme Microb. Technol. 42, 278-283 (2008). [ Links ]
30. Bornscheuer, W.T. Microbial carboxyl esterase: classification, properties and applications in biocatalysis. FEMS Microbiol. Rev. 26, 73-81 (2002). [ Links ]
31. Flipsen, J.A.C., van der Hijden, H.T.W.M. & Verheij, H.M. Action of cutinase at the triolein-water interface. Characterization of interfacial effects during lipid hydrolysis using the oil-drop densitometer as a tool to lipase kinetics. Chem. Phys. Lipids 84, 105-115 (1996). [ Links ]
32. Gonçalves, A.M. et al. Stability studies of a recombinant cutinase immobilized to dextran and derivatized silica supports. Enzyme Microb. Technol. 24(1-2), 60-66 (1999). [ Links ]
33. Carvalho, C.L.M., Serralheiro, M.L.M, Cabral, J.M.S. & Aires-Barros, M.R. Application of factorial design to the study of transesterification reactions using cutinase in AOT-reversed micelles. Enzyme Microb. Technol. 21(2), 117-123 (1997). [ Links ]
34. Sharma, R., Chisti, Y. & Banerjee, U.C. Production, purification, characterization, and applications of lipases. Biotechnol Adv. 19(8), 627-662 (2001). [ Links ]
35. Pio, T.F. & Macedo, G.A. Cutinases: properties and industrial applications. Adv. App. Microbiol. 66, 77-95 (2009). [ Links ]
36. Regado, M.A. Flavour development via lipolysis of milkfats: changes in free fatty acid pool. Int. J. Food Sci. Technol. 42, 961-968 (2007). [ Links ]
37. Langrand, G., Triantaphylides, C. & Barratti, J. Lipases catalized formation of flavour esters. Biotechnol. Lett. 10(8), 549-554 (1988). [ Links ]
38. Welsh, W.W., Murray, W.D. & Williams, R.E. Microbiological and enzymatic production of flavor and fragrance chemicals. Crit. Rev. Biotechnol. 9, 105-169 (1989). [ Links ]
39. Barros, D.P.C., Fonseca, L.P. & Cabral, J.M.S. Cutinase-catalized biosynthesis of short chain alkyl esters. J. Biotechnol. 131,109-110 (2007). [ Links ]
40. Stamatis, H., Sereti, V. & Kolisis, F.N. Studies on the enzymatic synthesis of lipophilic derivatives of natural antioxidants. J. Am. Oil Chem. Soc. 76(12), 1505-1510 (1999). [ Links ]
41. Figueroa-Espinoza, M.C. & Villeneuve, P. Phenolic acids enzymatic lipophilization. J. Agric. Food Chem. 53, 2779-2787 (2005). [ Links ]
42. Peña-Montes, C. et al. Differences in biocatalytic behavior between two variants of StcI esterase from Aspergillus nidulans and its potential use in biocatalysis. J. Mol. Catal. B:Enzym. 61(3-4), 225-234 (2009). [ Links ]
43. Stamatis, H., Sereti, V. & Kolisis, F.N. Enzymatic synthesis of hydrophilic and hydrophobic derivatives of natural phenolic acids in organic media. J. Mol. Catal. B: Enzym. 11, 323-328 (2001). [ Links ]
44. Kolattukudy, P. et al. Cutinase cleaning compositions. US patent 4,981,611 (1991). [ Links ]
45. Flipsen, J.A.C., Appel, A.C.M., van der Hijden, H.T.W.M. & Verrips, C.T. Mechanism of removal of immobilized triacylglycerol by lipolytic enzymes in a sequential laundry wash process. Enzyme Microb. Technol. 23 (3-4), 274-280 (1998). [ Links ]
46. Rettich, F. Residual toxicity of wall-sprayed organophosphates, carbamates, and pyrethroids to mosquito. J. Hyg. Epidemiol. Microbiol. Immunol. 24, 110-117 (1980). [ Links ]
47. Indeerjeet, K., Mathur, R.P., Tandon, S.N. & Prem, D. Identification of metabolites of malathion in plant, water and soil by GC-MS. Biomed. Chromatogr. 11, 352-355 (1997). [ Links ]
48. Senanayake, N. & Karalliedde, L. Neurotoxic effects of organophosphorus insecticides. N. Engl. J. Med. 316, 761-763 (1987). [ Links ]
49. El-Dib, M.A., El-Elaimy, I.A., Kotb, A. & Elowa, S.H. Activation of in vivo metabolism of malathion in male Tilapianilotica. Bull. Environ. Contam. Toxicol. 57, 667-674 (1996). [ Links ]
50. Kavlock, R. et al. NTP Center for the evaluation of risks to human reproduction: phthalates expert panel report on the reproductive and developmental toxicity of di-it n-hexyl phthalate. Reprod. Toxicol. 16, 709-719 (2002). [ Links ]
51. Kim, Y.H. et al. Enhanced degradation and toxicity reduction of dihexyl phthalate by Fusarium oxysporum f. sp. pisi cutinase. J. App. Microbiol. 102, 221-228 (2007). [ Links ]
52. Kim, H.Y., Lee, J. & Moon, S.H. Degradation of an endocrine disrupting chemical, DEHP (di-2-ethylhexyl)-pfthalate), by Fusarium oxysporum f. sp. pisi cutinase. Appl. Microbiol. Biotechnol. 63(1), 75-80 (2003). [ Links ]
53. Sung, H.H., Kao, W.Y. & Su, Y.J. Effects and toxicity of phthalate esters to hemocytes of giant freshwater prawn, Macrobrachium rosenbergii. Aquat. Toxicol. 64, 25-37 (2003). [ Links ]
54. Ahn, J.Y., Kim, Y.H., Min, J. & Lee, J. Accelerated degradation of dipentyl phthalate by Fusarium oxysporum f. sp. pisi cutinase and toxicity evaluation of its degradation products using bioluminescent bacteria. Curr. Microbiol. 52(5), 340-344 (2006). [ Links ]
55. Kim, H.Y., Min, J., Bae, K.D., Gu, M.B. & Lee, J. Biodegradation of dipropyl phthalate and toxicity of its degradation products: a comparison of Fusarium oxysporum f. sp. pisi cutinase and Candida cylindracea esterase. Arch. Microbiol. 184(1), 25-31 (2005). [ Links ]
56. Huang, S.J. Polymer waste management-biodegradation, incineration, and recycling. J. Macromol. Sci. Pure A 32, 593-597 (1995). [ Links ]
57. Maeda, H. et al. Purification and characterization of a biodegradable plastic-degrading enzyme from Aspergillus oryzae. Appl. Microbiol. Biotechnol. 67, 778-788 (2005). [ Links ]
58. Masaki, K., Kamini, N.R., Ikeda, H. & Iefuji, H. Cutinase-like enzyme from the yeast Cryptococcus sp. strain S-2 hydrolyzes polylactic acid and others biodegradable plastics. Appl. Environ. Microbiol. 71(11), 7548-7550 (2005). [ Links ]
59. Vasudevan, P.T. & Briggs, M. Biodiesel production-current state of the art and challenges. J. Ind. Microbiol. Biotechnol. 35,421-430 (2008). [ Links ]
60. Jeong, G-T. & Park, D-T. Lipase-catalyzed transesterification of rapeseed oil for biodiesel production with tert-butanol. Appl. Biochem. Biotechnol. 148,131-139 (2008). [ Links ]
61. Cambou, B. & Klibanov, A. Preparative production of optically active esters and alcohols using esterase-catalyzed stereospecific transesterification in organic media. J. Am. Chem. Soc. 106 (9), 2687-2692 (1984). [ Links ]
62. Ohnishi, K., Toida, J., Nakazawa, H. & Sekiguchi, J. Genome structure and nucleotide sequence of lipolytic enzyme gene of Aspergillus oryzae. FEMS Microbiol. Lett. 126, 145-150 (1995). [ Links ]
63. Rubio, M.B., Cardoza, R.E., Hermosa, R., Gutiérrez, S. & Monte, E. Cloning and characterization of the Thcutlgene encoding a cutinase of Trichoderma harzianum T34. Curr. Genet. 54, 301-312 (2008). [ Links ]
64. Macedo, G. & Fraga, L. Production of cutinase by Fusarium oxysporum in solid-state fermentation using agro-industrial residues. J. Biotechnol. 131(2), S212 (2007). [ Links ]
65. Fett, W.F., Wijey, C., Moreau, R.A. & Osman, S.F. Production of cutinase by Thermonospora fusca ATCC 27730. J. Appl. Microbiol. 86, 561-568 (1999). [ Links ]
66. Rispoli, F.J. & Shanh, V. Mixture design as a first step for optimization of fermentation medium for cutinase production from Colletotrichum lindemuthianum. J. Ind. Microbiol. Biotechnol. 34, 349-355 (2007). [ Links ]
67. McQueen, D.A.R. & Schottel, J.L. Purification and characterization ofa novel Extracellular esterase from pathogenic Streptomyces scabies that is inducible by zinc. J. Bacteriol. 169(5), 1967-1971 (1987). [ Links ]
68. Fett, W.F., Gerard, H.C., Moreau, R.A., Osman, S.F. & Jones, L.E. Screening of nonfilamentous bacteria for production of cutindegrading enzymes. App. Environ. Microbiol. 58(7), 2123-2130 (1992). [ Links ]
69. Du, G.C. Zhang, S.L., Hua, Z.Z., Zhu, Y. & Chen, J. Enhanced cutinase production with Termobifida fusca by two-stage pH control strategy. Biotechnol. J. 133, 942-951 (2007). [ Links ]
70. van der Vlugt-Bergmans, C.J.B., Wagemakers, C.A.M. & van Kan, J.A.L. Cloning and expression of the Cutinase A gene of Botrytis cinerea. Mol. Plant-Microbe Interact. 10(1), 21-29 (1997). [ Links ]
71. Fett, W.F., Wijey, C., Moreau , R.A. & Osman, S.F. Production of cutinolytic esterase by filamentous bacteria. Lett. Appl. Microbiol. 31, 25-29 (2000). [ Links ]
72. Fan, C-Y. & Köller, W. Diversity of cutinases from plant pathogenic fungi:diferential and sequential expression if cutinolytic esterase by Alternaria brassicicola. FEMS Microbiol. Lett. 158, 33-38 (1998). [ Links ]
73. Hawthorne, B.T., Rees-George, J. & Crowhurst, R.N. Induction of cutinolytic esterase activity during saprophytic growth of cucurbit pathogens, Fusariumsolanif. sp. cucurbitaeraces one and two (Nectria haematococca MPI and MPV, respectively). FEMS Microbiol. Lett. 194, 135-141 (2001). [ Links ]
74. Pio, T.F. & Macedo, G. A. Optimizing the production of cutinase by Fusarium oxysporum using response surface methodology. Enzyme Microb. Technol. 41, 613-619 (2007). [ Links ]
75. Rispoli, F. & Shanh, V. Optimization of the media ingredients for cutinase production from Colletotrichum lindemuthianum using mixture design experiments. Biotechnol. Prog. 24, 648-654 (2008). [ Links ]
76. Griswold, K.E., Mahmood, N.A., Iverson, B.L. & Georgiou, G. Effects of codon usage versus putative 5'-mRNA structure on the expression of Fusarium solani cutinase in the Escherichia coli cytoplasm. Protein Exp. Purif. 27, 134-142 (2003). [ Links ]
77. Calado, C.R., Almeida, C., Cabral, J.M. & Fonseca, L.P. Optimisation of culture conditions and characterization ofcutinase produced by recombinant Saccharomyces cerevisiae. Enzyme Microb. Technol. 31, 161-170 (2002). [ Links ]
78. http://www.pdb.org [ Links ]
79. Nyon, M.P. et al. Catalysis by Glomerella cingulata cutinase requires conformational cycling between the active and inactive states of its catalytic triad. J Mol Biol 385, 226, 235 (2009). [ Links ]
80. Kodama, et al. Crystal structure and enhanced activity of a cutinase-like enzyme from Cryptococcus sp. strain S-2. Proteins 77, 710-717 (2009). [ Links ]
81. Liu, et al. Structural and functional studies of Aspergillus oryzae cutinase: enhanced thermostability and hydrolytic activity of synthetic ester and polyester degradation. J. Am. Chem. Soc. 131(43), 15711-15716 (2009). [ Links ]
82. Longhi, S., Czjzek, M., Lamzin, V., Nicolas, A. & Cambillau, C. Atomic Resolution (1.0 Â) crystal structure of Fusarium solani cutinase: stereochemical analysis. J. Mol. Biol. 268, 779-799 (1997). [ Links ]
83. Ollis, D.L. et al. The α/β hydrolase fold. Protein Eng. 5(3), 197-211 (1992). [ Links ]
84. Nardini, M. & Dijkstra, B.W. α/β hydrolase fold enzymes: the family keeps growing. Curr. Opin. Struct. Biol. 9, 732,737 (1999). [ Links ]
85. Egmond, M.R. & de Vlieg, J. Fusarium solani cutinase. Biochimie 82, 1015-1021 (2000). [ Links ]
86. Longhi, S. & Cambillau, C. Structure-activity of cutinase, a small lipolytic enzyme. Biochim. Biophys. Acta 1441, 185-196 (1999). [ Links ]
87. Kazlauskas, R.J. Elucidating structure-mechanism relationships in lipases: prospects for predicting and engineering catalytic properties. BIBTECH 12, 464-472 (1994). [ Links ]
88. Jaeger, J.E., Dijkstra, B.W. & Reetz, M.T. Bacterial biocatalysts: molecular biology, three-dimensional structures, and biotechnological applications of lipases. Annu. Rev. Microbiol. 53, 315-351 (1999). [ Links ]
89. Kraut, J. Serine proteases: structure and mechanisms of catalysis. Annu. Rev. Biochem. 46, 331-358 (1977). [ Links ]
Información sobre los autores
Denise Castro-Ochoa
Denise Castro-Ochoa es egresada de la Licenciatura en Ingeniería Bioquímica del Instituto Tecnológico de los Mochis (2000). Es Maestra en Ciencias en Ingeniería Bioquímica por el Instituto Tecnológico de Veracruz (2003) y actualmente es candidata a Doctora en Ciencias Bioquímicas por la Facultad de Química de la UNAM donde obtendrá el grado a corto plazo. Ha publicado 4 artículos en re\ islas indizadas y tiene otros en preparación. Sus estudios están enfocados al área de biología molecular, así como regulación, producción y caracterización bioquímica de carboxilesterasas microbianas.
Carolina Peña-Montes
Carolina Peña-Montes es Química de Alimentos y Doctora en Ciencias Bioquímicas por la Facultad de Química de la UNAM. Trabajó en el Instituto Mexicano de la Propiedad Industrial como examinadora de Patentes de alimentos y biotecnología. Realizó una estancia de investigación de dos años con beca del DAAD en el Instituto de Bioquímica Técnica de la Universidad de Stuttgart. Alemania, bajo la dirección del Dr. Rolf Schmid. Actualmente es Técnico Académico Titular de Tiempo Completo de la Facultad de Química, Departamento de Alimentos y Biotecnología de la UNAM. Ha publicado 4 artículos en revistas indizadas y tiene otros en preparación. Su trabajo de investigación se desarrolla en las áreas de biología molecular, fisiología microbiana, enzimas y su aplicación en biocatálisis.
Amelia Farrés
Amelia Farrés es Bióloga egresada de la Facultad de Ciencias de la UNAM y estudió el Doctorado en Biotecnología en la UACPyP, CCH. UNAM. Actualmente es Profesor Titular de tiempo completo definitivo. Jefa del Departamento de Alimentos y Biotecnología. Facultad de Química. UNAM y es Investigador del Sistema Nacional de Investigadores nivel II. Ha impartido cursos de licenciatura y posgrado y dictado varias conferencias. Ha recibido distinciones como: Mención Honorífica) Medalla Gabino Barreda en Licenciatura y Doctorado. Premio Nacional en Ciencia y Tecnología de Alimentos 1998 y 2005. En la formación de recursos humanos, ha formado a 6 Doctores, 17 Maestros y 32 licenciados. Tiene 30 publicaciones indizadas, 17 capítulos en libros y memorias in extenso. 22 publicaciones nacionales y de difusión. 10 Normas mexicanas en el Sistema Producto Leche (autor) y 28 como revisor. Sus líneas de investigación son: aplicación de enzimas en alimentos, genética y fisiología microbiana, determinación de diversidad biológica por métodos moleculares y caracterización bioquímica de proteínas funcionales de interés en alimentos.














