Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agrociencia
versión On-line ISSN 2521-9766versión impresa ISSN 1405-3195
Agrociencia vol.49 no.6 Texcoco ago./sep. 2015
Ciencia animal
Comparación de la exactitud de valores genómicos de animales predichos a través del análisis con dos modelos alternativos
Comparison of the accuracy of genomic values of animals predicted through analysis with two alternative models
Baldomero Alarcón-Zúñiga, Fernanda Ramírez-Flores, Agustín Ruíz-Flores*, Rodolfo Ramírez-Valverde, Luis A. Saavedra-Jiménez, José L. Zepeda-Batista
Posgrado en Producción Animal. Departamento de Zootecnia. Universidad Autónoma Chapingo. 56230. Chapingo, Estado de México. * Autor responsable (arf@correo.chapingo.mx)
Recibido: marzo, 2015.
Aprobado: junio, 2015.
Resumen
El valor genómico es la mejor predicción del valor genotípico de un animal, cuya exactitud varía en función de varios factores. Los objetivos de este estudio fueron, mediante simulación, comparar la exactitud de los valores genómicos de animales predichos a través del análisis con dos modelos alternativos, y obtener la correlación genética entre estos y los valores genéticos verdaderos simulados. Una población con tamaño efectivo de 800 individuos fue simulada y se usaron 100 generaciones para generar desequilibrio de ligamiento. Después se simuló otra población con 14 generaciones, un panel de 53 010 polimorfismos de nucleótido simple (SNP's) ubicados aleatoriamente en 30 cromosomas y 540 loci de características cuantitativas. También se simularon los genotipos y fenotipos de 6400 animales usando una heredabilidad=0.40 y considerando solamente los efectos aditivos. Cuatro conjuntos de valores moleculares fueron entrenados con los marcadores SNP's de las generaciones 7 a 10, y los correspondientes valores genéticos ajustados. Cuando la población de entrenamiento (PEn) tuvo tamaño mayor, los estimadores de exactitud (R2) fueron mayores. Similarmente, cuando la población de evaluación (PEv) estuvo más cerca a la PEn, los estimadores de R2 fueron mayores y se fueron reduciendo cuando la distancia entre PEn y PEv se incrementaba. El estimador de la varianza del error de predicción (PEV) fue menor (0.15±0.01) cuando PEn y PEv estuvieron más cerca, independientemente del tamaño de PEn, generación 10 para PEn y generación 11 para PEv. Por el contrario, el PEV mayor (0.29±0.02) se obtuvo cuando la PEn incluyó los animales de la generación siete y se evaluó la generación 14; es decir, cuando la distancia entre PEn y PEv era mayor.
Palabras clave: Valor genómico predicho, exactitud del valor genómico, selección genómica.
Abstract
The genomic value is the best prediction of the genotypic value of an animal, whose accuracy varies in function of several factors. The objectives of this study were to compare, through simulation, the accuracy of the genomic values of animals predicted through analysis with two alternative models, and to obtain the genetic correlation between these and the true genetic values simulated. A population with an effective size of 800 individuals was simulated and 100 generations were used to generate linkage disequilibrium. Then, another population was simulated with 14 generations, a panel of 53 010 single nucleotide polymorphisms (SNPs), placed randomly in 30 chromosomes and 540 quantitative traits loci. The genotypes and phenotypes of 6400 animals were also simulated using a heritability=0.40, and considering only the additive effects. Four sets of molecular values were trained using the SNP markers of generations 7 to 10, and the corresponding adjusted genetic values. When the training population (TP) was larger in size, the accuracy estimators (R2) were higher. Similarly, when the evaluation population (EP) was closer to the TP, the R2 estimators were higher and they became smaller when the distance between TP and EP increased. The estimate of the prediction error variance (PEV) was lower (0.15±0.01) when TP and EP were closer, regardless of the size of the TP, generation 10 for TP and generation 11 for EP. On the contrary, the highest PEV (0.29± 0.02) was obtained when the TP included animals from generation seven and generation 14 was evaluated; that is, when the distance between TP and EP was greater.
Keywords: Predicted genomic value, accuracy of the genomic value, genomic selection.
INTRODUCCIÓN
Un objetivo común en programas de mejoramiento genético es usar la información del ADN para aumentar el progreso genético al reducir el intervalo entre generaciones y aumentar la exactitud de las predicciones. Ahora se puede determinar el genotipo de animales para decenas de miles de polimorfismos de nucleótido simple (SNP's, siglas en inglés; Goddard y Hayes, 2007). Estos SNP's pueden usarse para obtener los valores genómicos de los animales (Meuwissen et al., 2001).
La evaluación genómica de animales puede efectuarse en una (Misztal et al., 2009; Aguilar et al., 2011), o en dos etapas (Hayes et al., 2009; Van Raden et al., 2009). Cuando se realiza en dos etapas, en la primera se usa una población de entrenamiento (PEn) para obtener las ecuaciones de predicción, las cuales se usarán en una segunda etapa en la población de evaluación (PEv) para predecir los valores genómicos (VGn); en la PEv no se requiere tener los registros fenotípicos de los individuos, pero sí sus genotipos. Los animales utilizados en la PEv no deberán considerarse en la PEn (Garrick, 2011). En la fase de entrenamiento se predice la influencia de pequeñas regiones genómicas por regresión de la información observada del genotipo de los marcadores para una población dada. El genotipo deberá estar constituido por un gran número de marcadores (Garrick et al., 2009). Los animales en ambas poblaciones deberán ser de la misma raza (Kachman et al., 2013), y pueden evaluarse al obtener una muestra de su tejido para realizar el genotipado, lo cual permite reducir el intervalo entre generaciones y, como consecuencia, aumentar el progreso genético.
La exactitud de los valores genómicos predichos se define como la correlación entre el VGn y el valor genético verdadero (VG) (Garrick y Saatchi, 2011; Meuwissen et al., 2001). Un incremento en la exactitud de los VGn de los animales candidatos a selección incrementará el progreso genético, especialmente para características con baja heredabilidad (h2).
Habier et al. (2007) indicaron que tanto el desequilibrio de ligamiento entre los marcadores y los loci de características cuantitativas (QTL, por sus siglas en inglés) y las relaciones de parentesco entre individuos de las PEn y PEv, influyen en la exactitud de los VGn. Relaciones cercanas entre los individuos de PEn y PEv tienen mejores predicciones que las de individuos con relaciones más distantes (Habier et al., 2010; Saatchi et al., 2010; Saatchi et al., 2011). Otros factores que también influyen en la exactitud de los VGn son la distancia entre marcadores, tamaño de la PEn, número de animales o sementales en la PEn, heredabilidad de la característica, estructura genética, y número de marcadores usados para la predicción genómica (Goddard y Hayes, 1992; Meuwissen et al., 2001; Hayes et al., 2009).
Con información ajustada, de acuerdo con Garrick et al. (2009), se puede obtener una exactitud hasta 2.76 veces mayor que con el registro de un solo individuo. Ostersen et al. (2011) obtuvieron exactitudes 18 a 39 % mayores de los VGn, dependiendo de la característica evaluada, cuando utilizaron VG ajustados por los valores genéticos de los padres del animal (DEVG, siglas en inglés) en lugar de VG, como variable de respuesta. Garrick et al. (2009) mostraron que los DEVG con el promedio parental removido producen VGn predichos más exactos, por dos razones: 1) los DEVG como variable respuesta resultan en menos conteos dobles que con los VG porque los DEVG excluyen información de los ancestros, y si tanto la progenie como sus padres son genotipados, el grado de conteo doble decrece cuando se usan DEVG como la variable de respuesta; 2) al usar los VG como las variables de respuesta el grado de doble conteo en los VGn decrece, en particular cuando las confiabilidades de los valores genéticos son bajas.
La predicción del VGn para un animal utilizando un enfoque Bayesiano, como el Bayes C (Meuwissen et al., 2001; Habier et al., 2011), es la media a posteriori del valor genético del animal. La exactitud puede obtenerse elevando al cuadrado la correlación entre el VG verdadero y los VGn.
Los objetivos de este estudio fueron comparar la exactitud de los valores genómicos predichos con dos modelos de análisis utilizando datos simulados, y estimar la correlación genética entre los valores genéticos verdaderos obtenidos mediante simulación con los valores genómicos predichos con el programa Gen-Sel.
MATERIALES Y MÉTODOS
El desequilibrio de ligamiento se obtuvo simulando una población histórica con tamaño efectivo de 800 individuos y 100 generaciones. Después se simuló otra población con 14 generaciones discretas, 20 machos y 200 hembras seleccionadas aleatoriamente para producir la siguiente generación, un panel de 53 010 SNP's ubicados aleatoriamente en 30 cromosomas, cada uno separado 100 centiMorgans de longitud, y 540 QTL con efectos provenientes de una distribución gama (Weller et al., 2005). Los SNP's y los QTL se consideraron bialélicos con frecuencias iniciales aleatorias. También se simularon los genotipos y fenotipos de 6400 bovinos usando una heredabilidad de 0.4, y sólo se consideraron los efectos aditivos. Otros parámetros de la población simulada se indican en el Cuadro 1. Para generar la información genotípica y fenotípica se utilizó el programa QMSim (Sargolzaei y Schenkel, 2009).

Cuatro conjuntos de valores moleculares fueron entrenados usando los marcadores SNP's de las generaciones 10 (n=1,000), 9 y 10 (n=1,400), 8 a 10 (n=1800), 7 a 10 (n=2200), y los correspondientes DEVG. Posteriormente se predijeron los valores genéticos con un modelo mixto que incluyó los efectos aleatorio del animal y fijos de sexo del animal y generación mediante el programa ASReml (Gilmour et al., 2009) y los DEVGs se obtuvieron siguiendo la metodología de Garrick et al. (2009). La ponderación para el i-ésimo animal se obtuvo con la ecuación wi= (1 —h2)/[(c+(1 —r2)/r2)h2], donde c es la falta de ajuste de la ecuación de predicción o la parte genética no explicada por los marcadores, se asumieron valores de c=0.1 y h2=;0.4; y r2 fue la confiabilidad de los DEVG para el i-ésimo animal. El modelo usado para obtener los efectos de los marcadores en la población de entrenamiento fue:
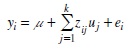
donde yi es el DEVG para el i-ésimo animal, μ es la media poblacional, k es el número de loci de los marcadores en el panel, zij es el genotipo del i-ésimo animal (número de copias del alelo 1) en el j-ésimo locus del marcador, ui es el efecto aleatorio del j-ésimo SNP muestreado de ui~N(0, σ2u) con probabilidad 1 —π o 0 con probabilidad p, y ei es el residual con varianza σ2c/wi, dependiendo de la exactitud del DEVG para el i-ésimo individuo. Los valores a priori para σ2a y σ2c fueron 0.4 y 0.04, respectivamente.
Las ecuaciones de predicción genómica para obtener los valores genómicos se derivaron de un análisis ponderado para los animales de las generaciones 11 a 14, para lo cual se usó la función BayesCp del programa Gen-Sel (Fernando y Garrick, 2009) a través de la plataforma bioinformática para implementar selección genómica (Bioinformatics to Implement Genomic Selection, BIGS) de la Universidad Estatal de Iowa (EE.UU.)1. La opción BayesCp se seleccionó por sus ventajas sobre otras opciones de Bayes, al ajustar un modelo similar a BayesB (Meuwissen et al., 2001; Habier etal., 2011), además de estimar la proporción de loci no informativos en el modelo (π), y el supuesto de una varianza común para todos los loci con efectos no cero, en lugar de la varianza individual para cada locus (Habier et al., 2011). La cadena usada tenía una longitud de 41,000, se utilizaron las últimas 40 000 muestras para obtener los estimadores a posteriori de los efectos medios y varianzas, se quemaron las primeras 1000 iteraciones, y p se fijó en 0.95.
El valor genómico para el i-ésimo animal se obtuvo como la suma sobre todos los marcadores de las medias a posteriori de los efectos predichos de los SNP's, multiplicados por el número de copias del alelo 1, usando la ecuación:
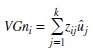
donde VGni es el valor genético molecular, valor genómico, para el i-ésimo animal, zij es el genotipo del j-ésimo marcador en el i-ésimo animal, y ûj es la media a posteriori del efecto del SNP para el j-ésimo marcador.
Los modelos utilizados para obtener los valores genómicos en Gen-Sel fueron: 1) uno sin considerar los efectos de sexo y número de generación, y 2) otro que sí los incluyó. Los criterios de comparación utilizados fueron la exactitud, estimada a través del coeficiente de determinación múltiple (R2) y la varianza del error de predicción de los valores genómicos (PEV). La correlación genética entre los valores genéticos verdaderos simulados obtenidos con el programa QMSim (Sargolzaei y Schenkel, 2009) y los valores genómicos predichos con el programa Gen-Sel (Fernando y Garrick 2009) se estimó con el programa ASReml (Gilmour et al., 2009).
RESULTADOS Y DISCUSIÓN
Los estimadores de R2 fueron mayores cuando PEn fue mayor (incluyó más generaciones) que PEv. De manera similar, los estimadores de R2 fueron mayores cuando disminuía la distancia entre PEv y Pen, y se fueron reduciendo cuando aumentaba la distancia entre ambas poblaciones (Cuadro 2).

Los resultados de este estudio son similares a los encontrados por Habier et al. (2010), Saatchi et al. (2010) y Saatchi et al. (2011), quienes observaron que con relaciones cercanas entre los individuos de PEn y PEv se obtienen mejores predicciones, en comparación con los obtenidos cuando estas poblaciones son más distantes.
En congruencia con los resultados para R2, el estimador de PEV fue menor, 0.15±0.01 cuando la PEn y la PEv estuvieron más cercanas, independientemente del tamaño de la PEn, generación 10 para PEn y generación 11 para PEv. Al contrario, el mayor PEV (0.29± 0.02) se obtuvo cuando la PEn incluyó los individuos de la generación siete y se evaluó la generación 14; es decir cuando era mayor la distancia entre PEn y PEv. En el Cuadro 3 se muestran los estimadores de R2 y PEV para las diferentes poblaciones de entrenamiento y de evaluación, obtenidos con el modelo que no incluyó los efectos de sexo del animal y generación. Cuando la PEn fue de mayor tamaño (más generaciones incluidas), los estimadores de R2 fueron mayores. De manera similar, cuando PEv estuvo más cerca de PEn, los estimadores de R2 fueron mayores.

El estimador de PEV fue menor (0.15± 0.01), cuando era menor la distancia entre PEn y PEv, independientemente del tamaño de la PEn, generación 10 para PEn y generación 11 para PEv. Por el contrario, el mayor PEV (0.29±0.02) se obtuvo cuando la PEn incluyó los animales de la generación siete y se evaluó la generación 14. Estos resultados son similares a los encontrados por Habier et al. (2010), Saatchi et al. (2010) y Saatchi et al. (2011), quienes muestran que relaciones cercanas entre los individuos en PEn y PEv tienen mejores predicciones que con grupos más distantes. Los resultados también concuerdan con los de Meuwissen et al. (2001) y Hayes et al. (2009), quienes señalan que el tamaño de la PEn también influye en la exactitud de la predicción de los VG.
Saatchi et al. (2010) obtuvieron aumentos en R2 de 39.7 y 24.2 % cuando incluyeron las generaciones 51 a 58 y 55 a 58 en la PEn, y la PEv estuvo compuesta por las hembras de la generación 59 y h2 fue 0.5. Cuando estos autores usaron h2=0.1, los incrementos en R2 fueron 88.9 % (de 0.112 a 0.2116) y 41.1 % (de 0.112 a 0.2116). Este efecto también fue reportado por Meuwissen et al. (2001) y Saatchi et al. (2012). Estos resultados pueden ser una expresión de varios factores, como lazos genéticos débiles entre PEn y PEv, un incremento en la tasa de recombinación, un cambio en la estructura genotípica y una reducción en el desequilibrio de ligamiento entre marcadores y QTLs debido a un aumento en el número de generaciones entre ambas poblaciones (Saatchi et al., 2010).
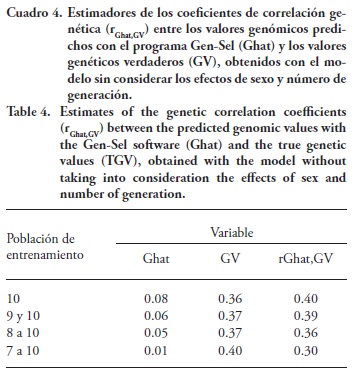
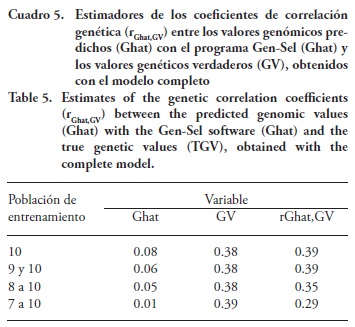
Los estimadores de los coeficientes de correlación genética (rGhat,GV) entre los valores genómicos predichos (Ghat) con el programa Gen-Sel y los valores genéticos verdaderos (VG), obtenidos con el modelo sin considerar los efectos de sexo y número de generación y con el modelo completo, son relativamente bajos, aunque es notoria la manera en que su valor disminuye a medida que PEn incluye generaciones más alejadas, de 0.40 a 0.30, y de 0.39 a 0.29 para las PEn que incluyen solamente la generación 10 y para la que incluye de la generación siete a la 10, con los modelos sin considerar los efectos de sexo y número de generación y el completo (Cuadro 4 y 5). Estas correlaciones son menores a las encontradas por Meuwissen et al. (2001), quienes reportan valores de 0.318±0.018 a 0.848±0.012, obtenidos con varios métodos de predicción de valores genómicos (mínimos cuadrados, BLUP, BayesA y BayesB).
Los valores en este estudio son similares a los obtenidos en ganado Hereford por Saatchi et al. (2013), con estimadores de correlaciones genéticas entre valores genómicos predichos y DEVG para 10 características de 0.20±0.06 a 0.52±0.04 con el método BayesB, y de 0.18±0.06 a 0.45±0.04 con el método BayesC. Además, estos autores reportan correlaciones genéticas de 0.13±0.06 a 0.32±0.05 entre los valores genéticos deregresados y los valores genéticos obtenidos con las metodologías tradicionales de evaluación mediante BLUP. En un estudio de simulación, Spangler et al. (2007) obtuvieron para marmoleo de la carne estimadores de correlaciones de 0.256 a 0.859, entre los valores genéticos verdaderos y los predichos, a partir de conjuntos de datos con diferente proporción de información disponible.
Según Kizilkaya et al. (2010), para una característica con heredabilidad de 0.5 y usando aproximadamente 1000 animales en PEn, se obtienen correlaciones mayores de 0.8 para características controladas por aproximadamente 500 loci, suponiendo que los loci causales son conocidos y genotipados.
La tendencia a disminuir de los estimadores de correlación genética entre los valores genómicos predichos y los valores genéticos verdaderos es clara, en la medida que aumenta la distancia entre PEn y PEV, lo cual sucedió en ambos modelos (Cuadros 4 y 5).
CONCLUSIONES
Las exactitudes obtenidas con los dos modelos de predicción de valores genómicos comparados fueron similares.
Con los dos modelos de predicción comparados, las exactitudes de los valores genómicos predichos fueron mayores en la medida que las poblaciones de entrenamiento incluyeron más generaciones; esto es, que fueron de mayor tamaño. El mismo resultado se obtuvo cuando la distancia entre las poblaciones de entrenamiento y evaluación era menor.
Las varianzas del error de predicción fueron mayores cuando aumentaba la distancia entre las poblaciones de entrenamiento y evaluación.
Los estimadores de las correlaciones genéticas entre los valores genómicos predichos con los modelos comparados y los valores genéticos verdaderos fueron bajos. La disminución de estos estimadores fue notoria a medida que aumentaba la distancia entre las poblaciones de entrenamiento y de evaluación.
LITERATURA CITADA
Aguilar, I., I. Misztal, A. Legarra, and S. Tsuruta. 2011. Efficient computation of the genomic relationship matrix and other matrices used in single-step evaluation. J. Anim. Breed. Genet. 128:422-428. [ Links ]
Fernando, R., and D. J. Garrick. 2009. GenSel — user manual for a portfolio of genomic selection related analyses, créate 9.1. http://bigs.ansci.iastate.edu/bigsgui/ReleaseNotes/Version-9_1-ReleaseNotes.1.pdf. (Consulta: Enero, 2013). [ Links ]
Garrick, D. J. 2011. The nature, scope and impact of genomic prediction in beef cattle in the United States. Genet. Sel. Evol. 43:17-27. [ Links ]
Garrick, D. J., J. F. Taylor, and R. Fernando. 2009. Deregressing estimated breeding values and weighting information for genomic regression analyses. Genet. Sel. Evol. 41:55-62. [ Links ]
Garrick, D. J., and M. Saatchi. 2011. Opportunities and challenges for genomic selection of beef cattle. Rev. Bras. Zootec. 40:310-316. [ Links ]
Gilmour, A. R., B. J. Gogel, B. R., Cullis, and R. Thompson. 2009.ASReml User Guide Release 3.0 VSN International Ltd, Hemel Hempstead, HP1 1ES, UK. http://www.vsni.co.uk. (Consulta: Enero, 2013). [ Links ]
Goddard, M. E., and B. J. Hayes. 1992. Mapping genes for complex traits in domestic animals and their use in breeding programmes. Nat. Rev. Genet. 10:381-391. [ Links ]
Goddard, M. E., and B. J. Hayes. 2007. Genomic selection. J. Anim. Breed. Genet. 124:323-330. [ Links ]
Habier, D., R. L. Fernando, and J. C. M. Dekkers. 2007. The impact of genetic relationship information on genome-assisted breeding values. Genetics 177:2389-2397. [ Links ]
Habier, D., J. Tetens, F-R Seefried, P. Lichtner, and G. Thaller. 2010.The impact of genetic relationship information on genomic breeding values in German Holstein cattle. Genet. Sel. Evol. 52:5-16. [ Links ]
Habier, D., R. L. Fernando, K. Kizilkaya, and D. J. Garrick. 2011.Extension of the Bayesian alphabet for genomic selection. BMC Bioinformatics 12:186-197. [ Links ]
Hayes, B. J., P. J. Bowman, A. J. Chamberlain, and M. E. Goddard. 2009. Invited review: Genomic selection in dairy cattle: Progress and challenges. J. Dairy Sci 92:433-443. [ Links ]
Kachman, S. D., M. L. Spangler, G. L. Bennett, K. J. Hanford, L. A. Kuehn, W. M. Snelling, R. M. Thallman, M. Saatchi, D. J. Garrick, R. D. Schnabel, J. F. Taylor, and E. J. Pollak. 2013. Comparison of molecular breeding values based on within- and across-breed training in beef cattle. Genet. Sel. Evol. 45:30-38. [ Links ]
Kizilkaya, K., R. L. Fernando, and D. J. Garrick. 2010. Genomic prediction of simulated multibreed and purebred performance using observed fifty thousand single nucleotide polymorphism genotypes. J. Anim. Sci. 88:544-551. [ Links ]
Meuwissen, T. H. E., B. J. Hayes, and M. E. Goddard. 2001. Prediction of total genetic value using genome-wide dense marker maps. Genetics. 157:1819-1829. [ Links ]
Misztal, I., A. Legarra, and I. Aguilar. 2009. Computing procedures for genetic evaluation including phenotypic, full pedigree, and genomic information. J. Dairy Sci. 92:4648-4655. [ Links ]
Ostersen, T., O. F. Christensen, M. Henryon, B. Nielsen, G. Su, and P. Madsen. 2011. Deregressed EBV as the response variable yield more reliable genomic predictions than traditional EBV in pure-bred pigs. Genet. Sel. Evol. 43:38-43. [ Links ]
Saatchi, M., S. R. Miraei-Ashtiani, A. Nejati Javaremi, M. Moradi-Shahrebabak, and H. Mehrabani-Yeghaneh. 2010. The impact of information quantity and strength of relationship between training set and validation set on accuracy of genomic estimated breeding values. Afr. J. Biotechnol. 9:438-442. [ Links ]
Saatchi, M., M. C. McClure, S. D. McKay, M. M. Rolf, J. Kim, J. E. Decjer, T. M. Taxis, R. H. Chapple, H. R. Ramey, S. L. Northcut, S. Bauck, B. Woodward, J. C. M., Dekkers, R. L. Fernando, R. D. Schnabel, D. J. Garrick, and J. F. Taylor. 2011. Accuracies of genomic breeding values in American Angus beef cattle using K-means clustering for cross-validation. Genet. Sel. Evol. 43:40-55. [ Links ]
Saatchi, M., R. D. Schnabel, M. M. Rolf, J. F. Taylor, and D. J Garrick. 2012. Accuracy of direct genomic breeding values for nationally evaluated traits in US Limousin and Simmental beef cattle. Genet. Sel. Evol. 44:38-47. [ Links ]
Saatchi, M., J. Ward, and D. J. Garrick. 2013. Accuracies of direct genomic breeding values in Hereford beef cattle using national or international training populations. J. Anim. Sci. 91:1538-1551. [ Links ]
Sargolzaei, M., and F. S. Schenkel. 2009. QMSim: a large-scale genome simulator for livestock. Bioinformatics. 25:680-681. [ Links ]
Spangler, M. L., J. K. Bertrand, and R. Rekaya. 2007. Combining genetic test information and correlated phenotypic records for breeding value estimation. J. Anim. Sci. 85:641-649. [ Links ]
Van Raden, P. M., C. P. Van Tassell, G. R. Wiggans, T. S. Sonstegard, R. D. Schnabel, J. F. Taylor, and F. S. Schenkel. 2009. Invited Review: reliability of genomic predictions for North American Holstein bulls. J. Dairy Sci. 92:16-24. [ Links ]
Weller, J. I., M. Shlezinger, and M. Ron. 2005. Correcting for bias in estimation of quantitative trait loci effects. Genet. Sel. Evol. 37:501-522. [ Links ]














