INTRODUCCIÓN
México es reconocido como centro de origen del género Agave, el uso de este género va desde la obtención de fibras, alimento, forraje y principalmente la producción de bebidas alcohólicas (Ortega et al., 2000). Esta última actividad representa un importante porcentaje del producto interno bruto de México y de ingresos para la población dedicada a las actividades relacionadas con el cultivo, desde la plantación hasta la cosecha, comercialización e industrialización de Agave.
El tequila y el mezcal poseen una denominación de origen y son reguladas bajo las Normas Oficiales Mexicanas NOM-006-SCFI-2012 (SE, 2012) y NOM-070-SCFI-2016 (SE, 2017), respectivamente, donde se establece que el tequila se elabora sólo a partir de piñas de Agave tequilana Weber cv azul, a diferencia del mezcal, el cual puede ser elaborado de 14 especies, siempre y cuando éstas no sean utilizadas para la elaboración de otras bebidas con denominación de origen del mismo estado, y en cumplimiento a la normativa en vigor. El Agave angustifolia es la especie más utilizada en la producción de mezcal, y con mayor avance en su paquete tecnológico, a diferencia de las otras especies que son en su mayoría silvestres, cultivadas o semicultivadas (Carrillo, 2007).
La propagación de los principales Agaves utilizados en la producción del tequila y el mezcal es mediante la reproducción asexual a través del rizoma (hijuelos), que en teoría permiten obtener clones fenotípica y genéticamente idénticos a la planta madre (Torres-Morán et al., 2006), tal es el caso de A. tequilana y A. angustifolia. La edad óptima de un agave para reproducirse es entre tres y cinco años, y puede dar origen anualmente a entre uno y dos hijuelos. Dependiendo de la especie, algunas se propagan solamente por semillas, tal es el caso de A. karwinskii y A. potatorum que se propagan de manera sexual, vía que resulta más compleja ya que los agricultores cortan la inflorescencia al inicio de su desarrollo para promover la acumulación de azúcares en el tallo, evitando su reproducción y disminuyendo las poblaciones silvestres (Aguirre-Dugua y Eguiarte, 2013; Pérez-Negrón y Casas, 2007).
Ante el surgimiento de nuevos productores de tequila y mezcal, se ha generado la necesidad de contar cada vez con mayor superficie establecida del cultivo de Agave, actualmente existen más de 144 mil hectáreas de A. tequilana Weber cv azul utilizadas para la producción de tequila (CRT, 2017) y más de 20 mil hectáreas registradas ante el consejo regulador del mezcal (CRM, 2019), generando con esto la necesidad de contar con material propagativo sano, vigoroso y correctamente identificado de acuerdo con la especie de interés.
La identificación de especies de Agave fundamentada en medidas anatómicas no es una labor sencilla (Verduzco-Martínez et al., 2009) y en las piñas cosechadas que llegan a las destilerías resulta imposible, ya que pierden todos sus descriptores varietales. Los marcadores moleculares han resultado ser una herramienta útil en la caracterización de especies de Agave (Gil-Vega et al., 2006; Rodriguez-Garay et al., 2009) es una alternativa rápida y de fácil acceso en especies de difícil identificación (Davis, 1996).
Los trabajos de variabilidad genética en Agave han sido apoyados por el uso de marcadores moleculares, los cuales se han consolidado como una herramienta confiable, que ha permitido un desarrollo exponencial en el conocimiento de las bases genéticas de diversas respuestas y procesos presentes en los organismos (Astorga, 2008). Entre las técnicas más utilizadas se encuentran los AFLP (Amplified Fragment Length Polymorphism), RFLP (Restriction Fragment Length Polymorphism), RAPD (Random Amplified Polimorphic DNA), ISSR (Inter Simple Secuence Repeats); estos últimos utilizados en trabajos de relaciones genéticas de Agave cocui y otras especies del mismo género, así como en la determinación de la variabilidad genética entre individuos de las especies A. cocui Trel. A. angustifolia Haw. y A. tequilana (Dávila, 2007). Los marcadores ISSR se utilizan por su rápida implementación y por su bajo costo al establecer diversidad genética (Awasthi et al, 2004; Jin et al., 2003; Zizumbo-Villarreal et al., 2005). La interpretación de este tipo de marcadores se realiza con la lectura de geles, donde las amplificaciones repetitivas idénticas con secuencias de anclaje diferentes que proporcionan patrones y son analizados como marcadores dominantes, lo que significa que son bialélicos con bandas presentes o ausentes, las cuales se registran como los dos alelos de un locus (Wolfe y Liston, 1998).
En las plantas de Agave, pertenecientes a la familia Asparagaceae, se han utilizado diferentes tipos de marcadores moleculares como los RAPDs (Gil et al., 2001; Rodriguez-Garay et al 2009) en la caracterización de A. tequilana, los AFLPs en estudios de variabilidad de A. fourcroydes (Infante et al., 2003), en diferentes variedades de A. tequilana (Gil et al 2001), así como también los espaciadores inter-secuencias (ITS) de los genes ribosomales nucleares usados por Bogler y Simpson (1996) para estudios filogenéticos en la subfamilia. Las ventajas que ofrece la técnica de ISSR sobre otros marcadores son principalmente sobre la alta variación que detecta, así como en su reproducibilidad debida a las altas temperaturas de alineación utilizadas en la PCR; asimismo, no son necesarias altas concentraciones de ADN y no es necesario conocer la secuencia del genoma del organismo en estudio (Reddy et al., 2002).
El objetivo del presente estudio fue comparar la caracterización morfológica de hijuelos y el empleo de marcadores moleculares ISSR en la diferenciación de A. angustifolia, A. karwinskii, A. potatorum y A. tequilana, especies utilizadas en la producción de tequila y mezcal, con la finalidad de contar con una herramienta precisa, accesible y rápida en la identificación de especies de Agave.
MATERIALES Y MÉTODOS
Material genético
Se colectaron cinco plantas por cada especie de Agave bajo estudio; las plantas de Agave karwinskii (Cuixe) y A. potatorum (Tobalá) tuvieron su origen en la zona de Miahuatlán, Oaxaca, provenientes de la germinación de semillas en vivero por un periodo de 9 a 12 meses, el A. angustifolia (Espadín) provino de hijuelos de un calibre de piña de 5 cm del mismo municipio, mientras que los hijuelos de A. tequilana “0” (5 cm de diámetro de piña) fueron colectados en el municipio de Tequila, Jalisco, dentro del área de la denominación de origen (DOT) (Figura 1).
Evaluación morfológica
Se evaluaron nueve caracteres cuantitativos de cada planta: longitud de planta en cm, diámetro de piña en cm, en la hoja se midió el ancho y la longitud en cm; en la espina apical la longitud en mm, distancia entre espina apical y lateral en mm, largo y ancho de espina lateral en mm y el número de espinas en 10 cm (Figura 2). Se realizó análisis multivariado de componentes principales de los datos obtenidos mediante el programa estadístico Infostat versión V2.0. 2020.
Caracterización molecular
La extracción de ADN se realizó con el método de CTAB (Bromuro de cetil trimetil amonio) (Wagner et al., 1987). Se realizó el corte de dos trozos de 1 cm2 de la hoja más joven, lavada en una solución de cloro comercial 6 %. Se mezclaron 50 mg de tejido vegetal previamente pulverizado con nitrógeno líquido con 700 µL de CTAB 4 % (NaCl 1.4 M, β-mercaptoetanol 0.2 %, EDTA 20 mM, Tris-HCl 100 mM). La mezcla se agitó suavemente por inversión y se incubó por 60 min agitando cada 15 min; luego, la muestra se enfrió a temperatura ambiente y se agregaron 700 µL de cloroformo:alcohol isoamílico (24:1), se agitó vigorosamente y se centrifugó a 14,000 rpm por 10 min; cuidadosamente se extrajo la fase acuosa y se depositó en un nuevo tubo. Se adicionaron 500 µL de isopropanol frío al tubo y se mezcló suavemente, incubándose a -20 °C por 30 min; se centrifugó a 14,000 rpm por 5 min y se retiró el sobrenadante; se realizaron dos lavados con 500 µL de etanol 70 % centrifugando entre cada uno de ellos a 14,000 rpm por 5 min; finalmente, el pellet obtenido se dejó secar y se suspendió con una solución de Tris-HCl 10 mM, pH 8.0. Se verificó la pureza mediante la relación de absorbancia A260/A280, así como la integridad de éste a través de la visualización en geles de agarosa 1.2 %.
Se evaluó un total de seis iniciadores ISSR (Cuadro 1) bajo las siguientes condiciones: para una reacción de 12.5 µL se agregó MgCl2 1.5 mM, dNTPs 0.2 mM, 0.5 U de Taq ADN polimerasa y 10 pmol de iniciador ISSR. La amplificación se realizó en un termociclador empleando 40 ciclos de 94 °C por 1 min, 38 °C por 1 min y 72 °C por 2 min, y una extensión final de 72 °C por 7 min. Los productos de amplificación se visualizaron en geles de agarosa 1.2 % teñidos con gelred (Biotium) a una concentración de 1X en una solución buffer TBE 1X (108 g Tris base, 55 g ácido bórico, 40 mL EDTA 0.5 M, pH 8.0).
Cuadro 1 Iniciadores ISSR utilizados en identificación de especies de Agave.
| Iniciador | Secuencia | Productos Amplificados | Tm °C | Número de loci polimórficos |
| (GA)8YC | GAGAGAGAGAGAGAGAYC | 23 | 48.0 | 17 |
| G(AG)7AC | GAG AGA GAG AGA GAG AC | 16 | 49.0 | 10 |
| (GA)8C | GAG AGA GAG AGA GAG AC | 17 | 47.5 | 14 |
| (GA)8YG | GAG AGA GAG AGA GAG AYG | 19 | 49.5 | 14 |
| C(AG)7AG | CAC ACA CAC ACA CAC AG | 15 | 48.0 | 12 |
| DDB(CCA)5 | DDB CCA CCA CCA CCACCA | 14 | 49.5 | 6 |
Tm: temperatura de alineamiento, R: G o A, Y: C o T.
Los productos de PCR con los diferentes iniciadores fueron leídos directamente de los geles tomando en cuenta bandas de entre 200 y 1200 pb medido con marcador de 1 kb (Invitrogen) y los patrones de bandeo ISSR fueron convertidos a código binario de 1 y 0 para presencia y ausencia, respectivamente. Los datos binarios fueron utilizados para generar una matriz de similitud usando el índice de Jaccard y se construyó un dendrograma a partir de la matriz empleando el método UPGMA (Método de grupos de pares no ponderados con media aritmética), así como un análisis de componentes principales para las variables morfológicas empleando el programa INFOSTAT V2.0.2020.
RESULTADOS Y DISCUSIÓN
Evaluación morfológica
Los parámetros morfológicos evaluados presentaron una diferenciación a través de análisis de componentes principales (CP) de los hijuelos de A. angustifolia, A. karwinskii, A. potatorum y A. tequilana, coincidiendo con la delimitación de especies desarrollada por Gentry (1982). Se observa que las plantas son separadas en cuatro grupos, de acuerdo con la variación morfológica, mostrando que es posible a través del análisis de CP diferenciar a A. tequilana de A. angustifolia, tal como lo reportaron Rodriguez-Garay et al., (2009) en estudios previos sobre caracterización morfológica de ambas especies.
El análisis de componentes principales (CP) explicó un 70 % de variación acumulada entre los componentes CP1 y CP2, valor cercano a lo reportado por Castañeda-Nava et al. (2019), en el cual el amplio porcentaje de variación permitió distinguir entre dos subespecies y dos variedades de A. salmiana. Las variables de mayor relevancia para CP1 fueron la longitud de la planta, número de hojas y longitud de espina apical, lo que coincide con el trabajo de Mora-López et al. (2011) (Figura 3). En el CP2 la variable de mayor influencia fue el número de espinas en 10 cm y el ancho de hoja, caracteres referidos como adecuados en la identificación de las especies A. angustifolia, A. tequilana y A. salmianae (Alfaro et al., 2007; Rodríguez-Garay et al., 2009), cabe señalar que los trabajos previos mencionados, han sido desarrollados en plantas adultas y no en hijuelos, indicando con esto la factibilidad de utilizar parámetros varietales para caracterización de especies desde edades tempranas o a lo largo de su ciclo de vida.
Caracterización molecular
La pureza y calidad del ADN obtenido fue fundamental para tener una buena resolución de los marcadores ISSR. Con el método utilizado en este estudio se obtuvo una buena integridad del ADN, sin la presencia de contaminantes como carbohidratos o ARN. La pureza se obtuvo en el intervalo de 1.8 a 2.0 en la relación de longitud de onda 260/280 nm, presentando una calidad aceptable.
Con base en el patrón de amplificación obtenido en Agave tequilana, se seleccionaron los seis iniciadores que presentan mayor eficiencia en la determinación de variabilidad genética entre especies. Es importante resaltar que a pesar de que existe similitud en las secuencias de los oligonucleótidos utilizados, la temperatura de alineamiento fue fundamental en la expresión del polimorfismo de las diferentes especies (Cuadro 1).
Los seis oligonucleótidos evaluados detectaron en total de 104 loci, el número de loci polimórficos fue de 73, lo que representa un porcentaje de 70 %. Los productos de amplificación mostraron un tamaño de entre 200 y 2000 pb, lo que coincide con estudios previos realizados en A. tequilana (Ángeles-Espino et al., 2020) (Figura 4). Koskinen et al. (2004) y Nybom (2004) mencionaron que es posible observar diferencias entre individuos a nivel de nucleótidos, de acuerdo con el número de bandas amplificadas, entre mayor sea el número, mayor será la confiabilidad de los resultados.
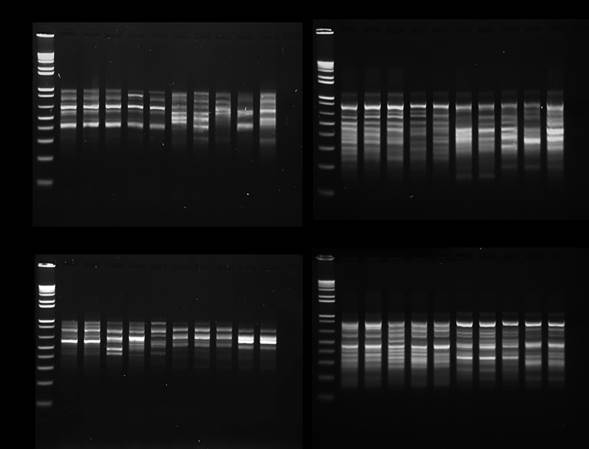
Figura 4 Amplificación ISSR en cuatro especies de Agave empleando el iniciador (GA)8YC (superior e inferior izquierdo) y el iniciador (GA)8C (superior e inferior derecho). T 1-5: Agave tequilana, K1-5: A. karwinskii, E1-5: A. angustifolia, P1-5: A. potatorum. M: marcador de peso molecular de 1 kb.
Se sugiere utilizar varios marcadores de manera simultánea en estudios de variabilidad genética con el fin de establecer ventajas entre otros métodos como AFLPs o ratificar los resultados obtenidos con diferentes tipos de marcadores; por lo general, de tipo dominante como los RAPD e ISSR (Awasthi et al., 2004; Bruneau et al., 2005; Ducarme y Wesselingh, 2005); sin embargo, en el presente estudio el uso de sólo seis iniciadores fue suficiente para la diferenciación de las especies de Agave estudiadas.
Los datos obtenidos de los geles de agarosa fueron utilizados para la construcción de la matriz binaria, para obtener una matriz con el coeficiente de similitud de Jaccard, y un agrupamiento mediante el método UPGMA (Figura 5).

Figura 5 Dendrograma construido mediante el método UPGMA a partir del análisis ISSR para las muestras de Agave. (E) A. angustifolia, (K) A. karwinskii, (P) A. potatorum, (T) A. tequilana.
De manera general, es posible diferenciar los grupos de cada una de las cuatro especies incluidas en el estudio: A. tequilana (T1-5), A. karwinskii (K1-5), A. angustifolia (E1-5) y A. potatorum (P1-5) y el agrupamiento de cada una de ellas; ésto coincide con estudios de Dos Santos et al (2015), donde los marcadores ISSR fueron efectivos en la separación de genotipos estudiados de A. sisalana, contribuyendo al conocimiento biológico de la especie en apoyo a su conservación.
Además de generarse una separación de las especies, se observa la formación de dos grupos G1 y G2, el primero compuesto por A. angustifolia, A. karwinskii y A. tequilana (Figura 5). Estudios realizados para determinar la variación de especies de Agave a través de caracteres morfológicos sugieren a A. angustifolia como antecesor de A. tequilana (Colunga-GarcíaMarín et al., 1996), por lo cual se esperaría identificar esta relación en el dedrograma de la Figura 5; sin embargo, se observa un subgrupo (SB1) donde se aprecia que la relación genética es mayor entre las especies de A. karwinskii y A. tequilana, y no entre A. angustifolia y A. tequilana; este comportamiento coincide con lo reportado por Dávila et al. (2007), donde mostraron que existe una relación genética más estrecha entre A. angustifolia y A. cocui que con A. tequilana, explicada como un proceso de derivación genética por el uso extensivo del cultivo.
El G2 separó las plantas A. potatorum, y las señaló como las menos relacionadas con las otras especies evaluadas; este comportamiento podría estar explicado por el hecho de tratarse de una especie propagada por semilla. La mayor similitud se presentó en las plantas de A. tequilana, comportamiento que ha sido mencionado con anterioridad en el estudio de Gil et al. (2001).
Los oligonucleótidos (CA)n usados en este estudio han sido empleados anteriormente en las amplificaciones de ISSR. Este tipo de marcadores, al ser multilocus dominantes, requiere un numero bajo de iniciadores para proporcionar información relevante en el estudio del polimorfismo dentro y entre poblaciones de plantas (Xia et al., 2007).
CONCLUSIONES
La caracterización morfológica de hijuelos utilizados en la producción de tequila y mezcal, así como la caracterización mediante marcadores ISSR son de utilidad en la diferenciación de las especies de Agave analizadas en este estudio cuando se utilizan hijuelos como mterial biológico, presentando un alto polimorfismo y fácil interpretación de los resultados.











 text new page (beta)
text new page (beta)





