Introducción
La técnica de la reacción en cadena de la polimerasa en su versión cuantitativa, acoplada a la transcriptasa reversa (RT-qPCR) es utilizada frecuentemente en la investigación científica. Su relativa facilidad para la cuantificación de ácidos nucleicos ha proporcionado información valiosa en la elaboración de regímenes de tratamiento y en la definición de perfiles de expresión genética en diferentes contextos (VanGuilder et al., 2008). Los niveles de expresión genética se pueden reportar de forma absoluta o relativa. En el primer caso se calcula el número de copias presentes usando como referencia una curva estándar (Chen et al., 2005). En el segundo caso se toma como referencia uno o más genes calibradores o controles internos, contra los cuales se compara la expresión de otros genes (Dorak, 2006; Schmittgen y Livak, 2008).
En la mayoría de los estudios de expresión genética es necesario indicar cuántas veces se incrementó la expresión relativa del gen de interés con respecto al control, por lo que generalmente se utilizan métodos de cuantificación relativa, siendo el Método del Ct Comparativo, conocido también como el método ΔΔCt (Livak y Schmittgen, 2001), uno de los más utilizados. El Ct es el valor de fluorescencia umbral o "threshold", por arriba del cual se considera que una reacción de PCR tiempo real es positiva. El componente fundamental de toda reacción de PCR cuantitativa son los oligonucleótidos específicos para el gen que se desea analizar. Cuando se aplica el método del Ct comparativo para evaluar los niveles de expresión genética existen dos características que deben evaluarse en los oligonucleótidos que se usarán en la reacción de qPCR: eficiencia de la amplificación y rango dinámico.
La eficiencia se refiere al nivel en que el proceso de amplificación ocurre de manera exponencial durante la reacción de PCR. Factores como las interacciones homo y heterodiméricas, la diferencia en la temperatura de fusión (Tm) entre los oligonucleótidos, la falta de complementariedad en alguna base o el pegado inespecífico, así como la formación de estructuras secundarias en el producto de la amplificación, reducen la eficiencia de la amplificación. La temperatura de fusión (Tm) de una secuencia de DNA se refiere a la temperatura a la cual 50 % de las copias de esa secuencia presentes en una reacción se encuentra en forma monocatenaria y 50 % en forma bicatenaria, que interactúan con su secuencia complementaria. El rango dinámico se refiere al intervalo de concentraciones del DNA o cDNA que se emplean como molde y que han sido previamente purificados, en el que se garantiza que se obtienen valores de Ct exactos. Cuando se usa el método del Ct comparativo, los rangos dinámicos del gen a evaluar y del gen control deben ser comparables. La eficiencia debe ser preferentemente ≥ 90 % y el rango dinámico debe cubrir al menos tres órdenes de magnitud.
El tomate rojo o jitomate (Solanum lycopersicum) es la hortaliza de mayor importancia en México. Su cultivo, cosecha y comercialización genera 72 mil empleos directos y aproximadamente 10.7 millones de empleos indirectos. De acuerdo con el Sistema de Información Agrícola y Pesquera (SIAP, 2009) la superficie sembrada en 2009 ascendió a 101,328 ha. La producción nacional en 2013 se ubicó en 2,727,691 toneladas. En los últimos años, por ventas al extranjero de esta hortaliza ingresaron al país en promedio más de mil millones de dólares anuales (SIAP, 2009).
Recientemente, el fitopatógeno Ralstonia solanacearum ha causado importantes pérdidas en cultivos de tomate en diversas regiones de México (Perea et al., 2011; Hernández-Romano et al., 2012). Esta bacteria ataca alrededor de 200 especies vegetales de 33 familias diferentes, entre las que se incluye el tomate (Genin, 2010). Es un bacilo que no forma esporas, Gram negativo, aeróbico, con un flagelo polar y presenta tal diversidad genética que se clasifica como un complejo de especies que se dividen en biovares y filotipos con base en sus perfiles metabólicos y la secuencia de genes específicos, respectivamente (Denny, 2006; Fegan y Prior, 2005).
La comprensión de las interacciones moleculares que se presentan entre R. solanacearum y S. lycopersicum puede proporcionar información que contribuya a la elaboración de nuevas estrategias de control. Los perfiles de expresión genética permiten conocer el comportamiento de diferentes genes de la planta y de la bacteria durante el proceso infectivo. Acceder a esta información podría ayudar a identificar genes cruciales para la defensa del tomate frente a la invasión por R. solanacearum, o bien identificar factores de virulencia de la bacteria necesarios para la inducción de enfermedad en la planta.
Diversos trabajos han evaluado los perfiles de expresión genética del tomate frente a la infección por los fitopatógenos bacterianos Clavibacter michiganensis subsp. michiganensis (Balaji et al., 2008; Chalupowicz et al., 2010) y R. solanacearum (Milling et al., 2011; Addy et al., 2012; Jacobs et al., 2012). La mayoría de estos trabajos valoran la calidad de los oligonucleótidos utilizados. En algunos casos, con los reactivos utilizados y bajo las condiciones de nuestro laboratorio se obtienen resultados distintos a los previamente reportados, lo que resalta la necesidad de evaluar cada par de oligonucleótidos bajo las condiciones de cada laboratorio, de manera que los resultados obtenidos reflejen de manera confiable la respuesta biológica que se está presentando durante la interacción de estos dos organismos.
Con el objetivo de identificar pares de oligonucleótidos que bajo nuestras condiciones experimentales muestren un desempeño adecuado, en este trabajo se evalúan 40 pares de oligonucleótidos asociados a 16 genes del tomate relacionados con resistencia y de 10 genes de R. solanacearum relacionados con virulencia, algunos de ellos previamente reportados por otros grupos de investigación (Chalupowicz et al., 2010; Milling et al., 2011; Addy et al., 2012).
Materiales y métodos
Cepa bacteriana y condiciones de crecimiento
La cepa bacteriana de Ralstonia solanacearum utilizada se aisló en el municipio de Coatlán del Río, Morelos (Hernández-Romano et al., 2012), se cultivó durante 24 h en 3 mL de medio Caldo nutritivo-Extracto de levadura (NBY) (Atlas, 2005) a 28 °C ± 2 °C y en agitación.
Material vegetal
Las plantas de tomate utilizadas fueron de la marca Harris Moran, variedad El Cid, presentaban entre 10 y 15 hojas al momento de ser procesadas.
Extracción y cuantificación de DNA genómico bacteriano y de tejido vegetal
Para la extracción de DNA genómico (DNAg) de R. solanacearum se utilizó el kit de purificación Wizard Genomic DNA de Promega (Cat. #A1120), de acuerdo con las instrucciones del fabricante. Se utilizó 1.0 mL de un cultivo de 24 h de crecimiento. El DNAg obtenido se resuspendió en un volumen final de 100 μL, la solución resultante se cuantificó y se verificó su pureza por duplicado con el espectrofotómetro EPOCH (BiotekTM), esta solución se utilizó para realizar los experimentos de evaluación de todos los oligonucleótidos. La extracción de DNAg a partir del tejido vegetal se realizó con el kit DNeasy Plant Mini Kit de Qiagen (Cat. #69104), en aproximadamente 100 mg de hojas, las cuales se maceraron y luego se siguió el protocolo señalado por el fabricante. Con esta solución se realizaron los ensayos de estandarización de oligonucleótidos correspondientes a los genes del tomate cuyos oligonucleótidos se encontraban en un mismo exón.
Obtención de cDNA del tomate
Para la obtención de cDNA de tomate se utilizó el kit GoScript Reverse Transcription System de Promega (Cat. #A500). Se determinó la concentración del RNA por espectrofotometría con el equipo EPOCH (BiotekTM) y se evaluó su pureza en integridad por electroforesis en gel de agarosa. Para efectuar la retrotranscripción, se mezclaron 5 μg de RNA total, oligo (dT) (0.5 μg/reacción) y agua libre de nucleasas para completar un volumen de 5 μL. La mezcla se calentó a 70 °C por 5 min y se mantuvo en hielo mientras se preparó una segunda mezcla que consistió en 4 μL de buffer de reacción (5X), 2 μL de MgCl2 (2.5 mM), 1 μL de la mezcla de dNTPs (0.5mM), 1 μL de inhibidor de RNasas (20 unidades), 1 μL de la enzima transcriptasa reversa y 6 μL de agua libre de nucleasas. Ambas mezclas se combinaron y el producto se llevó a 25 °C por 5 min y después a 42 °C por 1 h. Se inactivó la enzima colocando el tubo de reacción a 70 °C por 15 min, y se almacenó después a -80 °C hasta el momento de ser utilizada. Con esta muestra se evaluó el desempeño de los oligonucleótidos del tomate que se encontraban en diferentes exones.
Análisis in silico de los oligonucleótidos
Los genes seleccionados para este trabajo y sus correspondientes oligonucleótidos se muestran en los Cuadros 1 y 2. Se utilizaron las bases de datos de Ensembl (Cunningham et al., 2015), de GeneBank (Benson et al., 2013) y secuencias genómicas parciales de la cepa morelense de R. solanacearum (Com. pers.1) para obtener las secuencias de los genes; sus claves pueden consultarse en el Cuadro S1 (material suplementario, http://www.revistafitotecniamexicana.org/documentos/39-2/suplementario.pdf). Para el diseño de nuevos oligonucleótidos se utilizó el "software" Vector NTI (Lu y Moriyama, 2004), considerando un tamaño de amplicón ≤ 250 pb y una temperatura de fusión (Tm) de 55 a 60 °C. Con este mismo "software" se evaluó la presencia de interacciones homodiméricas, heterodiméricas y estructuras de asa y tallo. Para cada par de oligonucleótidos se evaluó su especificidad con la herramienta BLAST (Altschul et al., 1990).

Cuadro 1: Oligonucleótidos de los genes de S. lycopersicum. En negritas se resaltan los que presentaron un desempeño adecuado. Se indica el nombre del oligonucleótido (Nombre oligonuc.), su secuencia, el tamaño esperado del amplicón, la temperatura de alineamiento (Tm), la eficiencia y la pendiente de la evaluación del rango dinámico.
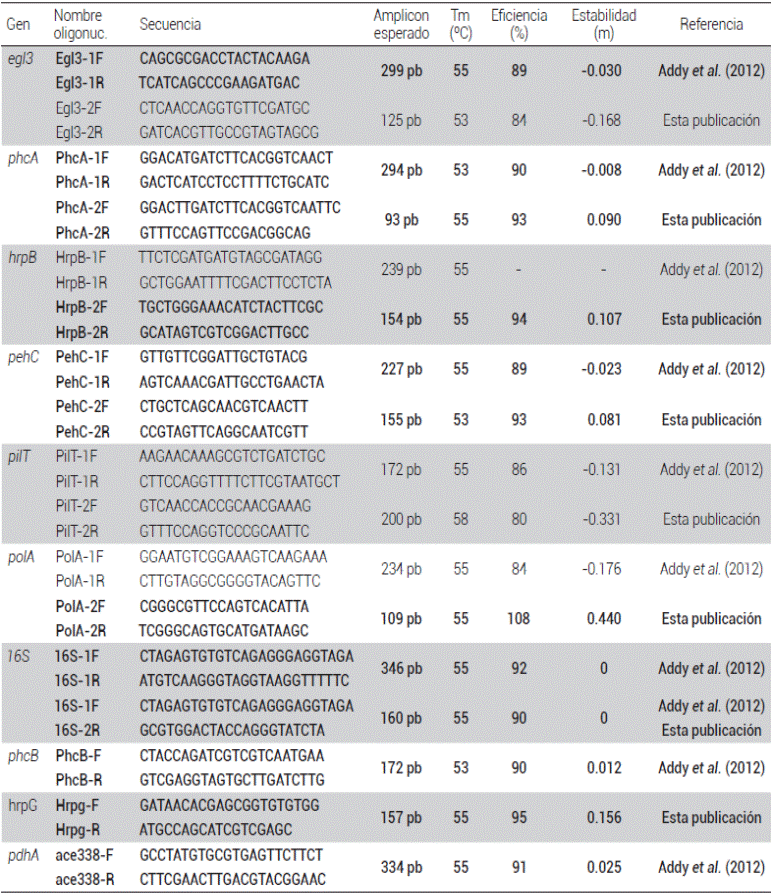
Cuadro 2: Oligonucleótidos de genes de R. solanacearum. En negritas se resaltan los que presentaron un desempeño adecuado. Se indica el nombre del oligonucleótido (Nombre oligonuc.), su secuencia, el tamaño esperado del amplicon, la temperatura de alineamiento (Tm), la eficiencia y la pendiente de la evaluación del rango dinámico.
Evaluación de la eficiencia de la amplificación y del rango dinámico
Para evaluar la eficiencia de cada par de oligonucleótidos se utilizaron los reactivos del kit Maxima SYBR Green qPCR Master Mix (2x) (Thermo Scientific, Cat. K0252). Cada muestra se amplificó por triplicado en un equipo Rotor-Gene Q 5plex (Qiagen) mediante el empleo de volúmenes de reacción de 10 μL en tubos de 0.1 mL (Qiagen, Cat. 981103). Las concentraciones de DNA en las muestras a utilizar como fuente de DNA molde de R. solanacearum y de tomate fueron de 459 ng μL-1 y 641 ng μL-1, respectivamente. A partir de estas soluciones se realizaron cuatro diluciones decimales, las cuales fueron utilizadas en los experimentos posteriores, de las cuales se emplearon 1.0 μL por reacción.
En el caso de las reacciones que emplearon cDNA, de la muestra de cDNA obtenida que contenía el equivalente a 5 μg de RNA total en 20 μL, se prepararon cuatro diluciones decimales, las cuales fueron utilizadas en los experimentos posteriores. Para el cálculo de la eficiencia se tomaron los valores de Ct generados por el equipo para cada dilución y se graficó el Ct promedio vs. Log10 (concentración de DNA molde), y luego se aplicó la fórmula
Para la evaluación del rango dinámico se aplicó la fórmula ΔCt = (Ct del gen control)-(Ct del gen a evaluar) y se graficó ΔCt vs. Log10 (concentración de DNA molde) (Livak y Schimmittgen, 2001). Si los valores de Ct del gen control y del gen evaluado son comparables en el intervalo de concentraciones utilizado, la pendiente de esta curva debe ser cercana a cero, con valores que oscilan preferentemente entre -0.1 y 0.1; además de ser un indicador del rango dinámico, esta pendiente muestra también el desempeño del operario, puesto que variaciones en el pipeteo pueden provocar cambios en la pendiente. Como genes control se seleccionaron el ribosomal 16S y el de la gliceraldehído-3-fosfato-deshidrogenasa (GAPDH), para R. solanacearum y tomate, respectivamente.
La adecuada preparación de las muestras se evaluó observando la diferencia de Ct entre las distintas diluciones; puesto que se manejaron diluciones decimales, la diferencia teórica está dada por log2(10) y deberá ser cercana a 3.32. La mezcla de reacción de 10 μL contenía: 5 μL de Master mix (2x), 2 μL de una solución 2.5 μM de cada oligonucleótido (directo y reverso), 1 μL de cDNA o DNAg y 2 μL de agua libre de nucleasas. Cada concentración de DNA molde se evaluó por triplicado. El equipo se programó con corridas de tres pasos, y se evaluaron tres temperaturas de alineamiento (53, 55 y 58 °C), que incluyó un análisis de curva de fusión al final del proceso de amplificación.
Cada corrida incluyó una fase de desnaturalización a 95 °C por 10 s, una fase de alineamiento (53, 55 o 58 °C) por 15 s (una temperatura por cada programa) y una fase de elongación a 72 °C por 20 s, en la que se registró la fluorescencia de la muestra. La etapa de obtención de la curva de fusión inició a 60 °C y terminó en 95 °C, con incrementos de 1 °C en cada paso, con 5 s de espera entre cada uno. La adquisición fue en el canal verde. La optimización de la ganancia del equipo se colocó con valores de 5 y 8.
Resultados y discusión
Oligonucleótidos previamente reportados no tienen homología con secuencias del tomate o de R. solanacearum
De 46 oligonucleótidos evaluados, 41 presentaron la máxima identidad con el tomate; sin embargo, cuatro oligonucleótidos (CHIT-1F, CHIT-1R, Pr1A-F, Pr1A-R) presentaron la mayor identidad con secuencias de Nicotiana tabacum (Cuadros S2 y S4). El oligonucleótido 1b1-F reportado por Chalupowicz et al. (2010), presentó la mayor identidad con Peromyscus maniculatus (ratón ciervo), mientras que con el gen pr1b1 del tomate, para el cual estaba teóricamente diseñado, presentó seis nucleótidos no idénticos; esto posiblemente se asocie con la actualización y mejora de las secuencias contenidas en las bases de datos. Para R. solanacearum, 33 de 34 oligonucleótidos fueron específicos. El oligonucleótido HrpB-1R, reportado por Addy et al. (2012), no presentó homología con R. solanacearum ni con alguna otra bacteria filogenéticamente cercana, sino que la mayor identidad fue con una secuencia de la mosca doméstica (Musca domestica L.) (Cuadro S3).
Oligonucleótidos reportados en otros trabajos, muestran un desempeño no aceptable bajo las condiciones de nuestro ensayo
De los nueve pares de oligonucleótidos del tomate previamente reportados por Chalupowics et al. (2010), cuatro mostraron un mal desempeño bajo las condiciones del presente estudio. Los pares CHIT-1F/CHIT-1R y GPDH-2F/ GPDH-2R no generaron ningún producto de PCR, respuesta que puede explicarse por la presencia de nucleótidos no idénticos (Cuadro S4), que pueden afectar de manera importante el desempeño de los oligonucleótidos (Stadhouders et al., 2010). Adicionalmente, variaciones en el empalme alternativo podrían afectar la detección. El par CHIT2-1F/CHIT2-1R tampoco generó un producto de amplificación, aun cuando presentó 100 % de identidad con la correspondiente secuencia del tomate y ambos se localizaban en el mismo exón.
Los oligonucleótidos 1b1-F/1b1-R, aún con la presencia de seis nucleótidos no idénticos en 1b1-F, todas sesgadas hacia el extremo 3', incluyendo una interacción no complementaria de A con C en la posición 1 del extremo 3', generaron una buena señal en la reacción de qPCR (valores de Ct entre 6.77 y 18.31) y una curva de fusión con un solo pico bien definido. Sin embargo, la reproducibilidad entre las réplicas no fue adecuada, la eficiencia fue de 81.3 % y la pendiente de la evaluación del rango dinámico fue de 0.308, lo que indica que los valores de Ct reportados no son exactos ni comparables con los que se obtienen con el gen control en el intervalo de concentraciones del DNA molde evaluado.
De los nueve pares de oligonucleótidos de genes de tomate reportados por Milling et al. (2011), sólo dos no mostraron un desempeño aceptable en el presente estudio. El par LoxA-1F/LoxA-1R no generó ningún producto de amplificación, a pesar de que no presentaba nucleótidos no idénticos con su secuencia blanco. El empalme alternativo, la expresión genética diferencial y condiciones no óptimas de amplificación, podrían explicar este resultado. El par Pr1A-F/Pr1A-R, homólogo a secuencias de N. tabacum, presentó una eficiencia de 53 % y una pendiente de 1.809 en la evaluación del rango dinámico.
De los nueve pares de oligonucleótidos para genes de R. solanacearum reportados por Addy et al. (2012), tres presentaron un desempeño inadecuado en nuestro laboratorio. El par HrpB-1F/HrpB-1R falló en generar un producto de amplificación, posiblemente debido a que el oligonucleótido reverso presentó homología con la mosca doméstica. Los pares PilT-1F/PilT-1R y PolA-1F/PolA-1R presentaron valores de eficiencia apenas por debajo de lo esperado (86 y 84 %, respectivamente) y sus valores de pendiente también estuvieron ligeramente alejados del intervalo aceptable (-0.131 y -0.176).
Los pares PilT-1F/PilT-1R y PolA-1F/PolA-1R no presentaron más de un nucleótido no idéntico con las secuencias blanco en la cepa morelense de R. solanacearum y tampoco se presentaron interacciones homo o heterodiméricas de importancia; sin embargo, al analizar los amplicones con el programa MFold (Zuker, 2003), el cual detecta posibles estructuras secundarias, se presentan estructuras de asa y tallo que involucran importantes interacciones (Figura S1, material suplementario), estas estructuras podrían afectar la eficiencia de la reacción. Llama la atención el caso del par PehC-1F/ PehC-1R, ya que ambos oligonucleótidos presentan múltiples nucleótidos no idénticos con la secuencia de la cepa morelense de R. solanacearum, varias de ellas localizadas en el extremo 3' (Cuadro S5, material suplementario); sin embargo, este par de oligonucleótidos mostró un desempeño adecuado (Cuadro 2). Para el gen pehC se tienen dos pares de oligonucleótidos, por lo que será interesante evaluar si ambos pares generan la misma información al valuar los niveles de expresión genética.
Estos resultados descartan a seis pares de oligonucleótidos del tomate y a tres de R. solanacearum previamente mencionados como candidatos para medir expresión genética bajo las condiciones del presente estudio, aun cuando se han utilizado con esta finalidad en otros trabajos.
El diseño cuidadoso in silico de los oligonucleótidos ayuda, pero no garantiza, el adecuado desempeño de los oligonucleótidos
Tanto para el tomate como para R. solanacearum se diseñaron oligonucleótidos de novo para tratar de sustituir aquellos que presentaron un desempeño inadecuado, cuidando que existiera 100 % de identidad con la secuencia blanco, que no existieran interacciones homo y heterodiméricas de importancia y que el amplicón fuera menor a 200 pb. Para el tomate se diseñaron cinco pares de oligonucleótidos, ninguno de los cuales presentó un desempeño adecuado (Cuadro 1), pero no se pudieron identificar causas que expliquen este comportamiento, salvo posibles estructuras secundarias con un bajo número de interacciones G-C y A-T.
Para R. solanacearum se sintetizaron ocho pares de oligonucleótidos y un oligonucleótido reverso (16S-2R) para usarse en combinación con otro que ya se tenía. De estos nueve pares de oligonucleótidos que involucraban secuencias no reportadas, seis presentaron un desempeño adecuado (Cuadro 2). De los dos pares de oligonucleótidos evaluados para el gen egl3, el par Egl3-1F/Egl3-1R genera un amplicón de 299 pb, el cual podría considerarse de tamaño elevado para ser utilizado en una reacción de qPCR; sin embargo, tanto la eficiencia como la estabilidad presentaron mejores valores que los oligonucleótidos Egl32F/Egl3-2R, lo que indica que el tamaño del amplicón o la presencia de nucleótidos no idénticos no siempre correlacionan con el desempeño de los oligonucleótidos.
Los oligonucleótidos localizados en el megaplásmido de R. solanacerum presentan más nucleótidos no idénticos que aquellos localizados en el cromosoma bacteriano
Una característica de Ralstonia solanacearum es su enorme diversidad genética, razón por la cual se ha clasificado como un complejo de especies (Fegan y Prior, 2005). Los oligonucleótidos reportados por Addy et al. (2012) fueron diseñados para cepas del Filotipo I, mientras que la cepa que se utilizó en este trabajo, la cual se aisló en el estado de Morelos, México, pertenece al Filotipo II y se tiene secuenciada en más de 90 %. Se evaluó el nivel de conservación de la secuencia de los oligonucleótidos en cepas representativas de cada filotipo (GMI1000 y CFBP2957), para las cuales se ha reportado el genoma completo (Remenant et al., 2010), así como en la cepa de Morelos.
El Cuadro S6 indica el número de nucleótidos no idénticos ("mismatches") entre los oligonucleótidos y la secuencia genómica de las cepas GMI1000, CFBP2957 y la cepa morelense utilizada en este trabajo. Se sumó el número total de nucleótidos no idénticos identificados para cada gen y se dividió entre el número de oligonucleótidos evaluados para cada gen, y así se obtuvo el promedio de nucleótidos no idénticos por oligonucleótido. Se puede observar que los oligonucleótidos localizados en el megaplásmido presentan un mayor número de nucleótidos no idénticos comparados con los que se localizan en el cromosoma.
Esto señala que, para organismos con alta variación genética de una región geográfica a otra, es aún menos recomendable tomar oligonucleótidos reportados por otros grupos de investigación. De preferencia se debe contar con la secuencia de la cepa con que se trabajará, puesto que incluso al interior de cada filotipo existen diferencias, como ocurre con los oligonucleótidos del gen hrpG, los cuales presentan baja similitud con los de las cepas GMI1000 y CFBP2957 (Cuadro S6).
Los oligonucleótidos generaron un único producto de amplificación. De los 35/40 pares de oligonucleótidos que generaron una señal fluorescente, todos generaron un amplicón mayoritario. En la Figura 1A se muestra la curva de fusión correspondiente al amplicón generado con los oligonucleótidos PR1-F/PR1-R, que evidencia la presencia de un único máximo al obtener la derivada de fluorescencia respecto de la temperatura. Curvas similares se obtuvieron en 28 de 35 pares de oligonucleótidos; en los restantes siete pares de oligonucleótidos se observó un segundo pico de menor altura localizado entre los 60 y 67 °C, lo que posiblemente indica la presencia de dímeros de oligonucleótidos (Figura S2).
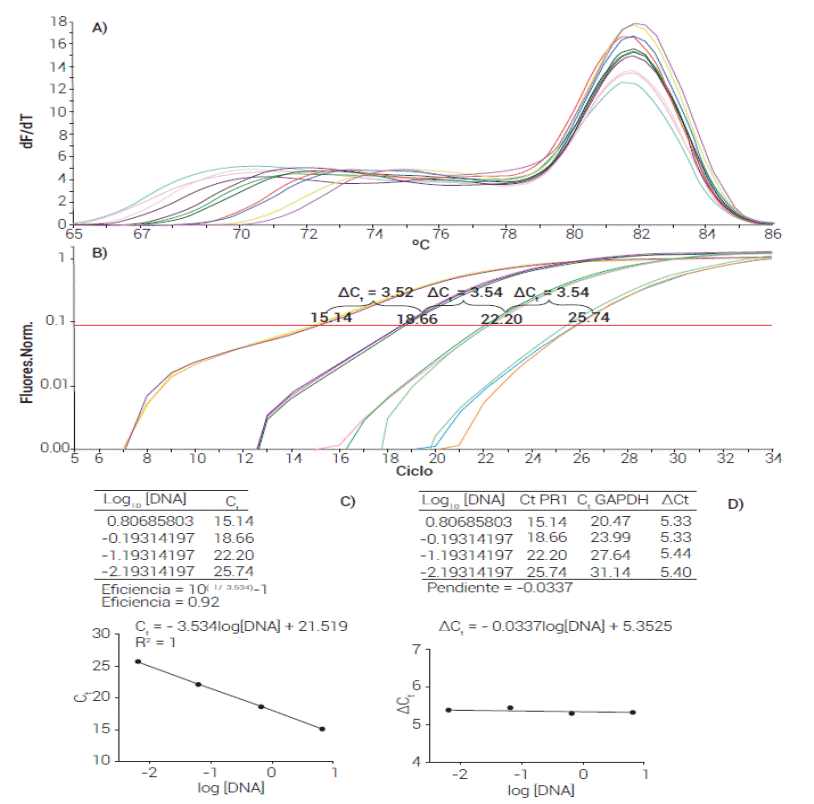
Figura 1: Curvas de fusión (A) y amplificación (B) obtenidas con los oligonucleótidos PR1-F/PR1-R. En A se observa un máximo, que indica que se obtuvo un solo producto de amplificación. En B, las diferencias entre los valores de Ct promedio de los triplicados de cada dilución decimal del DNA molde son constantes, lo que indica una manipulación técnica adecuada y eficiencias de amplificación constantes en el intervalo de concentraciones de DNA utilizado en el ensayo. Cálculo de la eficiencia (C) y estabilidad (D) del amplicón obtenido con los oligonucleótidos PR1-F/PR1-R. La eficiencia para este par de oligonucleótidos fue de 92 %, y la pendiente de la curva de estabilidad se localizó dentro del intervalo esperado (-0.1 a 0.1).
Estos perfiles de la curva de fusión se explican debido a que durante el proceso de calentamiento de la muestra durante la fase de fusión ("melting"), el amplicón o amplicones producidos durante la reacción de qPCR generan un punto de inflexión (cambio en la pendiente) en la curva de fusión al alcanzar su Tm. De esta manera, al obtener la primera derivada del cambio de fluorescencia respecto al cambio de temperatura, los puntos de inflexión aparecen como máximos cuyo valor coinciden con la Tm del amplicón, y su altura se asocia con la abundancia del producto correspondiente.
El registro de la fluorescencia se realizó a 72 °C, por lo que el efecto de estos máximos adicionales no afecta al cálculo de la eficiencia y el rango dinámico, así como tampoco a la eventual medición de la expresión genética. La Figura 1B muestra las curvas de amplificación para los oligonucleótidos PR1-F/PR-1R, donde se puede observar que las diferencias entre los valores de Ct promedio entre diluciones consecutivas son constantes (ΔCt = 3.5 ciclos), lo que señala, por un lado, que la manipulación técnica durante la preparación de las reacciones fue adecuada, y por otro que la eficiencia de la amplificación es constante en el intervalo de concentraciones de DNA molde utilizado.
La diferencia con el valor esperado (ΔCt = 3.32 ciclos) sugiere la presencia de un error sistemático, posiblemente asociado con la calibración del instrumental volumétrico utilizado. Las Figuras 1C y 1D ejemplifican el cálculo de la eficiencia y estabilidad para los oligonucleótidos PR1-F/ PR-1R.
Conclusiones
De los 17 pares de oligonucleótidos evaluados para R. solanacearum, 12 cumplieron con las características de eficiencia y estabilidad para ser usados en estudios de qPCR y se asociaron con nueve genes. De los 23 pares de oligonucleótidos evaluados en el tomate, 12 presentaron una adecuada eficiencia y estabilidad en la amplificación y podrán ser utilizados para la evaluación cuantitativa de la expresión de 11 genes del tomate. El desempeño de un oligonucleótido está influenciado de manera determinante por la complementariedad de bases con su secuencia blanco, por lo que es crucial verificar esta característica antes de su incorporación en un experimento.
La evaluación experimental de la eficiencia y el rango dinámico determinará si un par de oligonucleótidos es adecuado para medir expresión genética bajo ciertas condiciones experimentales, y siempre se deberá tomar en cuenta que cualquier modificación en las condiciones de reacción puede afectar el desempeño de los mismos, y la veracidad de los resultados obtenidos. Los oligonucleótidos a utilizar para evaluar niveles de expresión genética con la técnica de qPCR deben ser evaluados bajo las condiciones que se utilizan en cada laboratorio. La evaluación y selección de oligonucleótidos realizadas en este estudio, permitirá que los perfiles de expresión genética obtenidos sean un reflejo fiel de lo que ocurre durante la interacción del tomate con R. solanacearum, lo que proporciona cimientos sólidos sobre los cuales se construirá el conocimiento de este patosistema.











 text new page (beta)
text new page (beta)


