Introducción
El mango (Mangifera indica L.) es una planta perteneciente a la familia Anacardiaceae, nativa del sur de Asia, especialmente del Oriente de India; actualmente ha alcanzado una gran distribución por su cultivo en alrededor de 100 países (Galán, 2009), debido a su desarrollo en climas cálidos, y su adaptación a una gama de condiciones ambientales amplias (Asohofrucol y Corpoica, 2013).
Mangifera indica es una de las especies más apreciadas a nivel mundial, destacándose por la propagación y aprovechamiento del fruto, su utilidad para fines comerciales (Human y Rheeder, 2004) y farmacéuticos (Márquez et al., 2010), y por sus propiedades organolépticas y nutricionales (Kessel, 2008). Por sus atributos, M. indica es considerada de gran importancia comercial y con potencial económico (Galán, 2009), siendo cultivada en casi todas las zonas tropicales y subtropicales, principalmente en India, Brasil y México. La vasta distribución geográfica y ambiental de esta especie, ha sido un factor que ha propiciado la amplia diversidad genética de la misma, generando genotipos que exhiben diferencias notorias, particularmente en caracteres del fruto (García et al., 2010).
En Colombia, el cultivo de mango se produce en alrededor de 23 mil hectáreas (Espinal-García et al., 2005), presentes en 16 departamentos, y cuenta con aproximadamente 100 variedades (Espinal-García et al., 2005). Los mangos criollos son variedades de tamaño pequeño, reproducción poliembriónica, mayor vigor en las plantas e igual calidad que la planta madre (Vieccelli et al., 2016). Se considera que la mayoría de los cultivares de mango criollo tienen un origen poliembriónico (Vieccelli et al., 2016).
Los cultivares de mango, al igual que otras especies de árboles frutales, son identificados con base en rasgos morfológicos sustentados en descriptores (IPGRI, 2006). Sin embargo, la determinación basada exclusivamente en caracteres morfológicos es inexacta debido a la influencia de los factores ambientales y al número limitado de caracteres distinguibles. De esta manera, la identificación molecular de los cultivares de mango se ha llevado a cabo con diferentes sistemas moleculares como isoenzimas (Degani et al., 1990; Aron et al., 1997), repeticiones en tándem de número variable (VNTR) (Adato et al., 1995; Krishna y Singh, 2007; Begum et al., 2012; Sennhenn et al., 2014; Wang et al., 2016), polimorfismos de longitud en fragmentos amplificados (AFLPs) (Eiadthong et al., 2000; Kashkush et al., 2001; Yamanaka et al., 2006; Santos et al., 2008; Gálvez-López et al., 2009), ADN polimórficos amplificados al azar (RAPDs) (Souza et al., 2011; Samal et al., 2012; Hossain et al., 2016; Pruthvish y Chikkaswamy, 2016; Galal et al., 2017) y repetición de secuencias discretas (SSRs) (Surapaneni et al., 2013; Ravishankar et al., 2015; Azmat et al., 2016; Bajpai et al., 2016; dos Santos Alves et al., 2016; Nazish et al., 2017). Los resultados revelan diferencias entre las accesiones de mango sin importar el sistema de marcador utilizado, su origen geográfico o su situación genética (cultivares, razas autóctonas o variedades).
Las repeticiones de secuencia simple (SSRs) constituyen una de las herramientas más específicas, eficaces y rápidas para analizar la diversidad genética de las especies (Azofeifa-Delgado, 2006), se trata de secuencias de ADN de 1 a 6 nucleótidos dispuestos en grupo, distribuidos en todo el genoma y cuya herencia es segregante (Becerra y Paredes, 2000). Estos marcadores presentan múltiples ventajas como codominancia, multialelismo y alta heterocigosidad; además, requieren una cantidad mínima de ADN para su estudio y realizan una discriminación precisa entre individuos altamente emparentados gracias a su alto nivel polimórfico (González, 2003).
El conocimiento sobre la diversidad genética de las poblaciones de mango en Colombia es pobre, destacándose el estudio realizado en seis poblaciones de Colombia (Díaz-Matallana et al., 2009), por lo que se desconoce la diversidad genética poblacional de Mangifera indica a nivel de Valencia, Córdoba. En este sentido, esta situación afecta al sector agrícola y a la comunidad científica ante la carencia de información para seleccionar variedades y poblaciones de mango promisorias para el mejoramiento genético o selección de cultivares con mayor productividad.
Por lo anterior, este trabajo tiene como objetivo determinar en el municipio de Valencia, departamento de Córdoba, la diversidad genética de 35 accesiones de Mangifera indica, empleando 12 marcadores microsatélites, como requerimiento básico para los programas de mejoramiento de mango, conservación y selección de materiales promisorios (Krishna y Singh, 2007), para la industria alimentaria a nivel nacional.
Materiales y Métodos
Material vegetal
El material vegetal estudiado fue colectado en cinco fincas del municipio de Valencia, en el departamento de Córdoba, Colombia (Fig. 1). Se muestrearon 35 accesiones distribuidas en subpoblaciones de la siguiente forma: Villanueva: 8°20'27.97''N; 76°07'0848''O (n=6), Valencia: 8°15'25.77''N; 76°10'27.79''O (n=8), El Reposo: 8°10'41.44''N; 76°10'’13.51''O (n=6), San Rafael del Pirú: 8°11'06.89''N; 76°16'40.56''O (n=8), Santo Domingo: 8°08'36.88''N; 76°19'34.29''O (n=7).
Las accesiones muestreadas fueron deshidratadas con silica gel y mantenidas bajo las condiciones del Herbario HUC de la Universidad de Córdoba.
Extracción de ADN genómico
Para cada muestra se colocaron fragmentos de tejido foliar seco en una cápsula de porcelana con nitrógeno líquido, se maceraron hasta obtener un polvo fino y se guardaron a -20 ºC hasta su empleo.
La extracción del ADN genómico se llevó a cabo en 40 mg del material macerado, usando el Dneasy Plant Mini Kit de extracción de QIAGEN® (QIAGEN Inc., Valencia, CA, USA).
Análisis genético de repeticiones de secuencia simple (SSRs)
La concentración de ADN se determinó mediante un espectrofotómetro NanoDrop®ND-1000 (Thermo Fisher Scientific, Wilmington, EUA) a 260 y 280 nm de longitud de onda, como lo señalan Valadez y Kahl (2000).
Se emplearon 12 loci microsatélites utilizados por Ravishankar et al., (2011). La amplificación de los mismos se realizó mediante PCR en un volumen de mezcla de reacción de 12 μl que incluyó 0.24 μl de dNTPs 10 μM, 1.8 μl de amortiguador 10X, 0.72 μl de MgCl2 1.5 μM, 0.12 μl de cebadores específicos (forward y reverse) 0.5 μM, 0.12 μl de enzima Taq polimerasa a una concentración de 0.05 U/μl, 2 μl de ADN genómico a una concentración de 3.33 ng/μl y 6.88 μl de agua estéril.
La reacción de PCR se realizó en un Termociclador Bio-Rad T100™ (Applied Biosystems, Foster City, EUA) y consistió de una fase de desnaturalización de 95 °C durante 3 min, una segunda fase de 35 ciclos de 30 s de desnaturalización a 95 °C, 30 s a la temperatura de alineamiento (Cuadro 1), seguido por una tercera fase de extensión a 72 °C por 1 min y finalmente, una fase de extensión de 5 min a 72 °C.
Cuadro 1: Microsatélites tipificados de Mangifera indica L. (Ravishankar et al., 2011), secuencia de iniciadores, numero de alelos y rango alélico (pb).
| Marcador | Secuencia de iniciadores | Temperatura de alineamiento (ºC) | No. de alelos | Rango alélico (pb) |
|---|---|---|---|---|
| MiIIHR12 | F: GCCCCATCAATACGATTGTC R: ATTTCCCACCATTGTCGTTG |
55 | 9 | 154-185 |
| MiIIHR13 | F: CCCAGTTCCAACATCATCAG R: TTCCTCTGGAAGAGGGAAGA |
55 | 5 | 189-201 |
| MiIIHR15 | F: TAACCATTCGGCATCCTCT R: TGTGATAGAATGGCAAAAGAA |
55 | 5 | 153-164 |
| MiIIHR17 | F: GCTTGCTTCCAACTGAGACC R: GCAAAATGCTCGGAGAAGAC |
57 | 7 | 258-294 |
| MiIIHR18 | F: TCTGACGTCACCTCCTTTCA R: ATACTCGTGCCTCGTCCTGT |
55 | 5 | 170-181 |
| MiIIHR19 | F: TGATATTTTCAGGGCCCAAG R: AAATGGCACAAGTGGGAAAG |
56 | 7 | 192-224 |
| MiIIHR23 | F: TCTGACCCAACAAAGAACCA R: TCCTCCTCGTCCTCATCATC |
55 | 12 | 112-150 |
| MiIIHR24 | F: GCTCAACGAACCCAACTGAT R: CCAGCATTCAATGAAGAAGTT |
55 | 8 | 245-274 |
| MiIIHR26 | F: GCGAAAGAGGAGAGTGCAAG R: TCTATAAGTGCCCCCTCACG |
56 | 8 | 135-156 |
| MiIIHR31 | F: TTCTGTTAGTGGCGGTGTTG R: CACCTCCTCCTCCTCCTCTT |
56 | 5 | 212-236 |
| MiIIHR34 | F: CTGAGTTTGGCAAGGGAGAG R: TTGATCCTTCACCACCATCA |
55 | 5 | 227-243 |
| MiIIHR36 | F: TCTATAAGTGCCCCCTCACG R: ACTGCCACCGTGGAAAGTAG |
56 | 8 | 222-250 |
| Promedio total | - | 7 | - |
Los productos de PCR se separaron mediante electroforesis vertical en gel de poliacrilamida a 10% (acrilamida: bisacrilamida, 29:1) desnaturalizado (6 mol L-1 de urea), en una cámara Mini-Protean II Biorad® (Applied Biosystems, Foster City, EUA) (Tsang et al., 1986). Las bandas se visualizaron por tinción con nitrato de plata (Qiu et al., 2012), utilizando DNA ladder, donde el rango del marcador de peso molecular utilizado osciló entre 50 y 500 pb. Los geles fueron fotografiados con una cámara CANON (ELPH180 IS, Tokio, Japón) y la determinación del tamaño alélico se efectuó mediante el programa ImageJ (Abràmoff et al., 2004) donde por pixelaje de las bandas amplificadas, se determinaron los tamaños alélicos utilizando el software Past 3.14 (Hammer et al., 2001).
Análisis estadístico
Mediante el software GenAlEx versión 6.5 (Peakall y Smouse, 2006) se calculó para cada locus: número de alelos observados, número efectivo de alelos, valores de heterocigosidad observada y esperada (Ho y He), distancia genética y equilibrio de Hardy-Weinberg. El PIC (Contenido de Información Polimórfica) de cada locus microsatélite se estimó mediante el programa CERVUS versión 3.0.7 (Kalinowski et al., 2007). Los índices de fijación de Wright: FIS, FIT y FST, se calcularon mediante el programa FSTAT versión 2.9.3.2 (Goudet et al., 2002). El dendrograma se construyó mediante el método UPGMA con un bootstrap de 80%, empleando el programa MEGA versión 7 (Kumar et al., 2016). Asimismo, para la identificación de duplicados se utilizó el software Excel Microsatellite Toolkit (Park, 2001).
Resultados
En total, 84 alelos fueron observados para la población de mangos en Valencia, Córdoba, Colombia, con un número promedio de siete alelos en el germoplasma del mango. El locus MiIIHR23 presentó el mayor número (12) de alelos y los loci que presentaron el menor número (5) de alelos fueron MiIIHR13, MiIIHR15, MiIIHR18, MiIIHR31 y MiIIHR34 (Cuadro 1).
El promedio del número efectivo de alelos fue de 4.5, el locus con el mayor número efectivo (7.9) de alelos fue MiIIHR23 y MiIIHR34 presentó el menor número efectivo (2.1) de alelos (Cuadro 2).
Cuadro 2: Parámetros básicos de diversidad genética calculados para el germoplasma de Mangifera indica L. en Valencia, Córdoba, Colombia. Número efectivo de alelos: Na, heterocigosidad observada: Ho, heterocigosidad esperada: He, contenido de información polimórfica: PIC, equilibrio de Hardy-Weinberg: EHW. *Desvío significativo del equilibrio de Hardy-Weinberg (P<0.05).
| Locus | Na | Ho | He | PIC | EHW |
|---|---|---|---|---|---|
| MiIIHR12 | 3.306 | 0.409 | 0.742 | 0.718 | 0.000*** |
| MiIIHR13 | 3.902 | 0.300 | 0.744 | 0.702 | 0.000*** |
| MiIIHR15 | 3.375 | 0.111 | 0.704 | 0.652 | 0.000*** |
| MiIIHR17 | 5.667 | 0.647 | 0.824 | 0.800 | 0.006** |
| MiIIHR18 | 3.945 | 0.214 | 0.768 | 0.729 | 0.000*** |
| MiIIHR19 | 4.167 | 0.500 | 0.760 | 0.730 | 0.020* |
| MiIIHR23 | 7.918 | 0.412 | 0.874 | 0.862 | 0.000*** |
| MiIIHR24 | 6.081 | 0.533 | 0.836 | 0.815 | 0.000*** |
| MiIIHR26 | 5.885 | 0.059 | 0.832 | 0.810 | 0.000*** |
| MiIIHR31 | 2.569 | 0.053 | 0.611 | 0.573 | 0.000*** |
| MiIIHR34 | 2.117 | 0.211 | 0.528 | 0.493 | 0.000*** |
| MiIIHR36 | 5.684 | 0.778 | 0.824 | 0.802 | 0.001** |
| Promedio total | 4.551 | 0.355 | 0.748 | 0.724 | - |
Para la heterocigosidad observada (Ho), los resultados obtenidos oscilaron entre 0.778 y 0.053 correspondientes a MiIIHR36 y MiIIHR31 respectivamente con un promedio de 0.355; mientras que los valores de heterocigosidad esperada (He) fueron de 0.874 y 0.528, correspondientes a MiIIHR23 y MiIIHR34 respectivamente con un promedio de 0.748 (Cuadro 2).
El contenido de información polimórfica (PIC) promedio obtenido para la población fue de 0.724 y sus valores oscilaron entre 0.862 y 0.493 (Cuadro 2), los cuales corresponden a los marcadores MiIIHR23 y MiIIHR34 respectivamente.
Los 12 marcadores microsatélites estudiados mostraron ausencia del equilibrio de Hardy-Weinberg (p<0.05) (Cuadro 2).
El valor promedio del estadístico FIS (Cuadro 3) fue de 0.345, con valores que oscilan entre 0.014 para el locus MiIIHR24 y 0.907 para el locus MiIIHR26; con relación al estadístico FIT, el valor promedio fue de 0.581, en rangos de 0.051 a 0.939 para los loci MiIIHR36 y MiIIHR26 respectivamente. Por otro lado, el valor promedio de FST obtenido fue de 0.371 con valores que oscilan entre 0.180 para el locus MiIIHR36 y 0.559 para el locus MiIIHR31.
Cuadro 3: Valores de los estadísticos F de Wright (FIS, FIT, FST) para cada marcador en la población de Valencia, Córdoba, Colombia.
| Locus | F IS | F IT | F ST |
|---|---|---|---|
| MiIIHR12 | 0.281 | 0.412 | 0.182 |
| MiIIHR13 | 0.525 | 0.682 | 0.331 |
| MiIIHR15 | 0.655 | 0.828 | 0.501 |
| MiIIHR17 | -0.322 | 0.185 | 0.383 |
| MiIIHR18 | 0.759 | 0.845 | 0.358 |
| MiIIHR19 | 0.183 | 0.456 | 0.335 |
| MiIIHR23 | 0.056 | 0.555 | 0.529 |
| MiIIHR24 | 0.014 | 0.547 | 0.540 |
| MiIIHR26 | 0.907 | 0.939 | 0.343 |
| MiIIHR31 | 0.784 | 0.905 | 0.559 |
| MiIIHR34 | 0.458 | 0.572 | 0.210 |
| MiIIHR36 | -0.158 | 0.051 | 0.180 |
| Media | 0.345 | 0.581 | 0.371 |
Atendiendo a los valores de distancia genética obtenidas (Cuadro 4), puede apreciarse que las subpoblaciones de Valencia y El Reposo resultaron ser las subpoblaciones más parecidas genéticamente y las más cercanas geográficamente, mientras que Villanueva y Santo Domingo fueron las subpoblaciones menos parecidas y las más alejadas geográficamente.
Cuadro 4: Matriz de distancia genética de Nei (1972) en las subpoblaciones estudiadas en Valencia, Córdoba, Colombia.
| Villanueva | Valencia | El Reposo | San Rafael del Pirú | Santo Domingo | |
|---|---|---|---|---|---|
| Villanueva | ------- | ||||
| Valencia | 0.830 | ------- | |||
| El Reposo | 0.705 | 0.456 | ------- | ||
| San Rafael del Pirú | 0.983 | 1.031 | 0.982 | ------- | |
| Santo Domingo | 1.951 | 1.592 | 1.343 | 1.508 | ------- |
En el dendrograma generado para el análisis de las subpoblaciones (Fig. 2), se muestra la asociación que se presentó entre las subpoblaciones Villanueva, Valencia, El Reposo y San Rafael del Pirú, las cuales comparten su cercanía geográfica. Además, se muestra la subpoblación de Santo Domingo como la más alejada, siendo esta la más distante geográficamente.
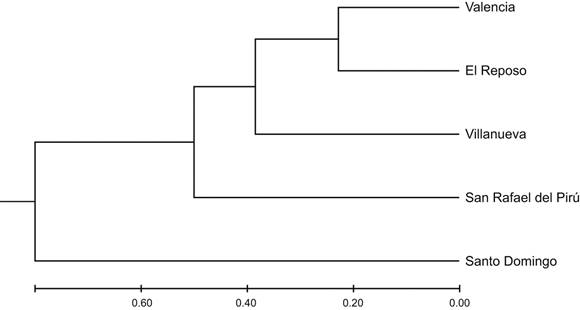
Figura 2: Dendograma basado en la distancia genética de Nei (1972) entre las subpoblaciones estudiadas en Valencia, Córdoba, Colombia, obtenido por el método UPGMA.
Discusión
El número medio de alelos por locus obtenido en esta investigación fue menor al obtenido por Dinesh et al. (2015) con una media de 12 alelos por locus. La obtención de un número de alelos inferior en nuestro estudio respecto al reportado se puede explicar por un descenso en la variabilidad del material vegetal analizado debido a un menor número de genotipos diferentes, y fue mayor al reportado por dos Santos Ribeiro et al. (2012), Armas-Moreno (2013) y Azmat et al. (2016) quienes registraron un promedio de 4, 2.6 y 3 alelos respectivamente, lo cual muestra que el material vegetal del presente estudio posee un mayor número de genotipos diferentes. Por otro lado, fue similar al obtenido por Bajpai et al. (2016) quien reportó un valor promedio de seis alelos.
Según Botstein et al. (1980), 11 de los 12 loci utilizados en este trabajo presentan valores del PIC superiores al valor de 0.5 indicado como umbral por encima del cual un marcador puede ser considerado muy informativo al momento de detectar la variabilidad genética y utilizarse eficazmente en estudios de diversidad genética en la población de mango estudiada. Nueve loci (MiIIHR12, MiIIHR13, MiIIHR17, MiIIHR18, MiIIHR19, MiIIHR23, MiIIHR24, MiIIHR26 y MiIIHR36) muestran un PIC superior a 0.7, valor por encima del cual el marcador es especialmente de utilidad para la construcción de mapas genéticos (Bandelj et al., 2004), y solo uno medianamente informativo, con valor de PIC mayor a 0.25. El PIC promedio obtenido en el presente estudio, utilizando marcadores STRs, resultó mayor al reportado por Begum et al. (2012) (PIC=0.66), Begum et al. (2013) (PIC=0.46), Tasliah et al. (2013) (PIC=0.548), Begum et al. (2014) (PIC=0.47), Bajpai et al. (2016) (PIC=0.438), dos Santos Alves et al. (2016) (PIC=0.550), Gitahi et al., (2016), (PIC=0.543) y Nazish et al. (2017) (PIC=0.398); menor al reportado por Ravishankar et al. (2015) (PIC=0.841) y similar al reportado por Hirano et al. (2010) (PIC=0.716) y Surapaneny et al. (2013) (PIC=0.717).
Los resultados obtenidos para la heterocigosidad observada (Ho) fueron menores a los reportados por Viruel et al. (2005) (Ho=0.69), Gálvez-López et al. (2009) (Ho=0.51), Hirano et al. (2010) (Ho=0.645), Ravishankar et al. (2015) (Ho=0.624), dos Santos Alves et al. (2016) (Ho=0.437) y Gitahi et al. (2016) (Ho=0.64), y los valores encontrados para la heterocigosidad esperada (He) fueron mayores a los reportados por Viruel et al. (2005) (He=0.65), Hirano et al. (2010) (He=0.709), Surapaneny et al. (2013) (He=0.390), dos Santos Alves et al. (2016) (He=0.624), Gitahi et al. (2016) (He=0.55) y Nazish et al. (2017) (He=0.262), y menores a los reportados por Gálvez-López et al. (2009) (He=0.92), Ravishankar et al. (2015) (He=0.858).
El número de alelos efectivos (Na) para la población de mango estudiada osciló entre 2.11 y 7.91 con un promedio de 4.55, valor representativo según Bakoumé et al. (2007), quienes proponen que se requiere de un alto número de alelos para la selección de individuos con caracteres deseables y evitar la pérdida de diversidad genética a través de planes de conservación. Este resultado (Na=4.55), es mayor al reportado por Viruel et al. (2005) (Na=3.3), Gitahi et al. (2016) (Na=3.0) y Nazish et al. (2017) (Na=1.8), y menor en comparación con el promedio obtenido por Hirano et al. (2011) (Na=6.5).
Los 12 marcadores microsatélites mostraron una desviación significativa con respecto al equilibrio de Hardy-Weinberg, lo cual puede deberse a endogamia, existencia de una posible estructura genética por subdivisión (Efecto Wahlund), emparejamiento selectivo o poblaciones pequeñas (Chapuis y Estoup, 2007). También pudo haberse dado un efecto fundador, donde los pocos individuos que forman la nueva población no representan el perfil genético de la de origen, sumado a la fuerte recombinación que pudo darse entre los escasos individuos iniciales de la población (Nichols et al., 2001).
Los valores obtenidos para los estadísticos FIS y FIT fueron positivos para todos los marcadores revelando un déficit de heterocigotos. El exceso de homocigotos en una población podría ser el resultado de eventos de endogamia, lo cual puede aumentar la probabilidad de que se pierdan alelos para la siguiente generación, llevando a la pérdida progresiva de diversidad genética con el paso del tiempo (Ellegren y Galtier, 2016).
De otra parte, la cercanía genética resultante entre las subpoblaciones Villanueva, Valencia, El Reposo y San Rafael del Pirú concuerda con lo expuesto por Rocha y Lobo (1996), en su trabajo con Enterolobium cyclocarpum Jacq., en donde la distancia genética resultó directamente proporcional a la distancia geográfica.
Conclusiones
El estudio de Mangifera indica en Valencia, Córdoba, Colombia, reveló una alta diversidad genética; además, presentó un elevado número de alelos y ausencia de equilibrio de Hardy-Weinberg. Los marcadores utilizados resultaron ser altamente informativos, atendiendo al PIC, razón por la cual son recomendables para posteriores análisis de diversidad genética en esta especie.











 text new page (beta)
text new page (beta)




