Introducción
Los conceptos de centro de origen de una especie, centro de domesticación y centro de diversidad genética juegan un papel importante en la priorización de áreas de conservación y manejo de recursos fitogenéticos. El centro de origen de una especie es el área geográfica en donde surge este taxon y desde el cual se dispersó y adquirió su distribución actual (Croizat et al., 1974). El centro de domesticación de una planta con manejo agrícola es aquella región en donde ocurrió la domesticación más temprana de las plantas silvestres que constituyen los sistemas de alimentación de los distintos pueblos (Engels et al., 2006; Boege, 2009). El centro de diversidad genética, por otra parte, es el sitio en donde existen tanto poblaciones silvestres como diferentes razas o variantes silvestres de una planta cultivada (Engels et al., 2006; Boege, 2009).
La domesticación y diversificación no son eventos históricos únicos que se desarrollen en un momento dado, sino que se trata de un largo proceso de dispersión y adaptación continua (Engels et al., 2006; Zeder et al., 2006; Boege, 2009; Piperno, 2011), a diferencia del origen de una especie que ocurre una sola vez, por lo que pueden existir varios centros de domesticación y diversidad genética para una especie cultivada. Los centros de origen, domesticación y diversidad genética son regiones importantes para la conservación del germoplasma original de las especies, porque los ancestros silvestres de las especies cultivadas son recursos fitogenéticos importantes que pueden contribuir a resolver problemas de alimentación presentes o futuros mediante planes de mejoramiento genético (Engels et al., 2006; Hernández-Verdugo et al., 2006; Boege, 2009; Jamnadass et al., 2009).
Adicionalmente, identificar los centros de domesticación y diversidad genética proporciona una información valiosa para entender cómo operaron los mecanismos de selección en plantas manejadas por el hombre (Zeder et al., 2006; Boege, 2009). Para establecer el centro de origen y los centros de domesticación de una especie, es necesario recopilar información utilizando evidencia paleoecológica, arqueológica y filogenética. Sin embargo, en los casos en los que no existen estudios paleoecológicos y/o registros arqueológicos, la información filogeográfica obtenida con marcadores moleculares puede ayudar a inferir tanto el centro de origen, como el número aproximado y sitio en donde ocurrió la domesticación de la especie (Jamnadass et al., 2009).
Uno de los enfoques más empleados para el estudio evolutivo de plantas domesticadas es el de la filogeografía (Schaal et al., 1998; Schaal y Olsen, 2000; Doebley et al., 2006; Engels et al., 2006; Jamnadass et al., 2009; Gross y Olsen, 2010), entendida como el estudio de los principios y procesos que rigen la distribución espacial de los linajes genealógicos dentro y entre especies (Avise, 2000). Los marcadores moleculares de DNA de cloroplasto son los que se han privilegiado en los estudios filogeográficos de las plantas, porque tienen mayor probabilidad de retener la información biogeográfica (Burger et al., 2008; Byrne y Hankinson, 2012; Nybom et al., 2014). En términos generales se asume que la diversidad genética de las especies cultivadas es menor que la de sus ancestros silvestres como consecuencia de la selección direccional y los cuellos de botella a que se someten las especies domesticadas. Sin embargo, existe evidencia de que tanto el flujo génico como la endogamia son inducidas por la domesticación. Por ejemplo, en plantas domesticadas en Mesoamérica, se propició el flujo génico a través de la dispersión deliberada de explantes de Stenocereus pruinosus (Otto ex Pfeiff.) Buxb. (Parra et al., 2012), o la endogamia por la selección de fenotipos que tienden a autopolinizarse con mayor frecuencia en Polaskia chichipe (Gosselin) Backeb. (Otero-Arnaiz et al., 2003).
Mesoamérica se reconoce como uno de los principales centros de origen, domesticación y diversidad biológica de plantas cultivadas para la alimentación humana y animal (Vavilov, 1951; Harlan, 1971; Perales-Rivera y Aguirre-Rivera, 2008), en donde se domesticaron más de 100 especies de plantas (Hernández-Xolocotzi, 1998; Casas et al., 2007). La ciruela mexicana (Spondias purpurea L.) es una especie semidomesticada, con niveles incipientes o intermedios de domesticación en la región Mesoamericana (Miller y Knouft, 2006; Ruenes-Morales et al., 2010) en la que las poblaciones cultivadas presentan una gran similitud genética con las poblaciones silvestres (Miller y Schaal, 2006). Estudios filogeográficos previos sugieren dos probables centros de domesticación de esta especie, uno en las selvas bajas caducifolias del sur de Jalisco y Colima, y otro en las selvas bajas de la costa sur de El Salvador (Miller y Schaal, 2005). Sin embargo, se desconoce cuál es el centro de origen de esta especie, y si existen otros sitios adicionales de domesticación a los reportados por Miller y Schaal (2005). Ruenes-Morales et al. (2010) sugieren a la Península de Yucatán como otro sitio de domesticación ya que se encuentra una gran diversidad de formas cultivadas de esta especie. La falta de información sobre sitios adicionales de domesticación puede deberse en gran medida a la falta de inclusión de ejemplares de sitios donde la especie se distribuye en México y que no fueron incluidos en los trabajos de Miller y Schaal, 2005 y Miller, 2008. Al incrementarse el número de muestras representativas de diferentes regiones es posible detectar un número mayor de eventos de domesticación, como se demostró en el caso del frijol lima “ib” (Phaseolus lunatus L.) (Andueza-Noh, 2012).
Para este estudio nos preguntamos lo siguiente: ¿cuál es el centro de origen de Spondias purpurea y cuáles son sus diferentes centros de domesticación y diversidad genética en Mesoamérica? Para lograr tal objetivo, se realizó un análisis filogeográfico empleando el espaciador intergénico trnS-trnG del cloroplasto. Las secuencias analizadas se obtuvieron de colectas llevadas a cabo por las autoras, ejemplares del banco de germoplasma de la red de ciruela del Sistema Nacional de Recursos Fitogenéticos para la Alimentación y la Agricultura (SINAREFI, de aquí en adelante) y las reportadas por Miller y Schaal (2006). Para su análisis las secuencias se agruparon en 54 poblaciones que se presentan en nueve de las 14 regiones biogeográficas reconocidas por Espinosa-Organista et al. (2008) para la región Mesoamericana de México.
Materiales y Métodos
Aislamiento, amplificación y secuenciación de DNA
El DNA genómico se obtuvo de tejido foliar de 66 individuos de Spondias purpurea que constituyen la colección viva que es el reservorio de germoplasma de la Red Ciruela SINAREFI, que se encuentran albergada en el Centro Regional Universitario de la Península de Yucatán-Universidad Autónoma Chapingo (CRUPY-UACh, de aquí en adelante). Las colectas para conformar este banco de germoplasma se realizaron en cuatro regiones productoras de ciruela mexicana: Vertiente Pacífico Norte (Jalisco, Nayarit, Sinaloa y Sonora); Vertiente Pacífico Sur (Chiapas, Guerrero, Michoacán y Oaxaca), Centro-Sur (Morelos, Puebla y Veracruz), y Sureste de México (Campeche, Quintana Roo, Tabasco y Yucatán).
En cada una de las zonas se colectaron estacas (1 m de altura y 5 cm de diámetro) de individuos en pie para las principales variedades tradicionales de la ciruela mexicana y de las poblaciones silvestres remanentes en las zonas, de acuerdo a las indicaciones de informantes clave. Las estacas colectadas se mantienen en las instalaciones del CRUPY-UACh y un respaldo en la Universidad Autónoma de Yucatán (UADY) y la información de las colectas (nombre de la variedad, colector(es), geo-referencias, usos de la planta) se mantiene en la base de datos del banco de germoplasma a cargo del SINAREFI. La distribución de las accesiones corresponde al siguiente número de individuos por estado de procedencia: 12 de Campeche, 15 de Morelos, uno de Nayarit, uno de Michoacán, dos de Jalisco, uno de Sinaloa, dos de Tabasco, tres de Veracruz y 27 de Yucatán (Apéndice 1).
El DNA se extrajo de muestras de 250 mg de tejido macerado con nitrógeno líquido empleando el método CTAB mini-prep modificado por Falcón y Valera (2007). La pureza del DNA genómico se evaluó mediante electroforesis en geles de agarosa borato de sodio a 0.8% (Brody y Kern, 2004) y se cuantificó usando un nano-espectro-fotómetro (Nanodrop 1000 Spectophotometer Thermo Scientific, Wilmington, EUA).
La amplificación de las muestras de DNA genómico se realizó utilizando los cebadores reportados por Miller y Schaal (2005) para el marcador del espacio intergénico trnS-trnG de DNA de cloroplasto. La reacción de PCR se hizo en 10 μl que incluyen 1.5 μl de DNA genómico (20 ng aproximadamente), 0.2 μl del cebador sentido (35 mM) y 0.2 μl del cebador contrasentido (35 mM), 4.5 μl de GoTaq (2X) y 3.6 μl de H2O. Las condiciones del termociclador (Techne-512, Barlorworld Scientific US, Burlington, EUA) para las PCR fueron las siguientes: desnaturalización 95 °C por 2 min; siete ciclos: 94 °C por 45s, 68 °C por 45s (disminuyendo 2 °C en cada ciclo), 72 °C por 60s, 30 ciclos, 94 °C por 45s, 48 °C por 45s, 72 °C por 60s y extensión final: 72 °C por 5 min. El producto de la PCR se purificó con el kit Clean Wizard (Promega) siguiendo las instrucciones del proveedor. Los amplicones purificados fueron enviados a secuenciar a The Centre for Applied Genomics del Sick Kids Hospital de la ciudad de Toronto, Canadá (TCAG por sus siglas en inglés) en el que se emplea un secuenciador ABI 3730XL (Applied Biosystems, Carlsbad, EUA).
Obtención de secuencias de GenBank
Para tener una mayor representatividad de especímenes cultivados y silvestres de Spondias purpurea y de las especies hermanas S. mombin var. mombin, S. mombin var. globosa J.D. Mitch. & D.C. Daly, S. radlkoferi Donn. Sm. y S. testudinis J.D. Mitch. & D.C. Daly, se obtuvieron las secuencias publicadas en GenBank (2015), con una búsqueda en la base Nucleotide empleando las palabras clave “Spondias” y “trnS-trnG”. La información de la localidad y el tipo de población de la que se obtuvo el ejemplar (silvestre, cerca viva, cerca natural, cultivado y parcelas) se recopiló en la base de datos Tropicos (TROPICOS, 2015) que alberga la información de las accesiones analizadas por Miller y Schaal (2005) que fueron depositadas en el herbario del Missouri Botanical Garden (MO). El tipo de manejo y la ubicación geográfica exacta para las colectas de Brasil no pudo obtenerse. La información de la ubicación geográfica, tipo de población, así como los números de accesiones del GenBank se reportan en el Apéndice 1.
Adicionalmente, se obtuvieron de GenBank: a) 51 secuencias de región intergénica trnA-trnH de DNAcp, 48 secuencias para siete especies del género Spondias, dos secuencias de la especie Pseudospondias microcarpa (A. Rich.) Engl. (Anacardiaceae) y una secuencia de Canarium balansae Engl. (Burseraceae), y b) 90 secuencias del marcador de DNA nuclear fosfoenol piruvato carboxilasa (PEPC): 51 secuencias para tres especies del género Spondias L. (Anacardiaceae) y 49 secuencias de 19 especies de Bursera Jacq. ex L. (Burseraceae) cuyas descripciones y número de accesión del GenBank se reportan en el Apéndice 2.
Agrupamiento de secuencias en provincias biogeográficas y por estatus de domesticación
Cada uno de los ejemplares para los que se obtuvieron secuencias se asignó a una de 54 poblaciones de acuerdo con su distribución geográfica (latitud y longitud) y éstas fueron numeradas de 1 a 54, de la distribución más austral hasta la más boreal (Apéndice 1). Las localidades de todos los ejemplares fueron mapeadas en Google Maps (figura no mostrada) con base en sus coordenadas geográficas, y ubicadas dentro de las áreas de distribución de las provincias biogeográficas modificadas por Espinosa-Organista et al. (2008). Las poblaciones de S. purpurea y las de sus especies hermanas están presentes en siete provincias biogeográficas: Eje Neovolcánico, Sierra Madre del Sur, Los Altos de Chiapas, Costa del Pacífico, Depresión del Balsas, Golfo de México y Península de Yucatán (Apéndice 1).
Los ejemplares se agruparon también por estatus de manejo empleando una categorización propuesta por Parra et al. (2012) quienes incluyen: 1) poblaciones silvestres, 2) manejo silvícola y 3) manejo agrícola. Estas tres categorías reconocen que los recursos fitogenéticos de Mesoamérica son colectados muchas veces de las poblaciones silvestres, mientras que en otros casos individuos silvestres son tolerados y mantenidos en vegetación primaria o secundaria, y otros tantos se cultivan en las parcelas o en los huertos de la casa donde se manejan con prácticas agrícolas (Casas et al., 2007; Parra et al., 2012). Los tres estatus de manejo para las poblaciones de ciruela corresponderían a 1) silvestres (en donde la ciruela puede o no ser recolectada), 2) manejo silvícola (toleradas en cercas vivas y cercas naturales) y 3) manejo agrícola (presentes en huertos familiares y cultivadas en parcelas).
Cabe destacar que debido al muestreo dirigido realizado por Allison Miller (Miller y Schaal, 2005) y los colectores asociados a la red frutal ciruela del SINAREFI, en cada población todos los ejemplares tienen solo uno de los estatus de manejo.
Alineación de las secuencias y prueba de neutralidad
Las secuencias se alinearon usando el algoritmo ClustalX con el paquete MEGA 5 (Tamura et al., 2011). El alineamiento final se revisó de manera visual. Para comprobar la neutralidad en la evolución molecular de las secuencias de la región del cloroplasto trnS-trnG, se usaron los estadísticos D de Tajima y D* de Fu y Li (Nei y Kumar, 2000) con el programa DnaSP 3.19 (Librado y Rozas, 2009).
Diversidad genética en poblaciones, regiones biogeográficas y por tipos de manejo
Los análisis de diversidad genética se realizaron solo para las secuencias de S. purpurea. Se registraron las frecuencias de los haplotipos para cada población, y los índices de diversidad haplotípica (Hd) y diversidad nucleotídica (p) se estimaron para cada provincia biogeográfica y para los datos agrupados en las tres categorías de manejo.
Análisis filogeográfico de las secuencias
El análisis filogeográfico para examinar las relaciones entre las áreas de distribución y los linajes genéticos de S. purpurea y sus especies hermanas se realizó obteniendo una red de haplotipos (Schaal et al., 1998; Avise, 2000). La red de haplotipos se obtuvo con la ayuda del programa TCS 1.21 (Clement et al., 2000) uniéndolos a todos hasta con un máximo de 70 diferencias. Los análisis de contingencia anidada para agrupar a los haplotipos (Templeton y Singt, 1993) se realizaron con el programa PopArt 1 (2014) en el módulo TCS. Se examinó la frecuencia de los diferentes haplotipos por especie y por poblaciones, así como tipo de manejo para cada grupo, con el fin de reconstruir la historia de los linajes genéticos de las cuatro especies de Spondias y para S. purpurea por separado.
Determinación de centros de origen, domesticación y diversidad genética
Se efectuaron análisis filogenéticos con: 1) región intergénica trnA-trnH de cloroplasto y 2) gen nuclear fosfoenol piruvato carboxilasa; para orientar la red de haplotipos de las especies de Spondias mesoamericanas. El alineamiento de las secuencias se realizó con el algoritmo ClustalW y una edición manual posterior con el programa MEGA 5.0 (Tamura et al., 2011), se obtuvieron dos alineamientos de longitud de 541 y 693 pares de bases, con 86 y 254 sitios variables, y 43 y 183 sitios parsimoniosamente informativos para la región intergénica trnA-trnH de DNA de cloroplasto y para el gen fosfoenol piruvato carboxilasa, respectivamente. Los modelos de sustitución nucleotídica: 1) Tamura 3 parámetros para la región intergénica trnA-trnH de DNA de cloroplasto y 2) mismo modelo más distribución gamma para los sitios (0.88) para el gen fosfoenol piruvato carboxilasa se seleccionaron con base a los valores de criterios de información Bayesiana y de información Akaike obtenidos en el módulo Modeltest del programa MEGA 5.0 (Tamura et al., 2011).
Los cladogramas se optimizaron con el método de máxima verosimilitud; el soporte de los grupos formados se evaluó con 1000 repeticiones con un método de bootstrap y el programa MEGA 5.0 (Tamura et al., 2011). Los cladogramas se emplearon para identificar la(s) especie(s) hermana(s) de S. purpurea y, con base en esta información, orientar la red de haplotipos.
La población silvestre de S. purpurea en la que se unió el grupo de haplotipos de S. purpurea al grupo de haplotipos de la(s) especie(s) hermana(s) se consideró como el centro de origen de la especie.
Los centros de domesticación se identificaron como las regiones dentro de las provincias biogeográficas en las que estuvieron presentes individuos tanto de poblaciones silvestres como aquellas con manejo silvícola y/o agrícola y que se determinaron como un grupo de haplotipos con base en el análisis de contingencia.
Los centros de diversidad genética se identificaron como las poblaciones silvestres, con manejo silvícola y/o agrícola, en que existen varios haplotipos, así como las provincias en las que los índices de diversidad genética fueran más altos.
Resultados y Discusión
Alineación de las secuencias y prueba de neutralidad
Las secuencias alineadas para el espacio intergénico trnS-trnG de DNA tuvieron una longitud de 592 pares de bases (pb), con 10 indels con longitudes de 1 a 219 pb, 43 sitios variables y 20 sitios parsimoniosamente informativos. El estadístico D de Tajima tuvo un valor de -0.89 y el D* de Fu y Li -1.23. Ambos fueron no significativos (P>0.10), lo que indica neutralidad en la evolución molecular del marcador. Las pruebas de neutralidad en el estudio del marcador trnS-trnG en Spondias (Miller y Schaal, 2005) fue similar en este estudio, indicando que el marcador es neutral. Por ello, su uso para inferir las relaciones filogenéticas entre los individuos evaluados, así como con otros marcadores de cloroplasto, es adecuado, como explican Shaw et al. (2007).
Red de haplotipos y análisis de contingencia anidada
De las 156 secuencias analizadas se identificaron 38 haplotipos diferentes para el marcador trnS-trnG, 20 pertenecientes a S. purpurea, 12 a S. mombin (en sus variedades mombin y globosa), dos a S. radlkoferi y tres a S. testudinis (Cuadro 1). Los haplotipos se agruparon en seis grupos en la red anidada de las especies de Spondias (Figs. 1A, C). El grupo 1 contiene 80% de las secuencias: los 17 haplotipos asignados de manera exclusiva a Spondias purpurea, tres haplotipos en los que están representados S. purpurea y S. mombin var. mombin, uno con S. purpurea y S. radlkoferi y uno con S. mombin var. mombin y S. testudinis (Cuadro 1). Este grupo se distribuye en las poblaciones 1-2, 5, 9-10, 12-14, 16, 18-21, 24-31, 34-42, 44-54 y en todas las provincias biogeográficas y en las poblaciones de Brasil (Cuadro 2, Fig. 1A); 58% de los ejemplares que pertenecen a este grupo tienen manejo agrícola, 35% provienen de poblaciones silvestres y 7% tienen manejo silvícola (Fig. 1B). Esta información sugiere que este grupo de haplotipos ha sido sometido a un proceso de domesticación y que no hay una asociación entre los diferentes haplotipos que le integran y la distribución geográfica de estos linajes.
Cuadro 1: Anidamiento de haplotipos (Hapl.) del marcador trnS-trnG de Spondias spp. en los grupos determinados en el análisis de contingencia anidada para todas las especies de Spondias L. (Grupo 1) y solo para S. purpurea L. (Sp, Grupo 2). Los haplotipos exclusivos para Spondias mombin L. var. mombin (Smm), Spondias mombin L. var. globosa J.D. Mitch. & D.C. Daly (Smg), S. radlkoferi Donn. Sm. (Sr) y S. testudinis J.D. Mitch. & Daly (St) no se incluyeron en el análisis del grupo 2.
| Hapl. | Especie | Grupos red 1 | Grupos red 2 | Hapl. | Especie | Grupos red 1 | Grupos red 2 | Hapl. | Especie | Grupos red 1 | Grupos red 2 |
| 1 | Sp | 1 | 1 | 14 | Smm, Sr | 2 | - | 27 | Smm | 2 | - |
| 2 | Sp | 1 | 2 | 15 | Sp | 1 | 2 | 28 | Smm | 2 | - |
| 3 | Sp | 1 | 1 | 16 | Sp | 1 | 5 | 29 | Smm | 3 | - |
| 4 | Sp | 1 | 2 | 17 | Sp, Smm | 1 | 1 | 29 | Smm, Smg | 3 | - |
| 5 | Sp | 1 | 2 | 18 | Sp, Smm | 1 | 3 | 30 | Smm | 3 | - |
| 6 | Sp | 1 | 1 | 19 | Sp | 1 | 3 | 31 | Smg | 3 | - |
| 7 | Sp | 1 | 2 | 20 | Sp | 1 | 1 | 32 | Smg | 3 | - |
| 8 | Sp | 1 | 1 | 21 | Sp | 1 | 3 | 33 | Smm | 3 | - |
| 9 | Sp | 1 | 1 | 22 | Smm | 1 | - | 34 | Sr | 4 | - |
| 10 | Sp | 1 | 3 | 23 | Smm | 2 | - | 35 | Sr | 5 | - |
| 11 | Sp | 1 | 3 | 24 | Smm | 2 | - | 36 | St | 1 | - |
| 12 | Sp | 1 | 4 | 25 | Smm | 2 | - | 37 | St | 1 | - |
| 13 | Sp, Sr | 1 | 1 | 26 | Smm | 2 | - | 38 | St | 1 | - |
Cuadro 2: Distribución de los haplotipos (Hapl.) de S. purpurea L., S. mombin L. var. mombin (Smm), S. mombin L. var. globosa J.D. Mitch. & D.C. Daly (Smg) S. radlkoferi Donn. Sm. (Sr) y S. testudinis J.D. Mitch. & Daly (St) en 54 poblaciones muestreadas (Pob) de provincia biogeográfica; más las de Brasil. Las secuencias se asignaron a 54 poblaciones de acuerdo con los datos de colecta presentados por Miller y Schaal (2006) y con las del banco de datos del SNICS-SINAREFI. CP=Costa del Pacífico; EN=Eje Neovolcánico; DB=Depresión del Balsas; SMS=Sierra Madre del Sur; GM=Golfo de México; LA=Los Altos de Chiapas; PY=Península de Yucatán. Se presenta además su estatus de manejo en la población (EM): silvestre (S), manejo silvícola (MS) y manejo agrícola (MA).
| Pob | 1 CP | 2 CP | 3 CP | 4 CP | 5 CP | 6 CP | 7 CP | 8 CP | 9 CP | 10 CP | 11 CP | 12 CP | 13 CP | 14 CP |
| Hapl. | 13 | 4 | Smm14 | Smm14 | 4 | Smm14 | Smm23 | Smm14 | 4 | 10 | Sr14 | 13 | 4 | 4 |
| Smm14 | Smm14 | 12 | Smm27 | Smm23 | Smm24 | Sr35 | Smm14 | 13 | ||||||
| Smm28 | Sr34 | Smm25 | Sr13 | 17 | ||||||||||
| Smm26 | Smm17 | |||||||||||||
| EM | MA | MA | MS | S | MA | S | S | MS | MA | MS | MS | S | S | S |
| Pob | 15 CP | 16 CP | 17 CP | 18 CP | 19 CP | 20 CP | 21 CP | 22 CP | 23 CP | 24 CP | 25 CP | 26 CP | 27 CP | 28 CP |
| Hapl. | Smm4 | 4 | Smm18 | 4 | 4 | 13 | 13 | Sm14 | Sr14 | 4 | 13 | 10 | 4 | 4 |
| Smm14 | 15 | 18 | ||||||||||||
| EM | S | MA | MS | MA | MS | S | MA | MS | S | MA | S | MA | MA | MS |
| Pob | 29 CP | 30 CP | 31 CP | 32 CP | 33 CP | 34 CP | 35 CP | 36 CP | 37 SMS | 38 LA | 39 LA | 40GM | 41 EN | 42 DB |
| Hapl. | 19 | 4 | 4 | Smm14 | Smm14 | 4 | 4 | 4 | 4 | 4 | 6 | 4 | 1 | |
| 10 | 16 | Smm14 | 13 | |||||||||||
| 13 | ||||||||||||||
| 20 | ||||||||||||||
| 21 | ||||||||||||||
| EM | MA | S | MA | S | MA | MA | MA | MA | MA,S | S | S | MA | S | MA |
| Pob | 43 GM | 44 PY | 45 EN | 46 EN | 47 CP | 48 PY | 49 CP | 50 PY | 51 PY | 52 CP | 53 CP | 54 CP | Brasil | Brasil |
| Hapl. | Sm14 | 4 | 2 | 5 | 2 | 11 | 1 | 4 | 4 | 4 | 1 | 4 | St36 | Smg 29 |
| 5 | 2 | 3 | St37 | Smm30 | ||||||||||
| 4 | St38Smm22 | Smm33 | ||||||||||||
| Smm29 | Smg 31 | |||||||||||||
| Smg 32 | ||||||||||||||
| EM | S | MA | MA | MA | S | MA | S | MA | MA,S | MA | S | MA | - | - |

Figura 1: Análisis de contingencia anidada de la red de haplotipos. A, C. Spondias mombin L. var. mombin, S. mombin L. var. globosa J.D. Mitch. & D.C. Daly, S. radlkoferi Donn. Sm., S. testudinis J.D. Mitch. & Daly y S. purpurea L.; B, D. Spondias purpurea. A, B. distribución de los individuos en las siete provincias biogeográficas (CP=Costa del Pacífico; EN=Eje Neovolcánico; DB=Depresión del Balsas; SMS=Sierra Madre del Sur; GM=Golfo de México; LA=Los Altos de Chiapas; PY=Península de Yucatán); C, D. estatus de manejo de la población: silvestre, silvícola, manejo agrícola.
El grupo 2 contiene 7 haplotipos de S. mombin var. mombin y uno que se registró en S. mombim var. mombin y S. radlkoferi (Cuadro 1); dos se distribuyen en las poblaciones 1-4, 6-8, 11-13, 15, 17, 22-23, 32-33, 37 y 43 que se ubican en las provincias de la Costa del Pacífico (en su porción sureste desde Oaxaca hasta Costa Rica) y en el Golfo de México (Cuadro 2, Fig. 1A); 50% de los ejemplares provienen de poblaciones silvestres y 50% de poblaciones con manejo silvícola (Cuadro 2, Fig. 1B). Esto sugiere que S. mombin está en un estado incipiente de domesticación a lo largo de su distribución en Mesoamérica y que los haplotipos que tienen manejo silvícola están todavía presentes en las poblaciones silvestres.
El grupo 3 está integrado por dos haplotipos de S. mombin var. mombin, dos de S. mombin var. globosa y uno que comparten las dos variedades (Cuadro 1). Todas las accesiones de este grupo provienen de Brasil (Cuadro 2, Fig. 1A) y no se tiene información sobre el manejo de estas especies. El grupo 4 contiene una secuencia de S. mombin var. mombin colectada en la población 6 silvestre, el grupo 5 una de S. radlkoferi de la población 7 con manejo agrícola y el grupo 6 una de S. radlkoferi de la población 11 con manejo silvícola (Cuadros 1, 2; Figs. 1A, B).
Los agrupamientos de haplotipos de las cinco especies estudiadas del género Spondias sugieren que los pobladores han introducido al cultivo un grupo de linajes genéticos presentes en poblaciones silvestres de S. purpurea (grupo 1) en menor proporción (Fig. 1B). Los linajes del grupo 1 han sido poco empleados en sistemas agroforestales donde se hace manejo silvícola (e.g. cercas vivas o vegetación natural o en ranchos para sombreado) (Fig. 1B). Las especies de S. mombin y S. radlkoferi son las que se prefieren para dichos usos, como lo sugieren las frecuencias similares de individuos de poblaciones silvestres y con manejo agrícola del grupo 2 (Fig. 1B). El agrupamiento de haplotipos representativos de la región sureste de la provincia de la Costa Pacífica y del Golfo de México (grupo 2), los de Brasil (grupo 3) y de los tres grupos con una secuencia única sugieren una diferenciación de los linajes genéticos que son particulares a la región sureste de Mesoamérica y Sudamérica (Fig. 1A). Por lo tanto, deben considerarse por separado en los esfuerzos de conservación de los recursos fitogenéticos del género Spondias.
El análisis de contingencia anidada para los haplotipos de Spondias purpurea permitió identificar 5 grupos en la red anidada (Figs. 1C, D). Tres de los cinco grupos de haplotipos agrupan más de cinco haplotipos, que en general tienen una amplia distribución en las diferentes poblaciones, mientras que dos grupos de haplotipos están confinados a la distribución más austral de la especie (Figs. 1B, D, 2). Las características de cada uno de estos grupos fueron útiles para la identificación de los centros de origen, de diversidad genética y domesticación como se discutirá en las siguientes secciones.

Figura 2: Red de haplotipos para cuatro especies de Spondias L. (S. mombin L. var. mombin, S. mombin L. var. globosa J.D. Mitch. & D.C. Daly, S. radlkoferi Donn. Sm., S. testudinis J.D. Mitch. & Daly, y S. purpurea L.). Los cuadros con bordes redondeados son haplotipos representados en poblaciones silvestres y con manejo agrícola; con bordes angulares corresponden a poblaciones con manejo silvícola y los óvalos a poblaciones silvestres.
Centro de origen de Spondias purpurea
El análisis de la red de haplotipos completa nos indica que S. mombin y S. radlkoferi forman un grupo diferenciado de S. purpurea (Fig. 2). Los haplotipos del grupo S. purpurea se caracterizan por tener en su mayoría individuos de esta especie; sin embargo, los haplotipos 13 y 17 se presentan en individuos de S. radlkoferi y de S. mombin var. mombin, respectivamente (Cuadro 1). Estos datos son apoyados por las reconstrucciones filogenéticas para los marcadores fosfoenol piruvato carboxilasa (Fig. 3A) y trnA-trnH (Fig. 3B), en los cuales la mayoría de individuos de S. purpurea forman un grupo monofilético y en los que algunas ramas terminales tienen agrupados individuos de S. mombim var. mombin y S. purpurea. Los resultados de ambos análisis sugieren: 1) un único origen para S. purpurea, 2) tiempo evolutivo corto para la divergencia de algunos linajes del género, 3) hibridación entre individuos de S. purpurea con S. mombin o S. radlkoferi en poblaciones locales. Ambas hipótesis deberán ser comprobadas o rechazadas ampliando el estudio con otros marcadores moleculares incluyendo más individuos de poblaciones en donde las especies crecen de manera simpátrica.

Figura 3: Reconstrucción filogenética (máxima verosimilitud) para: A. 99 secuencias del marcador de DNA nuclear fosfoenol piruvato carboxilasa para tres especies del género Spondias L. (Anacardiaceae; 51 secuencias, el par de letras finales en las ramas terminales que corresponden a S. purpurea son los haplotipos determinados por Miller (2008) y 19 especies de Bursera Jacq. ex L. (Burseraceae; 49 secuencias); B. región intergénica trnA-trnH de DNA de cloroplasto para siete especies del género Spondias L. (48 secuencias), Pseudospondias microcarpa (A. Rich.) Engl. (Anacardiaceae; 2 secuencias) y Canarium balansae Engl. (Burseraceae; 1 secuencia); Los valores para el bootstrap (1000 repeticiones) se presentan alrededor de los nodos.
La unión del grupo en que se incluyen las especies de S. mombin-S. radlkoferi y el grupo en que están incluidas S. purpurea-S. mombin-S. radlkoferi requiere 58 pasos (Fig. 2); por lo anterior, sugerimos que el marcador trnS-trnG es una región génica que nos permite diferenciar entre S. purpurea y sus dos especies hermanas. Cabe destacar que los haplotipos 14 (S. mombin-S. radlkoferi) y 2 (S. purpurea) son los que unen a los grupos por lo que podría considerarse a este último haplotipo como el ancestral para S. purpurea (Fig. 2). Si este es el caso, la región en la que se distribuye el haplotipo 2 (poblaciones 45, 47 y 49) (Cuadro 2, Fig. 4) puede considerarse como el centro de origen de la especie. Estas poblaciones se encuentran en la provincia del Eje Neovolcánico, en el occidente de México entre los estados de Jalisco y Michoacán (Fig. 4), regiones en donde se propone que se han originado el maíz y frijol, entre otras especies cultivadas importantes (Piperno, 2011).
Centros de domesticación
De acuerdo con el análisis de contingencia anidada para Spondias purpurea, el grupo 1 contiene nueve haplotipos distribuidos en las poblaciones: 1, 12-13, 20- 21, 25, 27-28, 30, 38-39, 41-42, 49 y 53, ubicadas en la provincia de la Costa del Pacífico, en Centroamérica (Nicaragua), el Eje Neovolcánico (Michoacán y Jalisco), la Sierra Madre del Sur y la Depresión del Balsas (Morelos) (Figs. 1B, 5); 73% de los ejemplares provienen de poblaciones silvestres, 23% son de poblaciones con manejo agrícola y 4% con manejo silvícola (Fig. 1D). Este linaje es el que ha sido, en proporción, menos empleado por los grupos humanos. Las regiones en las que existen poblaciones silvestres y con manejo agrícola y/o silvícola dentro de este grupo son: Nicaragua, poblaciones 20 y 21, y en Michoacán, Jalisco y Nayarit, 41, 49 y 53, respectivamente. Estos datos sugieren dos centros de domesticación para los linajes dentro de este grupo, lo que coincide con los resultados de Miller y Schaal (2005).

Figura 5: Distribución de haplotipos de Spondias purpurea L. incluidos en el grupo 1 del análisis de contingencia anidada de la red de haplotipos. El haplotipo 1 se repite en todos los paneles aunque pertenece al grupo 1.
El grupo 2 contiene 11 haplotipos distribuidos en las poblaciones 2, 5, 9, 13-14, 16, 18-19, 24, 27-28, 30-31, 34-38, 40, 42, 44, 45-47, 49-52 y 54 de todas las provincias biogeográficas (Fig. 4); 69% de los ejemplares provienen de poblaciones con manejo agrícola, 22% de poblaciones silvestres y 9% con manejo silvícola (Fig. 1D). Este linaje es el más extendido y es el que ha sido, en proporción, más empleado por los grupos humanos. Las regiones en las que existen poblaciones silvestres y con manejo agrícola y/o silvícola dentro de este grupo son: Costa Rica, poblaciones 9, 13 y 16; Honduras y Salvador, 24, 27 y 28; Guatemala, 30, 34 y 35; Jalisco, 45-47; y Yucatán, 50 y 51. Cabe destacar que el haplotipo 4, que comparten los individuos con manejo agrícola y los de las poblaciones silvestres, es el más frecuente de entre los 20 haplotipos de S. purpurea (Cuadro 2). Por esa razón, no se puede discernir si el linaje se domesticó en alguna de estas regiones y posteriormente se dispersó a las diferentes regiones, o si en cada uno de estos sitios tendríamos un centro de domesticación independiente.
El grupo 3 contiene siete haplotipos distribuidos en las poblaciones 10, 21, 26, 29, 30, 42 y 48 de las Provincias Costa del Pacífico (Centroamérica desde Nicaragua hasta Guatemala), Depresión del Balsas (Morelos) y Península de Yucatán (Campeche) (Figs. 1C, 6); 80% provienen de poblaciones con manejo agrícola y 20% de poblaciones silvestres (Fig. 1D). La región de Guatemala es la única en la que existen poblaciones silvestres y con manejo agrícola (29 y 30). Este resultado subraya nuevamente la importancia de la región de Guatemala como centro de domesticación de la ciruela mexicana (Miller y Schaal, 2005).
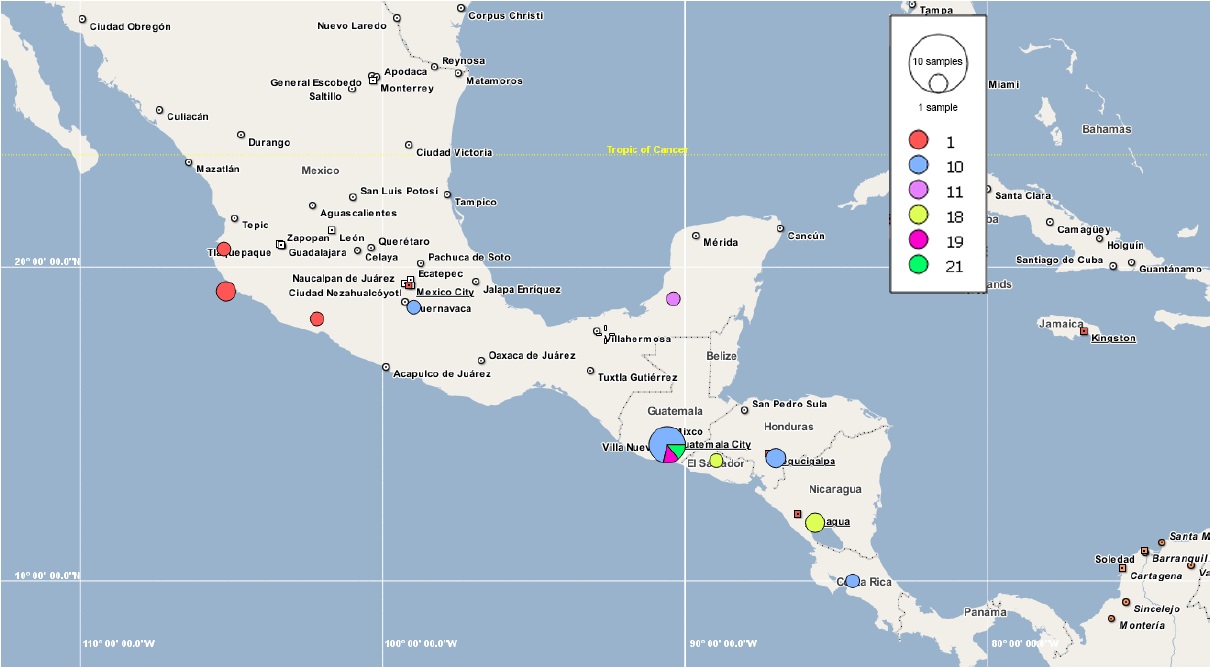
Figura 6: Distribución de haplotipos de Spondias purpurea L. incluidos en el grupo 3 del análisis de contingencia anidada de la red de haplotipos. El haplotipo 1 se repite en todos los paneles aunque pertenece al grupo 1.
El grupo 4 contiene un haplotipo de la población 5 y el grupo 5, uno de la población 35 ambas de la Provincia de la Costa del Pacífico (Panamá y Guatemala, respectivamente); y ambos haplotipos con manejo agrícola (Figs. 1C, D, 7).
Centros de diversidad genética
En 12 de las 54 poblaciones se distribuyen solo especies de S. mombim var. mombin, S. mombim var. globosa y/o S. radlkoferi, por lo que éstas no fueron incluidas en el análisis de diversidad por poblaciones y los porcentajes de poblaciones con diferentes haplotipos hacen referencia solo a 44 poblaciones de S. purpurea (Cuadro 2). De estas 44 poblaciones, 69.05% presentan un haplotipo, 21.43% dos haplotipos, 4.76% tres haplotipos, 2.38% cuatro haplotipos y 2.38% cinco haplotipos de S. purpurea. Las poblaciones denominadas con los números 13 (Parque Nacional Palo Verde, Costa Rica), 30 (Amatitlán, Guatemala), 42 (Villa de Ayala, México) y 49 (Aguacate, México) son las que tienen tres o más haplotipos y en conjunto albergan ocho haplotipos únicos. Por lo tanto, deben ser consideradas en acciones de conservación como centros de diversidad genética de la ciruela mexicana.
Las poblaciones silvestres albergan la mayor diversidad genética, seguidas por las poblaciones con manejo agrícola y silvícola (Cuadro 3). En conjunto los datos indican que las poblaciones silvestres tienen todavía una mayor diversidad genética que las que tienen manejo agrícola. En este sentido, es importante conservar las poblaciones 13 (Parque Nacional Palo Verde, Costa Rica), 30 (Amatitlán, Guatemala), 39 (Parque Nacional Cañón Sumidero, México), 47 (La Manzanilla, México), 49 (Aguacate, México) y 53 (Otates y Cantaranas, México) en las que se encuentran los siete haplotipos característicos de poblaciones silvestres (2, 3, 6, 17, 20 y 21) (Cuadros 1, 2).
Cuadro 3: Número de secuencias (Secuencias) y de haplotipos (Hapl.) analizados; y diversidad haplotídica (Hd) y nucleotídica (p) de las provincias biogeográficas y clasificadas de acuerdo a su estatus de manejo en la población. CP=Costa del Pacífico; EN=Eje Neovolcánico; DB=Depresión del Balsas; SMS=Sierra Madre del Sur; GM=Golfo de México; LA=Los Altos de Chiapas; PY=Península de Yucatán. S=silvestre; MS=Manejo silvícola; MA=Manejo agrícola.
| CP | EN | DB | SMS | GM | LA | PY | S | MS | MA | |
| Secuencias | 80 | 6 | 4 | 2 | 2 | 2 | 9 | 37 | 7 | 64 |
| Hapl. | 5 | 1 | 3 | 1 | 1 | 1 | 2 | 3 | 2 | 5 |
| Hd | 0.584 | --- | 0.833 | --- | --- | --- | 0.222 | 0.573 | 0.2857 | 0.471 |
| p | 0.002 | --- | 0.003 | --- | --- | --- | 0.001 | 0.001 | 0.0008 | 0.002 |
Se encontró también que las poblaciones con manejo agrícola que presentan haplotipos que ya no se encuentran en poblaciones silvestres fueron la 42 (Villa de Ayala, México) en la provincia de la Depresión del Balsas y la 48 (Tinúm, México) en la Península de Yucatán. En relación con este hallazgo cabe destacar a la población 42 en la que se observaron cuatro haplotipos que pertenecen además a tres de los cinco grupos identificados en el análisis de contingencia anidada (Cuadro 1, Fig. 1B). Los resultados sugieren que para ambas poblaciones existen linajes únicos en las poblaciones con manejo agrícola que derivan de otros que ya no se encuentran en su forma silvestre o que no han sido registrados en nuestro esfuerzo de muestreo ni en el de Miller y Schaal (2005) y Miller (2008). La conservación de estos haplotipos podría enfocarse a constituir bancos de germoplasma que alberguen representantes de S. purpurea de las diferentes poblaciones con características morfológicas distintivas, así como incentivar y promover el manejo de la especie en los solares o huertos para conservar a la especie in circa situm.
La provincia biogeográfica con una mayor diversidad de haplotipos y nucleotídica fue la Depresión del Balsas, seguida de la Costa del Pacífico y la Península de Yucatán. Las menos diversas fueron el Eje Neovolcánico, la Sierra Madre del Sur, el Golfo de México y Los Altos de Chiapas. Estas últimas contienen un solo haplotipo (Cuadro 3). Aunque se espera que la estimación de la diversidad genética esté subestimada para las provincias biogeográficas por la falta de un muestreo sistemático en algunas de ellas, los estimadores que se obtuvieron en este trabajo nos sugieren que en varias provincias existe una mayor diversificación de esta especie que en otras.
Estos datos nos indican que la Depresión del Balsas debe considerarse como un sitio de diversidad genética de la ciruela donde enfocar los esfuerzos de mejoramiento de este árbol tropical como proponen Jamnadass et al. (2009); sin dejar a un lado los centros de diversidad genética del cultivo de la Costa del Pacífico (Jalisco-Nayarit y Michoacán) y el de la Península de Yucatán, con el objetivo de descentralizar los esfuerzos de conservación y mejoramiento genético que requieren los árboles tropicales (Jamnadass et al., 2009). Las estrategias de conservación in situ del germoplasma en los centros de domesticación pueden ser una táctica que permita un entendimiento del proceso de la domesticación y la diversificación de S. purpurea, así como un manejo integral de este recurso fitogenético. Se requiere realizar un muestreo más extenso, particularmente en las regiones aledañas a las poblaciones identificadas como centro de origen, domesticación y diversidad genética de la ciruela mexicana.
Conclusiones
Los resultados de este estudio sugieren que en S. purpurea los linajes del grupo 2 y 3 son los que han sido introducidos con mayor frecuencia a los sistemas de cultivo, mientras que los del grupo 1 han sido moderadamente cultivados, con la mayor parte de sus representantes en las poblaciones silvestres, por lo que representan un recurso fitogenético importante. La presencia de estos haplotipos en que se encuentran tanto individuos silvestres, como con manejo silvícola y agrícola, sugieren varios eventos de domesticación de linajes particulares, todos ellos ocurridos en dos provincias biogeográficas: 1) Eje Neovolcánico, en su región occidente, y 2) Costa del Pacífico, en las intersecciones de Jalisco-Nayarit-Michoacán y de Guatemala-Nicaragua.
El análisis filogeográfico de las especies del género Spondias sugiere que la historia de domesticación de esta especie es compleja, con un centro de origen en la región occidental de la provincia del Eje Neovolcánico y centros de domesticación en la provincia de la Costa del Pacífico (Jalisco-Nayarit-Michoacán y Guatemala-Nicaragua). Los centros de diversidad genética de S. purpurea son además de los centros de domesticación, la Depresión del Balsas y la Península de Yucatán.











 text new page (beta)
text new page (beta)





