El área con mayor producción de durazno (Prunus persica) y ciruelo (Prunus domestica) en Michoacán, se localiza en el Municipio de Zinapécuaro al noroeste del estado, en las coordenadas 19° 52’ de latitud norte y 100° 50’ de longitud oeste, con una altitud de 2400 a 2700 msnm. El relieve lo constituye el Eje Volcánico Transversal, con clima templado con lluvias en verano. La precipitación pluvial anual promedio es de 622 mm y temperaturas entre los 3 y 34 °C (Villaseñor, 2005). El área productora de durazno y ciruelo se ha extendido en áreas de bosque mixto de pino-encino y abeto-pino, con una superficie sembrada de 1256 ha, que representa el 30 % de la superficie estatal y se concentra en cinco localidades: Ucareo, Jeráhuaro, Huajúmbaro, San Joaquín y La Galera, con una producción anual de 6950 t y un valor de la producción de $41.7 millones de pesos (SAGARPA, 2015). En Michoacán la superficie sembrada de ciruelo en 2014 fue 2827 ha, con una producción de 13,316 t y un valor de producción de 47.1 millones de pesos (SAGARPA, 2014).
En los últimos años, en algunas huertas comerciales, los árboles de durazno y ciruelo han sido afectados por un deterioro progresivo y la muerte paulatina de los mismos, la cual puede ocurrir en meses, presentándose los primeros síntomas durante la floración que pueden ser marchitez en hojas, amarillamiento de la copa del árbol, achaparramiento, cancro basal, exudado en el cuello del árbol, falta de vigor y muerte.
El género Armillaria incluye más de 40 especies, su identificación a partir de los caracteres morfológicos es complicada, debido a que en la mayoría de los casos, solo se encuentran estructuras asexuales o vegetativas, uno de los puntos que se ha discutido ampliamente en la literatura es la falta de estructuras sexuales para realizar la identificación taxonómica clásica (Kim et al., 2006; Brazee et al., 2011; Ross-Davis et al., 2012). En los últimos años, los datos moleculares, específicamente las secuencias de ADN, se están utilizado para identificar las especies de hongos y realizar estudios filogenéticos (Cai et al., 2011; Guo et al., 2016; Koch et al., 2017). En la taxonomía contemporánea de este hongo, las comparaciones de secuencias de ADN han llevado al descubrimiento de una serie de nuevas especies que no están relacionadas con especies morfológicas (Maphosa et al., 2006), lo cual ha tenido implicaciones en la identificación de este organismo.
Las especies del género Armillaria se han detectado en bosques naturales y huertas fructícolas (Cibrián et al., 2007) y se puede comportar como saprófitas o patógenas (Ross-Davis et al., 2012). Aún con el desplante de árboles infectados, el micelio sobrevive como saprófito, las raíces residuales pueden permanecer enterradas en el suelo y sirven como inóculo (Redfern y Filip, 1991). El inóculo puede persistir durante algunos años o décadas (Baumgartner y Rizzo, 2001), esto combinado con la falta de métodos eficientes para prevenir, controlar o curar (Adaskaveg et al., 1999, Aguin-Casal et al., 2006), contribuye a reducir significativamente los rendimientos durante toda la vida de una plantación infectada (Baumgartner, 2004) y la de sucesivas plantaciones.
En México, este hongo se distribuye en los estados de Baja California, Chiapas, Chihuahua, Durango, Estado de México, Guanajuato, Guerrero, Jalisco, Michoacán, Morelos, Nuevo León, Oaxaca, Puebla, Querétaro, Veracruz y en áreas forestales de la Ciudad de México (Valdés et al., 2004; Cibrián et al., 2007; Elías-Román et al., 2013; Klopfenstein et al., 2014; Fernández-Pavía, et al., 2015). El objetivo de este trabajo fue la identificación y caracterización molecular del patógeno asociado a la muerte descendente de los árboles de durazno y ciruelo. La hipótesis de este trabajo se basó en la presencia de Armillaria spp. como responsable de la muerte descendente de árboles de durazno y ciruelo en la zona productora de Zinapécuaro, Michoacán.
MATERIALES Y MÉTODOS
Muestreo
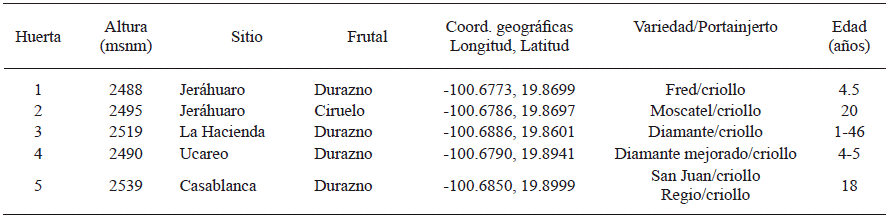
En 2015 y 2016, se realizaron recorridos de campo y se seleccionaron cinco sitios de muestreo en huertas comerciales de durazno y ciruelo, ubicadas en la parte alta, media y baja de la zona productora de Zinapécuaro (Cuadro 1). En cada sitio se identificaron árboles con diversos síntomas de deterioro, como marchitez en hojas, amarillamiento de la copa del árbol, achaparramiento, cancro basal, exudado en el cuello, falta de vigor y muerte. Se realizó un muestreo destructivo para tomar muestras de raíces primarias y secundarias y del tronco a una altura entre 15-30 cm. Se verificó la presencia de signos y síntomas de patógenos en el sistema radicular del árbol, encontrándose en algunos casos rizomorfos, estructura vegetativa típica de Armillaria y presencia de micelio de color blanco-marrón entre la corteza y la madera en forma de abanico. Se tomaron muestras de suelo a una profundidad de 30-60 cm.
Procesamiento de muestras vegetales y de suelo
Las muestras de material vegetal fueron lavadas exhaustivamente con la finalidad de eliminar el suelo, se cortaron segmentos de 1 cm de longitud y se colocaron en hipoclorito de sodio al 1.1 % durante 10 min en agitación suave, posteriormente se sumergieron en alcohol al 70 % y se lavaron con agua destilada estéril entre cada cambio. Los rizomorfos fueron lavados con peróxido de hidrógeno al 3 % por 5 min y dos lavados de agua destilada estéril, secados con toallas de papel estériles de acuerdo a lo descrito por Harrigton et al., (1992). En cajas de petri con medio cultivo selectivo, BDS (Benomyl-Dicloran- Estreptomicina), (Worrall, 1991; Aguín et al., 1998), se sembraron de 15 a 20 trozos de corteza con micelio y rizomorfos de color marrón a negro. Las cajas se cubrieron con papel aluminio y se guardaron dentro de bolsas de polipapel y se incubaron a 20 °C en oscuridad durante 4-5 semanas. Para explorar la población de la micoflora en suelo presente en los sitios de colecta, se utilizó la técnica de dilución de suelo PDA-TS (papa-dextrosa-agar tergitol) (Steiner y Watson 1965). Las cajas se mantuvieron a temperatura de 22-24 °C de 7 a 10 días, que fue el periodo en el que se desarrollaron los hongos. Los aislados se transfirieron a medio agua-agar para purificarlos mediante la técnica de punta de hifa y se sembraron por separado en cajas con medio agar-agua y PDA.
Identificación molecular. Extracción de ADN, amplificación específica y secuenciación
Una vez obtenidos los aislamientos puros de los hongos en estudio, se inició la caracterización molecular. La extracción del ADN se llevó a cabo con el Kit de extracción AxyPrep™ Multisource Genomic DNA Miniprep (Axygen) de acuerdo a las instrucciones del fabricante. Se realizó una PCR utilizando los iniciadores ARMEF-3-A (CGT GAY TTY ATC AAG AAC ATG) y ARMEF-R (TAC CCG TTC GGC GAT CAA TCT) diseñados para la región que amplifica para el gen Tef-1α (translation elongation factor 1α ) (Ross-Davis et al., 2012). Las reacciones de amplificación se realizaron en un termociclador TProfessional estándar (Biometra, Alemania) en un volumen final de 25 µL con los siguientes reactivos: Taq DNA Buffer PCR 1X, MgCl2 1 mM, dNTP’s 0.2mM, Platinum Taq DNA polymerase 1U (Invitrogen, USA), 10 µM de cada primer y 4 µL de ADN total. El programa de amplificación fue el siguiente: 96 °C por 3 min, 35 ciclos de 95 °C por 30 s, 58 °C por 30 s y 72 °C por 45 s, y una extensión final a 72 °C por 4 min. Se verificó la presencia del fragmento mediante electroforesis en geles de agarosa al 2 %, teñidos con bromuro de etidio (5 mg L-1) y los geles se documentaron en el fotodocumentador Gel Doc (Bio-Rad, USA). Los productos de PCR fueron purificados y secuenciados en ambas direcciones por la Compañía Macrogen Inc. (Seúl, Corea del Sur).
Alineamiento múltiple y análisis filogenético
Las secuencias del gen TEF-1α de los aislados fueron editadas y alineadas manualmente con el Software BioEdit 7.2.5 (Hall, 1999). Las secuencias se compararon con las disponibles en la base de datos del Centro Nacional para la Información Biotecnológica (NCBI por sus siglas en inglés) mediante el algoritmo BLAST®.
Para el análisis filogenético, todas las secuencias del gen EF-1α obtenidas de los aislados de Armillaria spp. de Michoacán se agruparon en una sola secuencia consenso. Además, se utilizaron secuencias de aislamientos de 34 especies de Armillaria caracterizadas a nivel mundial de acuerdo a lo reportado por Maphosa et al., (2006), Hasegawa et al., (2010), Ross-Davis et al., (2012), Elías-Román et al., (2013) y Guo et al., (2016). Se utilizó a Pleurotus ostreatus (número de accesión en el NCBI AY883432) como nodo raíz externo al grupo. Todas las secuencias fueron alineadas con el algoritmo CLUSTAL-W, y se analizaron con el método Neighbor-joining (NJ) (Saitou y Nei 1987) utilizando el software Molecular Evolutionary Genetics Analysis (MEGA 7). Se utilizaron 1000 réplicas de valores soporte (Bootstrap) para obtener un árbol consenso con una regla de mayoría del 50 % (Tamura et al., 2004). Se utilizó el modelo de sustitución Parametro-2 de Kimura (K2P) de acuerdo con Hasegawa et al., (2010).
RESULTADOS Y DISCUSIÓN
Identificación morfológica
Las cepas obtenidas de las huertas 1, 2 y 5 (Cuadro 1) provenientes de la corteza de árboles infectados con micelio del hongo, cuyo patrón de crecimiento fue en forma de abanico y los aislados de los rizomorfos obtenidos de las raíces, presentaron un crecimiento en medio de cultivo con aspecto algodonoso de color amarillo cremoso en el centro. Después de 20 días se observó la formación de rizomorfos con crecimiento dicotómico, a los 28 días formación de hifas, características del género Armillaria. No se encontraron en los sitios de estudio la presencia de basidiocarpos, antes, durante y después del periodo de lluvias.
En las muestras de material vegetal de las huertas 3 y 4, no se aisló Armillaria spp. En todos los sitios de muestreo en los aislados del suelo se encontraron los géneros, Trichoderma sp, Pilatoporus sp., Mucor sp., Penicillium sp., Aspergillus sp. y Fomitopsis sp., este último es un patógeno asociado que puede contribuir en la muerte de árboles sujetos a procesos de decaimiento (Cibrián et al., 2007). Además en la huerta 5 se detectó la presencia de Fusarium oxysporum, el cual es un patógeno de la raíz que ocasiona daños en el sistema vascular.
Identificación molecular. Alineamiento múltiple y análisis filogenético
Las secuencias de ADN obtenidas en este estudio no tienen una cobertura e identidad confiable (≥ 99 %) con ninguna de las especies conocidas del género Armillaria reportadas en el NCBI, por lo que aparentemente representan una nueva especie dentro del género. No obstante, las secuencias obtuvieron un 99 % de similaridad con aquellas reportadas como Armillaria spp. por Elías-Román et al., (2013), quienes a partir de aislados provenientes del Estado de México concluyeron que la pudrición de raíces en árboles de durazno es originada por una especie del género Armillaria no descrita. Las secuencias obtenidas en este estudio se depositaron en GenBank con los números de accesión KY611443 a KY611450 y están disponibles al público en general. Éstas secuencias y las reportadas por Elías-Román et al., (2013) corresponden al mismo organismo, el cual representa a especies aún no descritas en la literatura existente.
Basado en el alineamiento de 48 secuencias del gen EF-1a pertenecientes al menos a 34 especies del género Armillaria caracterizadas a nivel mundial, de acuerdo a lo reportado por Maphosa et al., (2006), Hasegawa et al., (2010), Ross-Davis et al., (2012), Elías-Román et al., (2013) y Guo et al., (2016), se realizó el análisis filogenético mediante el algoritmo Neighbor-joining con el cual se obtuvo un árbol consenso de probabilidades superior al 50 %, en el cual las especies de Armillaria se agruparon en cuatro clados principales (Figura 1). El gen EF-1a fue útil para separar a todas las especies. Las secuencias del estudio se agruparon en el mismo clado que las secuencias de la especie Armillaria mellea, pero en ramas diferentes (Figura 1), confirmando que se trata de especies nuevas que al parecer están filogenéticamente relacionadas con A. mellea. Sin embargo, tanto el análisis filogenético, el BLAST y el análisis de secuencias (polimorfismos de un solo nucleótido e inserciones y delecciones) separan claramente a ambos organismos como especies distintas, lo cual coincide con lo reportado por Elías-Román et al., (2013) con los aislados que obtuvieron en el Estado de México.

Figura 1 Filogenia del género Armillaria derivada mediante el algoritmo Neighbor-joining (NJ) inferida a partir de secuencias del gen EF-1α, incluyendo aislamientos colectados en Michoacán México. Una topología similar se obtuvo con ML y MP. Se utilizó a Pleurotus ostreatus como nodo raíz externo al grupo. Los valores de soporte Bootstrap (1000 réplicas) para NJ (≥ 50%) están simbolizados en los internodos con un círculo azul.
En el género Armillaria las especies que se agrupan en el mismo clado que A. mellea y A. ostoyae se asocian o se consideran como patógenos agresivos que regularmente pueden causar enfermedades en diversos árboles frutales y forestales, en comparación con las especies saprófitas o que se comportan como patógenos débiles que se agrupan en los otros clados (Ross-Davis et al., 2012; Guo et al., 2016). En este estudio el análisis filogenético agrupa a las secuencias en el mismo clado que aquellas de A. mellea reportadas para Norteamérica, Asia y Europa.
En un estudio paralelo a este, se calculó la incidencia acumulada de tres años de muerte descendente para la huerta 1, con un valor de 21.34 % (145/679 árboles) (Rivas-Valencia y Fernández-Montes et al. 2017), lo cual puede interpretarse como una epidemia de intensidad importante, la cual es ocasionada por una especie de Armillaria estrechamente relacionada con A. mellea.
La documentación de nuevas especies de Armillaria en México tiene sus antecedentes en lo reportado por Alvarado y Blanchette (1994), quienes en un estudio de las especies nativas de las regiones forestales del centro de México demostraron que algunos aislados obtenidos en el Estado de Puebla fueron incompatibles con cepas de referencia de especies conocidas en Norteamérica, sugiriendo que estos correspondían a una especie diferente. Debido a la falta de secuencias se desconoce si esos aislados corresponden a la misma especie biológica de esta investigación. Sin embargo, con el uso de las herramientas biotecnológicas y específicamente los datos de las secuencias de ADN, se puede identificar la especie de Armillaria, lo cual es fundamental para la evaluación del riesgo de la enfermedad y el manejo de la misma.
CONCLUSIONES
La muerte descendente de árboles de durazno y ciruelo en el municipio de Zinapécuaro, Michoacán está asociada a la presencia del genero Armillaria. Con base en el análisis de las secuencias de ADN se identifican dos aislados para Michoacán, diferentes a los aislados obtenidos en huertas de durazno en el Estado de México. Los aislados de Armillaria en Michoacán y Estado de México están relacionados genéticamente con A. mellea, el cual es considerado un patógeno agresivo. Actualmente, no se tienen identificados portainjertos tolerantes a Armillaria, se recomienda continuar con estudios de búsqueda de resistencia genética y manejo de la enfermedad en las huertas.











 text in
text in