El silenciamiento génico por ácido ribonucleico (ARN) es un sistema de inactivación de genes mediado por moléculas pequeñas de ARN interferentes (RNAi) que guían el corte endonucleolítico y la degradación o represión de la traducción de ARN mensajeros, o bloquean la transcripción (Cuperus et al., 2010). El silenciamiento génico tiene funciones esenciales en el desarrollo, la respuesta al estrés y en la expresión y el mantenimiento del genoma (Ding and Voinnet, 2007). Las funciones y las actividades del silenciamiento génico están conservadas en eucariotas. En plantas, nematodos e insectos, el silenciamiento génico es un componente esencial de la inmunidad antiviral (Ding and Voinnet, 2007).
Las plantas usan el silenciamiento génico para regular la expresión de genes en una manera temporal y específica en órganos y tejidos (Chitwood et al., 2009). Además, las plantas usan el sistema de silenciamiento génico para prevenir, restringir y eliminar infecciones virales (Garcia-Ruiz et al., 2010; Garcia-Ruiz et al., 2015; Ma et al., 2015). En interacciones plantas-virus, el establecimiento de enfermedad o resistencia depende en gran parte del balance entre el silenciamiento génico del virus por parte de la planta y la evasión o supresión de la defensa antiviral por parte del virus (Garcia-Ruiz et al., 2010; Garcia-Ruiz et al., 2015; Ma et al., 2015). El silenciamiento génico contra virus y contra ARN celular tienen varias componentes en común. La mayoría de los genes que participan en el silenciamiento de genes celulares forman parte del silenciamiento de virus (Ding and Voinnet, 2007).
En eucariotas, incluyendo plantas, el silenciamiento génico regula la expresión de genes a nivel transcripcional y post-transcripcional (Figura 1). El silenciamiento génico a nivel transcripcional inhibe la transcripción causando una reducción en la síntesis de ARN mensajero vía la metilación de ADN en cromosomas, plásmidos o minicromosomas virales (Raja et al., 2008; Ceniceros-Ojeda et al., 2016). El silenciamiento génico transcripcional es dirigido por los RNAi que guían la metilación enzimática del ADN (Ding and Voinnet, 2007). El silenciamiento génico a nivel post-transcripcional consiste en el corte endonucleolítico y degradación o la represión de la traducción de ARN mensajeros (Cuperus et al., 2010) (Figura 1). Todos los virus usan ARN para expresar sus genes, para replicarse o ambos. Este ARN es potencialmente percibido por la célula y activa defensas antivirales, tal como el silenciamiento génico (Ivanov et al., 2014). El silenciamiento génico antiviral a nivel transcripcional y pos-transcripcional ha sido demostrado para virus de ADN, virus de ARN, viroides y ARN satelitales (Cuadro 1).
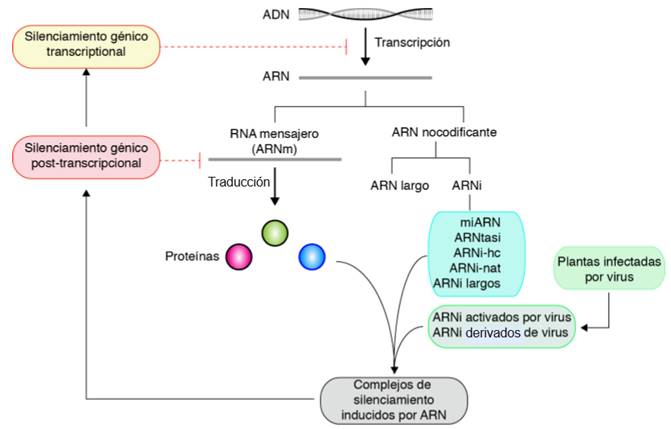
Figura 1. Flujo de información genética y silenciamiento génico en plantas. Complejos de silenciamiento inducido por ARN se forman entre proteínas y ARN interferentes (ARNi) derivados de transcritos que no codifican proteínas. El silenciamiento génico bloquea el flujo de información genética lo cual evita la transcripción, induce la degradación o evita la traducción. Plantas infectadas por virus acumulan ARNi derivados del ARN viral y ARNi celulares activados por la infección viral.
Cuadro 1. Componentes genéticos esenciales del silenciamiento génico con base en combinaciones planta-virus representativas y agrupadas siguiendo la organización genómica del virus.
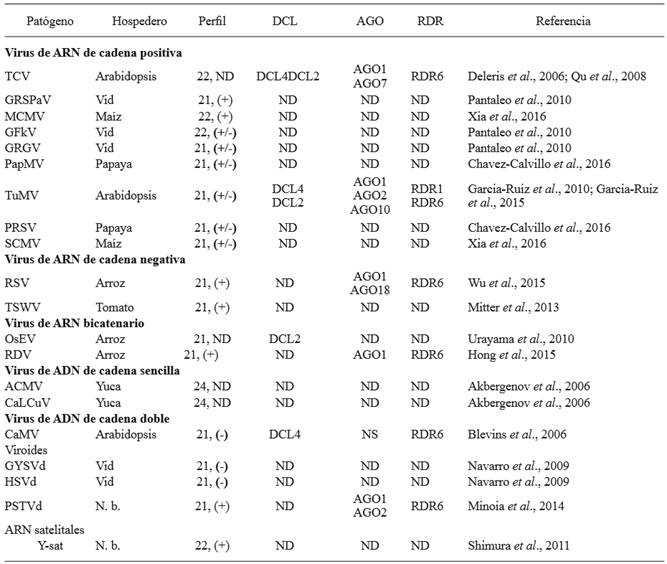
Patógenos. Virus del mosaico de la yuca africana (ACMV), Virus del enrollamiento de la hoja de col (CaLCuV), Virus del mosaico de la coliflor (CaMV), ARN satelital Y del Virus del mosaico del pepino (Y-sat), Virus de la mancha de la vid (GFkV), Virus asociado al globo de la vid roja (GRGV), Virus asociado a la picadura del tallo de la vid rupestre (GRSPaV), Viroide 1 del moteado amarillo de la vid (GYSVd), Viroide del enanismo del lúpulo (HSVd), Virus del moteado clorótico del maíz (MCMV), Endornavirus del arroz (OsEV), Virus del mosaico de la papaya (PapMV), Virus de la mancha anular de la papaya (PRSV), Viroide del tuberculo fusiforme de la papa (PSTVd), Virus del enanismo del arroz (RDV), Virus del rayado del arroz (RSV), Virus del mosaico de la caña de azúcar (SCMV), Virus del bronceado del tomate (TSWV), Virus del encrespado del nabo (TCV), Virus del mosaico del nabo (TuMV)
Hospederos. Arabidopsis (Arabidopsis thaliana), Yuca (Manihot esculenta), Vid (Vitis vinifera), Maíz (Zea mays), N. b. (Nicotiana benthamiana), Papaya (Carica papaya), Arroz (Oryza sativa), Tomate (Solanum lycopersicum)
Perfil de la población de ARNi derivados del virus. Tamaño en nt de los ARNi más abundantes derivados de los virus. Las poblaciones fueron caracterizadas por medio de sequenciacion de nueva generación, geles de ARN, o ambas. (+): Los ARNi de polaridad positiva son más abundantes que los de polaridad negativa. (-): Los ANRi de polaridad negativa son más abundantes que los de polaridad positiva. (+/-): Los ARNi de polaridad positiva y negativa son igualmente abundantes
La infección viral en plantas ocurre en dos fases: Tras entrar en la célula, los virus se replican, después se mueven localmente de una célula a otra y sistemáticamente a otros órganos y tejidos de la planta vía el sistema vascular (Ivanov et al., 2014). El silenciamiento génico dirigido contra ARN viral restringe la replicación del virus y su movimiento (Garcia-Ruiz et al., 2015). La defensa antiviral vía silenciamiento génico depende de la detección y degradación de ARN viral en la células infectadas y de la amplificación de la señal en células lejos del sitio de infección inicial (Ding and Voinnet, 2007).
Los virus modifican el ambiente celular para favorecer su replicación y movimiento dentro de la planta (Ivanov et al., 2014) usando mecanismos que incluyen la subversión, la reprogramación transcripcional y la inducción y supresión del silenciamiento génico (Ding and Voinnet, 2007; Garcia-Ruiz et al., 2015). La supresión del silenciamiento génico es esencial para que los virus se repliquen, establezcan infección y se muevan local y sistemáticamente (Garcia-Ruiz et al., 2015). Una mejor comprensión del mecanismo básico del silenciamiento génico tanto de ARN celular como viral es importante para explicar varios fenómenos que interesan a los fitopatologos, tales como la resistencia contra virus, su movimiento, su rango de hospedantes, su distribución en la planta, el sinergismo viral, las determinantes genéticas de resistencia a virus y la ingeniería genética de plantas resistentes a virus.
Funciones del silenciamiento genico
En eucariotas varios procesos biológicos, como el desarrollo, la formación de órganos y las respuestas al estrés, son regulados por silenciamiento génico (Chitwood et al., 2009). Por ejemplo, en plantas, la polaridad abaxial-adaxial de las hojas está regulada por gradientes de RNAi que se mueven entre células (Chitwood et al., 2009). Las proteínas ARGONAUTA (AGO) son componentes catalíticos del silenciamiento génico. En asociación con RNAi, las proteínas AGO cortan moléculas de ARN mensajero complementario (el blanco) a los ARNi (Carbonell et al., 2012; Cuperus et al., 2010; Cuperus et al., 2011). AGO7 y AGO10 se expresan específicamente en lo primordios de hojas y en los tejidos vasculares. AGO7 restringe la actividad del micro ARN390 (miR390) y dirige un gradiente de ARNi del lado adaxial al lado abaxial de las hojas (Chitwood et al., 2009). El papel antiviral del silenciamiento génico ha sido demostrado en varias especies de plantas (Cuadro 1) y los ARNi están involucrados también en las interacciones entre plantas y bacterias y entre plantas y hongos (Weiberg et al., 2014).
ARN no codificante y pequeños ARN interferentes en plantas
El genoma de las plantas contiene dos tipos de ARN no codificantes (Figura 1): ARN largo no codificante (ARNlnc) y pequeños ARNs interferentes (RNAi). Los ARNlnc se forman via transcripción bipolar y a partir de intrones o regiones intergénicas. Los ARNlnc no miden más de 200 nucleótidos (nt) y han sido implicados en la diferenciación de tejidos, el desarrollo reproductivo, la respuesta a estrés, la vernalización, el tiempo de floración y en la inmunidad de plantas (Shafiq et al., 2016). Los ARNi miden 21 a 24 nucleótidos (nt) se expresan de manera natural y son parte de los mecanismos de regulación temporal y espacial de genes mediante silenciamiento génico. Otros ARNi son producidos en respuesta al estrés biótico (infecciones virales, bacterianas o fungosas) o abiótico (estrés salino, hídrico, por calor o por frio) (Axtell, 2013). Los ARN monocatenarios que forman una estructura doble son precursores de pequeños ARNi: microARNs (miARNs) y otros ARNi. Los ARNs bicatenarios son precursores de ARNi heterocromáticos (ARNi-hc), ARNi antisentido (ARNi-nat) y ARNi secundarios (Figura 1) (Cuperus et al., 2010). Una revisión completa del tema está disponible en el artículo de Axtell (Axtell, 2013).
MicroARNs. Los miARNs juegan papeles críticos en el desarrollo de órganos y tejidos de eucariotas (Cuperus et al., 2011). En general, los miARNs de plantas miden 21 nt y son formados por la endonucleasa Dicer-Like 1 (DCL1) a partir de transcritos formados por la polimerasa II (Cuperus et al., 2011). En Arabidopsis thaliana, la mayoría de los miARNs se asocian con AGO1 mientras los demás miRNAs se asocian con AGO2 o AGO10 (Cuperus et al., 2010; Cuperus et al., 2011). Una alta complementaridad entre el miARN y el ARN mensajero guía el corte endonucleolítico del transcrito por las proteínas AGO y resulta en la degradación o la represión de la traducción del ARN mensajero (Carbonell et al., 2012). Los miARNs involucrados en el desarrollo están altamente conservados entre especies de plantas (Cuperus et al., 2011).
Los ARNs interferentes en trans (ARNtasi). Los ARNtasi regulan transcritos de la misma manera que los miARNs (Cuperus et al., 2010). Durante la biogénesis de los ARNtasi, miARNs específicos en asociación con proteínas AGO facilitan el corte de los transcritos precursores de ARNtasi y reclutan la polimerasa-dependiente de ARN número 6 (RDR6) al transcrito para sintetizar ARN bicatenario. Solamente algunos miARNs participan en la biogénesis de RNAi secundarios mediante amplificación por RDR6. El reclutamiento de RDR6 por los complejos AGO1-miARN necesita la presencia de miARNs que miden 22 nt o de híbridos asimétricos de miARNs/miRNA* que midan 21 nt (Cuperus et al., 2010).
ARN largos interferentes (ARNli). Los ARNli son inducidos por infecciones bacterianas u otras condiciones de estrés. Los ARNli miden de 30 a 40 nt y regulan varios genes de resistencia a bacterias y hongos (Padmanabhan et al., 2009).
ARN interferentes activados por virus. En Arabidopsis thaliana, infección con el Virus del mosaico del pepino (CMV) o el Virus del mosaico del nabo (TuMV) induce la biogénesis de ARNi derivados de aproximadamente 1,000 genes celulares vía un mecanismo que depende de la polimerasa-dependiente de ARN número I (RDR1). Estos ARNi se asocian con AGO1 y AGO2 y probablemente modulan la respuesta a infecciones virales (Cao et al., 2014).
ARN interferentes derivados del virus. Plantas infectadas acumulan ARNi derivados del virus (ARNiv) que miden de 21 a 24 nt y son formados por las proteínas Dicer-Like (DCL). Los ARNiv más abundantes miden 21 y 22 nt y son formados por DCL4 y DCL2, respectivamente (Deleris et al., 2006) (Cuadro 1). Los ARNi derivados del virus regulan la defensa antiviral mediante la inducción del silenciamiento génico transcripcional y posttranscripcional de genes virales (Brosseau and Moffett, 2015; Ceniceros-Ojeda et al., 2016).
Vias del silenciamiento genico antiviral
El silenciamiento génico antiviral inicia en una célula y se mueve a otras células y a otras partes de la planta a través de los plasmodesmos y el sistema vascular (Ding and Voinnet, 2007; Molnar et al., 2010). La vía consiste en cuatro partes: Inicio, degradación, amplificación y movimiento sistémico (Figura 2). Tras detectar el ARN viral, las proteínas DCL forman ARNi derivados del ARN bicatenario viral (Deleris et al., 2006). Durante la etapa de degradación, los ARNi derivados del virus se asocian con las proteínas ARGONAUTAS (AGO) para formar los complejos de silenciamiento inducidos por ARN (RISC) que llevaran a cabo el corte endonucleolítico de ARN monocatenario viral o la represión de la traducción (Brosseau and Moffett, 2015). El reconocimiento inicial del ARN viral es necesaria pero no es suficiente para restringir la infección de la planta por el virus (Garcia-Ruiz et al., 2010). Restricción de la infección viral requiere la amplificación del silenciamiento génico mediante las polimerasas dependientes de ARN (RDRs) (Garcia-Ruiz et al., 2010). Las RDRs celulares forman ARN bicatenario viral en células receptoras para amplificar el silenciamiento y establecer un estado antiviral (Figura 2). Esta amplificación contribuye a la especificidad y la fuerza del silenciamiento génico antiviral y es mediada por RDR1 y RDR6 para formar ARNi secundarios derivados del virus (Cuadro 1). Los virus de ADN expresan sus genes vía ARN mensajero. El silenciamiento génico contra virus de ADN se activa a nivel posttranscripcional resultando en la formación de ARNi que guían la detección del ARN viral. Además, los ARNi derivados del virus en asociación con las proteínas AGO y RDR guían la metilación de ADN viral resultando en el silenciamiento génico transcripcional del minicromosama viral, lo que impide la replicación y el movimiento del virus (Raja et al., 2008; Buchmann et al., 2009; Ceniceros-Ojeda et al., 2016).
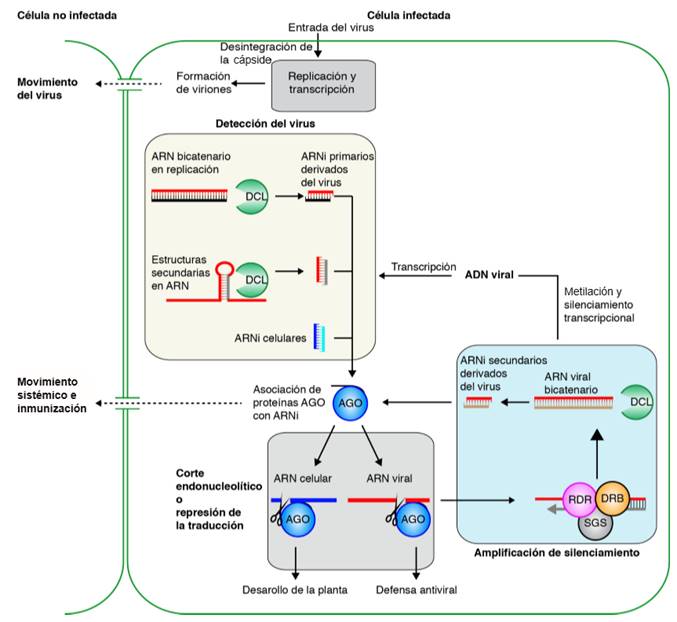
Figura 2. Componentes básicos del silenciamiento génico antiviral en plantas. Proteínas Dicer-like (DCL) cortan ARN bicatenario viral y forman ARNi primarios que se asocian con proteínas ARGONAUTA (AGO) y guían el silenciamiento de ARN viral. El corte endonucleolítico de ARN viral activa la amplificación de silenciamiento génico antiviral y resulta en la formación de ARN viral bicatenario por medio de polymerasas celulares dependientes de ARN (RDR) el cual es procesado por DCL y resulta en la formación de ARNi secundarios derivados del ARN viral. Amplificación de silenciamiento génico activa la metilación de ADN viral, estableciendo silenciamiento génico transcripcional. Los ARNi derivados del virus se mueven de una célula a otra y de manera sistémica, estableciendo un estado de inmunidad en toda la planta.
Poblaciones de ARN interferentes derivados de virus
En varias especies de plantas y virus, las poblaciones de ARNi derivados de virus han sido caracterizadas mediante sequenciación de nueva generación (Cuadro 1). Tres tipos de poblaciones han sido descritos: Poblaciones en las que moléculas de cadena positiva y negativa son igualmente abundantes, y en las que poblaciones de moléculas de cadena positiva son mas abundantes que moléculas de cadena negativa y viceversa. Para algunos virus fitopatógenos de ARN de cadena positiva, los ARNi derivados de virus de polaridad positiva son más abundantes que los de polaridad negativa y hay fragmentos del genoma viral que acumulan más RNAi que otros (Cuadro 1). Este patrón sugiere que las secuencias de ARN viral de cadena positiva, o sus estructuras, son reconocidas por las proteínas DCL. Para la mayoría de combinaciones de virus-plantas, las plantas infectadas acumulan RNAi derivados de virus de la totalidad del genoma viral en cantidades similares para ambas polaridades (Cuadro 1). Resulta muy interesante la observación de que las plantas mutantes que carecen de polimerasas de amplificación RDR1 o RDR6, acumulan cantidades reducidas de RNAi derivados de virus, implicando a las RDRs celulares en su biogénesis (Garcia-Ruiz et al., 2010).
Activacion de silenciamiento genico antiviral
La naturaleza del RNA viral que es reconocido por la célula y que activa el silenciamiento génico antiviral no se ha identificado. Con base en varios análisis genéticos (Cuadro 1), las fuentes de substratos virales para las proteínas DCL se puede dividir en dos grupos: Dependientes o independientes de las RDR celulares (Figura 2). Los RNA bicatenarios virales formados durante la amplificación de silenciamiento génico son dependientes de RDR celulares. Las secuencias auto-complementarias en el ARN genómico viral, productos intermedios de la replicación viral y los productos de transcripción bidireccional son independientes de RDR celulares (Ding y Voinnet, 2007). En general se asume, que estas poblaciones de ARN activan el silenciamiento génico antiviral y las proteínas DLC forman ARNi primarios derivados de virus (Ding y Voinnet, 2007). Alternativamente, o además, el ARN viral podría ser reconocido por represores de traducción que envían el ARN viral para ser procesado por proteínas DCL, o dirigidos a la síntesis de ARN bicatenario por RDR celulares (Luo y Chen, 2007). Un mecanismo de vigilancia también podría estar formado por proteínas AGO cargadas con ARNAi celulares con complementariedad al ARN viral y activar silenciamiento génico antiviral como sucede con los microARNs artificiales dirigidos contra ARN viral (Niu et al., 2006).
Inmunidad antiviral dirigida por ARN interferente
Los ARNi derivados de virus y que se asocian con proteínas AGO determinan la especificidad del silenciamiento génico antiviral guiando represión de la traducción o el corte endonucleolítico de ARN viral con complementariedad a los ARNis (Schuck et al., 2013). En una variación del sistema, en el caso de virus de ADN, los complejos de silenciamiento formados por ARNi derivados de virus y AGO4 guían la metilación de ADN viral, lo cual bloquea la transcripción (Raja et al., 2008). El resultado final es el establecimiento de un estado de resistencia viral que inhibe la replicación y movimiento virus.
En las interacciones virus-plantas, varios fenómenos pueden ser explicados con base en silenciamiento génico antiviral, tales como la protección cruzada, recuperación de síntomas, sinergismo y la resistencia de no hospedantes. La protección cruzada ocurre cuando la infección por una cepa de virus previene la infección posterior por un segundo virus de la misma especie o de una especie estrechamente relacionada (Kung et al., 2014). Aunque no todos los casos de protección cruzada se han explicado, de acuerdo con el modelo de silenciamiento génico antiviral, los ARNAi derivados del virus que causa la primera infección dirigen el corte o represión de la traducción de virus similares al intentar establecer una segunda infección, creando así un estado de inmunidad (Kung et al., 2014). La recuperación de los síntomas se ha descrito para varias combinaciones de virus-planta. Las plantas infectadas acumulan altos cantidades de virus y desarrollan síntomas visibles. Sin embargo, las hojas superiores de dichas plantas contienen bajas cantidades de virus y no desarrollan síntomas. Los ARNi derivados del virus y formados en la parte inferior de la planta se mueven sistémicamente al meristemo para guiar el corte o represión de la traducción de ARN viral lo cual evita la invasión del meristemo por el virus. Las hojas formadas después de estos eventos acumulan bajas cantidades de virus y no presentan síntomas (Ma et al., 2015).
El sinergismo viral se produce cuando una infección simultanea por dos virus resulta en una enfermedad con síntomas más severos que infección por un solo virus (Xia et al., 2016). La mayor parte de los casos documentados de sinergismo incluyen a los virus del género Potyvirus y dependen de los supresores de silenciamiento HC-Pro o P1 (Chávez-Calvillo et al, 2016; Xia et al., 2016). El supresor HC-Pro se asocia tanto con los ARNi celulares como con los ARNi derivados de virus (Garcia-Ruiz et al., 2015). Basado en el modelo de silenciamiento génico (Figura 2), los supresores del silenciamiento génico se unen a los ARNAi derivados de ambos virus presentes en la misma planta, bloqueando tanto el silenciamiento génico antiviral como el silenciamiento de genes celulares, lo cual resulta en síntomas severos, así como en mayor acumulación de los virus que acompañan al potyvirus (Xia et al., 2016).
Varios mecanismos que explican la resistencia de no hospedante, tales como silenciamiento génico antiviral y determinantes genéticos del hospedante necesarios para la replicación o movimiento de virus (Ivanov et al., 2014). Silenciamiento génico antiviral dirigido por ARNi celulares o activados por infecciones virales pueden ser responsables de la resistencia de no hospedante (Ding y Voinnet, 2007).
Supresion de silenciamiento genico por virus
Con el fin de replicarse y moverse de célula a célula y sistémicamente, los virus de plantas deben protegerse de las respuestas de defensa antivirales, incluyendo el silenciamiento génico. Se han descrito al menos dos mecanismos: la evasión y la supresión de silenciamiento génico. Los virus de ARN de cadena positiva se replican en vesículas membranosas que contienen ARN bicatenario que se forma durante la replicación. Los virus de ARN de cadena negativa y los virus de ARN bicatenario se replican en el núcleo o en vesículas membranosas. Estas estructuras pueden proteger el ARN viral del silenciamiento génico (Laliberté y Sanfacon, 2010).
El genoma de la mayoría de los virus de plantas y algunos virus de insectos codifica supresores que inactivan el silenciamiento génico mediante múltiples mecanismos, tales como la inhibición de la biogénesis de los ARNi, asociación con ARNi derivados de virus, degradación de proteínas AGO y DCL, o bloqueando la amplificación del silenciamiento (Cuadro 2). Los supresores virales de silenciamiento génico también se asocian con miARNAs y otros ARNi celulares. Del mismo modo, efectos sobre AGO, DCL, RDR y sus proteínas accesorias afecta la biogénesis y la actividad de los miARNs y ARNi celulares (Chapman et al., 2004). El silenciamiento génico regula el desarrollo de plantas. Por lo tanto, la perturbación de las rutas de silenciamiento génico conduce a cambios en la expresión génica del hospedante y es, al menos en parte, responsable del desarrollo de síntomas visibles en plantas infectadas por virus (Chapman et al., 2004). Dado que el silenciamiento génico antiviral resulta en la metilación de minicromosomas de virus de ADN, los geminivirus codifican proteínas supresoras que interfieren con la metilación del ADN (Cuadro 2).
Cuadro 2. Ejemplos representativos de proteínas virales supresoras de silenciamiento génico organizados con base en su mecanismo de actividad.
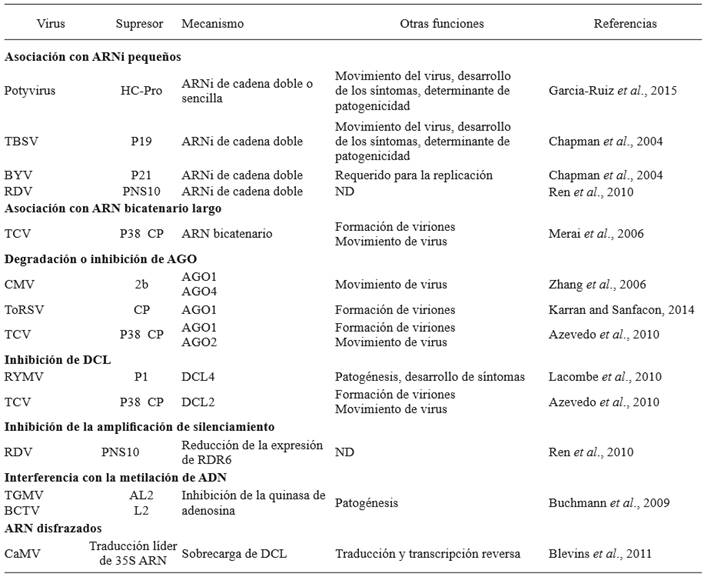
Virus. Virus del rizado de la remolacha azucarera (BCTV), Virus del amarillamiento de la remolacha azucarera (BYV), Virus del mosaico de la coliflor (CaMV), Virus del mosaico del pepino (CMV), Phytoreovirus del enanismo del arroz (RDV), Virus del moteado amarillo del arroz (RYMV), Virus del enanismo arbustivo del tomate (TBSV), Virus mosaico dorado del tomate (TGMV), Virus de la mancha anular del tomate (ToRSV), Virus del encrespado del nabo (TCV)
Ingenieria genetica de resistencia a virus
Para algunas combinaciones de virus-planta, fuentes de resistencia genética se han identificado e introducido mediante mejoramiento en variedades comerciales. Sin embargo, para muchas combinaciones de planta-virus, resistencia genética natural no ha sido identificada (Mehta et al., 2013; Cruz et al., 2014). Varios enfoques basados en el silenciamiento génico se han utilizado para obtener resistencia a virus en plantas utilizando transgenes que expresan ARN bicatenario, ARN monocatenario de cadena positiva o negativa, microARNs artificiales, o ARN interferentes en trans artificiales (Figura 3 y Cuadro 3). Las secuencias derivadas del virus se integran en cassettes de ADN bajo el control de un promotor constitutivo o específico de algún tejido. El transcrito forma ARN bicatenario que es procesado por proteínas DCL formando ARNi que programan el corte o represión de traducción de RNA viral. Estas plantas muestran un fenotipo que varía de inmunidad a tolerancia. Las fragmentos virales mas usados son la proteína de la cápside viral, la replicasa, los supresores de silenciamiento, o el sitio de iniciación de la replicación en geminivirus (Cuadro 3).
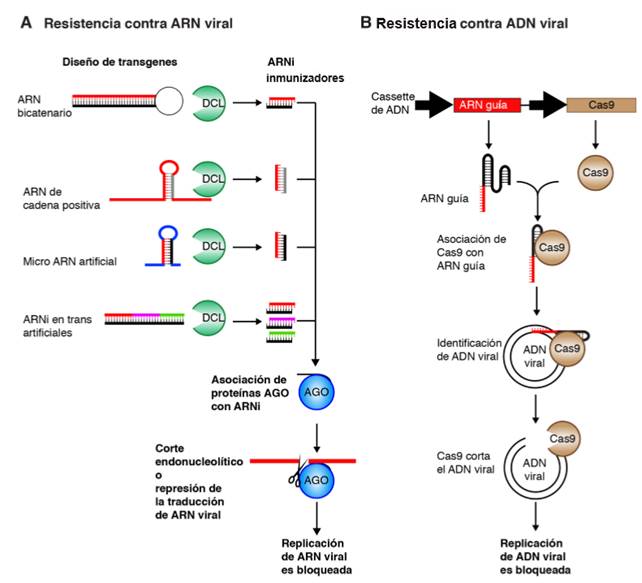
Figura 3. Ingeniería genética de resistencia antiviral en plantas por medio de silenciamiento génico o edición del genoma. A. Producción de ARNi inmunizadores activada por transgenes de origen viral que forma ARN bicatenario y es procesado por proteínas DCL. B. Diseño de resistencia antiviral contra virus de ADN usando el sistema CRISPR/Cas9. La proteína Cas9 se asocia con ARN guía (20 a 24 nt) y de manera específica corta ADN, evitando la replicación de virus de ADN.
Cuadro 3. Ejemplos representativos de plantas resistentes a virus desarrolladas mediante silenciamiento génico o edición del genoma. Los ejemplos están agrupados con base en el diseño del transgen.
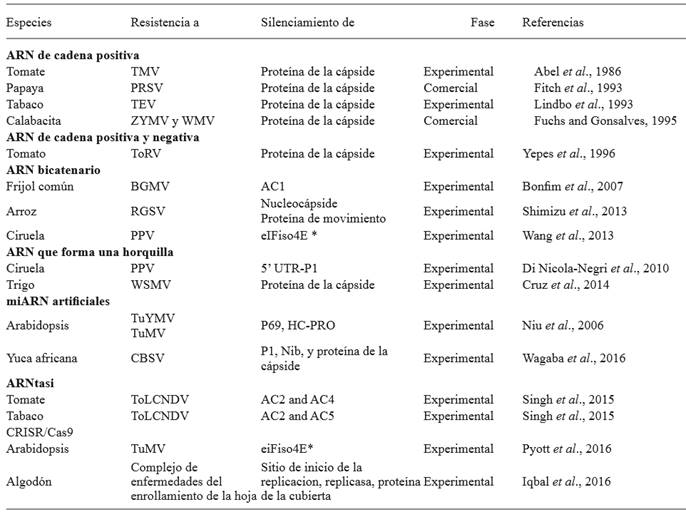
Virus. Virus del mosaico dorado del frijol (BGMV), Virus del rayado marrón de la yuca africana (CBSV), Virus del mosaico del pepino (CMV), Virus de la mancha anular de la papaya (PRSV), Virus de la sharka de la ciruela (PPV), Virus del enanismo del arroz (RGSV), Virus del grabado del tabaco (TEV), Virus del mosaico del tabaco (TMV), Virus de Nueva Delhi del rizado de la hoja del tomate (ToLCNDV), Virus de la mancha anular del tomate (ToRV), Virus del mosaico del nabo (TuMV), Virus del mosaico amarillo del nabo (TuYMV), Virus del mosaico de la sandia (WMV), Virus del mosaico estriado del trigo (WSMV), Virus del mosaico amarillo de la calabaza (ZYMV).
Complejo de enfermedades del enrollamiento de la hoja del algodón: Virus del enrollamiento de la hoja de algodón de Alabad (CLCuAlV), Virus del enrollamiento de la hoja de algodón de Bangalore (CLCuBaV), Virus del enrollamiento de la hoja de algodón de Kokhran (CLCuKoV), CLCuKoV-Bu(cepa de Burewala), Virus del enrollamiento de la hoja del algodón de Multan (CLCuMuV) y Virus del enrollamiento de la hoja del algodón (CLCuRaV)
Hospederos. Arabidopsis (Arabidopsis thaliana), Yuca africana (Manihot esculenta), Frijol comun (Phaseolus vulgaris), Algodón (Gossypium hirsutum), Papaya (Carica papaya), Ciruela (Prunus spp), Arroz (Oryza sativa), Calabacita (Cucurbita pepo), Tabaco (Nicotiana tabacum), Tomate (Solanum lycopersicum), Trigo (Triticum aestivum L.)
Gene silenciado. Nombre de los genes virales o de la planta (*) sometidos a silenciamiento génico o edición del genoma
Las repeticiones palindrómicas cortas agrupadas y regularmente interespaciadas (CRISPR) son segmentos de ADN bacteriano que contienen repeticiones cortas separadas por una secuencia espaciadora. Los CRISPR se derivan de virus bacteriófagos, o plásmido de ADN, integrado en el genoma bacteriano, y son procesados por nucleasas Cas para formar guías de ARN pequeño. La nucleasa Cas9 se asocia con RNAs guía y específicamente corta ADN con secuencia complementaria al ARN guía. Este sistema proporciona inmunidad antiviral en bacterias (Horvath y Barrangou, 2010) y ha sido adaptado para diseñar la herramienta de edición de genomas más poderosa y disponible en la actualidad con un sin fín de aplicaciones en la genómica funcional de eucariotas, incluyendo humanos, insectos, nematodos, hongos y plantas (Ali et al., 2015). El sistema CRISPR/Cas9 se ha utilizado recientemente para diseñar resistencia antiviral contra geminivirus (Cuadro 3) y será parte de los esfuerzos actuales para identificar o validar los genes de susceptibilidad en plantas, dirigiendo la ingeniería genética de resistencia antiviral sin necesidad de hacer plantas transgénicas.
Retos futuros
Los esfuerzos de investigación se centran actualmente en la identificación de los componentes genéticos del sistema de silenciamiento génico, así como en la identificación y caracterización de ARNs y proteínas virales supresoras de silenciamiento. Se espera que en un futuro cercano se definan los mecanismos básicos responsables de la detección inicial de ARN viral por la célula, los determinantes genéticos y el mecanismo de amplificación del silenciamiento génico, corte y degradación o represión de traducción de ARN viral, y los roles de los ARNi derivados de virus en la reprogramación transcripcional de las células infectadas. Un reto especial para los fitopatólogos es traducir el conocimiento básico en aplicaciones prácticas.











 text in
text in 


