Services on Demand
Journal
Article
Indicators
Related links
Share
Cuicuilco
Print version ISSN 0185-1659
Cuicuilco vol.20 n.58 México Sep./Dec. 2013
Dossier: antropología genética
Diversidad genómica en México. Pasado indígena y mestizaje
Andrés Moreno y Karla Sandoval
Departamento de Genética, Universidad de Stanford, California.
Resumen
El pasado indígena de México no sólo ha quedado preservado en los registros históricos, las ciudades precolombinas y la gran diversidad étnica y lingüística actual, también ha quedado impreso en nuestros genes. Los avances en genómica y el acelerado desarrollo de nuevas tecnologías de genotipado y secuenciación masiva han revolucionado la capacidad de analizar los genes e incluso genomas completos. Gracias al esfuerzo conjunto de investigadores nacionales y extranjeros hemos realizado la caracterización genómica más detallada que jamás se haya obtenido de la población mexicana. Analizando más de 100 000 posiciones genómicas en cerca de 500 individuos de 20 grupos étnicos, determinamos a fina escala la estructura de la población indígena y evaluamos su impacto en la población mestiza de diferentes regiones del país. Los resultados revelan que existe una correlación geográfica entre los componentes indígenas de las diferentes etnias y su respectiva contribución en la población mestiza de cada región. Las diferencias de ancestría entre individuos corresponden a una variación continua, mas no a una diferencia cualitativa, lo que cuestiona la distinción categórica entre indígena y mestizo. La autodeterminación responde a criterios socioculturales, mas no genéticos, por lo que la dicotomía conceptual que ha alimentado la segregación entre indígenas y mestizos carece de sustento a la luz de la genómica, dejando claro que los genes no apoyan las diferencias que socialmente se han fomentado entre los grupos humanos.
Palabras clave: Genoma humano, ancestría genética, polimorfismo, haplotipo, estructura poblacional, mestizaje, diversidad indígena, historia evolutiva.
Abstract
Mexico's indigenous past is preserved not only in historical records, pre-Columbian cities, and its current ethnic and linguistic diversity, but also in their genes. The advances in genomics and the accelerated development of new sequencing and genotyping technologies have revolutionized our capacity to analyze genes and even full genomes. Through a joint effort involving Mexican and International research groups, we have conducted the most detailed genomic survey of the Mexican people to date. By analyzing more than 100 000 genomic positions in nearly 500 individuals from 20 diverse Mexican ethnic groups, we have determined the fine-scale population structure of Native Mexicans and evaluated its impact into the composition of admixed Mexican genomes across the country. We found a close relationship between the different indigenous components and their proportional contribution into the admixed population, indicating that ancestral lineages have been retained with great geographical detail among different mestizo subpopulations. The variation in ancestry proportions between individuals are the result of a clinal pattern, rather than discrete differences, which questions the categorical distinction between indigenous and the so-called mestizos. Membership and self-identification must follow social and cultural criteria, but not genetic ones. Thus the use of such dichotomous terminology, which has historically reinforced social segregation between indigenous and mestizo, lacks genetic support; reassuring that genes do not support the social differences created among human population groups.
Keywords: Human genome, genetic ancestry, polymorphism, haplotype, population structure, admixture, indigenous diversity, evolutionary history.
Los mexicanos somos complicados con nuestra historia: hemos
aprendido a sentirnos orgullosos herederos de los pueblos prehispánicos
—algo que no ocurre en otras partes de América Latina—, pero
amamos más a los indígenas muertos que a los vivos.
Jorge Volpi
México es...: las mejores fotografías de mexicanismo.
El concepto de genoma se está volviendo cada vez menos enigmático y poco a poco más común en el vocabulario habitual del mundo académico y de la sociedad en general. Hace ya más de 10 años que se dio a conocer el primer ensamblaje de una secuencia completa del genoma humano, la cual ha servido de referencia y punto de partida para múltiples investigaciones, y desde entonces la palabra genoma ha inundado las comunicaciones científicas y de divulgación en prácticamente todos los ámbitos de la biología y la medicina en todo el mundo. Gracias a este impulso las tecnologías para analizar el ADN también han avanzado explosivamente y su disponibilidad se ha expandido como nunca antes. Las plataformas de genotipado masivo y de secuenciación se han convertido en herramientas prácticamente indispensables en todo tipo de laboratorios, incluyendo instituciones académicas, hospitales, compañías farmacéuticas, laboratorios clínicos y forenses, etc. Incluso sus costos se han reducido de manera tan dramática que la generación de datos genéticos se ha popularizado enormemente entre los grupos de investigación de todos los niveles. De hecho, en ocasiones la cantidad de datos genéticos disponibles supera nuestra capacidad para analizarlos y extraer conclusiones significativas que nos ayuden a entender mejor la diversidad humana a la luz de la genómica. En otras palabras, está claro que hoy en día el genoma está en boca de todos. Pero entonces surge de inmediato la pregunta: ¿qué hemos aprendido en nuestra sociedad gracias a la llegada de los datos genómicos?, ¿qué se sabe en México acerca de la composición genómica de su población? Y, ¿qué nos falta entonces por descubrir? Éstas son algunas de las cuestiones que abordaremos en las siguientes páginas, en las que además discutiremos el posible impacto de esta nueva fuente de información en la sociedad y en nuestra cotidianidad.
Los genes y la identidad actual mexicana
Hablar del genoma en México es, inevitablemente, hablar de su pasado y de la sociedad heterogénea que en la actualidad lo compone. La historia ha dejado múltiples registros de tipo material, arqueológico, osteológico, lingüístico y oral con los que ha sido posible revelar nuestros orígenes. Pero el paso del tiempo también ha dejado un importante registro biológico en los genes, y es precisamente a través de la información genética transmitida de generación en generación que podemos contribuir a la reconstrucción de nuestra historia y de la composición actual de la población. El pasado indígena de México es tan palpable y omnipresente que prácticamente forma parte de un subconsciente y de una realidad que no se cuestiona más. Es evidente que el pasado prehispánico de México está aún presente tanto en su gran diversidad cultural, lingüística y gastronómica como en su legado arqueológico y monumental. Sin embargo, al tratarse de nuestra identidad cultural suele formarse una idea generalizada que engloba todo este legado en un solo concepto: el de nuestra ancestría indígena, así sin más; ¿y cómo podría ser de otra manera si los detalles genealógicos se pierden rápidamente al intentar remontarnos unas cuantas generaciones atrás? La mayoría de los mexicanos sólo sabemos que parte de nuestra sangre es de origen indígena pero desconocemos los detalles de nuestro linaje, casi como si se tratara de algo tan distante o ajeno que la conexión pareciera meramente histórica en vez de algo contemporáneo. Tan es así, que la gran mayoría de la población actual no se considera indígena. La generalización del concepto "mestizo" (dejando de lado la discusión de si éste es o no un término adecuado) ha hecho que los detalles de nuestra ancestría indígena queden diluidos en un concepto que explícitamente significa mezcla. De manera complementaria, podríamos pensar que sólo dentro de las comunidades indígenas actuales sería posible trazar directamente dichos linajes nativos, dado que existe un mayor vínculo con sus antepasados, pues han conservado sus lenguas, tradiciones e incluso registros históricos y orales que dan cuenta de ello. Más adelante regresaremos a este punto para discutir si esto sería efectivamente una facultad exclusiva de la población indígena. Por ahora mencionemos que, más allá de las diferencias culturales y lingüísticas, la distinción entre la población mestiza e indígena también ha estado marcada históricamente por una gran diferencia socioeconómica y un alto grado de marginación que ha prevalecido durante la historia moderna de México. Esta situación y la dinámica demográfica de México durante los últimos 500 años (que efectivamente ha involucrado un alto grado de mezcla entre diferentes componentes ancestrales) han promovido una dicotomía conceptual entre quienes somos resultado de dicha mezcla, digamos por ahora "mestizos", y quienes son o se identifican como indígenas.
México es a todas luces un país donde prevalece el mestizaje, principalmente entre linajes indígenas y europeos, pero también de origen africano a raíz del intercambio de esclavos en tiempos coloniales [Bryc et al., 2010]. Sin embargo, a diferencia de otras regiones del mundo donde el colonialismo europeo dio lugar a una disminución significativa (o en algunos casos total) de la población nativa, en México la población indígena no sólo contribuyó en grandes proporciones al proceso de mestizaje, sino que actualmente constituye un segmento importante del total de habitantes. Según los censos más recientes [INEGI, 2010], basados principalmente en el número de habitantes que hablan alguna lengua indígena, alrededor de 10 % de la población mexicana pertenece a algún grupo indígena. Esto significa que al menos más de 10 millones de mexicanos pertenecen a alguna de las 62 etnias reconocidas oficialmente. Este hecho es promovido como motivo de orgullo nacional y estandarte político (y turístico) de la pluralidad y diversidad que México conserva. Pero también significa que la mayoría de los mexicanos (aproximadamente 90 %>) no son identificados como parte de esta diversidad autóctona, sino como mestizos. Esto quiere decir que es precisamente la mezcla y la riqueza de esta diversidad la que constituye la fuente de identidad para la mayoría de la población.
¿Esto quiere decir, entonces, que la causa de que no podamos descifrar el mosaico de nuestros componentes ancestrales es el alto grado de mezcla de la mayoría de la población? Y aquí no nos referimos a la respuesta que ya hemos venido enunciando acerca de nuestro pasado, en el que se combinaron nuestras raíces indígenas con raíces europeas y africanas, lo que hacemos, más bien, es cuestionarnos si con ayuda de herramientas modernas podemos o no descifrar los detalles de cada una de estas raíces. En realidad, si tuviéramos la suerte de contar con un árbol genealógico familiar muy detallado, tal vez lo podríamos hacer, pero, ¿y a nivel poblacional qué podemos decir más allá del conocido pasado indígena de México? y, ¿qué implicaciones tendría esto en la percepción global de la sociedad y de nuestra propia identidad? Al final de este artículo intentaremos dar respuesta a estas preguntas y veremos que es precisamente aquí donde el estudio de los genes y las herramientas genómicas desempeñan un papel importante y nos pueden ayudar a iluminar el camino.
Genética clásica antes de la era genómica
Las primeras respuestas a este tipo de preguntas han venido del análisis de pequeñas porciones del genoma, ya sea a través de marcadores genéticos en los cromosomas sexuales o de marcadores sanguíneos con electroforesis de proteínas [Lisker et al., 1965 y 1967; Córdova et al., 1967], mucho antes de la llegada de la llamada "era genómica". Por definición, el genoma es el conjunto del material hereditario de un individuo y, en consecuencia, un estudio genómico es aquel que considera la información genética proveniente de todos sus cromosomas. Sin embargo, antes de que las tecnologías hicieran posible el análisis sistemático de todo el genoma, los estudios clásicos de genética de poblaciones se centraron durante mucho tiempo en el análisis de los llamados marcadores uniparentales. Llamados así porque sólo se transmiten por vía materna o paterna, y se refieren a variantes genéticas localizadas en el ADN mitocondrial y en el cromosoma "Y", respectivamente. Éstos han sido ampliamente empleados durante años gracias a su reducido costo de análisis y a la gran utilidad de la información que aportan, y dado que sólo se transmiten uniparentalmente, es posible trazar la procedencia de los linajes maternos y paternos de la población. Desde su descubrimiento y aplicación, México ha gozado de una importante trayectoria en estudios dedicados al análisis de este tipo de marcadores [Rangel et al., 2008; Rubí et al., 2009; Sandoval et al., 2009 y Martínez et al., 2012, entre otros]. Mediante el análisis de unas cuantas mutaciones, ya sea en el ADN mitocondrial (ADNmt) o en el cromosoma Y, los investigadores pueden clasificar a los individuos de una población en grupos que comparten ciertas mutaciones. Dichos grupos se conocen como haplogrupos. Su distribución geográfica también ha sido ampliamente estudiada, de manera que es posible asignar el origen geográfico de los ancestros de un individuo por su haplogrupo. Así pues, diversos investigadores en México han descrito que existe un predominio de haplogrupos de origen europeo en los linajes paternos de la población mexicana y un predominio de haplogrupos de origen nativo en los linajes maternos [Mendizábal et al., 2008 y Sandoval et al., 2012]. Esto nos habla de que el proceso de mestizaje ha sido asimétrico, es decir, la mayoría de los padres que han dejado descendencia hasta nuestros días son de origen europeo, mientras que la mayoría de las madres son de origen indígena.1 Sin embargo, esto nos dice muy poco acerca de lo que ocurre en el resto del genoma, ya que cada segmento cromosómico se comporta como un linaje independiente debido al proceso conocido como recombinación (el cual no ocurre en la mayoría del cromosoma Y ni en el ADN mitocondrial, lo que hace posible trazar "sin interrupciones" su origen ancestral). De este modo, si por ejemplo la ascendencia indígena de un individuo se remonta por vía materna a muchas generaciones atrás, su ADN mitocondrial seguirá perteneciendo a un haplogrupo nativo, pero no necesariamente la ancestría del resto de su genoma; más bien, las proporciones de su ancestría global estarán dadas por el origen de sus ancestros más recientes. Es aquí, entonces, donde es sumamente valioso contar con la información del genoma completo para definir los detalles de la composición genética de un individuo y de la población.
Genética moderna durante la era genómica
Actualmente existen varios métodos de genotipado que obtienen información a lo largo de todo el genoma, cuyos detalles técnicos escapan al espacio y al objetivo de este artículo, sin embargo, creemos que es importante mencionar y comparar sus características principales. El objetivo común de estas técnicas es analizar múltiples posiciones a lo largo de todo el genoma simultáneamente para conocer la combinación de variantes (o alelos) de un individuo en una determinada posición genómica. Esto se conoce como genotipo, de allí la acción de "genotipar" a un individuo. Actualmente existen dos grandes grupos de técnicas para llevar a cabo este proceso a gran escala: el genotipado masivo y la secuenciación. En el primer caso se parte de un catálogo de posiciones genómicas que previamente se han reportado como polimórficas (que varían) dentro de una determinada población y, por lo tanto, se asume que es probable que también sean polimórficas en nuestra población de estudio. Recordemos que la mayoría —99.9 %— de la secuencia de cualquier par de genomas humanos es idéntica, con lo cual, interrogar sólo aquellas posiciones que de antemano se sabe que varían significa un gran ahorro de recursos. La cantidad de polimorfismos de un solo nucleótido (o SNP por sus siglas en inglés), que las técnicas de genotipado masivo han logrado incluir en un solo ensayo, han cambiado drásticamente durante los últimos 10 años. La automatización por medio de ensayos multiplex y de microarreglos ha permitido migrar desde 48 SNPS (como en las primeras versiones de SNPlex), pasando por unos cuantos miles (como en la versión 10K de los microarreglos de Affymetrix), hasta varios cientos de miles o incluso un par de millones de SNPS (como en las versiones más modernas de los microarreglos de Affymetrix e Illumina). En el segundo caso (la secuenciación) no se parte de una lista prediseñada de posiciones a analizar, sino que se determina nucleótido a nucleótido la secuencia completa de un segmento genómico. La secuenciación no es un método nuevo, de hecho es anterior a la llegada de los microarreglos de alta densidad. Lo que ha cambiado es la automatización de las técnicas de secuenciación y su capacidad para generar un creciente número de lecturas, de allí que se hable de técnicas de secuenciación de nueva generación; éstas producen tantas lecturas de diversos fragmentos genómicos (del orden de cientos de millones dependiendo de la plataforma específica) que, aunque sean cortos, alcanzan a cubrir la totalidad de la extensión del genoma humano varias veces. Este número de veces es lo que llamamos cobertura o profundidad (por ejemplo 2x, 10x, etc.). A diferencia del genotipado masivo, la secuenciación reporta todas las variantes del genoma de un individuo y, por lo tanto, arroja mucha más información; sin embargo, a pesar del desplome de sus costos en los últimos años, sigue siendo un método significativamente más caro que los microarreglos comerciales. Una solución intermedia ha sido el diseño de librerías de secuenciación dirigidas a una fracción del genoma, por ejemplo, secuenciar sólo las regiones que codifican para proteínas, conocidas como exoma, y que representan menos del 5 % del total del genoma. En la tabla 1 resumimos las características de los métodos más comúnmente utilizados en la actualidad; y en la figura 1 ilustramos de manera esquemática la cobertura que alcanza cada método a lo largo del genoma.

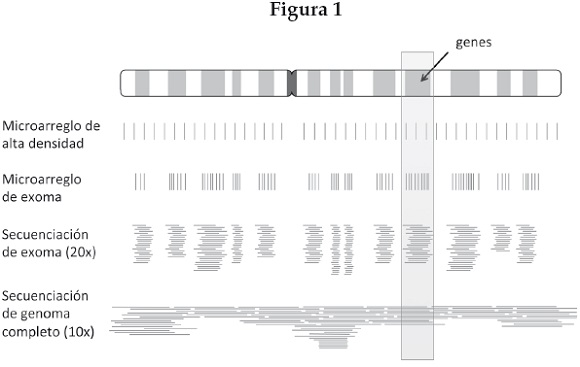
Genómica humana en México
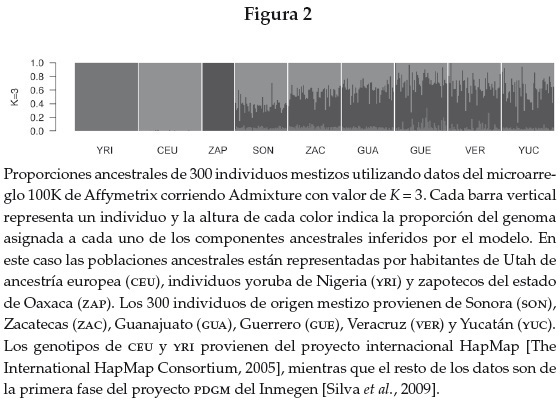
La utilización de estos métodos en México se encuentra en aumento y es cada vez más común ver este tipo de datos en laboratorios de investigación nacionales. Uno de los esfuerzos pioneros en la caracterización de la población mexicana por medio del genotipado masivo de SNPS ha sido el Proyecto de Diversidad Genómica de Poblaciones Mexicanas (PDGM) del Instituto Nacional de Medicina Genómica (INMEGEN). Durante el curso de dicho proyecto se han colectado cientos de muestras de participantes en diversos estados del país que han sido genotipadas utilizando una combinación de varios microarreglos de alta densidad, incluyendo Affymetrix 100K, 500K e Illumina 550K. En su primera fase se analizaron alrededor de 100 000 SNPS, incluidos en el microarreglo 100K de Affymetrix con el que fueron genotipados 300 individuos de origen mestizo provenientes de seis estados de la república, cubriendo sus principales áreas geográficas,2 así como 30 individuos de origen indígena muestreados en una comunidad zapoteca de Oaxaca [Silva et al., 2009]. Los resultados de dicho estudio mostraron que el promedio de ancestría indígena entre los individuos muestreados es de 53 %>, mientras que 42 %> de su genoma y el restante 5 %> son de origen europeo y africano, respectivamente. Sin embargo, de manera individual, los valores de ancestría de la población varían a lo largo de un rango amplio, desde individuos con casi total ancestría indígena hasta otros con casi total ancestría europea. El patrón de distribución de dichos valores no es aleatorio: los individuos muestreados en los estados del norte del país, por ejemplo Sonora, tienden a mostrar valores más altos de ancestría europea, mientras que aquellos muestreados en estados del sur, como Guerrero, tienen mayor ancestría indígena. Por su parte, los valores de ancestría africana, a pesar de ser constantemente menores, presentan excepciones que sólo se observaron en estados costeros como Guerrero y Veracruz, donde algunos individuos tienen hasta 30 % de ancestría africana. Para calcular los valores de ancestría los autores analizaron un subconjunto de 1 814 marcadores informativos de ancestría (AIMS por sus siglas en inglés). Para el presente trabajo reproducimos un análisis similar empleando un algoritmo análogo al del programa utilizado por Silva Zolezzi [2009] —Structure [Pritchard et al., 2000]—, pero diseñado para la manipulación de un número mayor de marcadores —Admixture [Alexander et al., 2009]—, lo que nos permitió utilizar el set completo de 102166 SNPS del microarreglo 100K, cuyos genotipos se encuentran disponibles en la siguiente dirección electrónica: <ftp://ftp.inmegen.gob.mx>. En la figura 2 mostramos las proporciones del genoma de cada individuo provenientes de cada ancestría, asumiendo un modelo en el que cada individuo (o fracciones de su genoma) puede pertenecer a un máximo de K componentes ancestrales (en este caso K = 3), representados por habitantes de Utah de ancestría europea (CEU), individuos del grupo yoruba de Nigeria (YRI) y zapotecos del estado de Oaxaca (ZAP). En este tipo de gráficos cada barra representa un individuo cuyos componentes ancestrales se muestran en diferentes colores y apilados verticalmente. El modelo identifica claramente cada una de las tres poblaciones parentales como un grupo separado en el que cada individuo alcanza cerca de 100 % de asignación a un único componente ancestral. En contraste, el resto de los individuos muestra proporciones variables de estos tres componentes, cubriendo un amplio rango de variación. Además, hemos ordenado intencionalmente los estados en dirección norte-sur, evidenciando un patrón clinal de ancestría europea, la cual es mayor en los estados del norte y menor en los del sur, sustituyéndose por un componente e indígena que predomina en los estados del sur.
Con el fin de visualizar la extensión geográfica de estos componentes más allá de las coordenadas de muestreo, utilizamos los valores obtenidos para cada individuo y construimos un mapa de contornos por medio de métodos de interpolación de datos. Para contar con puntos adicionales de muestreo hemos incorporado en nuestro análisis los genotipos de 50 individuos de Guadalajara, Jalisco, genotipados también con microarreglos de Affymetrix como parte del proyecto Popres [Nelson et al., 2008]. En la figura 3 se muestran los mapas de contornos tanto para el componente europeo como para el componente indígena. Dado que el componente africano es relativamente constante a lo largo de casi todas las regiones, son el componente indígena y el europeo los que dictan los principales patrones de mestizaje. Así pues, observamos que las distribuciones geográficas de los componentes indígena y europeo son casi inversamente proporcionales y nos ayudan a tener una idea general de las zonas donde un componente se sustituye por el otro. Un caso particular es la península de Yucatán, ya que, a pesar de su distancia con la zona norte, muestra una mayor proporción de ancestría europea comparada con sus regiones vecinas del sureste. Esto puede deberse a un mayor intercambio genético con individuos de ancestría europea en tiempos recientes, a pesar de ser una zona altamente indígena. De hecho, estudios previos han reportado que incluso la población maya de Yucatán suele mostrar un grado importante de mestizaje europeo [Li et al., 2008]. Éste no es un caso aislado, ya que el mismo patrón se observa en otras comunidades indígenas, como los grupos nahua [Mao et al., 2007], quienes también ocupan territorios extendidos, incluso a veces dentro de zonas urbanas y, por lo tanto, han tenido mayor posibilidad de contacto con individuos de cierta ancestría europea. Por otro lado, estos ejemplos contrastan con los de otras comunidades que se han mantenido históricamente más aisladas después del contacto europeo. Tal es el caso de la población zapoteca incluida en nuestro análisis, donde no hay indicios de ancestría europea en ninguno de los individuos muestreados (véase la figura 2).

Estructura subcontinental de la población mexicana
Hasta ahora hemos analizado la estructura de la población asumiendo un modelo de tres componentes ancestrales con diferentes proporciones de contribución al mestizaje en México. Éste no es un modelo incorrecto ni mucho menos, pero sí un tanto limitado en el sentido de que sólo es capaz de distinguir entre diferencias ancestrales a nivel continental; es por esta razón que hasta ahora no hemos cuestionado la inclusión de las poblaciones zapoteca, yoruba y de Utah como panel de referencia para la estimación de proporciones ancestrales. Pero hagámoslo ahora: ¿por qué elegir precisamente estas poblaciones y no otras que también fuesen de origen indígena, africano y europeo? En realidad, la respuesta breve y sin entrar mucho en detalles es que, en efecto, podríamos utilizar casi cualquier otro grupo de poblaciones continentales y muy probablemente las estimaciones de K = 3 serían muy similares. La razón es que la diferenciación de grupos continentales por lo general es mayor a cualquier diferencia subcontinental, de manera que un modelo de K = 3 en un individuo con ancestría proveniente de tres grupos tan divergentes tiene mucha robustez, casi sin importar las poblaciones ancestrales de referencia, e incluso a veces en ausencia de éstas. Sin embargo, si queremos ir más allá y explorar la estructura sub-B continental, entonces la inclusión de un panel más diverso de poblaciones se convierte en un factor crítico. Veamos un ejemplo: a continuación retomamos los datos publicados por Silva et al. [2009], que incluye 300 mestizos mexicanos genotipados con el microarreglo de 100K SNPS, y aplicamos el mismo modelo de Admixture pero aumentando al doble el número permitido de componentes ancestrales (K = 6). Como podemos observar en la figura 4, los componentes de las tres poblaciones continentales (YRI, CEU y ZAP) se mantuvieron intactos, mientras que dentro del grupo de mestizos aparecieron nuevos componentes. En particular, llama la atención un componente que parece ser exclusivo de Yucatán, ya que sólo se encuentra en altas proporciones entre los individuos de la muestra yuc y se pierde gradualmente en dirección norte. De igual manera, aparece un componente con altas proporciones en Sonora, que también disminuye gradualmente pero en dirección sur. También llama la atención que la mayoría de la ancestría indígena del resto de las muestras se asigna a un nuevo componente compartido que, a diferencia de K = 3, no es identificado como zapoteca; es decir, la muestra ZAP se sigue identificando como un componente independiente, pero ya no representa la mayoría de la ancestría de los genomas mestizos, probablemente porque éstos se diferencian de región en región. Es importante hacer hincapié en que la interpretación de estos resultados no debe hacerse al pie de la letra, ya que es un modelo que está sujeto a la información con que lo alimentamos. Antes de tomar nota de las proporciones exactas de cada componente, lo que el modelo nos está diciendo es: 1) que existe una importante subestructura en la muestra de mestizos que no está siendo capturada por las tres poblaciones ancestrales incluidas en el modelo, 2) que los nuevos componentes tienen una distribución regional delimitada y 3) que probablemente responden a diferencias en su origen indígena. Para comprobar esto y obtener una fotografía de mayor resolución es evidente que lo que deberíamos hacer es estudiar más ampliamente la diversidad de las poblaciones indígenas y entender su estructura genética.

Con esta motivación, y el respaldo de múltiples instituciones con las que trabajamos actualmente tanto en México como en el extranjero (incluyendo la Escuela Nacional de Antropología e Historia, el Instituto Nacional de Ciencias Médicas y Nutrición, la Universidad Nacional Autónoma de México, la Universidad de Guadalajara, la Universidad de Stanford y la Universidad de California en San Francisco), los autores de este artículo iniciamos un proyecto en colaboración con una red multidisciplinaria de 7 investigadores para describir la diversidad de poblaciones indígenas mexicanas a nivel genómico [Moreno, Gignoux et al., en prensa]. Este proyecto se encuentra en curso gracias a la participación de experimentados antropólogos y genetistas con amplia experiencia en el trabajo de campo y que han hecho posible reunir y genotipar cerca de 500 muestras de más de 15 e poblaciones indígenas, cubriendo gran parte de la diversidad lingüística, étnica y geográfica del país. Utilizamos un microarreglo de Affymetrix que interroga alrededor de un millón de posiciones genómicas y combinamos los datos generados con otras fuentes de datos genómicos de poblaciones nativas recientemente publicados [Mao et al., 2007 y Reich et al., 2012].
Los primeros resultados son contundentes: observamos un alto grado de diferenciación entre las poblaciones nativas de México y la existencia de patrones geográficos de subestructura poblacional. En particular documentamos al menos tres componentes regionales que se extienden a través de varias poblaciones de una misma área geográfica: norte, centro/sur y sureste; además de varios componentes que parecen estar restringidos a poblaciones individuales y aisladas. Es probable que estos últimos, precisamente por su restricción geográfica, no hayan contribuido de manera importante al mestizaje de la población general. Sin embargo, los componentes más extendidos están representados por grupos étnicos numerosos, como los grupos nahua o los grupos maya, que comparten el territorio con zonas urbanas y cosmopolitas. Por lo tanto, es probable que sus genes hayan contribuido en mayor proporción a la mezcla que conforma a la población mestiza. Esto querría decir que, a través del análisis de genomas mestizos, y aplicando técnicas de análisis de ancestría como las que hemos venido desplegando, deberíamos ser capaces de reconocer el origen indígena de la población mexicana (si sus poblaciones parentales están lo suficientemente diferenciadas, lo cual acabamos de comprobar). En este caso ya no estamos hablando de reconocer el origen indígena como un componente común a todo aquello que no es europeo ni africano, sino de intentar ubicar su origen subcontinental, lo que nos ayudaría a trazar nuestras raíces dentro de México.
Impacto de los componentes indígenas en la población general
Para responder a este objetivo unimos esfuerzos con el proyecto PDGM del Instituto Nacional de Medicina Genómica, el cual ha generado en su segunda fase una gran cantidad adicional de datos genómicos utilizando microarreglos de mayor densidad que en su primera fase (por ejemplo 500K y 550K SNPS). Esto ha permitido que la intersección de ambas bases de datos (de indígenas y mestizos) cuente con cerca de medio millón de SNPS en común y que podamos realizar análisis de muy alta definición. Además, en su segunda fase, el proyecto PDGM también ha incluido un número mayor de muestras, aumentando a 10 el número de estados representados y a tres el número de poblaciones indígenas muestreadas. La base de datos global cuenta entonces con cerca de 1 000 individuos (aproximadamente 500 de origen indígena y el resto de origen mestizo), incluyendo 20 poblaciones indígenas de todo el país (véase la tabla 2). En consecuencia, el estudio cuenta con una amplia cobertura geográfica y étnica de gran parte de la diversidad poblacional de México. Cabe mencionar que este esfuerzo conjunto representa hasta ahora la mayor cantidad de datos genómicos que jamás se hayan generado de la población mexicana, y probablemente de la de ningún otro país de cualquier región del mundo.

El mapa de la figura 5 indica la localización de los puntos de muestreo tanto en poblaciones indígenas (asteriscos) como mestizas (cruces). Como puede observarse, en la mayoría de los casos contamos con un punto de muestreo en comunidades indígenas cercano a cada población mestiza, lo que nos ha permitido evaluar con mucha mayor resolución el impacto de los componentes indígenas en la población general. En un primer análisis conjunto de los datos, incluimos las 20 poblaciones indígenas como potenciales grupos ancestrales y evaluamos diferentes modelos de Admixture o desde K = 2 hasta K = 20. El modelo con mayor probabilidad de ajustarse a los patrones de diferenciación de frecuencias alélicas con el menor error posible está dado por el agrupamiento de las muestras en nueve componentes (K = 9). Seis de estos componentes son de origen indígena, dos de origen europeo (dado por diferencias entre muestras del sur y norte de Europa) y uno de origen africano. Como antes comentamos, los componentes indígenas muestran una distribución geográfica bien delimitada, con tres de ellos extendiéndose ampliamente en la zona norte, centro-sur y sureste del país, mientras que el resto se encuentra restringido a comunidades aisladas. Nuestra sorpresa fue mayúscula cuando observamos que la contribución de estos mismos componentes en los genomas mestizos sigue un patrón proporcionalmente similar al observado en sus poblaciones parentales. Por ejemplo, el componente indígena de las poblaciones nativas del norte está representado en mayor porcentaje entre los individuos de Sonora y Durango, mientras que el componente maya está presente en altas proporciones en los individuos de Campeche y Yucatán, pero disminuye drásticamente fuera de estas regiones. Lo mismo ocurre con los componentes aislados, que a pesar de su mínima contribución general en la población mestiza, también muestran que la única población mestiza en la que se conservan en frecuencias perceptibles es precisamente aquella formada por los individuos (mestizos) del mismo estado o región en donde se ubica la comunidad indígena aislada. Tal es el caso de los lacandones en Chiapas y de los seri o comcaac en Sonora. En definitiva, queda en evidencia que la distribución geográfica de los componentes indígenas se reproduce en el patrón de las proporciones ancestrales de cada población mestiza. Por supuesto que los valores porcentuales de estos componentes nativos son menores que en los genomas indígenas, pues los componentes europeos y africanos representan una proporción importante de los genomas mestizos, pero gracias a la aplicación de métodos lo suficientemente sensibles hemos podido identificar estas señales aun cuando la ancestría indígena es más limitada.3

Relevancia e implicaciones sociales
Estos resultados demuestran que la estructura de la población indígena se ha transferido y conservado dentro de los componentes ancestrales de la población a la que hoy nos referimos como mestiza, a tal grado que una es el espejo de la otra. Dicho de otro modo, las raíces indígenas de los mexicanos han quedado registradas en nuestro genoma con mucho más detalle de lo que suponíamos. Y es precisamente por esto por lo que creemos que la difusión de este concepto tiene importantes repercusiones sociales, puesto que nos permite hablar, en vez de un origen anónimo, que hasta ahora solemos englobar como "indígena" o "prehispánico", de un origen diferencial (e identificable) de cada individuo mestizo con respecto a sus raíces indígenas. Pero, ¿por qué habría de ser esto algo novedoso?, ¿qué no es obvio decir que nuestro pasado está registrado de alguna manera en nuestros genes? Sí, por definición así es, pero sólo recientemente contamos con las herramientas de laboratorio y computacionales para explorar con mayor detalle nuestra información genética en busca de los linajes que explican nuestro pasado y nos conectan como sociedad. De hecho, la inquietud por conocer la relación entre las poblaciones nativas de América y la población mestiza es una antigua pregunta de investigación en antropología. En 2008 un estudio basado en 678 marcadores autosómicos [Wang et al., 2008] ya había descrito que suele haber una relación estrecha entre los componentes nativos locales y la población mestiza de diferentes países de América Latina. Sin embargo, esta descripción corresponde a una escala continental y, si tomamos en cuenta que 678 marcadores determinan la resolución de nuestro lente, nos daríamos cuenta de que, si quisiéramos tomar una fotografía a nivel regional, en este caso dentro de México, tal vez obtendríamos una imagen borrosa y, en consecuencia, las interpretaciones no serían concluyentes. Es decir, la definición de la estructura poblacional es una pregunta constante con respuestas que cada vez adquieren mayor resolución con el uso de un número mayor de marcadores (análogos al número de pixeles de una fotografía) y de un número mayor de muestras en áreas geográficas más delimitadas (análogo a la capacidad de un zoom de una cámara fotográfica). Así pues, combinando ambos factores (con una resolución de casi un millón de marcadores y cerca de 1 000 individuos genotipados) hemos reconstruido la imagen de la estructura genética de México con mayor resolución obtenida hasta ahora, y ésta nos habla de la íntima relación que guardan los asentamientos indígenas con las poblaciones mestizas de su alrededor, a pesar de que el territorio nacional haya sido sujeto de un súbito e intenso proceso de mestizaje.
Para contextualizar la importancia de este hecho imaginemos una hipótesis alternativa: bien podría ser que, precisamente debido al proceso de mestizaje,4 los componentes ancestrales (de origen indígena) fuesen indistinguibles uno de otro dentro de un genoma mestizo, lo que provocaría la pérdida de conexión con las raíces nativas de la población general. Esto no quiere decir que se perdería la ancestría indígena, pero sí el rastro de su identidad. Sin embargo, nuestros resultados muestran lo contrario: la población mestiza ha retenido gran parte de la composición de sus ancestros y su identidad está reflejada en cada genoma mestizo descendiente del mismo linaje. Pero, entonces, si comparten un linaje, ¿por qué seguimos distinguiendo entre indígenas y mestizos?, ¿por qué no nos referimos a ambos como una misma población? Esta pregunta causa polémica y seguramente tiene muchas respuestas, pero, desde el punto de vista evolutivo, efectivamente indígenas y mestizos comparten una historia estrecha y reciente, por lo que nos cuestionamos si el empleo de dicha terminología dicotómica es adecuado (conveniente lo es, pues nos ayuda a describir la procedencia de un individuo en un determinado contexto, pero la categorización —y discriminación— a la que se ha extendido es lo que parece carecer de sustento). Tampoco pretendemos insinuar que no existen diferencias entre las características de la población indígena y la población general. Por supuesto que las hay, incluso abismales, pero éstas son de tipo cultural, lingüístico, socioeconómico y demás, pero no biológicas, raciales o de cualquier otro tipo que sustente la existencia de categorías. Incluso en el plano genético las diferencias son graduales y siguen un patrón clinal en el que es imposible delimitar cualquier tipo de frontera entre ambos grupos. Por ejemplo, ¿cómo distinguimos entre un individuo indígena de Oaxaca con cierto grado de ancestría europea y un mestizo oaxaqueño con altas proporciones de ancestría indígena? No es posible hacerlo. Genéticamente son indistinguibles.5 O, por ejemplo, individuos de origen europeo que han sido recibidos y aceptados como miembros de comunidades indígenas, ¿no deberían tener el derecho de identificarse como indígenas? Es decir, la pertenencia a uno u otro grupo no puede determinarse con base en fundamentos biológicos o genéticos, ya que entonces entraríamos en el absurdo debate de cuántos genes indígenas son suficientes para certificarse como indígena. Sería ridículo establecer un umbral por arriba o por debajo del cual uno es o deja de ser indígena. La proporción recibida de cada ancestría en cada individuo es simplemente el resultado de su propio linaje y, en cada población, es el resultado de su historia evolutiva. Pero la autodeterminación de los pueblos es libre y no nos corresponde a los genetistas darle o quitarle validez. Tampoco, como investigadores, nos corresponde sentar las bases de la identidad de un grupo o individuo, al contrario, nosotros somos lectores del registro de su historia e identidad, que se ha transmitido en cada linaje hasta nuestros días. Sin embargo, creemos que sí es nuestra responsabilidad dar a conocer que las evidencias genéticas no sustentan las diferencias sociales que por siglos han alimentado la segregación entre mestizos e indígenas, y que tanto dañan el tejido de la sociedad mexicana al igual que el de muchas otras regiones de América Latina.
Los pueblos indígenas son los portadores de la historia antigua del territorio en el que ahora vivimos y nos han heredado una rama de su linaje, misma que seguimos transmitiendo a nuestros hijos. Por lo que es importante darnos cuenta de que compartimos una historia en común, no sólo con aquel pasado prehispánico y distante, sino, más aún, con aquellas comunidades indígenas cercanas a nuestro entorno y con quienes interactuamos hoy en día. Tal vez deberíamos olvidarnos de esa dicotomía entre indígena y mestizo con la que la sociedad parece sentirse cómoda, pero que en realidad refuerza tendencias y conceptos raciales (y racistas). Tal vez la diversidad cultural y étnica (y genética) de México no debería ser simplemente motivo de orgullo nacional, como si de un recurso turístico se tratase, sino una fuente de evidencia para tomar conciencia de la cercanía genealógica que tenemos con quienes han sido olvidados y relegados del "México moderno". En este plano, la genómica puede convertirse en una herramienta poderosa para proporcionar evidencias adicionales en torno a conceptos que han estado condicionados al conocimiento procedente de ciencias más tradicionales. Estamos convencidos de que, así como la tecnología ha revolucionado y modernizado al mundo científico, también debe modernizar nuestra mentalidad y, por lo tanto, nuestra actitud para con la sociedad en la que vivimos. Asimismo, la terminología de las ciencias genómicas deberá formar poco a poco parte de nuestro vocabulario cotidiano, y los conceptos derivados del conocimiento de nuestro genoma deberán formar parte del conocimiento general de la población y no sólo de un grupo de especialistas. Para ello es importante que desde ahora los científicos nos demos a la tarea de divulgar nuestros hallazgos y acercarlos a una audiencia plural. Pero también es necesario que se tomen decisiones políticas para priorizar la ciencia como fuente de conocimiento, así como para incorporar conceptos derivados de investigaciones científicas en los censos poblacionales y en los programas escolares básicos. Estos cambios no ocurrirán de un día para otro pero, si estudios como éste, además de su beneficio en el mero campo científico, son capaces de aportar un pequeño grano de arena a esta toma de conciencia, habremos hecho mucho desde nuestra trinchera en forma de laboratorio por la lucha contra las desigualdades sociales.
Agradecimientos. Queremos agradecer a los cientos de voluntarios anónimos de las comunidades participantes, sin quienes este trabajo no habría sido posible. La presente contribución ahonda particularmente en las implicaciones antropológicas y sociales de nuestra investigación, cuyos resultados son el fruto de una colaboración en la que participan múltiples instituciones e investigadores con quienes compartimos crédito en una publicación conjunta y a quienes queremos reiterar nuestro agradecimiento. En particular, agradecemos al doctor Carlos D. Bustamante, de la Universidad de Stanford, y al doctor Esteban González Burchard, de la Universidad de California, en San Francisco al doctor Héctor Rangel Villalobos, de la Universidad de Guadalajara, al maestro Víctor Acuña Alonzo, de la Escuela Nacional de Antropología e Historia y al doctor Samuel Cañizales Quintero, del Instituto Nacional de Ciencias Médicas y Nutrición, Salvador Zubirán, por su apoyo en la generación de datos, obtención de muestras y coordinación. Asimismo, agradecemos a los investigadores del INMEGEN y a todo el equipo de científicos involucrados por su importante contribución tanto en la generación de datos como en el análisis conjunto de los mismos. En particular, al doctor Gerardo Jiménez, a la doctora Irma Silva Zolezzi, al doctor Alfredo Hidalgo, al doctor Jesús Estrada, al maestro en ciencias, Juan Carlos Fernández, a la doctora Alejandra Contreras, al ingeniero Rodrigo García, a la doctora Sandra Romero, a la doctora Alessandra Carnevale y al doctor Xavier Soberón. J Durante el curso de la investigación fueron claves las aportaciones analíticas de los doctores. Christopher Gignoux y Fouad Zakharia, de quienes hemos aprendido enormemente. Los costos de genotipado fueron principalmente financiados con fondos del laboratorio del doctor Carlos Bustamante por parte de la Universidad de Stanford, y del Gobierno Federal de México por parte de los datos generados en el INMEGEN. Parte del apoyo para investigación también ha sido provisto por fondos del premio Rosenkranz 2012 concedido a Andrés Moreno. Agradecemos a Arturo y Cristina Moreno por sus comentarios a versiones tempranas de este manuscrito, así como a la doctora Rocío Vargas Sanders, del Instituto de Investigaciones Antropológicas de la UNAM, y al historiador Andrés Aubry, a quienes debemos gran parte de nuestra motivación y cuya memoria sigue acompañando nuestros pasos. Por último quisiéramos dedicar este trabajo a nuestra hija Romina, cuyo año de nacimiento coincidirá con el de esta publicación. Escribimos estas páginas mientras tú crecías dentro de mamá y aquí estarán esperando a que crezcas un poco más para que puedas leerlas. ¡Gracias por estar con nosotros!
Bibliografía
Alexander, David H., John Novembre y Kenneth Lange 2009 "Fast Model-Based Estimation of Ancestry in Unrelated Individuals", Genome Research, vol. 19, núm. 9, pp. 1655-1664. [ Links ]
Bryc, Katarzyna, Christopher Vélez, Tatiana Karafet, Andrés Moreno Estrada, Andy Reynolds, Adam Auton, Michael Hammer, Carlos D. Bustamante y Harry Ostrer 2010 "Colloquium Paper: Genome-Wide Patterns of Population Structure and Admixture Among Hispanic/Latino populations", Proceedings of the National Academy of Sciences of the United States of America, núm. 107, supl. 2, pp. 8954-8961. [ Links ]
Córdova, M. Soledad, Rubén Lisker y Alvar Loria 1967 "Studies on Several Genetic Hematological Traits of the Mexican Population. XII. Distribution of Blood Group Antigens in Twelve Indian Tribes", American Journal of Physical Anthropology, vol. 26, núm. 1, pp. 55-65. [ Links ]
Instituto Nacional de Estadística, Geografía e Informática (INEGI) 2010 Censo de población y vivienda, México, INEGI. [ Links ]
Li, Jun Z., Devin M. Absher, Hua Tang, Audrey M. Southwick, Amanda M. Casto, Sohini Ramachandran, Howard M. Cann, Gregory S. Barsh, Marcus Feldman, Luigi L. Cavalli Sforza y Richard M. Myers 2008 "Worldwide Human Relationships Inferred from Genome-Wide Patterns 5 of Variation", Science, vol. 319, núm. 5866, pp. 1100-1104. [ Links ]
Lisker, Rubén, Alvar Loria y M. Soledad Córdova 1965 "Studies on Several Genetic Hematological Traits of the Mexican Population. 8. Hemoglobin S, Glucose-6-Phosphate Dehydrog enase Deficiency, and other Characteristics in a Malarial Region", American Journal of Human Genetics, vol. 17, pp. 179-187. [ Links ]
Lisker, Rubén, Graciela Zárate y Elizabeth Rodríguez 1967 "Studies on Several Genetic Hematological Traits of the Mexican Population. XIV. Serum Polymorphisms in Several Indian Tribes", American Journal of Physical Anthropology, vol. 27, núm. 1, pp. 27-32. [ Links ]
Mao, Xianyun, Abigail W. Bigham, Rui Mei, Gerardo Gutiérrez, Ken M. Weiss, Tom D. Brutsaert, Fabiola León Velarde, Lorna G. Moore, Enrique Vargas, Paul M. McKeigue, Mark D. Shriver y Esteban J. Parra 2007 "A Genomewide Admixture Mapping Panel for Hispanic/Latino Populations", The American Journal of Human Genetics, vol. 80, núm. 6, pp. 1171-1178. [ Links ]
Martínez Cortés, Gabriela, Joel Salazar-Flores, Laura G., Rodrigo Fernández Rodríguez, Rubí Castellanos, Carmen Rodríguez Loya, Jesús S. Velarde Félix, José F. Muñoz Valle, Isela Parra Rojas y Héctor Rangel Villalobos 2012 "Admixture and Population Structure in Mexican-Mestizos Based on Paternal Lineages", Journal of Human Genetics, vol. 57, núm. 9, pp. 568-574. [ Links ]
Mendizábal, Isabel, Karla Sandoval, Gemma Berniell Lee, Francesc Calafell, Antonio Salas, Antonio Martínez Fuentes y David Comas 2008 "Genetic Origin, Admixture, and Asymmetry in Maternal and Paternal Human Lineages in Cuba", BMC Evolutionary Biology, núm. 8, p. 213. [ Links ]
Moreno, Gighoux et al. En prensa "The Genetics of Mexico Recapitulates Native American Substructure and Impacts Biomedical Traits", Science. [ Links ]
Nelson, Matthew R., Katarzyna Bryc, Karen S. King, Amit Indap, Adam R. Boyko, John Novembre, Linda P. Briley, Yuka Maruyama, Dawn M. Waterworth, Gérard Waeber, Peter Vollenweider, Jorge R. Oksenberg, Stephen L. Hauser, Heide A. Stirnadel, Jaspal S. Kooner, John C. Chambers, Brendan Jones, Vincent Mooser, Carlos D. Bustamante, Allen D. Roses, Daniel K. Burns, Margaret G. Ehm y Eric H. Lai 2008 "The Population Reference Sample, Popres: A Resource for Population, Disease, and Pharmacological Genetics Research", The American Journal of Human Genetics, vol. 83, núm. 3, pp. 347-358. [ Links ]
Pritchard, Jonathan K., Matthew Stephens y Peter Donnelly 2000 "Inference of Population Structure Using Multilocus Genotype Data", Genetics, vol. 155, núm. 2, pp. 945-959. [ Links ]
Rangel Villalobos, Héctor, J. F. Muñoz-Valle, A. González-Martín, A. Gorostiza, M. T. Magaña y L. A. Páez-Riberos 2008 "Genetic Admixture, Relatedness, and Structure Patterns Among Mexican Populations Revealed by the Y-chromosome", American Journal of Physical Anthropology, vol. 135, núm. 4, pp. 448-461. [ Links ]
Reich, David, Nick Patterson, Desmond Campbell, Arti Tandon, Stephane Mazieres, Nicolás Ray et al. (57 autores más) 2012 "Reconstructing Native American Population History", Nature, vol. 488, 1 núm. 7411, pp. 370-374. [ Links ]
Rubí Castellanos, Rodrigo, Gabriela Martínez Cortés, José F. Muñoz Valle, Antonio González Martín, Ricardo M. Cerda Flores, Manuel Anaya Palafox y Héctor Rangel Villalobos 2009 "Pre-Hispanic Mesoamerican Demography Approximates the Present day Ancestry of Mestizos throughout the Territory of Mexico", American Journal of Physical Anthropology, vol. 139, núm. 3, pp. 284-294. [ Links ]
Sandoval, Karla, Andrés Moreno Estrada, Isabel Mendizábal, Peter A. Underhill, María López Valenzuela, Rosenda Peñaloza Espinosa, Marisol López López, Leonor Buentello Malo, Heriberto Avelino, Francesc Calafell y David Comas 2012 "Y-Chromosome Diversity in Native Mexicans Reveals Continental Transition of Genetic Structure in the Americas", American Journal of Physical Anthropology, vol. 148, núm. 3, pp. 395-405. [ Links ]
Sandoval, Karla, Leonor Buentello Malo, Rosenda Peñaloza Espinosa, Heriberto Avelino, Antonio Salas, Francesc Calafell y David Comas 2009 "Linguistic and Maternal Genetic Diversity are not Correlated in Native Mexicans", Human Genetics, vol. 126, núm. 4, pp. 521-531. [ Links ]
Silva Zolezzi, Irma, Alfredo Hidalgo Miranda, Jesús Estrada Gil, Juan Carlos Fernández López, Laura Uribe Figueroa, Alejandra Contreras, Eros Balam Ortiz, Laura del Bosque Plata, David Velázquez Fernández, César Lara, Rodrigo Goya, Enrique Hernández Lemus, Carlos Dávila, Eduardo Barrientos, Santiago March y Gerardo Jiménez Sánchez 2009 "Analysis of Genomic Diversity in Mexican Mestizo Populations to Develop Genomic Medicine in Mexico", Proceedings of the National Academy of Sciences of the United States of America, vol. 106, núm. 21, pp. 8611-8616. [ Links ]
The International HapMap Consortium 2005 "A Haplotype Map of the Human Genome", Nature, vol. 437, núm. 7063, pp. 1299-1320. [ Links ]
Wang, Sijia, Nicolás Ray, Winston Rojas, María V. Parra, Gabriel Bedoya, Carla Gallo, Giovanni Poletti, Guido Mazzotti, Kim Hill, Ana M. Hurtado, Beatriz Camarena, Humberto Nicolini, William Klitz, Ramiro Barrantes, Julio A. Molina, Nelson B. Freimer, María Catira Bortolini, Francisco M. Salzano, María L. Petzl Erler, Luisa T. Tsuneto, José E. Dipierri, Emma L. Alfaro, Graciela Bailliet, Néstor O. Bianchi, Elena Llop, Francisco Rothhammer, Laurent Excoffier y Andrés Ruiz Linares 2008 "Geographic Patterns of Genome Admixture in Latin American Mestizos", PLoS Genetics, vol. 4, núm. 3, p. e1000037. [ Links ]
1 Esto debido al proceso de colonización, en el que se exterminaron más hombres nativos que mujeres indígenas.
2 Un cuestionamiento común a los estudios de la genética de poblaciones es la representatividad de su muestra. Muchos han argumentado que unos pocos cientos de individuos no son representativos de la población general. Si la expectativa es capturar el perfil del total de individuos que conforman una población, por supuesto que una muestra será insuficiente; ésa es la función de un censo, mas no de un muestreo. Sin embargo, si al muestrear un número reducido de individuos, digamos 25 o 50 por cada grupo, éstos muestran afinidades que comparten entre ellos, pero que los distinguen de otros grupos, entonces podemos inferir que sí son representativos de las características de su región.
3 Para ello utilizamos una batería de métodos que nos permiten distinguir los componentes ancestrales de cada segmento cromosómico por separado a lo largo de todo el genoma. Estos métodos se conocen como técnicas de estimación de ancestría local (en contraste con la ancestría global calculada para todo el genoma de un individuo). Siguiendo este abordaje por ventanas logramos estimar la ancestría local a lo largo de todo el genoma de cada individuo mestizo, lo que nos permitió identificar y enmascarar todas aquellas ventanas o segmentos cromosómicos cuya ancestría no sea indígena. Esto es equivalente en términos computacionales a "desnudar" selectivamente el genoma y dejar expuesta sólo su porción de origen indígena (o de cualquier otro componente ancestral de interés).
4 El mestizaje tiene diferentes efectos en la población según el tiempo que ha transcurrido desde el inicio del mismo. El efecto inicial es el de una población heterogénea debido al encuentro de segmentos provenientes de grupos previamente diferenciados. Sin embargo, con el paso del tiempo el efecto final es el de una eventual homogeneización de la población. México es hoy en día un país plural, sumamente diverso y con sus raíces conservadas. Pero si las tradiciones, la cultura y, por lo tanto, su gente y sus genes, no se conservan, eventualmente dichas raíces e identidades terminarán por perderse con el paso de las generaciones, debido al efecto continuo de la migración entre poblaciones que acaban por asimilarse en una sola población homogénea.
5 El mestizaje no debe referirse exclusivamente a la mezcla de ancestría europea. De hecho, desde el punto de vista técnico, también todos los indígenas son mestizos: si algo hemos aprendido de nuestro estudio es que los componentes de ancestría indígena se han mezclado entre diferentes comunidades nativas, de modo más gradual, pero genéticamente el proceso también es de mestizaje.














