Introducción
Las infecciones pulmonares por micobacterias no tuberculosas (MNT) son un desafío emergente de salud pública, que generalmente afectan a individuos con padecimientos pulmonares preexistentes. Otras aflicciones causadas por estas bacterias son contagios en la piel o una infección diseminada, particularmente en individuos inmunocomprometidos.1 Las características inmunológicas del hospedero y la especie micobacteriana influyen en la susceptibilidad y en las manifestaciones de la infección.
El género Mycobacterium, descubierto en 1882 por Robert Koch, abarca múltiples especies y subespecies, algunas de las cuales están incluidas en «complejos» por su gran similitud filogenética. El término MNT en general se refiere a micobacterias que no pertenecen al complejo de M. tuberculosis y M. leprae. Las MNT se encuentran en el ambiente: agua, suelo, alimentos y animales. Pueden estar presentes en superficies corporales y secreciones, sin causar enfermedad.2 Sin embargo, las MNT se han identificado como agentes causantes de infecciones oportunistas en humanos. Pueden adquirirse por inhalación, ingestión de agua o inoculación directa en la piel, e incluso se ha reportado transmisión de persona a persona.3,4
Los padecimientos infecciosos causados por MNT presentan un cuadro clínico similar al que se exhibe en la tuberculosis pulmonar (TBP), con la que también pueden presentarse como una infección concomitante, por lo que es importante realizar un diagnóstico diferencial entre ambos tipos de bacterias a fin de indicar el manejo adecuado, ya que los fármacos utilizados para tratar contaminaciones por M. tuberculosis son ineficaces contra las MNT. Por ello, el objetivo de esta revisión es describir el diagnóstico, el tratamiento y los mecanismos de resistencia a diversos antibióticos con el fin de tener más herramientas para el cuidado del paciente con enfermedad pulmonar causada por MNT.
Características generales y clasificación de las micobacterias no tuberculosas
Las MNT son bacilos rectos o ligeramente curvos, no móviles, ácido-alcohol resistentes, aerobios a microaerofílicos, con un alto contenido de G+C (guanina+citocina) en su genoma (57-73 %). Su pared celular es gruesa e hidrofóbica, es rica en ácidos micólicos complejos, lo que la hace impermeable a compuestos hidrofílicos y resistente a metales pesados, desinfectantes y antibióticos.5 Comúnmente, las MNT forman biopelículas, lo que contribuye a la resistencia a desinfectantes y antibióticos.5,6 La mayoría de las especies son invulnerables a temperaturas altas y son relativamente resistentes a pH bajo.7
Se han descrito más de 200 especies de MNT, la mayoría de las cuales son patógenas oportunistas.8 La gran mayoría de estas bacterias se han aislado de pacientes portadores del virus de inmunodeficiencia humana (VIH), quienes generalmente son susceptibles a varias infecciones.3
La clasificación más utilizada para la identificación de las MNT es la de Runyon, la cual propone cuatro grupos con base en la morfología de las colonias, la velocidad de crecimiento y la producción de pigmento.9 Los tres primeros grupos son de crecimiento lento, los cuales tardan más de siete días en formar colonias en medio sólido.10,11 El grupo I son bacterias fotocromógenas que producen pigmento amarillo cuando se exponen a la luz, mientras que el grupo II son escotocromógenas, que presentan pigmento amarillo brillante siempre visible en presencia de luz o en la oscuridad. El grupo III son no cromógenas, es decir, no producen pigmento o lo producen débilmente con coloración amarilla pálida y, cuando se exponen a la luz, no se intensifica. El grupo IV lo conforman bacterias de crecimiento rápido y no producen pigmento. En la Tabla 1 se enlistan las especies que más frecuentemente causan infecciones en humanos.
Tabla 1: Clasificación de las micobacterias no tuberculosas que causan infecciones en humanos más frecuentemente.
| Micobacterias de crecimiento lento* | Micobacterias de crecimiento rápido‡ |
|---|---|
| Grupo I. Fotocromógenas | Grupo IV No cromógenas |
| M. kansasii | M. chelonae |
| M. marinum | M. fortuitum |
| M. simiae | M. smegmatis |
| M. xenopi | M. abscessus |
| Grupo II. Escotocromógenas | M. peregrinum |
| M. scrofulaceum | M. cosmeticum |
| M. gordonae | M. gordonae |
| M. szulgai | |
| Grupo III. No cromógenas | |
| M. avium | |
| M. chimaera | |
| M. ulcerans | |
| M. malmoense |
*Lento: crecimiento visible en medio sólido en 21 días.
‡Rápido: crecimiento visible en medio sólido en menos de 7 días.
Epidemiología mundial de la enfermedad por MNT
Las MNT causaron principalmente infección pulmonar (67 %), mientras que los contagios extrapulmonares generalmente se localizaban en sangre y piel o tejidos blandos (31.8 %); 1.22 % de los pacientes presentaron infección pulmonar y extrapulmonar.12,13
La incidencia mundial de infecciones por MNT varía de 1.0 a 1.8 casos por 100,000 personas. La tasa anual en Estados Unidos es de 15.2 eventos por 100,000 personas, siendo la de enfermedad pulmonar de 5.6 casos, seguido por padecimientos de piel y de tejidos blandos con 0.9 y luego por linfadenitis con 0.3 incidencias por 100,000 personas, respectivamente.14 A diferencia de la tuberculosis, las contaminaciones pulmonares por MNT son más prevalentes en mujeres (59 %) y en adultos mayores de 65 años.15
Las especies más comunes de MNT en infecciones pulmonares pertenecen al complejo de M. avium (80.1 %), seguidas por M. chelonae y M. abscessus (12.1 %), M. fortuitum (5.6 %) y M. kansasii (5.5 %).16
La ocurrencia de MNT está aumentando en todo el mundo, con un espectro de especies muy diverso. Este aumento podría estar relacionado con la disponibilidad de nuevas técnicas de diagnóstico molecular y la búsqueda consciente e intencionada de las MNT en sus variadas manifestaciones clínicas. De hecho, 78 % de las especies identificadas se alcanzó en los últimos 20 años.13
Micobacteriosis causadas por MNT
La entidad nosológica depende de varios factores, entre ellos está el estado inmunológico de cada paciente.17 Las infecciones de la piel y tejidos blandos por MNT casi siempre son resultado de la inoculación iatrogénica o accidental causada por contaminación quirúrgica o traumatismos en afectados sin contagios previos. Las infecciones de pulmón se adquieren fácilmente en personas que padecen un trastorno pulmonar primario o una afección sistémica que los predispone.18,19 En la Tabla 2 se enlistan las especies más comunes según el sitio de infección.
Tabla 2: Especies de micobacterias no tuberculosas más comunes según la manifestación clínica.
| Sitios de infección | Especie |
|---|---|
| Piel | M. marinum |
| M. ulcerans | |
| M. abscessus | |
| Tejidos blandos y heridas quirúrgicas | M. chimaera |
| M. abscessus | |
| Sistémica y/o diseminada | M. avium (complejo) |
| M. abscessus | |
| Pulmonar | M. avium (complejo) |
| M. chelonae | |
| M. abscessus | |
| M. fortuitum | |
| M. kansasii | |
| M. xenopi | |
| M. malmoense |
Datos tomados a partir de: Johansen MD, Herrmann JL, Kremer L. Non-tuberculous mycobacteria and the rise of Mycobacterium abscessus. Nat Rev Microbiol. 2020;18(7):392-407. https://doi.org/10.1038/s41579-020-0331-1
Personas susceptibles a infección pulmonar por MNT
La infección del pulmón es la más recurrente y se adquiere fácilmente en personas que padecen un trastorno pulmonar primario o una afección sistémica que los predispone. Ocurre principalmente en tres grupos de pacientes: 1. individuos con anomalías pulmonares anatómicas que generalmente no tienen una base genética identificable, por ejemplo bronquiectasias secundarias por infiltraciones previas, enfisema y neumoconiosis; 2. individuos con trastornos inmunológicos genéticos que predisponen a bronquiectasias y/o infecciones de pulmón como fibrosis quística, discinesia ciliar primaria, deficiencia de alfa-1-antitripsina, síndrome de Williams-Campbell, Síndrome de Mounier-Kuhn, síndrome de Sjogren, proteinosis alveolar pulmonar e inmunodeficiencias primarias; y (3) sujetos sin anormalidades pulmonares o inmunológicas conocidas.20
Diagnóstico de infecciones pulmonares por MNT
El diagnóstico apropiado de la enfermedad pulmonar por MNT requiere que se integren datos clínicos, radiográficos y microbiológicos; los síntomas principales son tos crónica, esputo, hemoptisis, fatiga, malestar general y pérdida de peso, aunque a menudo son inespecíficos. En general, los dolientes presentan un trastorno pulmonar subyacente, como bronquiectasias o enfermedad pulmonar obstructiva crónica (EPOC). Los aquejados sintomáticos deben cumplir con los criterios microbiológicos específicos, a fin de ser diagnosticados con una infección pulmonar por MNT.21 En la Tabla 3 se enlistan los criterios diagnósticos propuestos por la Sociedad Americana de Tórax (ATS, por sus siglas en inglés, American Thoracic Society).
Tabla 3: Criterios diagnósticos de enfermedad pulmonar por micobacterias no tuberculosas*.
| Clínicos |
| 1. Síntomas pulmonares, opacidades nodulares o cavitarias en la radiografía de tórax, o tomografía de alta resolución que muestre bronquiectasias multifocales con múltiples nódulos pequeños |
| 2. Exclusión apropiada de otros diagnósticos. |
| Microbiológicos |
| 1. Cultivos positivos en al menos dos expectoraciones tomadas por separado. Si los resultados de las muestras iniciales no son diagnósticos, considere repetir el cultivo y realizar una tinción de BAAR |
| O bien |
| 2. Cultivo positivo en al menos un cepillado o lavado bronquial |
| O bien |
| 3. Biopsia pulmonar, transbronquial u otra, con características histopatológicas de micobacterias (inflamación granulomatosa o tinción de BAAR) y cultivo positivo para MNT o biopsia con características histopatológicas de micobacterias (inflamación granulomatosa o tinción de BAAR) y uno o más esputos o lavados bronquiales con cultivos positivos para MNT |
| 4. Se debe consultar a un experto cuando las MNT recuperadas sean poco frecuentes o generalmente representen contaminación ambiental |
| 5. Los pacientes con sospecha de infección pulmonar por MNT que no cumplan con los criterios diagnósticos deben tener seguimiento hasta que se confirme o excluya el diagnóstico. |
| 6. Hacer el diagnóstico de enfermedad pulmonar por MNT no necesariamente implica establecer un tratamiento, la decisión debe estar basada en los posibles riesgos y beneficios de la terapia para cada paciente |
*Propuestos por American Thoracic Society, 2007.
BAAR = bacilos ácido-alcohol resistentes, MNT = micobacterias no tuberculosas.
Es importante aplicar los criterios en la búsqueda del diagnóstico y posteriormente se debe llevar a cabo la identificación de la cepa causante de la infección, con el fin de dar tratamiento específico para cada paciente y evitar la generación de cepas resistentes. La identificación de las MNT se realiza por métodos bioquímicos, cromatográficos y moleculares. Para la caracterización de las MNT de crecimiento tardado se utilizan los métodos bioquímicos, cuya interpretación es difícil por la variabilidad del fenotipo y porque dependen del crecimiento óptimo del cultivo; algunos ejemplos son la prueba de catalasa semicuantitativa, catalasa termoestable, producción de niacina, reducción de nitratos, ureasa, hidrólisis de Tween 80, arilsulfatasa, fosfatasa ácida, pirazinamidasa, crecimiento en medios con cloruro de sodio, tiacetazona, picrato y oleato, entre otras.22,23 La cromatografía líquida de alta eficiencia de gases o en capa fina también se utiliza buscando la identificación de MNT,24 mediante los patrones de ácidos micólicos que son diferentes dentro de cada especie.23
Además del cultivo y la identificación bioquímica de la cepa, se deben considerar otras pruebas, como la secuenciación de ácidos nucleicos, una de las herramientas más utilizadas es la secuenciación del gen rRNA 16s, el cual ofrece un criterio importante hacia complementar los sistemas de clasificación fenotípica.25 La división de MNT de crecimiento acelerado y lento se mantiene en la filogenia molecular. En la mayoría de los casos, las MNT de crecimiento lento contienen una copia única del operón ribosómico, mientras que las de crecimiento veloz contienen dos. Con base en esto, se ha planteado la hipótesis de que el número de operones ribosómicos en las micobacterias se correlaciona con la tasa de crecimiento.
Las especies de MNT se pueden identificar secuenciando el gen rRNA 16s completo, con algunas excepciones.8,26 Generalmente, se espera una identidad de 100 % en la secuencia rRNA 16s contra la cepa tipo utilizada para identificación.27 Muchas MNT se pueden identificar empleando sólo los primeros 500 pb (que contienen dos regiones hipervariables), por ejemplo, las micobacterias de crecimiento rápido clínicamente significativas: M. fortuitum o M. abscessus. En algunas especies existen ambigüedades en la secuencia, por ello, se utilizan genes secundarios que evolucionan más rápido que los genes ribosomales y que tienen un mayor poder discriminatorio. Verbigracia, para la identificación de cepas de M. chelonae se requiere el gen rRNA 16S completo y/o un gen alternativo para la identificación hasta especie.28 Esto es práctico al procurar distinguir entre especies o complejos emparentados que no pueden ser identificados por rRNA 16s. Una región de 441 pb del gen hsp6529 y la región V del rpoB30 son las más utilizadas y se usan de manera rutinaria en algunos laboratorios.31,32
Mecanismos de resistencia natural y adquirida a los antibióticos más utilizados para tratar contagios por MNT
Con la meta de llevar a cabo un buen manejo de la dolencia, se debe hacer un diagnóstico correcto; luego, realizar la identificación completa de la cepa causante de la infección y posteriormente determinar la susceptibilidad de cada MNT a los antibióticos. Es importante conocer los componentes de defensa intrínseca o natural de las MNT y las posibles mutaciones recurrentes que les confieren esa resistencia adquirida. Esto es indispensable al procurar darles un manejo adecuado a los pacientes y asegurar el éxito.
Isoniazida
Este fármaco es un agente antituberculoso y la mayoría de las MNT son naturalmente resistentes a la isoniazida. La isoniazida es un profármaco que, al entrar a la micobacteria, se modifica a su forma activa e interacciona con NAD+ en presencia de Mn(2)+y O(2) y forma un aducto con NAD+ (isoniazida-NAD), ejerciendo su efecto bactericida. La formación del aducto isoniazida-NAD es catalizado por la enzima catalasa-peroxidasa, KatG.33 La función principal de la KatG en el complejo M. tuberculosis es catabolizar los peróxidos formados durante el estallido oxidativo generado por los fagocitos, antagonizando así el mecanismo inmunitario del hospedero.34
La isoniazida activa inhibe la síntesis de ácidos micólicos; se une a la enzima enoil acil reductasa (inhA), la cual está involucrada en la síntesis de ácidos grasos de fase II, evitando la formación de la pared celular. Se demostró, por análisis de cristalografía, que el aducto isoniazida-NAD se une a la inhA bloqueando su actividad.35
La resistencia intrínseca hacia la isoniazida de la mayoría de las MNT se debe a que la KatG tiene baja eficacia en la activación de la misma. El análisis de la secuencia de la KatG muestra que 20 % de los aminoácidos son exclusivos del complejo M. tuberculosis, pero conservado en las otras micobacterias que muestran resistencia parcial o total. La utilización de la isoniazida en el tratamiento antituberculoso ejerce presión evolutiva que está llevando a la resistencia, generando más variantes de KatG en M. tuberculosis , que no activan a la isoniazida.36 Otras mutaciones que generan insensibilidad en M. tuberculosis están asociadas al gen que codifica a la inhA. En general, causan mayor expresión de inhA o disminuyen la afinidad a NAD+, evitando formar el aducto.37
Rifampicina
Este es el fármaco antituberculoso que también se utiliza para la medicación contra M. kansasii y M. marinum, y es uno de los tres medicamentos más utilizados en un régimen oral a base de macrólidos contra infecciones por M. avium. La rifampicina se une a la subunidad β de la RNA polimerasa codificada por rpoB evitando la síntesis de RNA.38 La resistencia a la rifampicina en M. tuberculosis se asocia con cambios en el gen rpoB en una región de 81 pb entre los codones 507 a 533.29 Se han identificado cambios compensatorios en los genes rpoA y rpoC en aislados resistentes a la rifampicina.39 En M. kansasii, se han reportado variaciones adquiridas similares a las descritas en M. tuberculosis.40
Etambutol
Este fármaco es un agente para combatir la tuberculosis, pero es utilizado como complemento frente a las MNT de crecimiento lento. Por ejemplo, es muy común en el abordaje contra contaminaciones causadas por M. avium y M. kansasii, mientras que su uso es limitado contra MNT de crecimiento rápido.41 El etambutol inhibe la creación de la pared celular, evita la síntesis de arabinogalactanos y de lipoarabinomananos. El etambutol inhibe tres arabinosiltransferasas: EmbA, EmbB y EmbC.42
La resistencia al etambutol se ha asociado con mutaciones adquiridas en el operón embCAB que codifica estas arabinosiltransferasas.43 En M. tuberculosis se han reportado otros cambios en el gen ubiA, el cual codifica para la enzima decaprenil-fosfato 5-fosforibosiltransferasa sintasa, involucrada en la síntesis de la pared celular.44,45 La resistencia adquirida a etambutol en M. avium está asociada con la sobreexpresión del operón embCAB.46 En M. smegmatis y M. kansasii se han reportado mutaciones puntuales en embB.4
Pirazinamida
Es un derivado de amida del ácido pirazinóico sintético; es un agente antituberculoso de primera línea, pero se usa sólo en combinación con otros medicamentos antituberculosos como la isoniazida o la rifampicina. Se ha demostrado que al menos M. avium y M. abscessus son naturalmente resistentes.
La pirazinamida es un profármaco que, al entrar a la bacteria, se convierte en ácido pirazinóico por la enzima pirazinamidasa, PncA (Rv2043c).47 El ácido pirazinóico se une a la enzima aspartato decarboxilasa (PanD), responsable de la formación de β-alanina de L-aspartato, que es parte de la vía biosintética del pantotenato, esencial para la vitamina B5 y la coenzima A.48,49 Se ha verificado que el ácido pirazinóico es un inhibidor competitivo de PanD y se une con alto grado de complementariedad al sitio activo de la enzima. La afinidad de unión es consistente con la potencia de PZA contra M. tuberculosis.50 El antagonismo a pirazinamida en M. tuberculosis está relacionado con mutaciones en las enzimas PncA y en PanD.48,50 La mayoría de las mutaciones que confieren resistencia a pirazinamida están asociadas a PanD, ubicadas cerca del centro catalítico, interactuando con aminoácidos que encierran el sitio activo.50 Se piensa que la resistencia intrínseca en MNT se debe a que poseen proteínas de secreción que eliminan al ácido pirazinóico (POA, por sus siglas en inglés) evitando la inhibición de la enzima PanD.51
Aminoglucósidos
Los aminoglucósidos son azúcares hidrofílicos que incluyen kanamicina, amikacina, gentamicina y tobramicina. Éstos inhiben la síntesis de proteínas y se utilizan como fármacos de segunda línea para atender la tuberculosis.16 Además, son empleados a modo de complemento contra MNT de crecimiento lento. La amikacina y la tobramicina se utilizan comúnmente en enfermos con lesiones pulmonares cavitarias extensas o enfermedad diseminada por MNT, al igual que en aquellos incidentes donde ha fallado la terapia farmacológica previa.52 Su uso es común para el tratamiento de infecciones causadas por M. avium y M. kansasii, mientras que es limitado contra MNT de crecimiento acelerado.41
Los aminoglucósidos se unen a la subunidad ribosómica bacteriana 30s, causando falla en la traducción proteica y provocando la muerte celular. Éstos se unen al ribosoma cerca del sitio A, lo que altera la unión al tRNA, obstaculizando la decodificación de mRNA, aunque algunos aminoglucósidos pueden inhibir la translocación ribosómica del tRNA.53,54 Los aminoglucósidos igualmente interrumpen la función de revisión en el sitio A, lo que puede conducir a errores de cambio de marco de lectura, interfiriendo en la elongación proteica.55,56 El mecanismo primario de resistencia obtenida a los aminoglucósidos en micobacterias son mutaciones generadas en la subunidad 30S del ribosoma, en el gen rRNA 16s o el gen rpsL, que codifica a la proteína ribosómica S12.57,58 A pesar de la similitud química entre los diferentes aminoglucósidos, la resistencia a un agente no necesariamente resulta en insensibilidad a todos.59 Por ejemplo, una mutación en la posición 1408 del gen rRNA 16s en M. abscessus le confiere tolerancia ante amikacina y kanamicina, pero aún es sensible a la estreptomicina.60 Las variaciones del gen rRNA y rpsL representan hasta el 90 % de la resistencia adquirida a aminoglucósidos.61
Macrólidos
Son un grupo de antibióticos caracterizados por tener un anillo macrocíclico de lactona con 14 a 16 átomos, como la eritromicina, o la claritromicina y la azitromicina que son sus derivados sintéticos.62 Los macrólidos se utilizan comúnmente como método complementario en infecciones por MNT.63 Se consideran agentes bacteriostáticos en concentraciones clínicamente útiles y actúan inhibiendo la síntesis de proteínas, uniéndose en el sitio de salida del péptido en el ribosoma, evitando así la elongación de la cadena polipeptídica naciente.62
Los mecanismos de mutación adquirida en micobacterias están relacionados con modificaciones en el gen del rRNA 23s.64 Recientemente, se han descrito elementos de resistencia innata en micobacterias, conferida por los genes erm que codifican para metilasas de rARN. Estas enzimas metilan adeninas en la región de la peptidiltransferasa del rRNA 23s, lo cual evita la unión de los macrólidos al ribosoma. Se han descrito diversas metilasas erm inducibles en varias especies de MNT, como: M. smegmatis, M. boenickei, M. houstonense, M. neworleansense, M. porcinum, M. abscessus, MAC, M. chelonae, M. fortuitum y M. kansasii.61
Fluoroquinolonas
Las fluoroquinolonas inhiben a la DNA girasa, una topoisomerasa tipo II que participa en relajar el superenrollamiento dependiente de ATP del DNA circular antes de la replicación. Al inhibir a la DNA girasa, el DNA permanecerá superenrollado, evitando su síntesis.65 La enzima es un tetrámero compuesto por dos subunidades A y dos B, que están codificadas por los genes gyrA y gyrB, respectivamente. La DNA girasa es inhibida por las quinolonas mediante la formación de un complejo ternario entre el DNA, la quinolona y la DNA girasa, lo que resulta en la inhibición de la síntesis de DNA.66,67 La topoisomerasa IV también se inhibe por las quinolonas; esta enzima separa las cadenas de DNA recién sintetizadas, de modo que las moléculas puedan segregarse durante la división celular.61 Los mecanismos de resistencia innata a las fluoroquinolonas no son claros, ya que usualmente éstas se unen a las DNA girasas de diferentes micobacterias,65,68 sin embargo, M. avium es naturalmente resistente, M. smegmatis es moderadamente susceptible, y M. fortuitum bv.peregrinum es susceptible a las fluoroquinolonas.
La resistencia nativa de cada especie micobacteriana a las quinolonas puede ser por la permeabilidad diferencial en la pared de las células. Se ha propuesto que las proteínas de secreción promuevan la salida de las quinolonas, generando insensibilidad a este antibiótico.69,70 Por ejemplo: la proteína de secreción LfrA está regulada por LfrR71 y, por lo tanto, es posible que el fenotipo de tolerancia al fármaco se correlacione con el nivel de expresión del gen lfrA. Existe evidencia de que algunas micobacterias pueden tener otras enzimas de secreción que expulsan a las fluoroquinolonas, pero aún no están caracterizadas.68
Bedaquilina
La bedaquilina es una diarilquinolina, un fármaco de segunda línea, efectivo en el tratamiento de tuberculosis multifarmacorresistente (MDR) que igualmente es utilizada para enfrentar infecciones por M. abscessus y micobacterias del complejo M. avium.72 La bedaquilina inhibe la actividad de la adenosín trifosfato (ATP) sintasa micobacteriana codificada por atpE (Rv1305), que participa en la síntesis de ATP, el suministro de energía esencial.73,74 Se reportó que sólo 28 % de las cepas de M. tuberculosis identificadas como resistentes a bedaquilina tenían modificaciones en atpE.75 Las mutaciones en los genes mmpR5 y pepQ, que están involucradas en la oposición a la clofazimina, también conducen a la resistencia cruzada a la bedaquilina en aislados clínicos de M. tuberculosis.76,77
En aislados clínicos de MNT, se han reportado cepas que adquirieron resistencia, principalmente en M. avium, M. intracellulare, M. kansasii, M. abscessus, M. massiliense y M. fortuitum.78 En cepas de M. intracellulare resistentes a bedaquilina se han identificado variaciones en mmpT5, que codifica un regulador transcripcional perteneciente a la superfamilia TetR.79 Un estudio reciente de 197 aislados clínicos de M. abscessus reportó que la mayoría presentaron menor sensibilidad a la bedaquilina y 72 % presentaron alteraciones en el gen MAB4384, que es homólogo a mmpR5 en M. tuberculosis.80
Delamanida
Este fármaco es un agente antituberculoso, utilizado en el combate contra la tuberculosis MDR, y asimismo tiene actividad contra aislados clínicos de MNT de crecimiento rápido y lento. Sin embargo, hay diferencias en la concentración mínima inhibitoria entre las especies, por lo que es necesaria la determinación de la susceptibilidad in vitro antes de la administración del medicamento en pacientes.81
La delamanida es un derivado de nitro-dihidro-imidazol que pertenece a la clase de nitroimidazoles e inhibe la síntesis de ácidos micólicos, evitando la formación de la pared celular.82 La delamanida es un profármaco inactivo, y en M. tuberculosis, se cree que es metabolizado por la nitrorreductasa Ddn (Rv3547), dependiente del cofactor F420 o deazaflavina para tener su forma activa; el cofactor F420 es reciclado en forma reducida por la glucosa-6-fosfato deshidrogenasa 1, Fgd1 (Rv0407).83 Se ha reportado que ciertas mutaciones en los genes ddn o fgd1 están asociadas con la resistencia a PA-824, un nitroimidazol, pero son alteraciones poco comunes.84 El cofactor F420 es sintetizado por productos de los genes fbiA, fbiB y fbiC;85 se encontró una mutación en fbiA (D49Y) en un aislado clínico de M. tuberculosis resistente a delamanida.86 En 30 aislados de M. bovis BCG resistentes seleccionados in vitro, todos tenían modificaciones en uno de los cinco genes, ddn, fgd1, fbiA, fbiB o fbiC, y dos aislados clínicos de M. tuberculosis tenían variaciones en ddn.83 Hasta el momento no se han reportado mudanzas en cepas de MNT que confieran insensibildiad a este antibiótico.
El conocimiento de los mecanismos intrínsecos de resistencia de las MNT a los fármacos antimicobacterianos permitirá realizar una elección adecuada del régimen a seguir en estas infecciones. En la Tabla 4 se indican los componentes de resistencia en las MNT.
Tabla 4: Mecanismos de resistencia en micobacterias no tuberculosas.
| Fármaco | Modo de acción | Molécula blanco | Mecanismo de resistencia |
|---|---|---|---|
| Isoniazida | Inhibición de la síntesis de ácidos micólicos | InhA | *Mutación en katG o inhA (pérdida de la activación del profármaco) |
| Rifampicina | Inhibición de la síntesis de RNA | RNA polimerasa | ‡Mutación en rpoB (inhibición de la ribosilación de ADP) |
| Etambutol | Inhibición de la síntesis de arabinogalactanos de la pared celular | Arabinosil tranferasa | ‡Mutación en embB, embR o en el operón emb |
| Pirazinamida | Inhibición de síntesis de la vitamina B5 y la coenzima A | PanD | *Sistema de secreción que elimina POA |
| Aminoglucósidos | Inhibición de la síntesis de proteínas | Ribosoma | ‡Mutación en el rRNA 16s, gen rpsL, aminoglucósido transferasa |
| Fluoroquinolonas | Inhibición de la síntesis de DNA | DNA girasa | * ,‡Mutación en gyrA y ‡mutación en lfrA, proteína de secreción |
| Bedaquilina | Inhibición de ATP | ATP sintasa | ‡Mutación en atpE, mmpR5, pepQ, mmpT5 |
| Delamanida | Inhibición de la síntesis de ácidos micólicos | Nitro reductasa | ‡Mutación en ddn, fgd1, fbiA, fbiB, fbiC |
*Mutación natural o intrínseca,
‡Mutación adquirida.
inhA = enoil acil reductasa; RNA = ácido ribonucleico por sus siglas en inglés; POA = ácido pirazinóico por sus siglas en inglés; PanD = aspartato decarboxilasa por sus siglas en inglés; DNA = ácido desoxiribonucléico por sus siglas en inglés; ATP = adenosín trifosfato.
La tolerancia a los antimicrobianos de M. tuberculosis se ha descrito desde 1994, año en el que la Organización Mundial de la Salud (OMS) inició el Proyecto Mundial de Vigilancia de la farmacorresistencia, a fin de sistematizar los métodos de laboratorio utilizados para determinar la insensibilidad y la prevalencia. Con este esquema de vigilancia se ha reportado una prevalencia de farmacorresistencia de más de 4 % de los nuevos casos de tuberculosis en Europa Oriental, América Latina, África y Asia.87 En 2018, se reportaron entre 417,000 a 556,000 incidentes de tuberculosis resistente a rifampicina, de los cuales 78 % eran multifarmacorresistentes, es decir, también resistentes a isoniazida. Estos eventos correspondieron a 3.4 % de los sucesos nuevos y a 18 % de aquellos con abordaje previo, según el reporte de la OMS.88 Es importante conocer la tolerancia a los antimicrobianos en tuberculosis y en la infección de pulmones generada por MNT, con el fin de personalizar el tratamiento, buscando determinar el tipo de fármacos y el tiempo de administración. A pesar de la insensibilidad descrita en tuberculosis, se sabe muy poco de la incidencia de casos de resistencia por MNT, quizá debido a que el impacto en la mortalidad no es tan grave como en el caso de la tuberculosis a nivel mundial.
A pesar de que se desconoce la prevalencia de farmacorresistencia en eventos de infección pulmonar por MNT, se han reportado eventos aislados de insensibilidad adquirida a rifampicina,40 etambutol43,44 y bedaquilina,78,79 así como para ciertos aminoglucósidos como amikacina y kanamicina.60
Tratamiento de la infección pulmonar por MNT
La decisión terapéutica en la enfermedad pulmonar por MNT debe tomarse en función de los riesgos y beneficios potenciales para cada paciente, considerando las comorbilidades y la edad, ya que implica el uso prolongado de múltiples antibióticos.21 El régimen se formula de acuerdo con las pautas establecidas, basadas en la opinión de expertos en lugar de ensayos clínicos aleatorios.89-91 A pesar del cumplimiento de las pautas actuales, el combate de la afección pulmonar por MNT es deficiente.92 En la Tabla 5 se enlistan algunos fármacos que han tenido respuesta favorable para el tratamiento de MNT, según las recomendaciones de la ATS y la Sociedad Americana de Enfermedades Infecciosas (IDSA, por sus siglas en inglés Infectious Diseases Society of America), en comparación con las emitidas por la Sociedad Británica de Tórax (BTS, por sus siglas en inglés, British Thoracic Society).16,93
Tabla 5: Fármacos utilizados para el tratamiento de infecciones por micobacterias no tuberculosas.
| Especie | Guía ATS/IDSA | Guía BTS |
|---|---|---|
| M. abscessus | Resección quirúrgica mínima | Aislados sensibles a claritromicina o con resistencia inducida a macrólidos |
| Macrólidos (claritromicina); amikacina intravenosa; estreptomicina o cefotaxima | Fase inicial: ≥ un mes | |
| Amikacina, tigeciclina o imipenem vía intravenosa; claritromicina o azitromicina oral | ||
| Fase de continuación: | ||
| Amikacina nebulizada y claritromicina o azitromicina más clofazimina oral; linezolida, minociclina, moxifloxacina o cotrimoxazol oral, según las PSA y la tolerancia del paciente | ||
| Aislados con resistencia intrínseca a macrólidos | ||
| Igual que el anterior, excepto que se omite claritromicina en ambas fases | ||
| Complejo M. avium | Infección no grave | Infección no grave |
| Claritromicina o azitromicina, etambutol y rifampicina | Rifampicina, etambutol, azitromicina o claritromicina. La administración es tres veces por semana | |
| Infección grave | Infección grave | |
| Claritromicina, azitromicina, etambutol, rifampicina, fármacos intravenosos como amikacina o estreptomicina | Rifampicina, etambutol, azitromicina o claritromicina; amikacina intravenosa o nebulizada hasta por tres meses | |
| Aislados resistentes a claritromicina | ||
| Rifampicina, etambutol, isoniazida (+piridoxina) o moxifloxacina; amikacina intravenosa o nebulizada hasta por tres meses | ||
| M. kansasii | Aislados sensibles a rifampicina | Aislados sensibles a rifampicina |
| Rifampicina, etambutol, isoniazida, piridoxina. | Rifampicina, etambutol, isoniazida (+piridoxina), azitromicina o claritromicina. La administración es diaria | |
| La administración es diaria | Aislados resistentes a rifampicina | |
| Aislados resistentes a rifampicina | Régimen de tres medicamentos con base en PSA | |
| Régimen de tres medicamentos con base en PSA | ||
| M. malmoense | Isoniazida, rifampicina, etambutol, con o sin quinolonas y macrólidos con base en PSA | Infección no grave |
| Rifampicina, etambutol, azitromicina o claritromicina | ||
| Infección grave | ||
| Igual que el anterior más amikacina intravenosa o nebulizada hasta por tres meses | ||
| M. xenopi | Isoniazida, rifabutina o rifampicina, etambutol y claritromicina, con o sin un curso inicial de estreptomicina más una quinolona, de preferencia moxifloxacina, para ser sustituido por uno de los fármacos contra TB | Infección no grave |
| Rifampicina, etambutol, azitromicina o claritromicina, moxifloxacina o isoniazida (+ piridoxina). La administración es diaria | ||
| Infección grave | ||
| Igual que el anterior más amikacina intravenosa o nebulizada hasta por tres meses |
*ATS/IDSA = American Thoracic Society/Infectious Disease Society of America,
‡BTS = British Thoracic Society, cPSA = prueba de sensibilidad a los antibióticos.
Coinfección de MNT y M. tuberculosis
En la infección de pulmón causada por MNT, muchos síntomas son similares a la infección causada por M. tuberculosis, por lo que el diagnóstico de tuberculosis pulmonar podría ser sobreestimado. Sin embargo, igualmente podría deberse a una infección múltiple por cepas de M. tuberculosis y MNT, las cuales no se diagnostican de manera rutinaria. Las coinfecciones de MNT y M. tuberculosis a menudo son difíciles de detectar, debido a que los síntomas clínicos no permiten distinguir una infección única de una coinfección; aunado a lo anterior, generalmente no se realizan pruebas microbiológicas para identificar los dos tipos de micobacterias, lo que lleva a un diagnóstico parcial y a un régimen médico inadecuado.94
Aunque las coinfecciones con ambos tipos de gérmenes suelen causar enfermedades pulmonares graves en sujetos inmunocomprometidos y en sujetos con trastorno pulmonar subyacente, asimismo se han reportado casos de coinfección en sujetos inmunocompetentes sin trastorno pulmonar previo.95-97 En la India se han identificado coinfecciones de cepas de MNT en personas infectadas con cepas de tuberculosis resistentes a fármacos de primera línea (MDR).98 Incluso, se han observado coinfecciones múltiples de MNT (M. avium y M. intracellulare) con M. tuberculosis.99 También en China se han reportado coinfecciones de MNT en pacientes infectados con cepas de micobacterias extensamente resistentes a los antibióticos (XDR), siendo la coinfección con M. intracellulare la más frecuente.100 Con base en los estudios anteriores, es esencial llevar a cabo un proceso estricto con tal de identificar a los afectados con coinfecciones por MNT y M. tuberculosis, para que se les pueda ofrecer un tratamiento adecuado y se cuente con el registro de los eventos, con el fin de reportar la prevalencia y la incidencia en cada país (Figura 1).
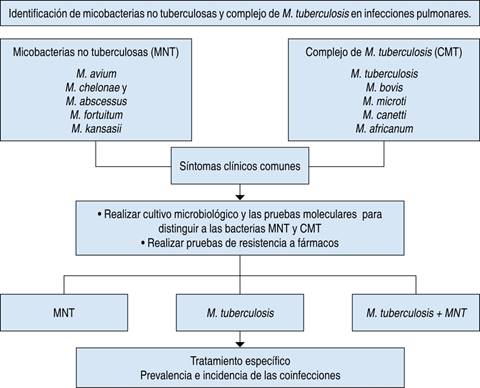
Figura 1: Las infecciones pulmonares por micobacterias no tuberculosas (MNT) y por el complejo Mycobacterium tuberculosis (CMT) son indistinguibles clínica y radiológicamente, por lo que es indispensable realizar cultivos, pruebas diferenciales y pruebas de sensibilidad a antibióticos para diagnóstico específico de cada especie, de manera que pueda ofrecerse un tratamiento dirigido a cada paciente. El conocimiento de la epidemiología de las coinfecciones entre micobacterias permitirá a los clínicos sospechar y tratar adecuadamente la enfermedad pulmonar causada por micobacterias. MNT = micobacterias no tuberculosas, CMT = complejo de M. tuberculosis.
Conclusión
Las infecciones por MNT representan un reto diagnóstico; en el caso de la enfermedad pulmonar, esto se debe a la gran similitud clínica y radiológica entre tuberculosis y las MNT, así como a la dificultad en la identificación microbiológica de cada especie, lo cual no se realiza de manera rutinaria en todos los centros hospitalarios, por la complejidad en el uso de los métodos bioquímicos o porque la infraestructura es insuficiente para desarrollar técnicas como la cromatografía y la biología molecular. De igual manera, el régimen es difícil, considerando las características genéticas, bioquímicas y metabólicas de cada especie de MNT, que les confieren resistencia natural o adquirida hacia los fármacos, y asimismo la discrepancia en la actividad antimicrobiana in vitro e in vivo, que puede entorpecer la decisión terapéutica inicial y llevar a esquemas prolongados de tratamiento, con el consecuente desarrollo de resistencia antimicrobiana y/o de efectos adversos indeseables en los pacientes. Finalmente, el conocimiento de la epidemiología de las coinfecciones entre micobacterias permitirá a los clínicos sospechar y tratar adecuadamente el padecimiento pulmonar causado por dichos patógenos, y llevar a cabo esquemas óptimos de medicación con base en la identificación de la cepa causante de la infección, en beneficio del paciente.











 text new page (beta)
text new page (beta)


