Introducción
Las garrapatas ocasionan pérdidas económicas en el ganado a nivel mundial, parasitan una amplia gama de huéspedes vertebrados y transmiten una gran variedad de agentes patógenos, más que cualquier otro grupo de artrópodos (Prudencio et al., 2010).
La garrapata Rhipicephalus microplus se alimenta de la sangre del ganado bovino, su hospedero definitivo, y es considerada como uno de los parásitos más dañinos en el ganado de zonas tropicales y subtropicales (Antunes et al., 2014).
La infestación por garrapata R. microplus afecta negativamente la productividad del ganado, transmite enfermedades tales como la babesiosis y la anaplasmosis (Jonsson, 2006). Las pérdidas económicas anuales en la industria ganadera son estimadas entre veintidós a treinta billones de dólares (Tabor et al., 2016), las cuales son resultado de la actividad directa en la alimentación de las garrapatas en su hospedero y del costo que ocasiona controlar tanto a las garrapatas como a las enfermedades producidas por los patógenos que ellas transmiten (Manjunathachar et al., 2014).
El método tradicional para el control de garrapatas está basado en el uso de químicos acaricidas a los cuales las garrapatas han desarrollado resistencia (Graf et al., 2004). Esto dificulta la erradicación y el control de las garrapatas, por lo que el uso de productos químicos no representa la mejor opción (Sonenshine y Roe, 2013). Tomando en cuenta la creciente preocupación por la seguridad del medio ambiente, la salud humana y la salud animal, se han vuelto necesarias las medidas de control alternativas (Abbas et al., 2014). Actualmente una estrategia para el control de la garrapata es el desarrollo de la inmunidad en el hospedero al administrar antígenos de la garrapata por medio de vacunas (Walker, 2009). La vacunación es considerada como una alternativa rentable, amigable con el medio ambiente, no genera contaminación residual en los productos de origen animal, es de fácil aplicación y es tecnología económica (Jonsson, 2006).
En los años noventa aparecieron comercialmente las vacunas contra la garrapata del ganado (Kaewmongkol et al., 2015). Estas vacunas están basadas en la forma recombinante del antígeno oculto Bm86 del intestino de R. microplus (García-García et al., 1998). Sin embargo, la eficacia de esta vacuna varía de acuerdo a los aislados existentes en las diferentes zonas geográficas (de la Fuente et al., 2007), lo cual puede ser debido a variaciones alélicas en el gen bm86 (De La Fuente y Kocan, 2006) (Sossai et al., 2005). Una diferencia del 3.4% en la secuencia de Bm86 entre las diferentes cepas puede ser suficiente para causar una respuesta inmune ineficiente (García-García et al., 1999). Se sugiere que existe una correlación inversa entre la eficacia de la vacuna y la variación del locus de bm86 (Freeman et al., 2010). Todo esto sugiere que un candidato vacunal debe ser conservado en su secuencia de aminoácidos entre distintas cepas o aislados para poder ser eficaz.
Existe un gran número de proteínas de garrapatas que han sido evaluadas como componentes para las vacunas, obteniendo diferentes porcentajes de protección, entre las que se encuentran BmVDAC y Subolesina (Taheri et al., 2014). La proteína BmVDAC está presente en las células del intestino de la garrapata, es una porina mitocondrial, interviene en regulación de la apoptosis en células infectadas, y tiene una sobre expresión importante durante la invasión de Babesia bigemina (Rodríguez-Hernández et al., 2015). La proteína Subolesina se localiza en diferentes órganos de la garrapata como los ovarios, las glándulas salivales y el intestino. Además, interviene en la digestión de la sangre, la reproducción y el desarrollo de la garrapata (de la Fuente et al., 2008). Un aspecto importante en la selección de antígenos que protejan contra las garrapatas, es la evaluación de la variabilidad alélica de los candidatos vacunales en las cepas de garrapatas de diferentes zonas geográficas; la identificación de secuencias peptídicas conservadas en estos candidatos vacunales permitirá obtener una vacuna con una protección elevada contra las diferentes cepas (de la Fuente y Kocan, 2003) (Manjunathachar et al., 2014). Derivado de lo anterior, el objetivo del presente trabajo fue analizar la variabilidad alélica en las secuencias de los antígenos vacunales BmVDAC y Subolesina de R. microplus en cepas provenientes de diferentes estados de México.
Método
Selección de proteínas de R. microplus
Se seleccionaron dos proteínas con base en su eficacia como antígenos candidatos vacunales, las cuales fueron: BmVDAC y Subolesina. Se obtuvieron las secuencias de referencia de la base de datos National Center of Biotechnology Information (NCBI), con los números de acceso GU994210.1y ABA62328.1, respectivamente.
Colección de garrapatas R. microplus de México
Los aislados seleccionados de R. microplus fueron colectados de los estados de Jalisco, Tabasco, Chiapas, Nayarit, Querétaro, Tamaulipas, Guerrero y Sinaloa, además de la cepa de referencia Media Joya. Estos se utilizaron para amplificar el gen bmvdac. Para el gen subolesina, se utilizaron los aislados provenientes de Yucatán, Nayarit, Chiapas, Tamaulipas, Querétaro, Sinaloa y Veracruz, además de la cepa Munoz utilizada como referencia. Por lo menos 30 hembras adultas de garrapatas R. microplus vivas fueron colectadas de distintos ranchos de cada estado. Fueron llevadas al laboratorio donde fueron lavadas y desinfectadas con benzal al 10%, se secaron con toallas de papel e inmediatamente fueron congeladas a -20C hasta su uso.
Diseño de iniciadores
Para el diseño de iniciadores para amplificar cada uno de los genes seleccionados se utilizaron los programas, Primer3web versión 4.1.0 (http://primer3.ut.ee/); el programa Primer Blast de la base de datos National Center of Biotechnology Information (NCBI) (www.ncbi.nlm.nih.gov/tools/primer-blast/) y finalmente, el programa GENtle (http://gentle.magnusmanske.de/).
Amplificación de los genes en los diferentes aislados
Los iniciadores utilizados para la amplificación de los genes en los diferentes aislados de R. microplus fueron, para el gen bmvdac: 5’ ATGGCTCCTCCGTGCTACGC 3’ (sentido) y 5’ CTTGTGTCCTCCCTGGTTGAA 3’ (antisentido), utilizando como referencia la secuencia de bmvdac reportada en GenBank (cepa Media Joya) número de acceso GU994210.1. Para la amplificación del gen subolesina, la secuencia fue obtenida del GenBank (cepa Munoz) con número de acceso ABA62328.1. Los iniciadores utilizados fueron: 5’ AAAGCGAACACATGACTGGG3’ (sentido) y 5’ TCCCGTATCTTGCTCTCTCG 3’ (anti sentido). Para amplificar el gen bmvdac, el ADN genómico fue extraído de los ocho diferentes aislados de R. microplus de México mediante el kit DNeasy Blood & Tissue (Quiagen, Hiden, Alemania), de acuerdo a las indicaciones del proveedor. Para el gen subolesina, la transcriptasa reversa (RT)-PCR fue utilizada para amplificar el mARN extraído utilizando el kit EZ-10 Spin Column Total RNA Miniprep (Bio Basic.inc, Toronto, Canadá) de los diferentes aislados de R. microplus provenientes de Yucatán, Nayarit, Tamaulipas, Chiapas, Querétaro, Sinaloa y Veracruz, el cADN fue sintetizado con SuperScript III Firs-Strand Synthesis System Kit (Invitrogen, Carlsbad, CA, USA) de acuerdo a las intrucciones del proveedor.
Para la reacción de PCR se utilizó un volumen de reacción total 20 μL: ADN 1 μL, (50 ng/μL), 1 μL de cada iniciador, 10 μL de PCR Master Mix, Promega, Wisconsin, USA) y 7 μL de agua libre de nucleasas.
La reacción se llevó a cabo bajo las siguientes condiciones: 1 ciclo de desnaturalización a 95°C por 3 minutos seguida de 34 ciclos de desnaturalización a 95°C por 30 segundos, alineamiento de 56°C por 30 segundos y una extensión a 72°C por 1 minuto y el paso final de extensión a 72°C por 5 minutos.
Los productos de PCR se utilizaron para realizar la electroforesis en gel de agarosa a una concentración de 1.5%. Los geles fueron observados en el fotodocumentador para verificar que las amplificaciones presentaran la medida correcta, 780 pb para bmvdac y 442 pb para subolesina. Las bandas de los amplicones esperados fueron cortados del gel de agarosa y purificados utilizando el kit comercial de purificación de ADN Wizard SV Gel and PCR Clean-Up System (Promega, USA). El ADN purificado fue enviado a secuenciar por el método automatizado de Sanger al Instituto de Biotecnología (IBT) de la Universidad Nacional Autónoma de México (Cuernavaca, Morelos, México).
Obtención de secuencias consenso, alineamiento múltiple y análisis bioinformático
A las secuencias de nucleótidos obtenidas del análisis y ensamblaje del resultado de la secuenciación se les realizó un análisis de búsqueda de marco de lectura abierto u ORF (Open Reading Frame) mediante el algoritmo bioinformático ORF finder (http://www.ncbi.nlm.nih.gov/projects/gorf/) que busca en la secuencia de nucleótidos la presencia de codones de inicio y de paro necesarios para su traducción a aminoácidos. De esta forma se obtuvo la secuencia de aminoácidos predicha correspondiente a cada gen de los diferentes aislados. Los análisis siguientes fueron realizados con estas secuencias.
Las secuencias de los diferentes aislados seleccionados fueron analizadas mediante el programa de alineamiento multiple Clustal Omega y el programa BLAST (Basic Logical Aligment Search Tool) para la identificación de las regiones conservadas.
Para el análisis de identidad y similitud entre las diferentes secuencias se utilizó el programa SIAS (Secuence Identites and Similarities) http://imed.med.ucm.es/Tools/sias.
Análisis filogenético
Los árboles filogenéticos fueron costruídos basados en el Método UPGMA (Unweighted Pair Group Method using Arithmetic averages) utilizando el programa MEGAX64 (Molecular Evolutionary Genetics Analysis software) (Tamura et al., 2013). Para evaluar la confiabilidad de las topologías del árbol inferidas, los análisis se realizaron utilizando el método estadístico Bootstrap con 10000 réplicas (Felsenstein, 1985). Para bmvdac se utilizó como grupo externo a Ixodes scapularis con número de acceso de GenBank XP_002408065.1 y Amblyomma variegatum con número de acceso BK007268.1. Para subolesina se utilizó como grupo externo a Ixodes ricinus con número de acceso de GenBank ABA62325.1. y Amblyomma cajennense con número de acceso AGI44599.1.
Resultados
Los aislados de R. microplus fueron obtenidos de diferentes zonas geográficas de México como se muestra en la Tabla 1.
Tabla 1 Localización de las zonas geográficas de México de aislados de garrapatas R. microplus incluidas en el presente estudio.
| Habitad de las garrapatas |
Estado | Genes amplificados | Localización (Latitud, longitud) |
|
|---|---|---|---|---|
| bmvdac | subolesina | |||
| Noreste | Tamaulipas | ✓ | ✓ | 22°39'35.6"N 98°15'14.1"W |
| Centro | Querétaro | ✓ | ✓ | 21°11'00.03"N 99°16'38.8"W |
| Sureste | Tabasco | ✓ | 17°48'35.6"N 93°23'42.8"W |
|
| Noroeste | Sinaloa | ✓ | ✓ | 23°17'18.8"N 106°25'02.3"W |
| Suroeste | Chiapas | ✓ | 16°18'11.8"N 91°55'29.9"W |
|
| Suroeste | Guerrero | ✓ | 18°22'01.0"N 100°37'57.6"W |
|
| Oeste | Jalisco | ✓ | 19°56'33.4"N 103°45'47.8"W |
|
| Oeste | Nayarit | ✓ | ✓ | 21°32'34.05"N 104°56'71.09"\* |
| Sureste | Yucatán | ✓ | 20°52'56.82"N 89°37'55.85"W |
|
| Este | Veracruz | ✓ | 20°05'12.5"N 97°02'43.7"W |
|
La secuencia utilizada como referencia para bmvdac corresponde a un fragmento de 819 pares de bases y va de los nucleótidos 1 al 819 de la cepa Media Joya de Tapalpa, Jalisco, reportada previamente (Rodríguez-Hernández et al., 2015), y de ese fragmento fueron amplificadas 750 pares de bases para el presente estudio. Este fragmento corresponde al 91.57% de la secuencia de referencia, y para subolesina la secuencia de referencia corresponde al fragmento de 442 pares de bases y va del nucleótido 1 al 442 de la cepa Munoz usada como referencia y publicada anteriormente (Almazán et al., 2010). De ese fragmento fueron amplificados 438 pares de bases que corresponden al 99.3% de la secuencia utilizada como referencia.
Para el alineamiento múltiple de bmvdac, se analizaron ocho aislados de R. microplus además de la secuencia de referencia reportada en GenBank (Fig. 1). En donde se observan las zonas conservadas entre las diferentes secuencias, los aislados que presentan diferencias en este alineamiento son, Jalisco, Tabasco, Sinaloa, Nayarit y Chiapas, las cuales presentan solamente un aminoácido diferente.

Fig. 1 Alineamiento múltiple de las secuencias de Jalisco, Tabasco, Chiapas, Nayarit, Querétaro, Tamaulipas, Guerrero, Sinaloa y la cepa de referencia GU994210.1. En amarillo están marcadas las regiones conservadas de las diferentes secuencias para el gen bmvdac. Se utilizó el programa CLUSTAL O (1.2.4) múltiple sequence alignment.
En promedio de observa un porcentaje del 99.79% de identidad y similitud. En seis de las nueve secuencias utilizadas provenientes de las diferentes zonas geográficas de México, se observó un porcentaje tanto de identidad como de similitud del 99.87% en las secuencias de aminoácidos de Chiapas, Nayarit, Media Joya, Querétaro, Tamaulipas y Guerrero. En las secuencias provenientes de Jalisco, Tabasco y Sinaloa presentaron el 99.56% en identidad y similitud. En promedio se observa un porcentaje de variabilidad de 0.21% entre las secuencias de bmvdac, como se indica en Tablas 2 y 3.
Tabla 2 Porcentajes de identidad entre las secuencias de aminoácidos de los aislados provenientes de los diferentes estados de la República Mexicana para bmvdac. Se utilizó el programa SIAS, Sequences Identites and Similarities.
| AISLADOS | QUERÉTARO | NAYARIT | JALISCO | GUERRERO | TABASCO | TAMAULIPAS | CHIAPAS | SINALOA | GU994210.1. |
| QUERÉTARO | 100% | ||||||||
| NAYARTT | 100% | 100% | |||||||
| JALISCO | 99.60% | 99.60% | 100% | ||||||
| GUERRERO | 100% | 100% | 99.60% | 100% | |||||
| TABASCO | 99 60% | 99.60% | 99.21% | 99.60% | 100% | ||||
| TAMAULIPAS | 100% | 100% | 99.60% | 100% | 99.60% | 100% | |||
| CHIAPAS | 100% | 100% | 99.60% | 100% | 99.60% | 100% | 100% | ||
| SINALOA | 99.60% | 99.60% | 99.21% | 99.60% | 99.21% | 99.60% | 99.60% | 100% | |
| GU994210.1. | 100% | 100% | 99.60% | 100% | 99.60% | 100% | 100% | 99.60% | 100% |
Tabla 3 Porcentajes de Similitud entre las secuencias de aminoácidos de los aislados provenientes de los diferentes estados de la República Mexicana para bmvdac. Se utilizó el programa SIAS, Sequences Identites and Similarities.
| AISLADOS | QUERÉTARO | NAYARIT | JALISCO | GUERRERO | TABASCO | TAMAULIPAS | CHIAPAS | SINALOA | GU994210.1. |
| QUERÉTARO | 100% | ||||||||
| NAYARTT | 100% | 100% | |||||||
| JALISCO | 99 60% | 99.60% | 100% | ||||||
| GUERRERO | 100% | 100% | 99.60% | 100% | |||||
| TABASCO | 99.60% | 99.60% | 99.21% | 99.60% | 100% | ||||
| TAMAULIPAS | 100% | 100% | 99.60% | 100% | 99.60% | 100% | |||
| CHIAPAS | 100% | 100% | 99.60% | 100% | 99.60% | 100% | 100% | ||
| SINALOA | 99.60% | 99.60% | 99.21% | 99.60% | 99.21% | 99.60% | 99.60% | 100% | |
| GU994210.1. | 100% | 100% | 99.60% | 100% | 99.60% | 100% | 100% | 99.60% | 100% |
En el alineamiento múltiple de subolesina con siete diferentes aislados provenientes de Yucatán, Nayarit, Querétaro, Chiapas, Sinaloa, Veracruz y Tamaulipas y la cepa Muñoz de referencia (Fig. 2), se observan las zonas conservadas entre las diferentes secuencias, los aislados que presentan diferencias en este alineamiento son, Sinaloa, Nayarit y Yucatán.

Fig. 2 Alineamiento multiple de las secuencias de Yucatán, Nayarit, Querétaro, Sinaloa, Veracruz, Tamaulipas y la cepa de referencia ABA62328.1. En amarillo se encuentran marcadas las regiones conservadas de las diferentes secuencias para el gen subolesina. Se utilizó el programa CLUSTAL O (1.2.4) múltiple sequence alignment.
Además, se observó un porcentaje promedio de identidad del 99.87% y similitud del 100% entre las secuencias de aminoácidos. De las ocho secuencias, siete de ellas presentaron procentaje de identidad del 99.91%, mientras que para la secuencia de Nayarit el porcentaje fue de 99.39% (Tabla 4 y 5). La variabilidad promedio observada entre las secuencias de aminoácidos fue del 0.13%.
Tabla 4 Porcentajes de identidad entre las secuencias de aminoácidos de los aislados provenientes de los diferentes estados de la República Mexicana para subolesina. Se utilizó el programa SIAS, Sequences Identites and Similarities.
| AISLADOS | ABA62328.1 | CHIAPAS | NAYARTT | QUERÉTARO | TAMAULIPAS | VERACRUZ | SINALOA | YUCATÁN |
| ABA62328.1 | 100% | |||||||
| CHIAPAS | 100% | 100% | ||||||
| NAYARTT | 99.31% | 99.31% | 100% | |||||
| QUERÉTARO | 100% | 100% | 99.31% | 100% | ||||
| TAMAULIPAS | 100% | 100% | 99.31% | 100% | 100% | |||
| VERACRUZ | 100% | 100% | 99.31% | 100% | 100% | 100% | ||
| SINALOA | 100% | 100% | 99.30% | 100% | 100% | 100% | 100% | |
| YUCATÁN | 100% | 100% | 99 30% | 100% | 100% | 100% | 100% | 100% |
Tabla 5 Porcentajes de similitud entre las secuencias de aminoácidos de los aislados provenientes de los diferentes estados de la República Mexicana para subolesina. Se utilizó el programa SIAS, Sequences Identites and Similarities.
| AISLADOS | ABA62328.1 | CHIAPAS | NAYARTT | QUERÉTARO | TAMAULIPAS | VERACRUZ | SINALOA | YUCATÁN |
| ABA62328.1 | 100% | |||||||
| CHIAPAS | 100% | 100% | ||||||
| NAYARTT | 100% | 100% | 100% | |||||
| QUERÉTARO | 100% | 100% | 100% | 100% | ||||
| TAMAULIPAS | 100% | 100% | 100% | 100% | 100% | |||
| VERACRUZ | 100% | 100% | 100% | 100% | 100% | 100% | ||
| SINALOA | 100% | 100% | 100% | 100% | 100% | 100% | 100% | |
| YUCATÁN | 100% | 100% | 100% | 100% | 100% | 100% | 100% | 100% |
Tanto los alineamientos multiples como los porcentajes de identidad y similitud resultantes en el presente estudio muestran la presencia de zonas conservadas entre las diferentes secuencias tanto para BmVDAC como para Subolesina; con base en lo reportado por García et al. (1999) y Freeman et al. (2010), referente a que el polimorfismo puede ser la causa de la variabilidad en la eficacia de la vacuna con el antígeno Bm86. BmVDAC y Subolesina, por su parte, presentan potencial para ser considerados como candidatos vacunales para el control de la garrapata R. microplus en México.
Los análisis filogenéticos mostraron similitudes muy elevadas en el árbol diseñado utilizando el método UPGMA. En el filograma construido para bmvdac con los aislados incluidos en el presente estudio se observan tres ramificaciones principales (Fig. 3), en la primera de ellas se encuentran, Chiapas, la cepa de referencia GU994210.1, Tamaulipas, Guerrero, Nayarit y Querétaro, las cuales presentan una misma secuencia genética localizada en diferentes zonas geográficas. Por otra parte, Sinaloa comparte una gran similitud con Tabasco y Jalisco. La segunda y tercera ramificación están conformada por las secuencias utilizadas como grupo externo (Amblyomma variegatum e Ixodes Scapuraris). El porcentaje obtenido del método Bootstrap de 10000 réplicas es mayor al 50% como se observa en los nodos respectivos (Xiong, 2006).
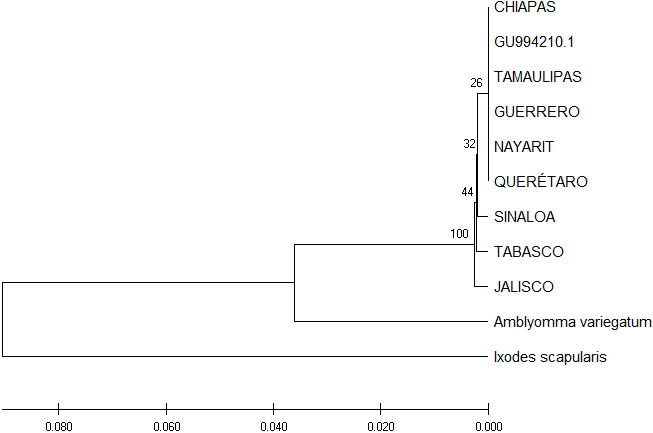
Fig. 3 Relación filogenética de los aislados de R. microplus para el gen bmvdac. El árbol filogenético fue construido utilizando el método UPGMA. Al lado izquierdo del nodo se encuentra el porcentaje de soporte del método Bootstrap utilizando 10000 réplicas.
El árbol filogenético para subolesina presenta tres ramificaciones (Fig. 4), en la primera, se encuentran una misma secuencia genética en los aislados de Sinaloa, Yucatán, Veracruz, Tamaulipas, Querétaro, Chiapas y la cepa de referencia ABA62328, por una parte y por la otra, se encuentra Nayarit. En la segunda y tercera ramificación se encuentra la secuencia de Amblyomma cajennense e Ixodes ricinus respectivamente, ambas utilizadas como grupo externo. El porcentaje obtenido del método Bootstrap de 10000 réplicas es del 82%.
Discusión y Conclusiones
Este reporte representa la primera investigación de la caracterización de la variabilidad alélica para para los genes bmvdac y subolesina, dos antígenos vacunales contra R. microplus entre aislados de diferentes estados en México. De acuerdo a lo reportado por Kaewmongkol et al., 2015, la variabilidad en la secuencia obtenida de la garrapata R. microplus para Bm86 es una posible explicación referente al rango de eficacia presentada en la vacuna contra este parásito, utilizando como antígeno la proteína recombinante Bm86. Debido a esto, para amplificar bmvdac y subolesina se seleccionaron aislados representativos de diferentes áreas geográficas de México, para de esta manera determinar la variabilidad entre las secuencias provenientes de diferentes estados del país.
De acuerdo a los datos publicados por García-García et al., 1999, la variabilidad superior al 3.4% entre las diferentes secuencias es suficiente para producir una respuesta inmune ineficiente, por lo que es importante destacar que la variabilidad entre las secuencias de aminoácidos de los aislados en el presente estudio es de un 0.21% para bmvdac y para subolesina se observa una variabilidad del 0.13%. Por consiguiente, con este análisis se considera que bmvdac y subolesina son antígenos que se encuentran conservados en las secuencias de las poblaciones de R. microplus de las diferentes zonas geográficas del país.
En el árbol filogenético para bmvdac se observa que las secuencias incluidas en la construcción del árbol presentan una misma secuencia genética localizada en seis diferentes zonas geográficas, lo cual indica que su alto porcentaje de similitud se encuentra disperso en las diferentes poblaciones de R. microplus; se observan diferencias en las secuencias de Jalisco, localizado en la zona oeste de México, Tabasco en el sureste y Sinaloa en el noroeste. En el árbol filogenético para subolesina, de las ocho secuencias incluidas para la construcción del árbol, siete de ellas provenientes de diferentes zonas geográficas comparten una misma secuencia genética, mientras que solo una de ellas, en este caso Nayarit localizado en la zona oeste del país, presenta variabilidad en su secuencia. Las diferencias observadas tanto bmvdac como en subolesina pueden ser debidas a la movilización del ganado del sur del país hacia los estados del norte y no a las diferentes zonas geográficas de donde provienen los aislados utilizados en el presente estudio, lo cual difiere de lo sugerido por De la Fuente et al., 2007. El porcentaje obtenido del método Bootstrap utilizando 10000 réplicas tanto para bmvdac como para subolesina es superior al 50%, lo cual indica un soporte estadístico fiable, de acuerdo a lo reportado por Xion et al., 2006.
Considerando la baja variabilidad alélica presentada entre las secuencias de los aislados de R. microplus de los diferentes estados de México, se concluye que tanto BmVDAC como Subolesina son antígenos que presentan secuencias altamente conservadas en aislados de México, por lo que pueden ser considerados como potenciales candidatos vacunales de acuerdo a lo reportado por Freeman et al., 2010, donde sugiere que existe una correlación inversa entre la eficacia de la vacuna y la variación del locus; por lo tanto, esta información es relevante para la selección de antígenos empleados en las vacunas contra garrapatas R. microplus. Se sugiere la realización de evaluaciones in vivo para determinar su efectividad como inmunógenos.











 text new page (beta)
text new page (beta)



