Introducción
Los rumiantes son mamíferos herbívoros y tienen el estómago compuesto de cuatro cavidades, por lo que también se les llama poligástricos, algunos ejemplos son la vaca, la cabra y la oveja (Servicio de Información Agroalimentaria y Pesquera, 2017). La ganadería de rumiantes tiene un doble papel, por un lado, el consumo de estos animales provee a los humanos de proteínas de alta calidad, vitaminas y minerales que no son fáciles de encontrar en otros alimentos (Wyness, 2016), pero también son la principal fuente antropogénica de metano (CH4), un gas de efecto invernadero (GEI) que tiene un poder de calentamiento 34 veces mayor que el dióxido de carbono (CO2) (Söllinger et al., 2018).
Se ha calculado que cada año estos animales liberan alrededor de 125 millones de toneladas de CH4 a la atmósfera (Seshadri et al., 2018). A pesar de esto, se estima que la producción de carne se ha cuadruplicado en los últimos 50 años y a nivel mundial se espera que las demandas de leche y carne de vaca se dupliquen para el año 2050 (Söllinger et al., 2018).
Entre los esfuerzos para generar una ganadería más sostenible, durante la Conferencia de las Naciones Unidas sobre el Cambio Climático-COP26 en noviembre 2021, se abordó este tema con el fin de crear conciencia sobre la importancia de una transición hacia sistemas de producción de alimentos más sustentables, que limiten las emisiones de gases provocadas por las liberaciones gastrointestinales y por el estiércol del ganado (Organización de las Naciones Unidas, 2021).
Los rumiantes fermentan el material vegetal que consumen produciendo acetato, butirato, propionato, CH4 y CO2. El CH4 es expulsado a la atmósfera mediante la exhalación o por el extremo terminal del tracto digestivo (Duin et al., 2016), este proceso es conocido como fermentación entérica y se lleva a cabo en el rumen, uno de los compartimentos esofágicos de los animales rumiantes. Los microorganismos presentes en el rumen además de degradar a los polisacáridos complejos que otorgan energía al animal, son responsables de la producción del CH4. Un hecho relevante es que la producción del CH4 representa una pérdida de energía para el animal de entre el 2 y 12%, e influye en la disminución del crecimiento animal o en la producción de leche, ambas importantes en la economía de la industria ganadera (Johnson & Johnson, 1995).
Las características funcionales del microbioma del rumen no han sido bien descritas debido a que la mayoría de los microorganismos no son cultivables en condiciones in vitro, pero el avance tecnológico permite que en la actualidad puedan ser estudiados mediante la metagenómica y la metatranscriptómica (Kaul, Sharma & Dhar, 2016). Es relevante hacer la distinción entre microbiota y microbioma, ya que el término microbiota se refiere al conjunto de microorganismos existente en una comunidad microbiana y el término microbioma hace referencia a la totalidad de genomas y transcriptomas existentes en el ambiente que se estudia (Li, Neves, Ghoshal & Guan, 2018).
Actualmente, se han identificado miles de filogenias microbianas utilizando técnicas moleculares cultivo-independientes para predecir las funciones de cada filotipo (Li et al., 2018), como se realizó en el proyecto pionero Hungate1000. El desarrollo de la metagenómica y la metatranscriptómica, en conjunto con métodos bioinformáticos, contribuyen a comprender mejor los mecanismos de fermentación ruminal con el fin de obtener información que pueda usarse como estrategia para mitigar la liberación del CH4, sin obstaculizar la productividad, ni dañar la función del animal (Wallace, Snelling, McCartney, Tapio & Strozzi, 2017), por consiguiente se exponen tanto el impacto que la industria ganadera tiene en el calentamiento global, como la importancia de los estudios que han utilizado la metagenómica y la metatranscriptómica.
Efecto de las emisiones de GEI por rumiantes
Descripción del Efecto Invernadero y sus consecuencias
Cuando el sol irradia energía a la Tierra en longitudes de onda cortas cargadas hacia la luz visible y ultravioleta, aproximadamente un tercio de ella se refleja de vuelta al espacio, los dos tercios restantes son absorbidos por la superficie terrestre y por la atmósfera en menor medida, Grupo Intergubernamental de Expertos sobre el Cambio Climático (IPCC), por sus siglas en inglés, 2007. La energía que es absorbida por la superficie terrestre calienta la Tierra y en consecuencia se emite radiación terrestre en longitudes de onda más largas, principalmente de la parte infrarroja del espectro (Caballero, Lozano & Ortega, 2007). Esa energía infrarroja emitida por la superficie terrestre es nuevamente detenida y absorbida por los GEI de la atmósfera, que como resultado calientan la troposfera que a su vez irradia calor, manteniendo a la Tierra a 33 grados Celsius más caliente de lo que estaría sin este fenómeno (Osnaya, Fernández Bremauntz & Martínez, 2004).
Los GEI incluyen el vapor de agua (H2O), el CO2, el CH4, el N2O y algunos productos químicos artificiales como los clorofluorocarbonos (CFC) (IPCC, 2007). Las propiedades de estos gases son parte fundamental para la regulación del clima en el planeta, no obstante, se ha incrementado su concentración debido a las actividades humanas, intensificando el efecto invernadero natural y contribuyendo al calentamiento global (Caballero et al., 2007). Las consecuencias al respecto incluyen un aumento en el nivel del mar e inundaciones costeras, la extinción de diversas especies, la acidificación del océano, riesgos para la seguridad alimentaria y periodos de calor extremo, por mencionar algunas (Hoegh-Guldberg et al., 2018).
Contribución del metano al efecto invernadero
El metano, también es emitido por (a) fuentes naturales que incluyen: el CH4 fósil de fuentes geológicas, emisiones por humedales, de termitas y del océano en pequeñas cantidades, al igual que por (b) fuentes antropogénicas como las emisiones causadas por fugas de la extracción, el uso de combustibles fósiles, quema de la biomasa vegetal (puede ser antropogénica y natural), agricultura de arrozales, tratamiento de desechos y (c) ganadería de rumiantes (Ciais et al., 2013). En la actualidad se estima que, del total de las emisiones de metano, entre el 50 y el 65% provienen de fuentes antropogénicas (Ciais et al., 2013) y se considera el segundo gas de efecto invernadero más abundante, solo por debajo del CO2 (Global Methane Emissions and Mitigation Opportunities [GMI], 2020). El CH4 tiene un tiempo de vida de solo 12 años en la atmósfera, pero debido a su gran poder de calentamiento en la actualidad ha contribuido aproximadamente con un tercio de los GEI (GMI, 2020).
Emisiones de los GEI por rumiantes
Para las emisiones de GEI generadas por la ganadería de rumiantes, se toman en cuenta diferentes actividades de la cadena de suministro de la industria. Entre ellas se encuentran las emisiones generadas por la producción de piensos (N2O y CO2), por la gestión del estiércol (N2O y CH4) y por la fermentación entérica (CH4) (Gerber et al., 2013).
De acuerdo con lo anterior, se cuantifica la huella del carbono contabilizando los diferentes GEI generados como equivalentes del dióxido de carbono, el valor se obtiene multiplicando la emisión de cada uno por su potencial de calentamiento global (GWP) en un periodo de 100 años (Ritchie & Roser, 2020).
Se estima que a nivel global los tres mayores gases de efecto invernadero emitidos por esta industria son el CH4, el CO2 y el N2O. Variaciones generales en las cantidades de GEI emitidos por estos animales dependen de aspectos como región, prácticas de manejo, calidad de la alimentación, salud y genética del animal (Opio et al., 2013). No obstante, se ha establecido que a nivel global el gas más emitido por la industria ganadera es el metano producido en la fermentación entérica (Opio et al., 2013).
De acuerdo con el Segundo Informe Bienal de Actualización ante la Convención Marco de las Naciones Unidas sobre el Cambio Climático (CMNUCC) del año 2018, las emisiones de México en el 2015 fueron de 699,564.28 Gigagramos de dióxido de carbono equivalente (Gg de CO2e), sin considerar a los sumideros de CO2 (Secretaría de Medio Ambiente y Recursos Naturales & Instituto Nacional de Ecología y Cambio Climático, 2018). De esta cantidad, el sector de la Agricultura contribuyó con el 15% (102,059.5 Gg de CO2e) de las emisiones totales del país, de las que el 10.1% (70,567.6 Gg de CO2e) fueron generadas por la ganadería. Para esta última, los GEI contabilizados fueron los generados en la fermentación entérica y en la gestión del estiércol (sin considerar las emisiones por la producción de piensos), que aportaron el 7.64% (53442.72 Gg de CO2e) y el 2.45% (17,124.88 Gg de CO2e) respectivamente. En la gestión del estiércol el 1.8% (12,688.80 Gg de CO2e) correspondió al CH4 y el 0.6% al óxido nitroso (4,436.08 Gg de CO2e) y en cuanto a la fermentación entérica la totalidad correspondió al metano (Secretaría de Medio Ambiente y Recursos Naturales & Instituto Nacional de Ecología y Cambio Climático, 2018) (Figura 1).

Figura 1 Porcentaje de emisiones de Gg de CO2e totales en México, modificado de Ritchie & Roser (2020).
Funcionamiento del sistema digestivo y meta-nogénesis ruminal
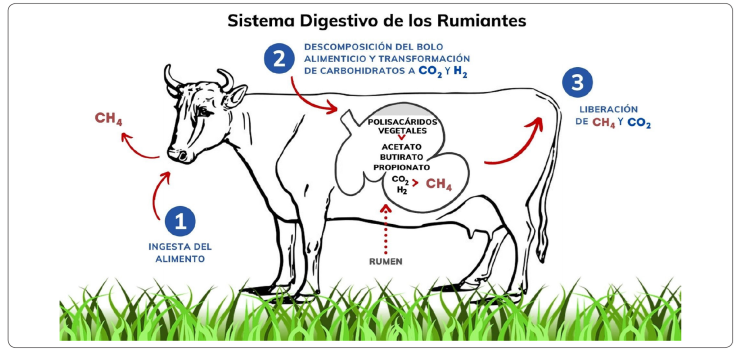
Los animales rumiantes presentan características morfológicas y fisiológicas digestivas que los distinguen de otro tipo de animales; la degradación de los alimentos que consumen se realiza en sus órganos localizados antes del duodeno (García, 2016). El funcionamiento digestivo de estos animales se lleva a cabo en dos etapas: el consumo de alimento y la rumia. Esta última etapa permite hacer un mejor aprovechamiento de los carbohidratos estructurales como la celulosa, hemicelulosa y pectina de las plantas consumidas (Del Rosario, 1999). El alimento se desplaza por el esófago hasta llegar al rumen (Parish, Rivera & Bolan, 2017), posteriormente, el rumiante regurgita el alimento hasta la boca para realizar el proceso de masticación, regresar el alimento al rumen y se lleva a cabo la digestión. Cuando el alimento regresa al rumen, este es fermentado y descompuesto, facilitando el aprovechamiento del alimento vegetal consumido por estos animales (Parish et al., 2017).
El estómago de un rumiante adulto puede llegar a ocupar el 75% de la cavidad abdominal representando el 30% de su peso vivo. Este se divide en cuatro cavidades, abomaso, omaso, retículo y rumen (García, 2016). Estos últimos tres son considerados como proventrículos debido a que están cubiertos por un epitelio queratinizado, mientras que el abomaso es conocido como “estómago verdadero”, ya que está cubierto por una membrana mucosa glandular (Giuliodori, Mattioli, Picco & Relling, 2013). El abomaso recibe compuestos de partículas no-fermentadas de alimentos, secreta enzimas digestivas y ácidos fuertes (Pérez & Sirias, 2007). En el omaso se separa el material sólido captado del contenido ruminal, permitiendo el reciclaje de agua y minerales que retornan al rumen y se absorben los residuos de ácidos grasos que hayan logrado pasar a su interior (Pérez & Sirias, 2007). El retículo tiene la función de movilizar el alimento digerido hacia el rumen en la regurgitación del bolo alimenticio después de la rumia, la cual inicia por el contacto del alimento grueso con los receptores nerviosos de la pared ruminal (Giuliodori et al., 2013). El rumen tiene la capacidad de funcionar como cámara de fermentación almacenando entre 180 y 225 litros de bolo alimenticio, internamente se encuentra dividido en saco dorsal y ventral, ambos funcionan simultáneamente para humedecer el bolo alimenticio con la microbiota del líquido ruminal y descomponerlo (Pérez & Sirias, 2007), además es el principal lugar de asimilación de los ácidos grasos volátiles (AGV) que son su principal fuente de energía.
Una vez que el bolo alimenticio se encuentra en el rumen, la degradación anaeróbica de la biomasa vegetal se lleva a cabo en tres pasos clave: (I) degradación de los polisacáridos vegetales complejos; (II) fermentación de los carbohidratos a AGV y (III) metanogénesis (Söllinger et al., 2018). En el rumen, esta conversión del material alimenticio a CH4 involucra las actividades integradas de diferentes especies microbianas que incluyen bacterias, arqueas, hongos y protozoarios, el paso final se lleva a cabo por estos microorganismos metanogénicos (Boadi, Benchaar, Chiquette & Massé, 2011).
Las bacterias, protozoos y hongos descomponen el bolo alimenticio hidrolizando proteínas y convirtiendo la celulosa, el almidón y otros nutrientes ingeridos en aminoácidos y azúcares como la glucosa (Boadi et al., 2011). La fermentación de la glucosa es un proceso oxidativo que ocurre en la vía Embden-Meyerhof-Parnas bajo condiciones anaerobias (Moss, Jouany & Newbold, 2000):
En este paso, los microorganismos fermentan a los carbohidratos produciendo hidrógeno (H+) como producto final, que resulta perjudicial, ya que es un proceso desfavorable para la fermentación desde el punto de vista termodinámico, además dificulta la regeneración de NAD+ a partir del cofactor reducido NADH, por estas razones es necesario eliminarlo (Castillo-González, Burrola-Barraza, Domínguez-Viveros & Chávez-Martínez, 2014). Para esto, el H+ puede ser utilizado por otras bacterias presentes en el ecosistema microbiano para la formación de AGV, H2 y CO2 como productos finales (Parish et al., 2017):
Los AGV son absorbidos como nutrientes por el animal huésped, mientras que el H2 y el CO2 son utilizados en el rumen por microorganismos pertenecientes al dominio Archaea generando CH4, que es expulsado al exterior por el animal (Parish et al., 2017): (Figura 2).
Durante este proceso, los metanógenos ruminales utilizan principalmente el H2 para reducir el CO2 a CH4, mediante una serie de reacciones, donde el CO2 es utilizado como fuente de carbono y el H2 como el principal donador de electrones (Ramírez, Posada Ochoa & Noguera, 2014) (Figura 3).
Para el rumiante es más eficiente energéticamente la vía del propionato, ya que a diferencia del acetato y el butirato, el propionato sirve como una vía competitiva para el uso del H2 y no promueve la formación del metano (Castillo, Abuelo & Hernández, 2016). Sin embargo, la metanogénesis es la vía más utilizada y la manera más sencilla de eliminar el hidrógeno (Moss et al., 2000).
Aunque la mayor parte del CH4 que se sintetiza es producto de la reducción que se da a partir de H2 y CO2, esta reducción también puede obtenerse mediante la oxidación del formiato, metanol, las metilaminas o el acetato (Sosa, Galindo & Bocourt, 2007), debido a que los metanógenos son capaces de utilizar diferentes sustratos y pueden ser clasificados en tres grupos: hidrogenotróficos, aceticlásticos y metilotróficos (Lyu, Shao, Akinyemi & Whitman, 2018).
Actualmente, los estudios del microbioma del rumen se encuentran a la vanguardia de la investigación agrícola animal, debido a la evidencia reciente que vincula la diversidad de la población con los rasgos fisiológicos del animal (Mizrahi & Jami, 2018).
Metasecuenciación: ¿Cuáles son los micro-organismos presentes?
Análisis y metodología general de la metasecuenciación
En años recientes, el desarrollo de las tecnologías cultivo-independientes han permitido un avance para describir y comprender la existencia o interacción de diversos microorganismos en los ecosistemas microbianos (Shakya, Lo & Chain, 2019). Entre estas alternativas moleculares cultivo-independientes se encuentra la identificación de especies basada en la secuenciación del DNA o “barcoding”, que se ha diseñado utilizando marcadores moleculares como los mitocondriales y nucleares, entre ellos el 16S rDNA/rRNA para las bacterias y las arqueas, el 18S rDNA/rRNA para los protozoos y el espaciador intergénico transcrito (ITS) para los hongos (Paz, González & Crawford, 2011). La secuenciación consiste en la determinación del orden de los nucleótidos que componen un fragmento del DNA o del RNA (Pacheco-Arjona & Sandoval-Castro, 2018). Este sistema ha permitido clasificar grupos de microorganismos mediante estos genes taxonómicamente informativos, en sus inicios estos marcadores moleculares eran secuenciados por el método de Sanger, que solo permitía identificar una especie a la vez, sin embargo, debido al avance de las técnicas de secuenciación ahora es posible identificar varias al mismo tiempo en muestras de alta diversidad microbiana, a menudo llamada metasecuenciación o “metabarcoding” (Pavan-Kumar, Babu & Lakra, 2015).
La técnica de metasecuenciación identifica múltiples especies con DNA extraído de una muestra ambiental, se realiza utilizando PCR con cebadores o primers universales específicos para la amplificación de regiones específicas en los genes, posteriormente estos fragmentos son secuenciados con tecnologías de alto rendimiento o de nueva generación (NGS). Entre estas técnicas de secuenciación, se encuentran: la pirosecuenciación 454 (454 Life Sciences), la secuenciación por síntesis (Illumina) y la secuenciación por ion semiconductor (Ion Torrent) (Reuter, Spacek & Snyder, 2015), por mencionar algunas.
Las lecturas o secuencias del DNA resultantes pasan por un control de calidad y una posterior agrupación en unidades taxonómicas operativas (OTUs, por sus siglas en inglés) con base en la similitud de su código genético (típicamente 97%) (Ruppert, Kline & Rahman, 2019). La asignación de OTUs a especies o taxones se lleva a cabo mediante la comparación con bases de datos de referencia públicas. Las más utilizadas para su identificación en el rumen han sido NCBI, Greengenes, SILVA y RDP (Li et al., 2018). Además, para estimar la comunidad de arqueas en el rumen se ha creado una base de datos específica llamada RIM-DB (Ruminal and Intestinal Methanogen Database) desarrollada por Janssen & Kirs (2008). Las herramientas bioinformáticas más utilizadas para realizar el análisis metasecuencial son: QIIME2 (dada2), MG-RAST, Galaxy y OBITools (Bikel et al., 2015).
Microbioma central del rumen
El estudio más completo hasta ahora utilizando la metasecuenciación, ha sido el proyecto Global Rumen Census, que ha demostrado la existencia en el mundo de un microbioma central (core microbiome) similar en la comunidad ruminal, sin importar la dieta o la especie del huésped. Los resultados fueron obtenidos tras el análisis de 742 muestras de la microbiota de 32 distintas especies de rumiantes y camélidos, en 35 países (Henderson et al., 2015).
En este estudio, las 7 bacterias más abundantes pertenecen a los géneros Prevotella, Butyrivibrio y Ruminococcus, así como especies no clasificadas incluidas en las familias Lachnospiraceae, Ruminococcaceae, Bacteroidales y Clostridiales, por esto se le considera el núcleo bacterial microbiano (Creevey, Kelly, Henderson & Leahy, 2014). Otros estudios también han demostrado, una gran dominancia del género Prevotella por encima de la comunidad bacteriana ruminal (Jami, Israel, Kotser & Mizrahi, 2013; Mizrahi & Jami, 2018; Stevenson & Weimer, 2007), a este género se le atribuyen funciones como la producción de enzimas degradadoras del almidón y la hemicelulosa en la pared celular de las plantas y la actividad proteolítica (Danielsson et al., 2017). En otro estudio, se analizó la comunidad bacteriana del rumen de bovinos desde el nacimiento hasta la edad adulta y se encontró que el género Ruminococcus estaba presente tanto en animales de corta edad como en el rumen funcional maduro, se identificó a este género como de gran importancia para el correcto funcionamiento del rumen (Jami et al., 2013).
Por otro lado, la mayoría de las arqueas presentes en el rumen fueron identificadas como metanógenas, con predominancia del género Methanobrevibacter spp. (Morgavi, Forano, Martin & Newbold, 2010). Las dos especies más abundantes identificadas en el Global Rumen Census son Methanobrevibacter gottschalkii y Methanobrevibacter ruminantium, representando el 74% de las arqueas totales (Henderson et al., 2015). El 77.7% de las arqueas fueron metanogénicas hidrogenotróficas y el 22.1% capaces de usar hidrógeno o grupos metilo derivados del metanol o metilaminas (Henderson et al., 2015). Los metanógenos capaces de usar acetato fueron muy pocos, menos del 0.015%, pertenecientes a los géneros Methanosaeta y Methanosarcina, este último capaz de utilizar también otros sustratos como el metanol, las metilaminas y en ocasiones el H2 y el CO2 (Maeder et al., 2006).
La tecnología de secuenciación de ampliación de regiones específicas en los genes por metasecuenciación, es útil para identificar a los microorganismos (Petrosino, Highlander, Luna, Gibbs & Versalovic, 2009). Así mismo, ha otorgado un panorama amplio y un comienzo en la comprensión del microbioma ruminal, pero aún no ha sido capaz de representar con precisión la dinámica microbiana del microbioma de los rumiantes y de proponer estrategias para el aumento de su producción y la disminución de emisiones del metano generado. Además, este análisis puede estar sesgado por la selección del cebador de la PCR, por las condiciones del ciclo de amplificación y está muy limitado para encontrar nuevos filotipos debido a que los cebadores se diseñan de acuerdo con secuencias ya conocidas (Li et al., 2018). Asimismo, la identificación de OTUs permitiría inferir sobre el fenotipo funcional cuando se tienen valores muy altos de similitud con los genomas de referencia, sin embargo, no se podría llegar a un resultado certero, ya que las diferentes cepas pueden tener cambios dramáticos en cuanto a la función de sus capacidades (Denman, Martínez- Fernández, Shinkai, Mitsumori & McSweeney, 2015). Otras formas de aplicación de la biología molecular son a través de la metagenómica y la metatranscriptómica prometedoras para avanzar en la comprensión del microbioma ruminal.
Metagenómica y Metatranscriptómica para la comprensión del Microbioma Ruminal
La metagenómica y la metatranscriptómica son útiles para la evaluación taxonómica y funcional, basada en el DNA (especies y genes) y en el RNA (metabolismo activo), respectivamente, del microbioma de un ecosistema (Suenaga, 2011), ya que proporcionan información más detallada para evaluar la dinámica de una comunidad microbiana y contestar las siguientes preguntas: ‘¿Quién está ahí?’, ‘¿en qué cantidad?’ y ‘¿qué están haciendo? Con la información obtenida se relacionan la composición microbiana, la diversidad de genes, las enzimas y su funcionamiento, con rasgos fisiológicos, como la eficiencia energética y la emisión del metano por el animal.
Con estos dos métodos, se obtiene una cantidad masiva de datos biológicos, por lo que la bioinformática y el análisis computacional son vitales para la metagenómica y la metatranscriptómica (por shotgun), algunas de las que se mencionan en este artículo aparecen en la (Tabla I).
Tabla I Herramientas bioinformáticas para el análisis metagenómico y metatranscriptómico
Metagenómica
La metagenómica es el análisis de los genomas contenidos en una muestra ambiental, tiene la capacidad de realizar vínculos entre microorganismos cultivables y no cultivables de la comunidad microbiana (ej. microbiota del rumen), de un huésped (ej. rumiante) y el fenotipo de su huésped (ej. emisión de metano por rumiante) (Thomas, Gilbert & Meyer, 2012). Se divide en dos tipos: (i) la metagenómica dirigida (metasecuenciación) y (ii) la metagenómica de secuenciación masiva o de escopeta, conocida también como “Shotgun Metagenomic Sequencing” (SMS) (Cortés-López, Ordoñez & Viveros, 2020). En sus inicios la metagenómica se basó en el análisis dirigido (i), que entre sus técnicas cuenta con la clonación del DNA en un vector (ej. fagos, fósmidos y cósmidos) (Suenaga, 2015).
El segundo tipo (ii) se ha desarrollado a partir de la reciente reducción de costos y al avance de técnicas de secuenciación masiva o de nueva generación (NGS). La metagenómica de secuenciación masiva o de escopeta shotgun se considera la más integral y completa para la evaluación de un microbioma, pues permite la secuenciación aleatoria de todo su contenido genómico (Bernal, 2015). En lugar de amplificar un marcador molecular o dirigir un locus genómico específico, todo el DNA se corta en pequeños fragmentos para secuenciarlos de forma independiente, con la finalidad de ensamblar genomas completos (Sharpton, 2014). Es decir, se captura todo el repertorio genómico de los microorganismos cultivables y no cultivables, se perfilan genes y se reconstruyen genomas microbianos individuales, para asociarlos con su perfil funcional y vías metabólicas (Petrosino et al., 2009).
Metatranscriptómica
La metatranscriptómica permite evaluar la actividad funcional del microbioma en conjunto y hacer aproximaciones del fenotipo a partir de los transcritos expresados (Shakya et al., 2019). Es una ciencia que implica un análisis comparativo de transcriptomas y ayuda a comprender la respuesta de las comunidades microbianas hacia un entorno cambiante (Kaul et al., 2016). El transcriptoma es el conjunto de todas las moléculas de RNA presentes en una célula o grupos de células en un punto de tiempo dado, es la parte activa de un microbioma, el RNA varía con el tiempo y de célula a célula, en contraste con el DNA que es fijo en el tiempo y en todas las células de un organismo. La metatranscriptómica proporciona información de los microorganismos activos y registra las transcripciones expresadas de una comunidad microbiana en un punto en el tiempo y bajo condiciones ambientales específicas (Aguiar-Pulido et al., 2016), esto es sumamente relevante, ya que las transcripciones expresadas representan una aproximación del fenotipo real. En contraste, la metagenómica solo distingue a los miembros que están presentes, sin explicar si estos realmente están contribuyendo al funcionamiento del ecosistema (Shakya et al., 2019). La metatranscriptómica shotgun se basa en la técnica de secuenciación RNA-Seq, que por lo regular utiliza bibliotecas de cDNA para llevarse a cabo, comúnmente se realiza utilizando Pirosecuenciación 454, o más recientemente a partir de Illumina (Toseland, Moxon, Mock & Moulton, 2014). Aunque la tecnología RNA-Seq continúa implementando mejoras, actualmente presenta muchas ventajas sobre otros métodos de análisis de RNA, puesto que no se limita al estudio de secuencias ya conocidas, sino que permite identificar nuevas, y con ello encontrar genes de especial interés para las distintas áreas de la biotecnología (Wang, Gerstein & Snyder, 2009).
En esta revisión se muestra un panorama general del flujo de trabajo de los proyectos que utilizan la metagenómica y la metatranscriptómica, para el análisis del microbioma del rumen (Figura 4).
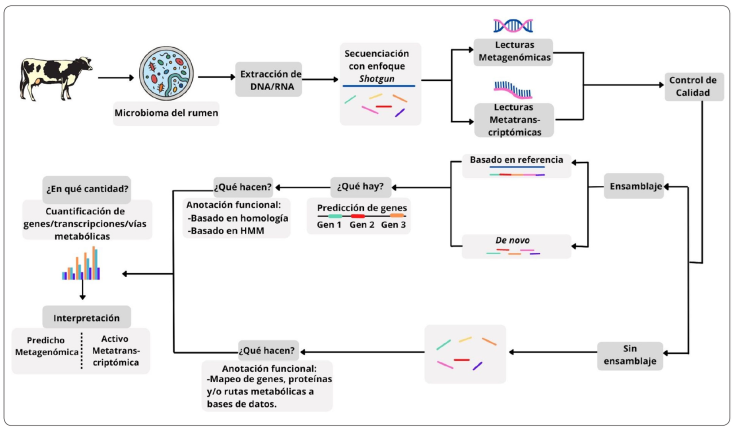
Figura 4 Flujo de trabajo general del estudio del microbioma ruminal mediante la metagenómica y/o la metatranscriptómica, modificado de McCann et al. (2014) y Choi et al. (2015).
Flujo de trabajo general para el análisis del microbioma
Extracción del DNA y su secuenciación
Para el análisis a través de la metagenómica shotgun se aborda como primer paso la extracción del DNA, es deseable obtener la mayor cantidad y calidad para una mayor representación de la muestra ambiental y evitar un sesgo que afecte el resultado final; para la extracción de muestras del DNA ruminal se encuentran disponibles diversos métodos (Henderson et al., 2013; Villegas-Rivera et al., 2013; Yu & Morrison, 2004). El DNA extraído se fragmenta en pequeños pedazos que se secuencian individualmente, como resultado se obtienen lecturas de secuencia; la preparación de librerías metagenómicas y la longitud de las lecturas varía de acuerdo con la plataforma de secuenciación utilizada (NGS), de éstas las más aplicadas en estudios metagenómicos del rumen han sido 454 Roche e Illumina. Es deseable obtener lecturas largas, ya que se facilita el ensamblaje y la posterior interpretación de los datos (Thomas et al., 2012).
De la misma forma, la secuenciación metatranscriptómica shotgun, comienza con la extracción del RNA de alta calidad, siguiendo protocolos disponibles (Wang et al., 2011). Para este segundo análisis, la extracción del RNA comienza con la evaluación de la calidad de los datos crudos, que incluye separar los fragmentos del RNA dependiendo de su naturaleza (Shakya et al., 2019): los RNAs mensajeros (mRNA) representan los patrones de expresión génica entre las especies, mientras que los RNAs ribosomales (rRNA) que representan aproximadamente el 90% del total del RNA permiten conocer la composición de la comunidad microbiana y son útiles para los análisis filogenéticos (Kopylova, Noé & Touzet, 2012). Trabajar con el RNA total de la muestra hace posible generar perfiles composicionales y funcionales utilizando los mismos datos metatranscriptómicos. No obstante, generalmente los rRNAs son de menor interés, por lo que son removidos antes de la secuenciación mediante procedimientos moleculares (Hrdlickova, Tolue & Tian, 2017). Además, la remoción de los rRNAs y el enriquecimiento de los RNAs mensajeros otorga información funcional más relevante (Li et al., 2018).
Control de calidad y ensamblaje de secuencias del DNA microbianas
Una vez obtenidas las secuencias del DNA o del RNA, deben pasar por un control de calidad que filtre secuencias residuales artificiales, bases de baja calidad, así como las secuencias del huésped (rumiante), pues éstas interfieren en el posterior ensamblaje de las secuencias microbianas y conducen a conclusiones erróneas (Li et al., 2018). Para evitar estas interferencias, se utilizan los programas Trimmomatic y TopHat2 (Bikel et al., 2015). Posteriormente, se lleva a cabo el ensamblaje de las secuencias cortas resultantes para obtener fragmentos más largos del DNA (contigs) o RNA y que reconstruyan la secuencia original antes de ser fragmentada (Choi, Lee & Sul, 2015). Existen dos tipos de ensamblaje, el ensamblaje basado en referencia y el ensamblaje de novo.
En la metagenómica, para el ensamblaje basado en referencia, las lecturas obtenidas se mapean a genomas de referencia localizados en bases de datos (ej. Joint Genome Institute, SILVA, RDP) para identificar taxones microbianos y detectar genes de interés (Hernández, Quijada, Rodríguez-Lázaro & Eiros, 2020); este ensamblaje se realiza con las herramientas bioinformáticas AMOS, MIRA y GEMmapper. Este método está limitado para las comunidades microbianas que no se encuentren bien caracterizadas en bases de datos y no es útil para encontrar nuevos microorganismos y genes (Jovel et al., 2016). Debido a lo anterior, la segunda estrategia de ensamblaje de novo es la más utilizada y recomendada para comunidades microbianas como las del rumen que son complejas y poco caracterizadas.
La estrategia de novo ensambla lecturas de secuencia sin utilizar información genómica de referencia y encuentra nuevos genomas y genes. Está basada en las gráficas de Bruijn y en el montaje de diseño superpuesto [Overlap Layout Consensus (OLC)] para reconstruir contigs (Bikel et al., 2015) . Las desventajas del ensamblaje de novo es que necesita una mayor capacidad computacional y puede generar ambigüedades; las herramientas bioinformáticas para este ensamblaje incluyen Meta-Velvet, SOAPdenovo2 y IDBA-UD (Hernández et al., 2020).
De la misma manera para la metatranscriptómica, posterior al control de calidad, se lleva a cabo el ensamblaje a partir de una referencia o con ensamblaje de novo. Para el ensamblaje de novo se pueden utilizar las plataformas de ensamblaje metagenómico, IDBA-UD o MEGAHIT, no obstante, podrían existir algunas inconsistencias, por lo que se han desarrollado nuevas plataformas y específicamente para la metatranscriptómica, IDBA-MT, IDBA-MTP, Trinity y Transcript Assembly Graph (TAG) (Shakya et al., 2019).
Interpretación funcional: ¿Qué hay?, ¿qué hacen? y ¿en qué cantidad?
Una vez ensambladas las secuencias, se realiza la interpretación de los datos obtenidos, los genomas y transcriptomas de los organismos presentes en el microbioma codifican las funciones colectivas que realizan sobre la fisiología de la comunidad microbiana, por ello la diversidad funcional puede ser cuantificada mediante la anotación de secuencias metagenómicas y metatranscriptómicas con funciones (Sharpton, 2014).
En la metagenómica, esto implica, como primer paso, identificar las lecturas que contengan secuencias de genes codificantes y utilizar las siguientes herramientas bioinformáticas: FragGeneScan, MetaGeneMark y MetaGeneAnnotator (Thomas et al., 2012). Como segundo paso, las secuencias que fueron identificadas como genes o elementos genómicos relevantes se comparan con bases de datos de genes, proteínas, o rutas metabólicas cuyas funciones se infieren de acuerdo con su similitud (Abubucker et al., 2012). Este segundo paso, se lleva a cabo a través de la búsqueda de una homología basada en secuencias de nucleótidos o de proteínas traducidas, utilizando los softwares USEARCH, BLAST y DIAMOND; para identificar secuencias con baja identidad a referencias se utilizan programas basados en Hidden Markov Models (HMM), (Li et al., 2018). Las bases de datos más aceptadas para la anotación funcional son KEGG, eggNOG, COG/KOG, SEED y UniProt.
Los datos que resulten de este análisis son evaluados mediante los recuentos de las lecturas asignadas a taxones o funciones y son utilizados para describir las funciones potenciales, abundancia y expresión relativa en el metagenoma (Nayfach & Pollard, 2016). El objetivo final del análisis metagenómico es identificar cómo diversos tratamientos (ej. dieta del rumiante) influyen en la composición funcional de una comunidad microbiana y revela aquellas funciones que se asocian con variables ambientales o fisiológicas específicas del huésped (Looft et al., 2012).
En el análisis metatranscriptómico, después del ensamblaje se pueden o no omitir las predicciones de los genes para la posterior anotación funcional, de acuerdo con el flujo de trabajo y los objetivos específicos del estudio. Para la anotación funcional, las secuencias del RNA obtenidas se comparan directamente con las bases de datos (ej. KEGG, eggNOG y UniProt). Una vez realizada la anotación, se lleva a cabo el análisis de las vías metabólicas mediante el mapeo de funciones enzimáticas utilizando las herramientas bioinformáticas Minipath e iPath (Shakya et al., 2019; Yamada, Letunic, Okuda, Kanehisa & Bork, 2011; Ye & Doak, 2009). Todo el análisis metatranscriptómico finaliza con la cuantificación de la expresión y la abundancia relativa, los valores resultantes proporcionan información para comprender qué genes y vías metabólicas son importantes para un fenotipo en particular (ej. emisión de metano), mediante la comparación de la expresión genética relativa entre condiciones específicas y un tiempo dado (Aguiar-Pulido et al., 2016).
También existe un enfoque del análisis sin ensamblaje, este depende en gran medida de la correcta y completa caracterización de la comunidad microbiana en las bases de datos, pues las lecturas de secuencia después de pasar por el control de calidad se alinean a genomas de referencia para su directa anotación funcional (Li et al., 2018). Este análisis se lleva a cabo utilizando los softwares MG-RAST, HUMAnN, Kraken2 y MEGAN. Para los estudios en el rumen este análisis es difícilmente factible, ya que las bases de datos no cuentan con la información genómica del funcionamiento de las especies y los genes presentes en el microbioma. No obstante, el proyecto Hungate1000, ha reunido una colección de genomas de referencia de todas las especies de arqueas y bacterias hasta ahora encontradas en la microbiota ruminal. Este catálogo engloba 501 genomas de referencia (480 bacterias y 21 arqueas), y ha logrado identificar el 75% de los taxones presentes hasta el nivel de género y asignar vías metabólicas a microorganismos individuales involucrados en el funcionamiento del rumen (Seshadri et al., 2018).
Estos proyectos tienen como objetivo desarrollar tecnologías de mitigación del metano y encontrar un equilibrio entre la producción de alimentos y emisiones de gases de efecto invernadero (RMG Network - Hungate1000, 2016). Los datos del proyecto Hungate1000 se encuentran actualmente disponibles en el portal del Joint Genome Institute https://img.jgi.doe.gov y facilitan los análisis metagenómicos y metatranscriptómicos.
El combinar ambos métodos ómicos en estudios integradores, ayuda a obtener mejores resultados y evita que los análisis metagenómicos sobrestimen o subestimen la relevancia funcional de la codificación de los genes en metagenomas, distingue entre la abundancia de genes a nivel metagenómico y a nivel metatranscriptómico (Bikel et al., 2015). Los hallazgos encontrados en diferentes estudios realizados desde lo metagenómico y metatranscriptómico para la comprensión del microbioma ruminal se resumen en la Tabla II.
Tabla II Estudios metagenómicos y metatranscriptómicos del microbioma ruminal.
| Título | Enfoque | Hallazgos principales | Herramientas de secuenciación | Referencias |
|---|---|---|---|---|
| The rumen microbial metagenome associated with high methane production in cattle | Metagenómica | El ganado vacuno con menor emisión de metano presentó mayor abundancia de Succinovibrionaceae. La abundancia de genes de arqueas se correlaciona con el nivel de emisiones del metano. | Illumina HiSeq | (Wallace et al., 2015) |
| Investigating the effect of two methane-mitigating diets on the rumen microbiome using massively parallel sequencing | Metagenómica | La dieta tiene un efecto significativo en los perfiles microbianos del rumen y de las heces fecales, a partir de esto es posible identificar patrones de la población microbiana asociados a bajas emisiones de metano. | Illumina HiSeq2000 | (Ross, Moate, Marett, Cocks& Hayes, 2013) |
| Methane yield phenotypes linked to differential gene expression in the sheep rumen microbiome | Metagenómica y metatranscriptómica | En grupos de ovejas tanto de alta como de baja emisión de metano, se observó una abundancia similar de los genes involucrados en la vía de la metanogénesis. Sin embargo, hubo un aumento significativo en la transcripción de estos en el grupo de alta emisión. | Ilumina HiSeq2000 | (Shi et al., 2014) |
| Comparative metagenomic and metatrans-criptomic analyses reveal the breed effect on the rumen microbiome and its associations with feed efficiency in beef cattle | Metagenómica y metatranscriptómica | La actividad del microbioma ruminal se ve mayormente influenciada por factores ambientales, mientras que el factor genético influye a un menor nivel. | Illumina HiSeq2000 | (Li, Hitch, Chen, Creevey& Guan, 2019) |
| Effect of dietary forage sources on rumen microbiota, rumen fermentation and biogenic amines in dairy cows | Metatranscriptómica | En las vacas, una dieta con alta proporción de heno alfalfa beneficia la producción de leche y mantiene una microbiota ruminal sana que atribuye a una mayor degradación de la materia orgánica en el rumen equilibrando proteínas y carbohidratos para un crecimiento microbiano ruminal óptimo. | 454 GS FLX | (Zhang, Zhu, Zhu, Liu & Mao, 2014) |
| Comparative analysis of microbial profiles in cow rumen fed with different dietary fiber by tagged 16S rRNA gene pyrosequencing | Metatranscriptómica | La pirosecuenciación del gen rRNA 16S en vacas, reveló una disminución de Fibrobacteraceae y Ruminococcaceae al llevar a cabo una dieta baja en fibra. | 454 GS FLX | (Thoetkiattikul et al., 2013) |
| High throughput whole rumen metagenome profiling using untargeted massively parallel sequencing | Metagenómica | Existe una mayor variación en el metagenoma del rumen entre animales que en las réplicas del mismo rumen. | Illumina GAIIx | (Ross et al., 2012) |
| Phage-bacteria relationships and CRISPR elements revealed by a metagenomic survey of the rumen microbiome | Metagenómica | Una pirosecuenciación aleatoria de metagenomas (viromas) aislados del líquido del rumen bovino, mostró que los bacteriófagos del rumen son diversos y únicos del mismo por lo que probablemente influyen en la comunidad microbiana del rumen. | Illumina GAIIx 454 GS FLX | (Berg Miller et al., 2012) |
| Next generation sequencing to define prokaryotic and fungal diversity in the bovine rumen | Metagenómica | Dentro del rumen, las comunidades arqueales y fúngicas son más consistentes en fracciones líquidas y sólidas en comparación con la comunidad bacteriana. | 454 GS FLX | (Fouts et al., 2012) |
| Holistic assessment of rumen microbiome dynamics through quantitative metatranscriptomics reveals multifunctional redundancy during key steps of anaerobic feed degradation | Metatranscriptómica | El aumento en las emisiones de metano tras la alimentación no se debe a cambios en la comunidad microbiana del rumen, sino a la respuesta que tiene el microbioma en los diferentes pasos de la degradación anaeróbica del alimento. | Illumina HiSeq 2500 | (Söllinger et al., 2018) |
| Bovine Host Genetic Variation Influences Rumen Microbial Methane Production with Best Selection Criterion for Low Methane Emitting and Efficiently Feed Converting Hosts Based on Metagenomic Gene Abundance | Metagenómica | Los genes de la metanogénesis (McrA y fmdB) se asociaron con las emisiones del metano, mientras que los genes de interferencia entre el microbioma del huésped (TSTA3 y FucI) se asociaron con la eficiencia de la digestión alimenticia. | Illumina HiSeq 2500 | (Roehe et al., 2016) |
| Metagenomic analysis of the rumen microbial community following inhibition of methane formation by a halogenated methane analog | Metagenómica | Al suministrar altas concentraciones de BCM a las cabras, la emisión de se redujo, las emisiones de aumentaron y la fermentación se desplazó hacia la vía de la producción de propionato de metilo. | 454 GS FLX | (Denman et al., 2015) |
| Metatranscriptomic Profiling Reveals Linkages between the Active Rumen Microbiome and Feed Efficiency in Beef Cattle | Metatranscriptómica | El microbioma ruminal activo es uno de los factores biológicos que pueden contribuir a las variaciones en la eficiencia alimentaria del ganado de carne. El estudio destaca la posibilidad de mejorar tanto la utilización de los nutrientes como la eficiencia de la alimentación mediante la alteración de las funciones microbianas del rumen. | Illumina HiSeq2000 | (Li & Guan, 2017) |
| Establishment of ruminal bacterial 2014 community in dairy calves from birth to weaning is sequential | Metagenómica | El microbioma del rumen se establece antes del consumo de los alimentos sólidos, pero la ingesta de los alimentos sólidos determina la composición del microbioma. | 454 GS FLX | (Rey et al., 2014) |
| The Rumen Metatranscriptome Landscape Reflects Dietary Adaptation and Methanogenesis in Lactating Dairy Cows | Metratranscriptómica | Los cambios inducidos por la dieta dieron como resultado una reducción de la emisión del metano. El análisis metatranscriptómico proporciona información sobre los mecanismos subyacentes clave y abre el camino a nuevos métodos racionales para reducir aún más la producción de metano de los animales rumiantes. | Illumina HiSeq 2500 | (Hornung et al., 2018) |
Conclusiones
Las ciencias ómicas otorgan información biológica detallada acerca de las interacciones que se desempeñan en una comunidad microbiana. Gracias al uso de la metagenómica y la metatranscriptómica se han llevado a cabo investigaciones que han encontrado la relación entre la estructura y función del microbioma del rumen con las emisiones de metano, a fin de encontrar estrategias para mitigarlas.
Entre estas estrategias se encuentra el estudio de diferentes variables como la dieta del animal, el uso de aditivos alimenticios o probióticos, así como el efecto de factores genéticos y ambientales. Lo anterior, para determinar cómo al modular estas variables, los agentes impulsores clave (ej. microorganismos y genes enzimáticos) afectan la dinámica del proceso ruminal y la relación huésped-microbioma, promoviendo vías metabólicas que incrementan o disminuyen las tasas de la metanogénesis (Choi et al., 2015). Considerando que la dieta proporciona el sustrato para el microbioma, esta variable permanecerá como un concepto fundamental para el diseño experimental y la interpretación de los datos (McCann, Wickersham & Loor, 2014).
A pesar de los avances en la comprensión del microbioma ruminal, la viabilidad, precisión y potencial de los estudios metatranscriptómicos y los metagenómicos continúan siendo limitados, ya que depende mucho de la calidad y cantidad de los datos de referencia disponibles. Actualmente sólo una pequeña cantidad de secuencias del RNAm del microbioma ruminal pueden mapearse contra bases de datos como KEGG (Li et al., 2018), por esta razón es de suma importancia fortalecer los esfuerzos por llevar a cabo la identificación del microbioma ruminal con proyectos como el Hungate1000, y generar bases de datos de referencia que contenga las secuencias genómicas de los microorganismos presentes en el rumen.
Asimismo, las herramientas bioinformáticas continúan en desarrollo, por lo que es necesario optimizar los algoritmos, los parámetros analíticos y estadísticos que permitan obtener resultados funcionales relevantes y reducir errores de acuerdo con los objetivos específicos de cada estudio.
Teniendo en cuenta el impacto negativo que genera la industria ganadera en el medio ambiente, la metagenómica y la metatranscriptómica abren una nueva oportunidad para crear una ganadería más sostenible, reducir en gran porcentaje la contaminación ambiental provocada por ésta y al mismo tiempo mantener o incluso incrementar la productividad animal.











 text new page (beta)
text new page (beta)