ABREVIATURAS
Aa |
aminoácidos |
Ac |
acetona |
AcAc |
acetoacetato |
ADN |
desoxirribonucleico |
AGNE |
ácidos grasos no esterificados |
Arg |
arginina |
ARNm |
ribonucleico mensajero |
ARNt |
ribonucleico de transferencia |
C |
carbono |
C=O |
grupo carbonilo |
C16:0 |
palmítico |
C3H3O3 |
piruvato |
Ca2+ |
ion calcio |
CO2 |
dióxido de carbono |
COCH3 |
grupo acetilo |
COOH |
grupo carboxilo |
Gln |
glutamina |
GLU |
glucosa |
H |
hidrógeno |
H2O |
agua |
HCO |
anión hidrógenocarbonato |
N |
nitrógeno |
NADPH+H+ |
nicotinamida adenina dinucleótido fosfato |
NH2 |
grupo amino |
NH + |
ion amonio |
O |
oxígeno |
OH |
grupo hidroxilo |
PO 2− |
grupo fosfato |
TAG |
triacilgliceroles |
β-HBA |
β-hidroxibutirato |
INTRODUCCIÓN
Las células eucariontes están compuestas de agua, iones inorgánicos y miles de moléculas orgánicas (Cooper, 2019b). Que participan en sistemas para extraer, transformar y utilizar energía del medio ambiente (Tortora et al., 2019b). Lo que permite a los organismos realizar trabajos mecánicos, químicos, osmóticos y eléctricos (Ameer et al., 2018; Rodwell, 2018a; Melo & Cuamatzi, 2019). La mayoría de estas moléculas orgánicas pertenecen a una de tres clases de polímeros: i) polisacáridos, ii) lípidos y iii) proteínas (Fails & Magee, 2018a). Estos polímeros constituyen entre el 80 y 90% del peso de la mayoría de las células (Pavlinov et al., 2019) y están formados por la unión (polimerización) de varios componentes químicos de bajo peso molecular: carbohidratos, ácidos grasos y aminoácidos, respectivamente (Guoyao, 2017c). La interacción entre estos componentes es dinámica; los cambios en un componente provocan cambios de coordinación o compensación en otro (Tortora et al., 2019b). La bioquímica es quien describe en términos moleculares, este conjunto de interacciones (Pol et al., 2014). Considerando dos vías metabólicas de manera principal: i) catabolismo para obtener acetil-Coenzima A (Tortora & Derrickson, 2018b) y ii) anabolismo para adquirir moléculas más grandes (Pol et al., 2014; Engelking, 2015; Menzies et al., 2016). Contribuyendo así, con conocimientos y aplicaciones prácticas en la medicina (Gundu, 2020), la agricultura (Milani et al., 2017), la nutrición (Preethi & Sekar 2021) y la industria (Wu et al., 2019). Pero su principal preocupación es la célula como organismo vivo (Cooper, 2019a).
Por lo tanto, esta revisión ofrece una descripción general de la dinámica molecular en la interfaz del metabolismo de polisacáridos, lípidos y proteínas, para fundamentar las bases de la biología celular.
PROPIEDADES FISICOQUÍMICAS DE LOS POLISACÁRIDOS
Los polisacáridos son moléculas orgánicas formadas por más de diez monosacáridos, unidos mediante enlaces O-glucosídicos (Yang et al., 2015; Guoyao, 2017c). Su fórmula general contiene átomos de carbono (C) hidratados con moléculas de agua (H 2 O) (Bender & Mayes, 2018cc. Por lo tanto, presentan solubilidad en este fluido y su clasificación se establece con base a la posición de su grupo carbonilo (C=O) (Chavarría & Cárabez, 2018). Formado por un átomo de C unido a un átomo de oxígeno (O) mediante un doble enlace (Cooper, 2019b). Si el grupo C=O se localiza en el extremo de la molécula, es una aldosa. Si el grupo C=O se localiza en medio de la molécula, es una cetosa (Mckee & Mckee, 2014a; Delbianco et al., 2016).
Los polisacáridos son la principal fuente biológica de almacenamiento y consumo de energía (Chavarría & Cárabez, 2018) y forman parte de la estructura orgánica de todos los seres vivos (Cooper, 2019c). Su ingreso en el organismo, es a partir del alimento y su hidrólisis (ruptura de enlaces O-glucosídicos) por amilasas producidas en las parótidas (Kumar & Chakravarty, 2018), y glucógeno fosforilasas y glucosa-6-fosfatasas, producidas por las células acinares del páncreas (Boticario & Cascales, 2012; Cárabez et al., 2018a). Posterior a esta hidrólisis se libera al monómero glucosa (GLU), con la fórmula química C6H12O6 (Bender & Mayes, 2018b), para ser absorbida por medio del epitelio intestinal (Fails & Magee, 2018a) y distribuida por el torrente sanguíneo a los diferentes tejidos (Dashty, 2013; Oosterveer & Schoonjans, 2014), donde presenta cinco principales vías metabólicas: i) glucogenogénesis, ii) ruta de las pentosas fosfato iii) glucogenólisis, iv) glucólisis y v) glucogénesis (Appleton et al., 2013a; Nelson & Cox, 2017b).
ANABOLISMO DEL GLUCÓGENO (GLUCOGENOGÉNESIS)
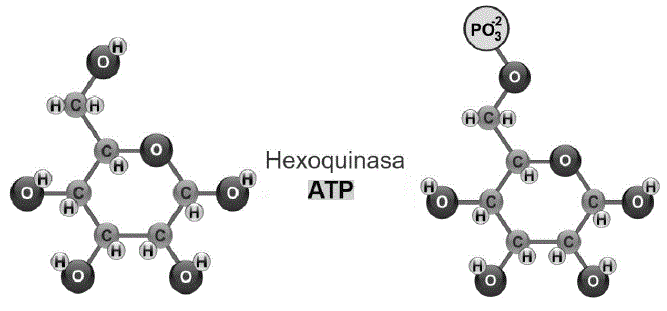
La glucogenogénesis se lleva a cabo en miocitos (Engelking, 2015) y hepatocitos (Tortora & Derrickson, 2018b), donde la GLU ingresa al citoplasma, para ser fosforilada [adición de un grupo fosfato (PO 4 2 −)], a partir de adenosina trifosfato (ATP) (Rui, 2014) (Figura 1).
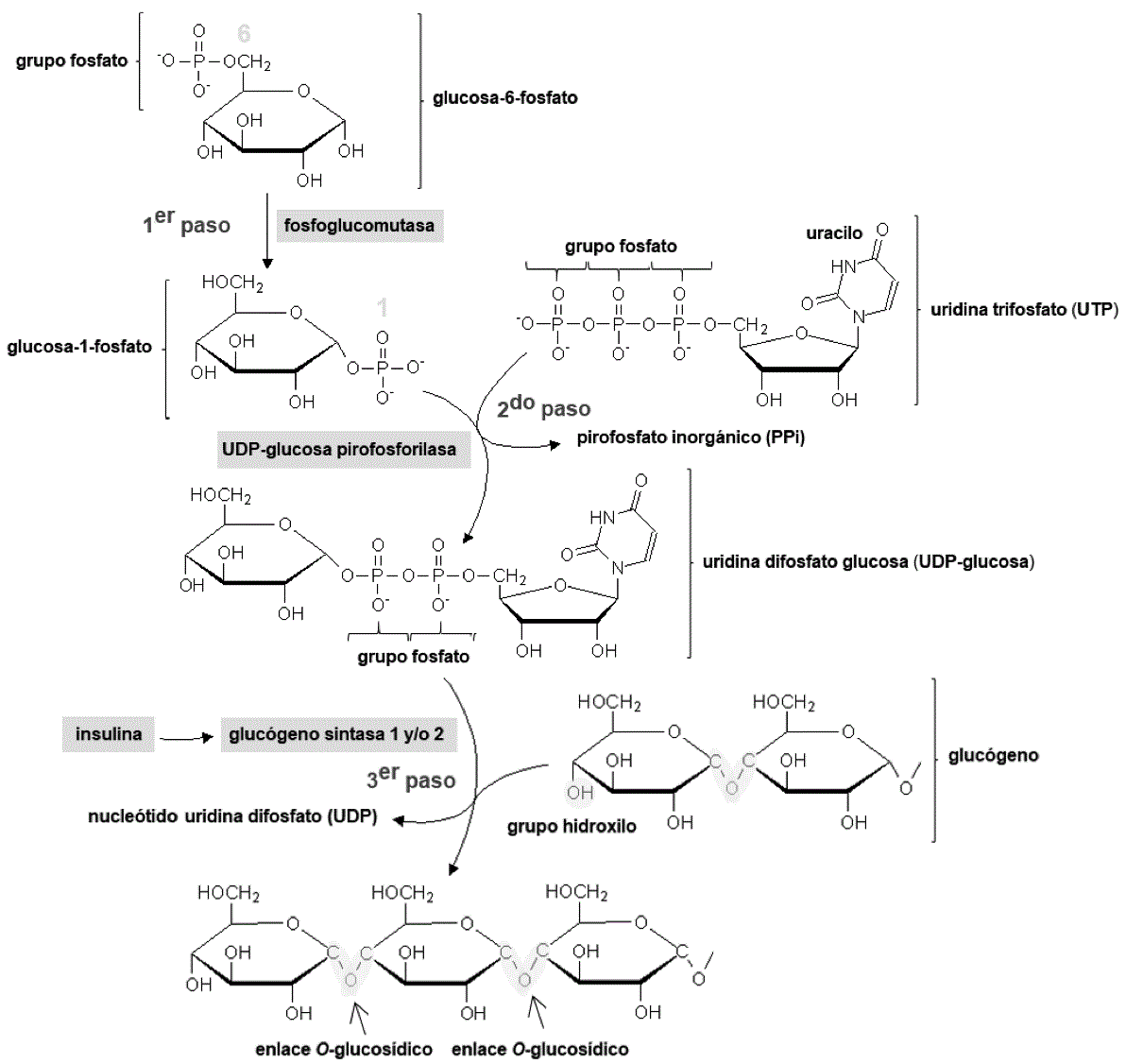
La glucosa-6-fosfato resultante, abunda en el citoplasma de todas las células (Litwack, 2018a) y cuando sus niveles son elevados, la fosfoglucomutasa transfiere el grupo PO4 2− del C6 al C1 sintetizando glucosa-1-fosfato (Delbianco et al., 2016). La uridina trifosfato, interacciona con glucosa-1-fosfato, formando uridina difosfato glucosa (Fox et al., 2017). La insulina, activa a la glucógeno sintasa 1 expresada en miocitos y/o la glucógeno sintasa 2 expresada en hepatocitos (Gadupudi et al., 2016), para que el grupo hidroxilo (OH) de la uridina difosfato glucosa se fije al glucógeno (creando un enlace O- glucosídico), alargando al polisacárido (Figura 2).
RUTA DE LAS PENTOSAS FOSFATO
Este proceso tiene lugar en el citoplasma y se divide en dos, la fase oxidativa y la fase no oxidativa (Tortora & Derrickson, 2018b). La fase oxidativa, presenta de tres reacciones: i) la glucosa-6-fosfato es deshidrogenada [pierde 2 hidrógenos (H)] (Nelson & Cox, 2017b). Como producto se obtiene 6-fosfogluconolactona y una molécula de nicotinamida adenina dinucleótido fosfato (NADPH+H+), y ii) la 6-fosfogluconolactona es hidrolizada y como producto se obtiene 6-fosfoglucanato (Lee et al., 2019) y iii) el 6- fosfoglucanato es descarboxilado [eliminación del grupo carboxilo (COOH)] (Mckee & Mckee, 2014b). Como producto se obtiene ribulosa-5-fosfato (cetopentosa), una molécula de NADPH+H+ y dióxido de carbono (CO 2) (Stincone et al., 2015).
Durante la fase no oxidativa la ribulosa-5-fosfato, puede presentar isomerización y ser transformada en otra molécula que posee los mismos átomos, pero dispuestos de forma distinta (Madigan et al., 2019a). En otra palabras, cambia de posición su grupo C=O para convertirse en a ribosa 5-fosfato (aldopentosa) (Cárabez et al., 2018b). Por lo tanto, las principales funciones de la ruta de las pentosas fosfato son: i) sintetizar monosacáridos de cinco C y ii) generar NADPH+H+ (Nelson & Cox, 2017b).
CATABOLISMO DEL GLUCÓGENO (GLUCOGENÓLISIS)
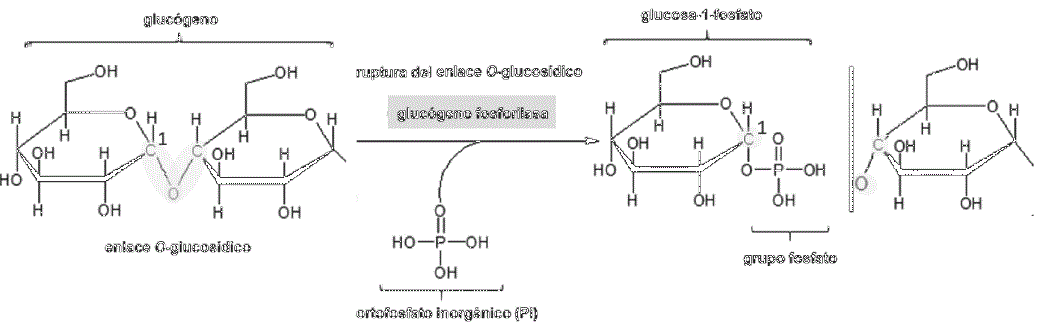
Este proceso tiene lugar en el citoplasma de casi todas las células, aunque de manera especial en los miocitos del músculo y en los hepatocitos del hígado (Mckee & Mckee, 2014c). Cuando los niveles de GLU en la sangre son bajos, la adrenalina o epinefrina en el músculo y el glucagón en el hígado, activan las proteínas quinasas (Ahern, 2019d), y éstas realizan la fosforilación a glucógeno fosforilasa, por lo que esta enzima se activa (Mckee & Mckee, 2014c). La glucógeno fosforilasa cataliza la transferencia de un ortofosfato inorgánico en el C1 del glucógeno (Fox et al., 2017), y este cambio rompe el enlace O-glucosídico y libera glucosa-1-fosfato (Figura 3). La glucosa-1-fosfato es transformada en glucosa-6-fosfato, transfiriendo el grupo PO4 2− del C1 al C6 (Ahern, 2019d).
CATABOLISMO DE LA GLUCOSA (GLUCÓLISIS)
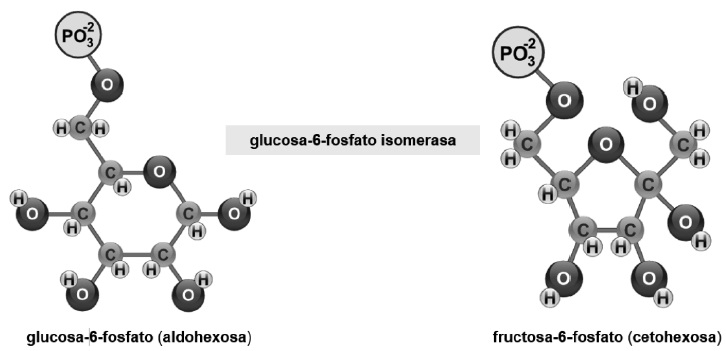
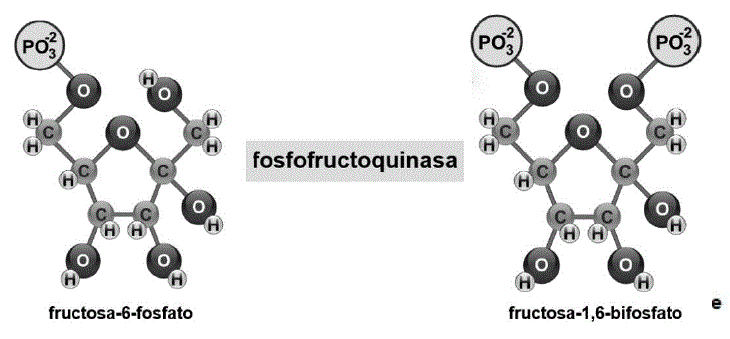
Este proceso consiste en la degradación de la glucosa-6-fosfato para obtener acetil- Coenzima A, a partir del piruvato (C3H3O3) (Ferrier, 2017b). Se lleva a cabo en el citoplasma donde la glucosa-6-fosfato (aldohexosa), presenta isomerización (Mckee & Mckee, 2014c) y es transformada en fructosa-6-fosfato (cetohexosa) al cambiar de lugar su grupo C=O (Figura 4).
La fructosa-6-fosfato, es fosforilada (Figura 5), a partir de ATP en los C1 y C6 (Tortora et al., 2019a), para dar lugar a la fructosa-1,6-bifosfato (Delbianco et al., 2016; Ferrier, 2017a).
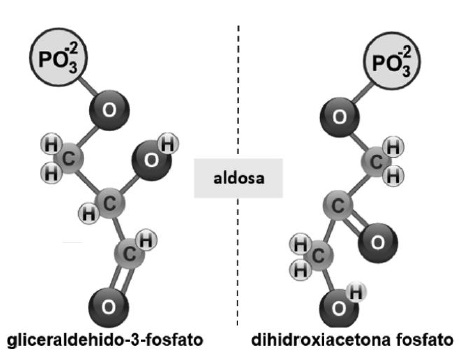
Posteriormente la fructosa-1,6-bifosfato (Figura 6) es dividida en dos: i) gliceraldehido- 3-fosfato y ii) dihidroxiacetona fosfato (Melo & Cuamatzi, 2019).
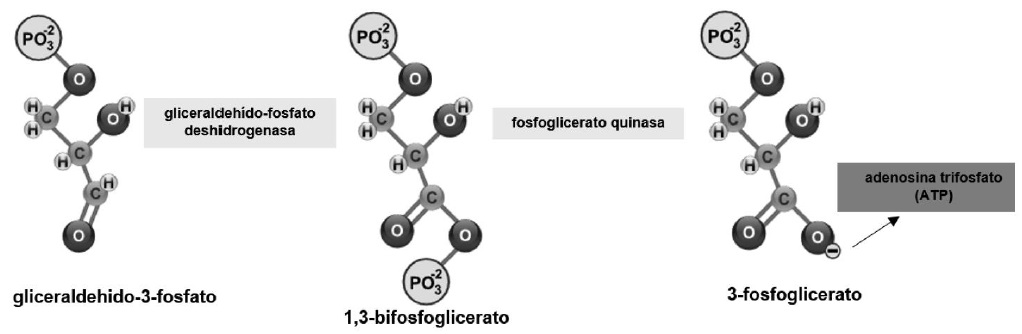
El gliceraldehido-3-fosfato es oxidado y fosforilado, en los C1 y C3 formando 1,3- bifosfoglicerato (Mckee & Mckee, 2014c) (Figura 7). Posteriormente, transfiere su grupo PO42−, para sintetizar ATP (Ahern, 2019b) y se transforma en 3-fosfoglicerato (Voet et al., 2016; Tortora & Derrickson, 2018b).
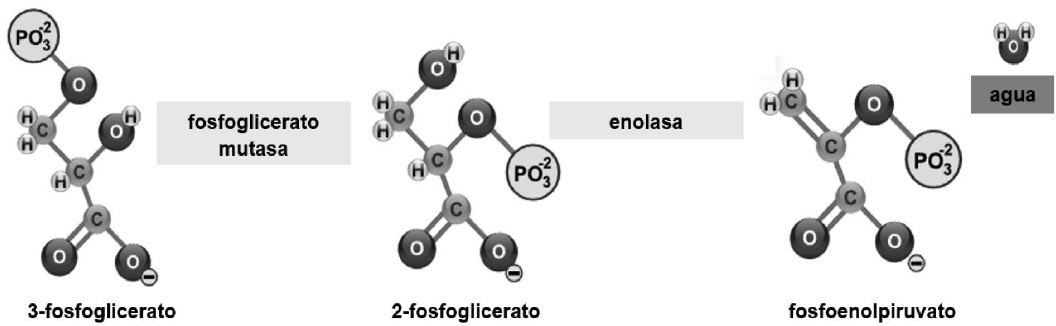
El 3-fosfoglicerato presenta isomerización y su grupo PO42− cambia del C3 al C2, transformado la molécula en 2-fosfoglicerato (Nelson & Cox, 2017b). A continuación la enolasa propicia la formación de un enlace doble (Voet et al., 2016), eliminando una molécula de H2O y formando fosfoenolpiruvato (Guoyao, 2017f; Bender & Mayes, 2018a) (Figura 8).
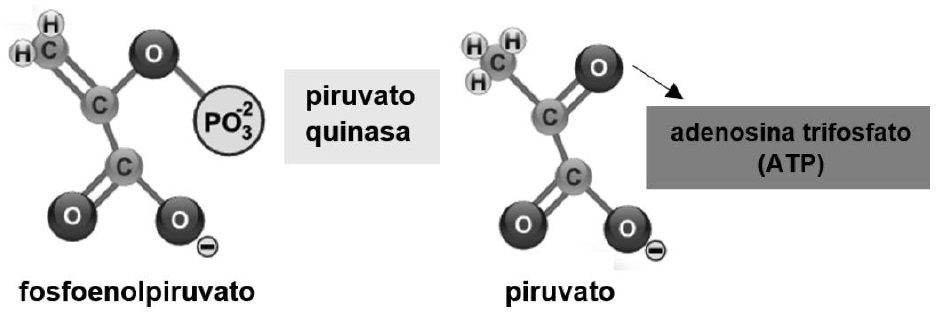
El fosfoenolpiruvato transfiere su grupo PO42− (Cárabez et al., 2018a), para sintetizar ATP (Ahern, 2019b) y se transforma en C3H3O3 (Botham & Mayes, 2018d), molécula que es atraída hacia la matriz mitocondrial, utilizando la fuerza protón-motriz generada por la cadena respiratoria (Fails & Magee, 2018b; Madigan et al., 2019c) (Figura 9).
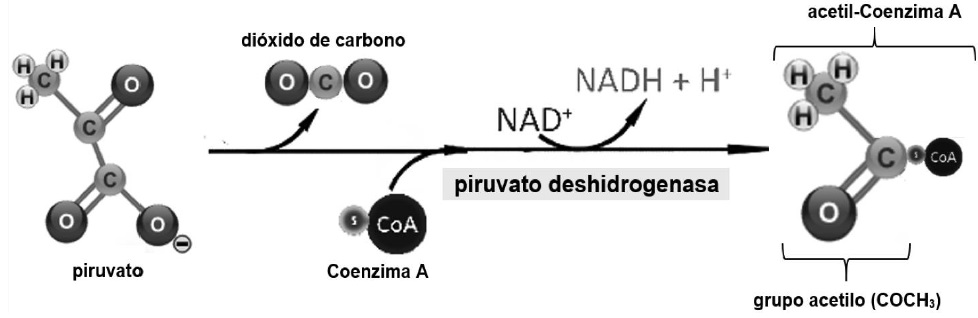
El destino del C3H3O3 que se produjo en la glucólisis, depende de la disponibilidad de O. En condiciones anaeróbicas el C3H3O3 presenta reducción adicionando átomos de H para formar láctico (Tortora & Derrickson, 2018b). En condiciones aeróbicas el C3H3O3 presenta descarboxilación y su grupo COOH se libera como CO2 (Stincone et al., 2015) , el resto de la molécula presenta oxidación, para formar el grupo acetilo (COCH3). Por último la Coenzima A, se transfiere al grupo COCH3 formando acetil-Coenzima A (Guoyao, 2017f) (Figura 10).
PROPIEDADES FISICOQUÍMICAS DE LOS LÍPIDOS
Los lípidos constituyen un depósito de almacenamiento energético en los adipocitos (Guoyao, 2017a). Participan en la formación de membranas fosfolipídicas de las células eucariontes y sus organelos (Schoeler & Caesar, 2019). En el torrente sanguíneo, transportan vitaminas liposolubles p. ej., la A para la formación de tejidos blandos y mucosas (Botham & Mayes, 2018c), la D para la absorción del ion calcio (Ca2+) (Jameson, 2017), la E como antioxidante y formación de eritrocitos (Madigan et al., 2019c) y la K que contribuye en la coagulación (Guoyao, 2017a). Además actúan como aislante térmico en los tejidos subcutáneos para retener el calor corporal (Mas, 2018b).
Su ingreso en el organismo, es a partir del alimento y su hidrólisis (ruptura de enlaces éster) por lipasas y esterasas producidas por las células acinares del páncreas (Ahern, 2019c). Posterior a esta hidrólisis se liberan ácidos grasos no esterificados (AGNE) y triacilgliceroles (TAG) (Tortora et al., 2019a), para ser absorbidos por medio del epitelio intestinal (Pol et al., 2014; Guoyao, 2017d), y transportados hacia los hepatocitos del hígado (Botham & Mayes, 2018c). Donde son empaquetados en lipoproteínas de muy baja densidad (Wadhera et al., 2016), para su posterior exportación hacia los tejidos periféricos (Wang et al., 2016). Los AGNE obtenidos durante dicho proceso, son necesarios para sintetizar acetil-Coenzima A (Appleton et al., 2013d).
ANABOLISMO DEL TRIACILGLICEROL (LIPOGÉNESIS)
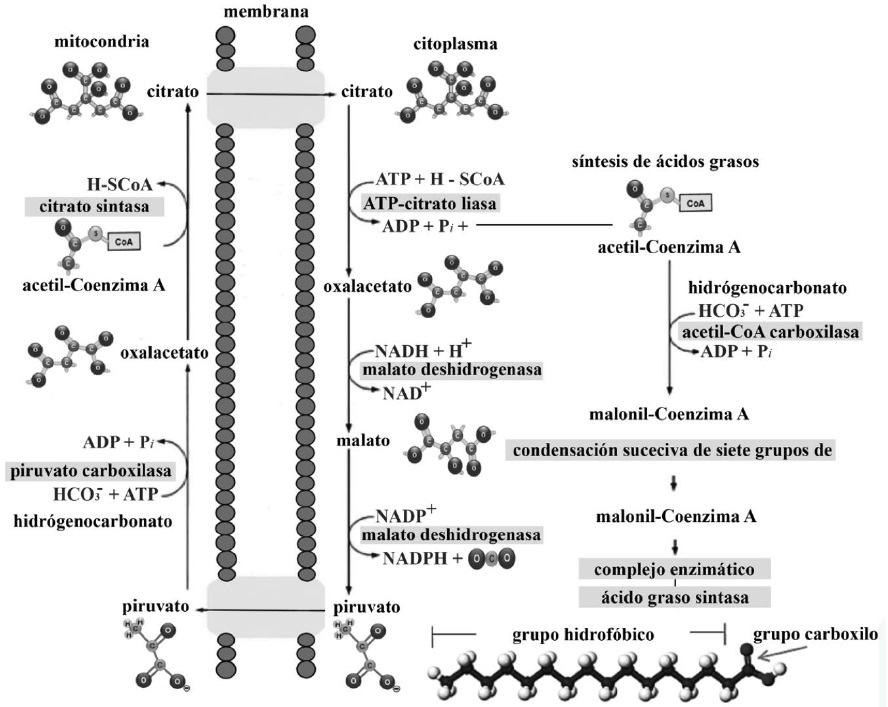
La lipogénesis inicia en la mitocondria, con la producción de acetil-Coenzima A (Cooper, 2019a). Debido a que la membrana de la mitocondria es impermeable al paso de acetil- Coenzima A (Friedman & Nunnari, 2014), se requiere del sistema tricarboxilato (Figura 11) y de la citrato sintasa para convertirla en citrato (Nunes-Nesi et al., 2013), por medio de la fijación de C (Ameer et al., 2018), de este modo se asegura su ingreso al citoplasma celular (Botham & Mayes, 2018c). A continuación, el citrato es trasformado nuevamente en acetil-Coenzima A por la ATP-citrato liasa (Nunes-Nesi et al., 2013; Botham & Mayes, 2018c), obteniendo oxaloacetato y adenosina difosfato (Mas, 2018a; Tortora & Derrickson, 2018a).
La lipogénesis es un proceso endergónico, por lo tanto, la acetil-Coenzima A debe ser activada mediante carboxilación a través de su unión con el anión hidrógenocarbonato (HCO3-) en una reacción que consume ATP (Botham & Mayes, 2018a), catalizada por la acetil-CoA carboxilasa (Cooper, 2019b). Como resultado, la acetil-Coenzima A se convierte en malonil-Coenzima A (Nelson & Cox, 2017c). Por su parte, el oxaloacetato es reducido por malato deshidrogenasa a malato, y éste a su vez convertido en C3H3O3 por medio de la enzima málica, produciendo NADPH+H+ (Appleton et al., 2013e; Dashty, 2013). Posteriormente el ácido graso requiere elongación, mediante el complejo proteico ácido graso sintasa (Pol et al., 2014). Este complejo realiza condensación, reducción, deshidratación y nuevamente reducción, acoplando grupos de malonil-Coenzima A con acetil-Coenzima A (Nelson & Cox, 2017c). Las dos reducciones mencionadas, requieren de NADPH+H+ (Dashty, 2013), y durante la elongación se van añadiendo grupos de dos C al ácido graso, sintetizando siempre al hexadecanoico o palmítico (C16:0), como producto final (Guoyao, 2017d). Posteriormente, el C16:0 es liberado del complejo proteico y puede ser elongado introduciendo C en su cadena, para producir otras moléculas de ácidos grasos más grandes (Botham & Mayes, 2018c), y/o insaturado introduciendo enlaces dobles en su cadena (Cooper, 2019a). La síntesis de TAG, se lleva a cabo en el retículo endoplasmático liso (Quintero, 2014).
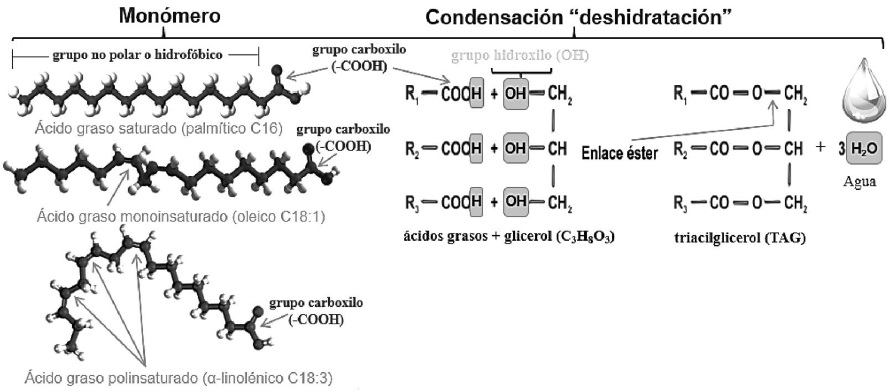
Una vez que se obtienen diferentes AGNE, el enlace éster de los lípidos, se establece mediante la unión de los tres grupos OH del glicerol (Nelson & Cox, 2017c) (Figura 12), y el grupo COOH (la parte polar) de tres ácidos grasos (Botham & Mayes, 2018cc). Este enlace es una condensación o deshidratación donde se pierden 3 moléculas de H2O (Smith, 2020b). Debido a esta unión, los grupos polares unidos al carbohidrato no son accesibles (Pratt et al., 2016). En consecuencia, se forman moléculas no polares o hidrofóbicas, altamente insolubles en agua (Dowhan & Bogdanov, 2016).
CATABOLISMO DEL TRIACILGLICEROL (LIPÓLISIS) Y CETOGÉNESIS
Cuando las reservas de glucógeno en el citoplasma de miocitos y hepatocitos disminuyen, se activa la carnitina palmitoiltransferasa (Longo et al., 2016), estimulando el transporte de AGNE hacia el interior de la mitocondria hepática (Merritt et al., 2020; Wang et al., 2020). Donde la β-oxidación, conduce a una descarboxilación de los AGNE (Wanders et al., 2020), el grupo COOH se libera como CO2 y el resto de la molécula presenta deshidrogenación, estableciendo el grupo COCH3 (Botham & Mayes, 2018b). La Coenzima A, se transfiere al grupo COCH3 y forma acetil-Coenzima A (Guoyao, 2017f). Esta molécula se combina con oxaloacetato para su ingreso al ciclo de Krebs (Appleton et al., 2013c). Si su oxidación es completa, se liberará CO2 y pares de átomos de H (Friedman & Nunnari, 2014), que donarán sus electrones para efectuar reacciones de óxido reducción (Madigan et al., 2019c), la formación de H2O y almacenamiento de energía en forma de ATP (Jump, 2011).
Sin embargo, si el oxaloacetato no es suficiente, la acetil-Coenzima A se acumula dentro de la mitocondria (Longo et al., 2016). Posteriormente dos moléculas de acetil-Coenzima A reaccionan para formar acetoacetil-CoA, en una reacción catalizada por tiolasa (Merritt et al., 2018). El acetoacetil-CoA se condensa con otra molécula de acetil-Coenzima A, para formar β-hidroxi-β-metilglutaril-CoA (Mas, 2018a). A partir de esta molécula se metaboliza acetoacetato (AcAc), cuerpo cetónico que sale de la mitocondria y en el citoplasma del hepatocito puede reducirse a β-hidroxibutirato (β-HBA) (Selvaraj et al., 2020) o descarboxilarse lenta y espontáneamente hasta acetona (Ac) (Deemer et al., 2020).
PROPIEDADES FISICOQUÍMICAS DE LAS PROTEÍNAS
De las tres biomoléculas discutidas, las proteínas son las únicas que contienen átomos de nitrógeno (N) (Ferrier, 2017c). Están constituidas por la combinación de 20 aminoácidos (aa) (Ahern, 2019a), unidos mediante un enlace peptídico (Guoyao, 2017b). Este enlace de tipo covalente, une el grupo amino (NH2) de un aa y el grupo COOH de otro, con la formación de una molécula de H2O (Madigan et al., 2019b). Las proteínas participan activamente en la homeostasis celular (Cooper, 2019b), p. ej., transportando O (Guoyao, 2017b), estructurando inmunoglobulinas (Kenneth & Casey, 2017) y constituyendo enzimas (Ahern, 2019c).
Su ingreso en el organismo, es a partir del alimento y su hidrólisis (ruptura de enlaces peptídicos) por peptidasas o proteasas y aminotransferasas, producidas por las células acinares del páncreas (Ahern, 2019c). Posterior a esta hidrólisis se liberan aa (Rodwell, 2018a), para ser absorbidos por medio del epitelio intestinal (Guoyao, 2017e; Piña & Flores, 2018), y transportados hacia los hepatocitos del hígado (Appleton et al., 2013b), para su posterior exportación hacia los tejidos periféricos (Fernández & Peimbert, 2018).
Dentro del citoplasma celular, los aa pueden perder su grupo NH2 y como esqueletos carbonados funcionar como: i) substrato para sintetizar C3H3O3 y posteriormente acetil- Coenzima A (Appleton et al., 2013d), ii) estructurar purinas y neurotransmisores (Rodwell, 2018b) y iii) participar en la proteogénesis (Rodwell, 2018a; Madigan et al., 2019b) o en la ureogénesis (Nelson & Cox, 2017a) principalmente.
ANABOLISMO DE LAS PROTEÍNAS (PROTEOGÉNESIS)
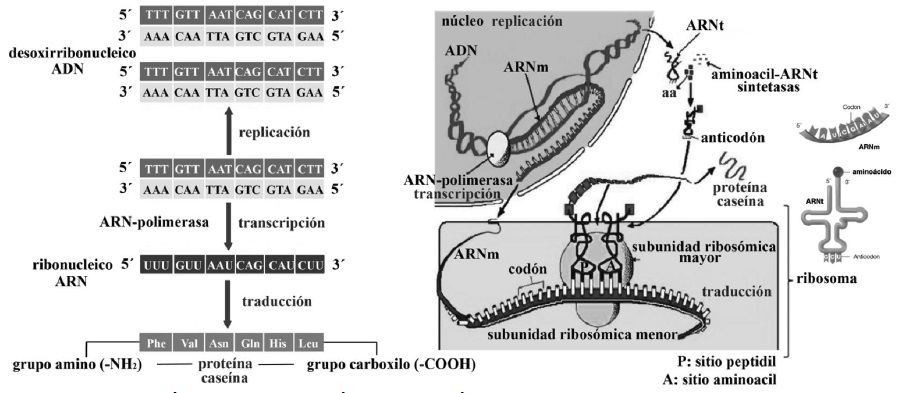
La proteogénesis (Figura 13), comienza en el núcleo celular (Noller, 2017), con la transcripción del ribonucleico de transferencia (ARNt) (Nelson & Cox, 2017d; Madigan etal., 2019d). Posteriormente, la enzima ARN-polimerasa realiza la transcripción del ribonucleico mensajero (ARNm) a partir de una secuencia de desoxirribonucleico (ADN) (Liu et al., 2013), que sirve como patrón o molde de la información genética (Litwack, 2018b). El ARNm se transporta hasta el retículo endoplasmático rugoso y a sus ribosomas (Weil, 2018b). Durante la iniciación, se forma un puente entre la subunidad ribosómica menor y la mayor (Weil, 2018a).
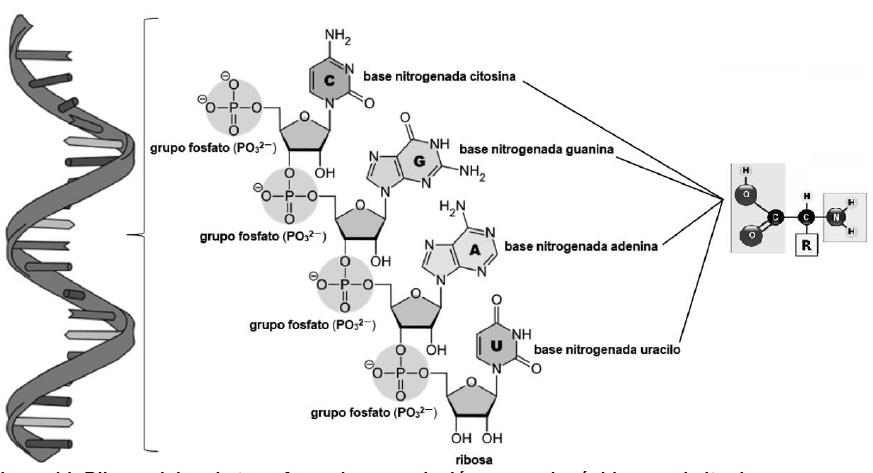
Por su parte, el ARNt (Figura 14), tienen que unirse con diferentes aminoacil-ARNt- sintetasas (Rodnina & Wintermeyer, 2016), para exponer el grupo NH2 de sus bases nitrogenadas (citosina, guanina, adenina y uracilo) y fijar el grupo COOH de los diferentes aa (Smith, 2020a).
Los aa transportados en el ARNt ingresan en el complejo ribosomal que presenta dos sitios de unión: i) el sitio P o peptidil y ii) el sitio A o aminoacil (Berk et al., 2006). La traducción se lleva a cabo en los ribosomas, mediante la lectura de tripletes (de tres en tres nucleótidos) llamados: codón para el ARNm y anticodón para el ARNt (Ingolia, 2014). La primera etapa de traducción, comienza cuando el extremo 5' del ARNm se inserta en la subunidad ribosómica menor (Nelson & Cox, 2017d), exponiendo el codón iniciador adenina-uracilo-guanina o AUG para su unión con el primer anticodón uracilo-adenina- citosina o UAC, en el sitio peptidil (Angov, 2011), originando metionina como primer aa (Madigan et al., 2019d).
Posteriormente, cuando el sitio peptidil y el sitio aminoacil están ocupados simultáneamente, la enzima peptidil transferasa establece un enlace peptídico entre los aa, insertando el primero en el segundo (Weil, 2018a). A continuación, en la elongación codón y anticodón se van asociando de manera precisa según la complementariedad de sus bases (Dutta & Nandi, 2012), y esta secuencia de pasos es repetida según el número de aa que contenga el polipéptido (Madigan et al., 2019b). Como terminación de este proceso, se traducen diferentes proteínas y enzimas principalmente hidrolasas (Swiderek et al., 2015).
CATABOLISMO DE LAS PROTEÍNAS (PROTEÓLISIS) Y UREOGÉNESIS
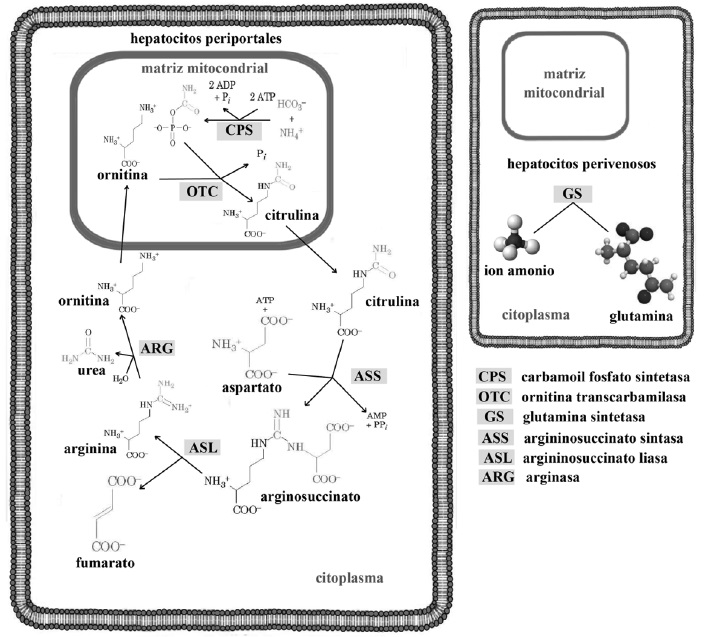
Posterior a la digestión gástrica y enzimática de las proteínas, la ruptura de sus enlaces peptídicos, y la liberación y absorción de aa (Piña & Flores, 2018), se obtiene también ion amonio (NH4+) (Rodwell, 2018a). Esta molécula viaja al hígado, donde su primer contacto es con los hepatocitos periportales (Guoyao, 2017e), que poseen en su estructura enzimas ureagénicas encargadas de la síntesis de urea (Figura 15). En la mitocondria de los hepatocitos periportales, se condensan HCO3-, NH4+ y ATP (Appleton et al., 2013b) para formar carbamil fosfato (Friedman & Nunnari, 2014). La ornitina ingresa a la mitocondria y el carbamil fosfato cede su grupo carbamilo para formar citrulina (Weiner et al., 2015).
La citrulina sale de la mitocondria al citoplasma, donde se une al aspartato, formando arginosuccinato (Menzies et al., 2016). El arginosuccinato es dividido en dos: i) arginina (Arg) y ii) fumarato. La Arg es hidrolizada por la arginasa liberando urea y ornitina (Nelson & Cox, 2017a). Esta última entra en la mitocondria para iniciar otra vuelta en el ciclo (Rodwell, 2018a). La urea por su parte, puede viajar al riñón (Guoyao, 2017b) y ser excretada en orina (Marini & van Amburgh, 2003). El ion NH4+ que no es metabolizado en urea, tiene contacto con los hepatocitos perivenosos, que poseen en su estructura glutamina sintetasa (Piña & Flores, 2018), que convierte ion NH4+ en glutamina (Gln). Este aa polar o hidrofílico, presenta afinidad por el H2O (Appleton et al., 2013b). Por lo tanto, favorece el transporte y excreción del ion NH4+ en la orina (Rodwell, 2018a).
ANABOLISMO DE ADENOSINA TRIFOSFATO (CICLO DE KREBS)
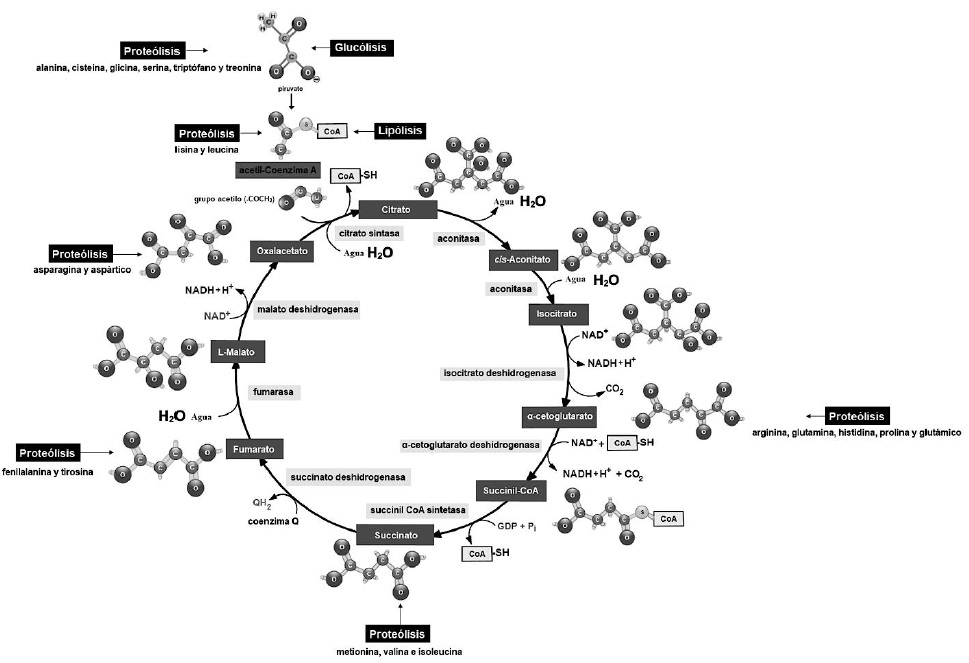
El ciclo de Krebs fue descubierto por Hans Adolf Krebs (Appleton et al., 2013c). Forma parte del intercambio gaseoso mitocondrial (Madigan et al., 2019c) y permite liberar la energía almacenada del acetil-Coenzima A en forma del nucleótido ATP (Botham & Mayes, 2018d). El acetil-Coenzima A sede su grupo COCH3 para unirse con oxaloacetato y formar citrato mediante una reacción de condensación (Menzies et al., 2016; Verschueren et al., 2019). Durante una vuelta completa del ciclo y a través de hidrólisis, descarboxilación oxidativa e hidratación (Figura 16), el citrato se convierte de nuevo en oxaloacetato (Appleton et al., 2013d).
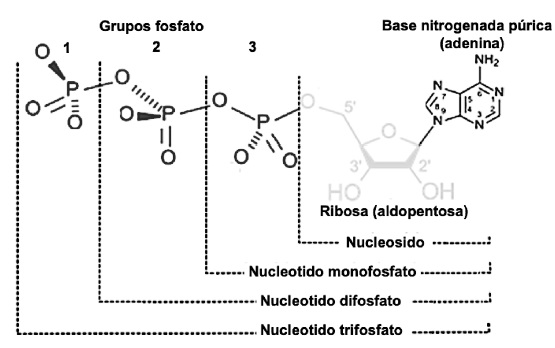
Los átomos de C que se liberan en el proceso forman CO2 (Madigan et al., 2019c). El ciclo de Krebs consume por cada vuelta un acetil-Coenzima A y tres NAD+ (Nelson & Cox, 2017e). Produce por cada vuelta dos CO2 y tres NADPH+H+ (Friedman & Nunnari, 2014). Por cada acetil-Coenzima A que ingresa en el ciclo de Krebs se producen 12 ATP (Appleton et al., 2013c), cada uno formado por una base nitrogenada púrica o purina (adenina), unida a una ribosa (aldopentosa) y a tres PO42− (Botham & Mayes, 2018a) (Figura 17).
Por cada GLU (C6H12O6) que ingresa en el ciclo se producen dos C3H3O3, que a su vez producen dos acetil-Coenzima A (Nelson & Cox, 2017e). Por lo tanto, por cada GLU (C6H12O6) que ingresa en el ciclo de Krebs se producen cuatro CO2, seis NADPH+H+ y 24 moléculas de ATP (Friedman & Nunnari, 2014).
La información presentada en párrafos anteriores, muestra cómo las biomoléculas que constituyen los organismos vivos, interactúan para mantener y perpetuar la vida, gobernada por las mismas leyes físicas y químicas que gobiernan el universo inerte. La frontera del conocimiento, se organizó en torno a principios o cuestiones centrales de la bioquímica y cómo las células utilizan un conjunto relativamente pequeño de metabolitos basados en carbono para crear moléculas poliméricas, estructuras supramoleculares y depósitos de información. La estructura química de estos componentes define su función celular, cuyo resultado final es la transformación y la autoperpetuación de esa compilación de biomoléculas, en resumen, la vida.
CONCLUSIONES
Las células eucariontes están compuestas de agua, iones inorgánicos y moléculas orgánicas. Contienen cadenas carbonadas con grupos funcionales hidroxilo, amino y carboxilo, responsables de la formación de tejido celular. Estas estructuras obedecen las leyes de la química y la física que determinan el metabolismo de los sistemas vivos. Los animales al poseer una elevada complejidad química y una robusta organización microscópica, constituyen en su anabolismo y catabolismo molecular, sistemas de extracción, transformación y aprovechamiento de monosacáridos, aminoácidos y ácidos grasos. Para la formación de acetil-Coenzima A y la liberación de su energía en el ciclo de Krebs. Así, la bioquímica del metabolismo celular, puede entenderse en términos de las estructuras y funciones de tres clases principales de moléculas orgánicas polisacáridos, lípidos y proteínas.











 texto en
texto en