INTRODUCCIÓN
La tortuga verde del Pacífico Oriental (Chelonia mydas) en peligro de extinción, localmente conocida como tortuga negra/prieta, se distribuye a lo largo de la costa del Pacífico americano, ocupando diferentes regiones geográficas durante cada una de las etapas de su ciclo de vida (Cliffton et al., 1982). Estos organismos son susceptibles a amenazas específicas de su ambiente y de origen antropogénico (Aguirre et al., 2006), por lo tanto, el estudio de las condiciones de salud de las poblaciones en vida libre, incluida la presencia de agentes infecciosos, está recibiendo cada vez mayor atención para su conservación.
Las tortugas marinas en vida libre albergan de forma natural una amplia variedad de bacterias en su tracto gastrointestinal; sin embargo, también se ha reportado su comportamiento oportunista y patogenicidad (Ahasan et al., 2018). En un organismo, las comunidades de microorganismos asociados son esenciales para una amplia variedad de funciones. El desarrollo del huésped y su estado de salud dependen de la presencia de una comunidad microbiana intacta, que juega un papel importante en todos los organismos vivos (Bloodgood et al., 2020). En diferentes especies, se ha demostrado que la microbiota promueve el desarrollo de órganos y tejidos, la producción de vitaminas y aminoácidos esenciales que afectan la utilización de grasas (Koropatnick et al., 2004), y la respuesta de la glucosa y linfocitos a las lesiones intestinales (Warwick et al., 2013). Las diferentes bacterias del tracto gastrointestinal de las tortugas marinas pueden proporcionar diversas funciones que aún se desconocen y que sugieren jugar un papel importante en la asimilación de los alimentos y su aprovechamiento (Ahasan et al., 2017). No obstante, estos microorganismos también pueden causar daño al huésped, por ejemplo, aumentando la susceptibilidad a inflamaciones intestinales y enfermedades infecciosas principalmente (Garner et al., 1995). Internacionalmente, se ha reportado la presencia de diversas bacterias en tortugas marinas tales como: Salmonella, Mycobacterium, Escherichia coli, Citrobacter freundii, Edwarsiella sp., Vibrio alginolyticus, Vibrio cholerae, Vibrio fluvialis, Vibrio furnisii, Vibrio parahaemolyticus, Aeromonas y Proteus, entre otras (Work et al., 2003; Orós et al., 2005; Santoro et al., 2006; Zavala- Norzagaray et al., 2015) y han sido identificados como oportunistas y potencialmente patógenas para las tortugas. Además, se han reportado algunos efectos adversos para la salud en humanos que consumen carne y huevos de tortugas marinas infectadas con patógenos zoonóticos, una práctica ilegal común en los países costeros de todo el mundo (Aguirre et al., 2006). En México, el conocimiento de los microorganismos relacionados con enfermedades en tortugas marinas aún es limitado. Por ello, es importante generar información veraz sobre los posibles riesgos para las tortugas y la salud humana asociados a su consumo ilegal. Este estudio tuvo como objetivo proporcionar la línea base sobre agentes bacterianos potencialmente patógenos para las tortugas y de tipo zoonótico, a través de cultivos bacteriológicos y métodos bioquímicos en muestras orales y cloacales de tortugas negras (C. mydas) en vida libre del complejo lagunar Ojo de Liebre, Baja California Sur (BCS), México.
MATERIAL Y MÉTODOS
Sitio de estudio
La laguna Ojo de Liebre (LOL) y la laguna Guerrero Negro (GNO) pertenecen al complejo lagunar Ojo de Liebre y son parte de la Reserva de la Biosfera “El Vizcaíno”, ubicada en el Pacífico norte de BCS, entre la latitud 27° 35′ y 28° 15′ Norte, y la longitud 113° 50′ y 114° 20′ Oeste. Ambas lagunas son hipersalinas y no tienen aportes de agua dulce; presentan un alto grado de evaporación y una circulación de agua lenta. En conjunto, las características oceanográficas y climatológicas de la región brindan un hábitat de alta riqueza, que representa uno de los principales sitios de alimentación y desarrollo de tortugas negras en sus estadios juveniles y adultos en el Pacífico mexicano (Reséndiz et al., 2018a).
Recolección y procesamiento de muestras
Se realizaron un total de cinco salidas de campo mensuales desde septiembre de 2019 a febrero de 2020, con capturas de tortugas negras en LOLy GNO. Todas las capturas se realizaron con redes de monofilamento tipo “Castillo” durante el día. Los animales capturados se sometieron a un examen físico (Norton, 2005), y a continuación se realizaron raspados orales y cloacales con hisopos estériles. Para ello, se realizaron movimientos circulares y rotatorios con el hisopo en la superficie interna de la boca y la superficie interna de la cloaca y se colocaron en medio de cultivo gel Stuart COPAN® para su transporte. Enseguida, se registró la longitud curva del caparazón (LCC; centímetros), la longitud recta del caparazón (LRC; centímetros) y el peso (kilogramos) (Bolten, 1999); se consideraron adultos aquellos organismos con un LCC superior a 77.5 cm (Márquez, 1996). Inmediatamente después, las tortugas fueron marcadas con placas metálicas de Inconel 625 en las aletas traseras (Balazs, 1999) y liberadas ilesas en el sitio de captura. Las muestras se refrigeraron a 4ºC y se trasladaron al laboratorio de Microbiología de la Universidad Autónoma de Baja California Sur, donde fueron sembradas en agar McConkey para la identificación de enterobacterias y bacilos gramnegativos (incubación durante 24h a 37ºC), y en agar tiosulfato citrato bilis sacarosa (TCBS) para la identificación de bacterias del género Vibrio (incubación durante 24h a 30ºC). Posteriormente, las colonias fueron aisladas y procesadas con el sistema de pruebas bioquímicas para la identificación de bacterias de la familia Enterobacteriaceae y otros bacilos API®20E. Las reacciones se leyeron de acuerdo con la tabla de identificación “Índice de perfil analítico API®20E”.
Análisis de datos
El índice de condición corporal (ICC) de cada organismo se calculó con la fórmula propuesta por Bjorndal et al., (2000):
Adicionalmente se calcularon las medias y las desviaciones estándar de LCC, LRC, peso e ICC. Se estimaron los porcentajes de abundancia relativa de cada agente bacteriano en la boca y cloaca con la siguiente fórmula:
Enseguida, se calculó la prevalencia de cada agente en los organismos con la fórmula:
Finalmente, se utilizó la prueba de Kruskal-Wallis para determinar la relación entre el tamaño de los organismos (LCC) y la presencia de las diferentes especies de bacterias. Los valores de p≤ 0.05 se consideraron estadísticamente significativos. Los análisis estadísticos se realizaron en R versión 3.6.2.
RESULTADOS
Se capturaron un total de 178 tortugas negras, las cuales midieron 72.37 ± 11.98 cm de LCC, 67.27 ± 11.26 cm de LRC y pesaron 44.61 ± 21.15 kg. Las tortugas se clasificaron como 125 juveniles y 53 adultas y su ICC fue de 1.38 ± 0.18. Se obtuvieron un total de 523 aislamientos asociados con enterobacterias gramnegativas de 7 especies diferentes (Tabla 1).
Tabla 1 Descripción cuantitativa de los agentes bacterianos aislados de tortugas negras (Chelonia mydas) juveniles y adultos en el complejo lagunar Ojo de Liebre, Baja California Sur, México.
| Familia | Especie | Aislamientos totales | Abundancia relativa (%) | Prevalencia (%) | Aislamientos en boca | Abundancia relativa en boca (%) | Aislamientos en cloaca | Abundancia relativa en cloaca (%) |
|---|---|---|---|---|---|---|---|---|
| Enterobacteriaceae | Citrobacter freundii | 154 | 29.45 | 86.52 | 101 | 29.79 | 53 | 28.80 |
| Klebsiella sp. | 107 | 20.46 | 60.11 | 107 | 31.56 | 0 | 0.00 | |
| Enterobacter sp. | 78 | 14.91 | 43.82 | 78 | 23.01 | 0 | 0.00 | |
| Pseudomonadaceae | Pseudomonas aeruginosa | 88 | 16.83 | 49.44 | 52 | 15.34 | 36 | 19.57 |
| Morganellaceae | Proteus sp. | 79 | 15.11 | 44.38 | 0 | 0.00 | 79 | 42.93 |
| Burkholderiaceae | Burkholderia cepacia | 8 | 1.53 | 4.49 | 1 | 0.29 | 7 | 3.80 |
| Vibrionaceae | Vibrio fluvialis | 9 | 1.72 | 5.06 | 0 | 0.00 | 9 | 4.89 |
| Total | 523 | 100 | 339 | 100 | 184 | 100 |
En la boca de las tortugas, las bacterias más abundantes fueron Kleibsiella sp. (31.56%), seguido de Citrobacter freundii (29.79%), Enterobacter sp. (23.01%), Pseudomonas aeruginosa (15.34%) y Burkholderia cepacia (0.29%), mientras que en la cloaca la más abundante fue Proteus sp. (42.93%), Citrobacter freundii (28.80%), Pseudomonas aeruginosa (19.57%), Vibrio fluvialis (4.89%) y finalmente Burkholderia cepacia (3.80%) (Figura 1).
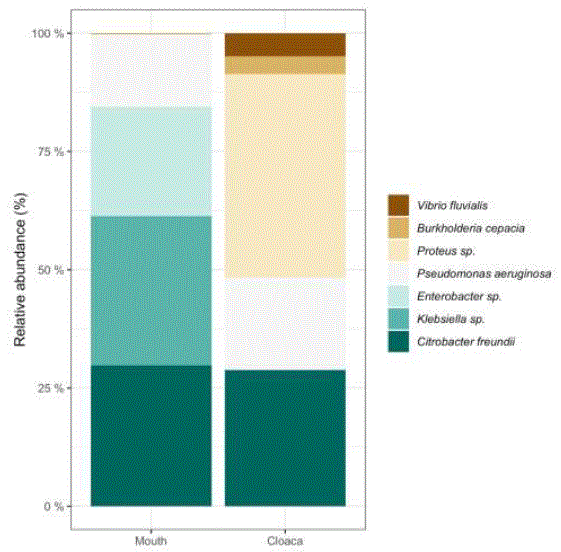
Figura 1 Abundancia relativa de agentes bacterianos en la boca y cloaca de tortugas negras (Chelonia mydas) en el complejo lagunar Ojo de Liebre, Baja California Sur, México
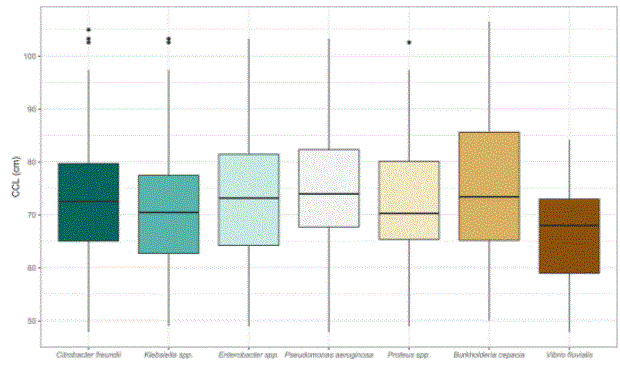
La prueba de Kruskal-Wallis no mostró diferencias significativas entre la presencia de los diferentes microorganismos y el LCC de las tortugas (Ji-cuadrada= 5.75, gl= 6, valor de p= 0.45) (Figura 2).
DISCUSIÓN
Las tortugas fueron clasificadas como juveniles y adultas de acuerdo a la talla reportada para el área (Márquez, 1996). Su ICC coincidió con lo reportado previamente para el área (Reséndiz et al., 2018b) indicando que las tortugas tenían un buen estado nutricional y presumiblemente la capacidad para un desempeño reproductivo favorable. El examen físico no mostró evidencia de signos clínicos, lesiones graves o enfermedades que comprometieran el funcionamiento de los órganos y sistemas de las tortugas ni que pusieran en riesgo sus actividades normales o atentaran contra su integridad (Norton, 2005).
Es importante considerar que las diferencias en las comunidades de microorganismos de las tortugas marinas se atribuyen principalmente a factores ecológicos y ontogénicos (nivel trófico, dieta, hábitat, etc.) (Ahasan et al., 2018), por lo que la composición bacteriana gastrointestinal difiere notablemente entre poblaciones en vida libre y organismos en rehabilitación (antes de la hospitalización y después de la rehabilitación) (Pace et al., 2019; Bloodgood et al., 2020). En general, la alta prevalencia de proteobacterias se ha asociado con disbiosis además del deterioro en el estado de salud de las tortugas marinas (Ahasan et al., 2018). Citrobacter freundii mostró la mayor prevalencia (86.52%). Esta bacteria gramnegativa de la familia Enterobacteriaceae ha sido reportada anteriormente en tortugas marinas internacionalmente (Santoro et al., 2006), nacionalmente en tortugas negras en Sinaloa (Zavala-Norzagaray et al., 2015) y en tortugas amarillas (Caretta caretta) en el Golfo de Ulloa, BCS (Reséndiz et al., 2019). Se considera un patógeno oportunista asociado a infecciones en animales juveniles e infecciones secundarias en animales inmunodeprimidos (Glazebrook y Campbell, 1990), y no es zoonótico (Johnson- Delaney, 2014). En tortugas marinas se desconoce la dosis infecciosa y el período de incubación. Se ha reportado que su transmisión es por vía fecal-oral al ingerir alimentos contaminados o por contacto directo entre el portador y organismos susceptibles e inmunosuprimidos (Johnson-Delaney, 2014), esta especie se encuentra en el tracto gastrointestinal de los animales y utiliza cuerpos de agua como reservorio (Work et al., 2003). Kleibsiella sp. tuvo una prevalencia de 60.11%. Esta bacteria gramnegativa de la familia Enterobacteriaceae se reportó anteriormente en tortugas marinas a nivel internacional (Warwick et al., 2013) y nacional en tortugas amarillas en el Golfo de Ulloa, BCS (Reséndiz et al., 2019). Este patógeno juega un papel importante como causante de enfermedades infecciosas oportunistas, principalmente en organismos inmunodeprimidos y frecuentemente en animales juveniles (Work et al., 2003). También se ha asociado con bacteriemia, infecciones de heridas e infecciones respiratorias y urinarias (Johnson-Delaney, 2014). Se desconoce la dosis infecciosa y el período de incubación en las tortugas marinas. No es zoonótica y se desconocen sus vectores, aunque se ha informado que su vía de transmisión más frecuente es a través de las heces (Jacobson, 2007). Este género se puede aislar del suelo, cuerpos de agua, piel, exudados nasofaríngeos, o bien del tracto gastrointestinal de tortugas portadoras (Glazebrook y Campbell, 1990) y puede sobrevivir en ambientes marinos durante varias horas o en animales clínicamente sanos durante largos períodos (Tan et al., 2009). Pseudomonas aeruginosa presentó una prevalencia del 49.44%. Estos bacilos gramnegativos pertenecen a la familia Pseudomonadaceae (Buller, 2004) y han sido estudiados en tortugas marinas a nivel internacional (Work et al., 2003; Santoro et al., 2006) y nacional (Reséndiz et al., 2019). Estos son patógenos oportunistas que sugieren un mayor riesgo de enfermedad en tortugas juveniles inmunodeprimidas y con fibropapilomatosis (Work et al., 2003); la mayoría de sus afecciones surgen de la colonización de las vías respiratorias y urinarias o de infecciones de diseminación profunda que pueden provocar neumonía, bacteriemia e infecciones respiratorias crónicas (Buller, 2004; Jorgensen y Ferraro, 2009). No son zoonóticos y en tortugas marinas no se conocen vectores (Work et al., 2003; Johnson-Delaney, 2014); la dosis infecciosa en las tortugas marinas también se desconoce y su período de incubación varía según la infección (Orós et al., 2005; Jorgensen y Ferraro, 2009). Esta especie se transmite por contacto directo con agua contaminada, aerosoles o aspiración por contacto de membranas mucosas con descargas de conjuntivas infectadas o tracto respiratorio superior de organismos infectados (Johnson-Delaney, 2014) y puede sobrevivir durante varios meses en el medio marino con nutrientes básicos (Jacobson, 2007). Proteus sp. tuvo una prevalencia de 44.38%. Estos bacilos gramnegativos de la familia Morganellaceae han sido reportados en otras especies de tortugas marinas sanas y enfermas a nivel internacional (Santoro et al., 2006), en México, en tortugas negras (Zavala-Norzagaray et al., 2015) y en tortugas amarillas clínicamente sanas (Reséndiz et al., 2019). Se les considera parte de la microbiota gastrointestinal de las tortugas marinas, sin embargo, pueden generar infecciones crónicas del tracto urinario como bacteriemia, neumonía y lesiones focales en organismos debilitados y emaciados (Work et al., 2003). No son zoonóticos y no se conocen vectores de transmisión; además, en las tortugas marinas, se desconoce la dosis infecciosa y el período de incubación no está bien establecido (Johnson-Delaney, 2014), aunque se ha reportado que causa infecciones al salir del tracto gastrointestinal (Jorgensen y Ferraro, 2009). Este género no se transmite por contacto directo entre organismos y se puede encontrar en cuerpos de agua (Jacobson, 2007). Sobrevive fácilmente fuera del huésped, especialmente en áreas donde existe descomposición de proteína animal (Buller, 2004; Jacobson, 2007). Enterobacer sp. mostró una prevalencia del 43.82%. Esta enterobacteria gramnegativa se reportó en tortugas amarillas en el Golfo de Ulloa, BCS (Reséndiz et al., 2019) y en otras especies de tortugas marinas a nivel internacional (Work et al., 2003; Zavala-Norzagaray et al., 2015). Se considera un patógeno oportunista que se ha asociado con brotes infecciosos como la fibropapilomatosis en tortugas juveniles (Work et al., 2003). Puede causar numerosas infecciones, como neumonía, sepsis en el tracto intestinal y en el tracto urinario, que pueden provocar bacteriemia (Work et al., 2003; Orós et al., 2005). No es zoonótico y no se conocen vectores (Glazebrook y Campbell, 1990; Warwick et al., 2013). Además, se desconoce su período de incubación y dosis infecciosa en tortugas marinas. No obstante, se sabe que se transmite por contacto directo o indirecto de las superficies mucosas con el agente infeccioso y también se puede transmitir por vía fecal-oral (Johnson-Delaney, 2014). Este género es colonizador del tracto gastrointestinal inferior de humanos, animales y se puede encontrar con frecuencia en plantas, suelo, cuerpos de agua y es capaz de sobrevivir con una fuente mínima de energía (Pace et al., 2019). Vibrio fluvialis tuvo una prevalencia del 5.06%. Este bacilo gramnegativo de la familia Vibrionaceae ha sido reportado en tortugas marinas enfermas y sanas a nivel internacional y nacional (Zavala-Norzagaray et al., 2015; Reséndiz et al., 2019). Se considera un patógeno emergente que se transmite al comer alimentos contaminados (Ahasan et al., 2017; Franco-Monsreal et al., 2014). Se desconocen todas sus consecuencias en las tortugas marinas, pero en los mamíferos causa diarrea similar al cólera, infecciones cutáneas asociadas a la exposición a ambientes acuáticos e incluso sepsis en individuos inmunodeprimidos (Franco-Monsreal et al., 2014). Se desconoce su período de incubación y dosis infecciosa en tortugas marinas. Este microorganismo es zoonótico (Johnson-Delaney, 2014) y puede sobrevivir durante largos períodos de tiempo en el medio marino, donde se distribuye ampliamente (Igbinosa y Okoh, 2010; Franco-Monsreal et al., 2014). Algunos reportes indican que también se ha aislado de aguas residuales, heces animales y humanas, así como mariscos, principalmente en moluscos bivalvos (Alton et al., 2006; Igbinosa y Okoh, 2010). Finalmente, Burkholderia cepacia tuvo una prevalencia del 4.49%. Esta bacteria gramnegativa de la familia Burkholderiaceae ha sido reportada previamente a nivel internacional y nacional en tortugas golfinas (Lepidochelys olivacea) (Santoro et al., 2006; Zavala-Norzagaray et al., 2015), tortugas verdes (C. mydas) con y sin fibropapilomatosis (Work et al., 2003) y en tortugas amarillas clínicamente sanas (Reséndiz et al., 2019). Este microorganismo patógeno puede ocasionar enfermedad pulmonar crónica con secreción mucopurulenta, caracterizada por múltiples abscesos en la piel y tejidos subcutáneos, o una sepsis grave con muerte en aproximadamente 7-10 días (Orós et al., 2005; Jacobson, 2007). En tortugas marinas se desconoce la dosis infecciosa, su período de incubación varía de 1 a 14 días (Johnson-Delaney, 2014) y se considera zoonótica por contacto directo o indirecto de la mucosa con descargas de lesiones de animales infectados (Warwick et al., 2013; Johnson-Delaney, 2014). Se ha reportado en mamíferos, reptiles y peces, considerando a los humanos como huéspedes accidentales (Buller, 2004; Jorgensen y Ferraro, 2009). Se puede encontrar en el suelo, cuerpos de agua y áreas cercanas a las actividades agrícolas, donde puede sobrevivir durante largos períodos a temperatura ambiente (Buller, 2004; Jacobson, 2007).
La presencia de estos agentes bacterianos en las tortugas marinas no se relacionó con su talla; la prueba de Kruskal-Wallis no mostró diferencias significativas entre la presencia de los diferentes microorganismos y el LCC de las tortugas, lo cual indica que las bacterias encontradas se presentaron indistintamente en animales juveniles y adultos. Dentro de las proteobacterias encontradas en las tortugas negras, Vibrio fluvialis y Burkholderia cepacia son patógenos zoonóticos, y aunque tienen una baja prevalencia, deben ser monitoreados regularmente para prevenir riesgos tanto en las tortugas como en la salud pública. Puesto que, a pesar de la prohibición federal de captura, consumo y comercio de tortugas marinas en México desde 1990, estos organismos continúan siendo capturados y consumidos. Este hecho representa un gran peligro potencial para la salud humana (Aguirre et al., 2006), pudiendo causar deshidratación extrema, vómitos, diarreas e incluso la muerte a los consumidores debido a la presencia de estos microorganismos (Alton et al., 2006), además de virus, parásitos o contaminantes en las tortugas marinas (Zavala-Norzagaray et al., 2015). La información generada advierte sobre posibles riesgos para la salud de los consumidores de tortugas marinas; además de ser una práctica ilegal, es potencialmente peligrosa para la salud pública y afecta a las poblaciones de las diferentes especies protegidas. Este estudio complementa las evaluaciones de salud de las tortugas negras en la zona y los planes de manejo y conservación de los organismos y sus ecosistemas en la Reserva de la Biosfera “El Vizcaíno”, junto con las autoridades locales.
CONCLUSIONES
Se reportaron siete agentes bacterianos potencialmente patógenos para las tortugas marinas en individuos de tortuga negra aparentemente sanos, de los cuales dos son zoonóticos. Se requiere evidencia clínica contundente para definir si estos microorganismos causan enfermedades y se necesitan más estudios específicos para aclarar las diferencias entre la microbiota y la patobiota de las tortugas marinas, especialmente con métodos moleculares.











 text in
text in