Introducción
La oveja Pelibuey es la raza de pelo más importante en México. Esta raza ingresó al país entre 1930 y 1940 procedente de Cuba1,2,3, y desde entonces no ha entrado al país material genético nuevo. Inicialmente, la oveja Pelibuey se distribuyó en las regiones tropicales. Sin embargo, actualmente se encuentra diseminada en todo el país4. La oveja Pelibuey no es una raza altamente productiva, pero su importancia biológica radica en su capacidad para adaptarse a diferentes ambientes y climas5 y su habilidad para reproducirse a lo largo del año6.
El amplio rango de ambientes en los cuales la oveja Pelibuey fue criada, llevaron al desarrollo de respuestas adaptativas a esos ambientes, las cuales son parte de su acervo genético. Sin embargo, en años recientes, esta raza ha sido objeto de cruzas indiscriminadas con otras razas especializadas en la producción de carne para hacerla más productiva3,7. Esto ha puesto en riesgo la diversidad genética original de la raza. Además, prácticas como la inseminación artificial y el flujo intenso de material genético entre los rebaños han acentuado el problema8.
Un paso inicial para la conservación de los recursos genéticos animales es la caracterización de las razas. La Organización de las Naciones Unidas para la Alimentación y la Agricultura (FAO) ha propuesto que la caracterización inicialmente debe consistir en una descripción fenotípica de la raza y el posterior uso de marcadores moleculares para llevar a cabo la caracterización genética de diferentes razas de ovinos9. Marcadores moleculares, como los microsatélites, se han utilizado en estudios de genética de poblaciones para caracterizar un gran número de razas9. Un conocimiento más profundo sobre la diversidad y variabilidad genética, así como la estructura poblacional en diferentes rebaños de México, permitirá determinar el grado de riesgo de la oveja Pelibuey y, en su caso, sugerir estrategias viables para su conservación. Por lo tanto, el objetivo de este trabajo es caracterizar genéticamente mediante microsatélites diferentes subpoblaciones de Pelibuey en México.
Material y métodos
Muestreo
Se recolectaron 119 muestras de sangre de oveja Pelibuey que incluyeron 23 rebaños nacionales y un rebaño cubano. Los rebaños nacionales se muestrearon en las zonas agro-ecológicas correspondientes al trópico húmedo, trópico seco y la región montañosa central. La localización geográfica de los rebaños nacionales muestreados se puede observar en la Figura 1. Once de los 24 rebaños muestreados pertenecen a universidades e institutos de investigación, los 13 restantes corresponden a productores particulares. Todos los rebaños incluidos en el estudio se manejaron de acuerdo a los lineamientos del Subcomité Interno para el Cuidado y Uso de los Animales de Experimentación (SICUAE) de la Facultad de Medicina Veterinaria y Zootecnia de la UNAM. Los rebaños pertenecientes al Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (INIFAP) que se localizan en Yucatán (INIFAP Mocochá=IN-MOC) y Puebla (INIFAP Las Margaritas= IN-MAR) fueron considerados como las subpoblaciones nacionales de referencia, porque fueron los primeros en establecerse en México y su manejo ha tenido como objetivo la conservación de la raza. Asimismo, ambos han servido como base para la formación de otros rebaños nacionales. Los criterios para incluir a los animales en el muestreo fueron los siguientes: que tuvieran una apariencia externa característica de la oveja Pelibuey y que fueran hembras, clínicamente sanas y no emparentadas entre sí.
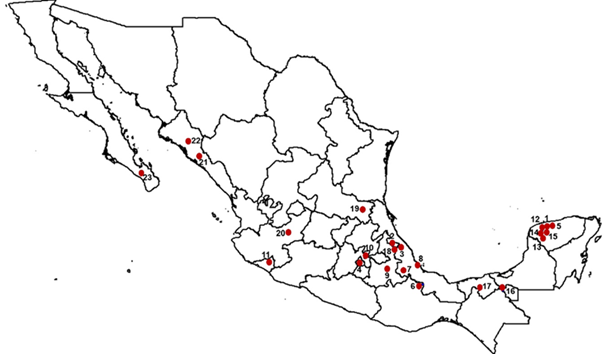
El orden de las subpoblaciones puede consultarse en el Cuadro 2. 1. INIFAP Mocochá (IN-MOC), 2. INIFAP Las Margaritas (IN-MAR), 3. Centro de Enseñanza, Investigación y Extensión en Ganadería Tropical (CEIEGT)-UNAM, 4. Centro de Enseñanza, Investigación y Extensión en Producción y Salud Animal (CEPIPSA)-UNAM, 5. Instituto Tecnológico de Conkal (ITC), 6. Universidad del Papaloapan (UNPA), 7. Colegio de Postgraduados Campus Córdoba (COLPOS-COR), 8. Universidad Veracruzana (UV), 9. Benemérita Universidad Autónoma de Puebla (BUAP), 10. Colegio de Postgraduados Campus Texcoco (COLPOS-TEX), 11. Universidad de Colima (UCOL), 12. Rancho San Alberto, 13. Rancho Garrido, 14. Rancho Belbesah, 15. Rancho Libertad, 16. Rancho Jalapa, 17. Rancho El Porvenir, 18. Rancho El Paraíso, 19. Rancho Santa Anita, 20. Posta El Cuatro, 21. Finca El Cielo, 22. Rancho La Fama y, 23. Rancho El Carrizal.
Figura 1 Localización de los rebaños nacionales de oveja Pelibuey que se incluyeron en el estudio
Cuadro 1 Estadísticas globales generadas con los nueve microsatélites
| Marcador | TA
(°C) |
RT
(bp) |
NA | NE | HO | HE | PIC | I | HWE | FIT | FST |
|---|---|---|---|---|---|---|---|---|---|---|---|
| OarCP34 | 58 | 96- 134 |
9 | 5.31 | 0.731 | 0.740 | 0.79 | 1.90 | N.S. | 0.111* | 0.099* |
| OarFCB304 | 58 | 140- 210 |
11 | 7.34 | 0.812 | 0.741 | 0.85 | 2.11 | 0.0285* | 0.075* | 0.154 |
| OarJMP29 | 56 | 110- 180 |
12 | 6.67 | 0.694 | 0.807 | 0.83 | 2.11 | 0.0002* | 0.185 | 0.056* |
| OarJMP58 | 58 | 130- 185 |
14 | 8.68 | 0.737 | 0.761 | 0.87 | 2.35 | N.S. | 0.173 | 0.148 |
| DYMS1 | 58 | 155- 225 |
11 | 8.53 | 0.697 | 0.778 | 0.87 | 2.26 | N.S. | 0.217* | 0.130 |
| ILSTS5 | 58 | 180- 235 |
9 | 6.65 | 0.781 | 0.664 | 0.84 | 2.00 | 0.0180* | 0.092* | 0.228* |
| SRCRSP5 | 58 | 146- 200 |
12 | 6.24 | 0.699 | 0.667 | 0.82 | 2.06 | N.S. | 0.180 | 0.215* |
| SRCRSP9 | 58 | 95- 135 |
10 | 7.65 | 0.657 | 0.723 | 0.86 | 2.14 | N.S. | 0.255* | 0.180 |
| MAF33 | 60 | 115- 170 |
11 | 5.73 | 0.730 | 0.707 | 0.80 | 1.97 | N.S. | 0.123 | 0.150 |
| Promedio | 11 | 6.97 | 0.726 | 0.732 | 0.84 | 2.1 | 0.158 | 0.151 |
Temperatura de alineamiento (TA), rango de tamaño de alelos (RT), número de alelos (NA), número efectivo de alelos (NE), heterocigosis observada (HO), heterocigosis esperada (HE), contenido de información polimórfica (PIC), índice de Shannon (I), desviación del equilibrio de Hardy-Weinberg (HWE), índice de fijación de la población total (FIT) e índice de diferenciación entre poblaciones (FST) de nueve marcadores microsatélites utilizados en la caracterización genética de la oveja Pelibuey
NS= no significativo; *P<0.05.
Cuadro 2 Número de animales (n), número total de alelos (TNA), número medio de alelos (MNA), número efectivo de alelos (NE), heterocigosis observada (HO), heterocigosis esperada (HE) y coeficiente de endogamia dentro de la población (FIS) en 24 subpoblaciones de ovejas Pelibuey, basado en el análisis de nueve marcadores microsatélites
| Población | Estado | n | TNA | MNA | NE | HO | HE | FIS |
|---|---|---|---|---|---|---|---|---|
| IN-MOC | Yucatán | 5 | 28 | 3.11 | 2.46 | 0.666 | 0.627 | -0.071 |
| IN-MAR | Puebla | 5 | 30 | 3.33 | 2.73 | 0.733 | 0.664 | -0.118* |
| CEIEGT | Veracruz | 5 | 30 | 3.33 | 2.62 | 0.689 | 0.651 | -0.064 |
| CEPIPSA | CdMx | 5 | 30 | 3.33 | 2.73 | 0.779 | 0.677 | -0.171* |
| ITC | Yucatán | 5 | 36 | 4.11 | 3.0 | 0.778 | 0.721 | -0.089* |
| UNPA | Oaxaca | 5 | 49 | 5.44 | 4.20 | 0.778 | 0.840 | 0.081 |
| COL-CORD | Veracruz | 5 | 37 | 4.11 | 3.72 | 0.756 | 0.782 | 0.038 |
| UV | Veracruz | 5 | 37 | 4.11 | 3.28 | 0.711 | 0.674 | -0.062 |
| BUAP | Puebla | 5 | 37 | 4.11 | 3.32 | 0.711 | 0.738 | 0.041 |
| COL-TEX | Edo. México | 5 | 43 | 4.78 | 4.10 | 0.800 | 0.803 | 0.003 |
| UCOL | Colima | 5 | 36 | 4.0 | 3.10 | 0.733 | 0.709 | -0.039 |
| Rancho San Alberto | Yucatán | 5 | 38 | 4.22 | 3.47 | 0.711 | 0.733 | 0.033 |
| Rancho Garrido | Yucatán | 5 | 38 | 4.22 | 3.20 | 0.689 | 0.751 | 0.091* |
| Rancho Belbesah | Yucatán | 5 | 39 | 4.33 | 3.58 | 0.733 | 0.758 | 0.036 |
| Rancho Libertad | Yucatán | 5 | 43 | 4.89 | 3.89 | 0.711 | 0.812 | 0.138* |
| Rancho Jalapa | Tabasco | 4 | 32 | 3.56 | 2.92 | 0.694 | 0.706 | 0.019 |
| Rancho El Porvenir | Tabasco | 5 | 37 | 4.11 | 3.32 | 0.733 | 0.733 | 0.000 |
| Rancho El Paraíso | Puebla | 5 | 34 | 3.78 | 2.77 | 0.733 | 0.686 | -0.077 |
| Rancho Santa Anita | SLP | 5 | 31 | 3.44 | 2.82 | 0.689 | 0.699 | 0.015 |
| Posta El Cuatro | Jalisco | 6 | 44 | 4.89 | 3.45 | 0.685 | 0.735 | 0.075 |
| Finca El Cielo | Sinaloa | 5 | 38 | 4.22 | 3.40 | 0.711 | 0.746 | 0.051 |
| Rancho La Fama | Sinaloa | 5 | 39 | 4.33 | 3.60 | 0.733 | 0.787 | 0.076 |
| Rancho El Carrizal | BCS | 4 | 36 | 4.0 | 3.26 | 0.694 | 0.778 | 0.122* |
| Cuba | La Habana | 5 | 37 | 4.11 | 3.27 | 0.779 | 0.738 | -0.060 |
| Total/Promedio | 119 | 36.6 | 4.08 | 3.25 | 0.726 | 0.731 | 0.0072 |
INIFAP: Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias; CEIEGT: Centro de Enseñanza, Investigación y Extensión en Ganadería Tropical; CEPIPSA: Centro de Enseñanza, Investigación y Extensión en Producción y Salud Animal; COLPOS: Colegio de Postgraduados; BUAP: Benemérita Universidad Autónoma de Puebla.
*Indica FIS diferente de cero (P˂0.05).
Siguiendo con las recomendaciones de la FAO, en el análisis se incluyeron cinco individuos de cada rebaño10. Debido a que el ADN de algunas muestras se degradó, en dos rebaños (Rancho Jalapa y Rancho El Carrizal) únicamente se incluyeron cuatro muestras.
Las muestras de sangre (6 ml) se obtuvieron mediante venopunción de la yugular, en tubos Vacutainer® con EDTA como anticoagulante y se identificaron al momento del muestreo. Las muestras se almacenaron a -80°C hasta su procesamiento y análisis.
Extracción de ADN y reacción en cadena de la polimerasa (PCR)
El aislamiento de ADN se llevó a cabo tomando como base un protocolo simple y barato para la extracción de ADN a partir de sangre de aves11, el cual posteriormente fue adaptado para sangre de ovinos12.
Los aislamientos se evaluaron para determinar su rendimiento y pureza por medio de espectrofotometría (NanoDrop Thermo Scientific, Wilminton, DE). Los resultados obtenidos se corroboraron mediante la elaboración de un gel de agarosa al 0.8%, utilizando 10 µl del ADN extraído. El ADN obtenido se almacenó a -80°C hasta su uso en la técnica de PCR.
Nueve marcadores microsatélites se amplificaron mediante PCR de acuerdo a las recomendaciones de la Sociedad Internacional de Genética Animal (ISAG) y de la FAO10,13. Las reacciones de PCR se llevaron a cabo en un volumen final de 25 µl y consistieron de 100 ng de ADN genómico (2 µl), 0.2 µM de cada uno de los iniciadores (Forward y Reverse; 0.5 µl), 200 µM de cada dNTP (2.5 µl), 2.5 mM de MgCl2+ (0.5 µl), 1.25 U de Taq DNA polimerasa (0.25 µl), amortiguador para PCR 10x (2.5 µl) y 16.25 µl de agua estéril.
Las reacciones de PCR se llevaron a cabo en un termociclador (Axygen Scientific Inc.). El protocolo de PCR fue como sigue: un paso inicial de desnaturalización a 95 °C por 5 min; 35 ciclos con fases de desnaturalización a 94 °C por 45 seg, alineamiento por 1 min a temperatura variable y extensión a 72 °C por 1 min; con un paso de extensión final a 72 °C por 10 min. Las reacciones de PCR se conservaron a 4 °C hasta su análisis.
Electroforesis
Las reacciones de PCR fueron sometidas a electroforesis en geles de poliacrilamida no desnaturalizantes al 12%. La electroforesis se llevó a cabo con TBE 0.5% como buffer de corrimiento. Para determinar el tamaño de los fragmentos de ADN, se utilizó un marcador de peso de 25 pb (Invitrogen Life Technologies, Carsbard, USA). Los geles se tiñeron con bromuro de etidio a una concentración de 0.5 µg/ml y fueron fotodocumentados en presencia de luz ultravioleta (Kodak Gel Logic 2200 Imaging System). Las imágenes de los geles se procesaron mediante el programa MyImageAnalysisSystemTM (Fisher).
Análisis estadístico
El número de alelos (NA), número efectivo de alelos (NE) número medio de alelos (MNA), índice de Shannon (I), heterocigosis observada (HO) y heterocigosis esperada (HE) para cada marcador fueron estimadas usando los programas POPGENE v.1.3114 y FSTAT15. El contenido de información polimórfica (PIC) se calculó usando el programa CERVUS v 3.0.716. Se realizaron pruebas exactas para determinar la desviación del equilibrio de Hardy-Weinberg (HWE) para cada marcador mediante técnicas de simulación Monte Carlo basadas en cadenas de Markov con el programa GENEPOP v 4.7.017,18.
La estructura genética se analizó por medio de los estadísticos F. Los valores del índice de endogamia dentro de la población (FIS), índice de fijación de la población total (FIT) e índice de diferenciación genética entre poblaciones (FST) se calcularon usando el programa GENEPOP v 4.7.0 realizando 1,000 permutaciones17,18. El análisis de alelos privados se realizó por medio del programa GENALEX v 6.519,20. El análisis de alelos compartidos entre las subpoblaciones consistió en la elaboración de una matriz binaria en la que se registró la presencia (1) y ausencia (0) de cada uno de los alelos en las subpoblaciones. Posteriormente, se calcularon las proporciones de las subpoblaciones que presentaron los alelos compartidos más comunes y los menos comunes.
El flujo génico entre las subpoblaciones fue medido a través del número de migrantes por generación (Nm) y calculado a partir del FST entre pares de subpoblaciones mediante el programa GENETIX v. 4.0521.
El aislamiento por distancia entre las subpoblaciones, se analizó mediante una prueba de Mantel22, la cual consistió en correlacionar las distancias genéticas (medidas a través de las distancias mínimas insesgadas de Nei23) y las distancias geográficas entre los pares de subpoblaciones usando el programa XLSTAT. Para la prueba se utilizó una significancia de 0.05 y 10,000 permutaciones.
Las relaciones espaciales entre las subpoblaciones se analizaron mediante un análisis de componentes principales (PCA), el cual se implementó en el programa PCAGEN24. La estructura de la población y el grado de mezcla se estimaron usando un modelo de agrupamiento bayesiano implementado en el programa STRUCTURE v 2.325. El análisis llevado a cabo involucró un modelo de mezcla con frecuencias de alelos correlacionadas. Para elegir el número apropiado de clústeres inferidos (K) para modelar los datos, se realizó la inferencia de 2-24 clústeres con 20 corridas independientes en cada uno. Todas las corridas usaron 100,000 interacciones (burn-in) seguidas por 1,000,000 interacciones (MCMC). El número más probable de K fue calculado con el algoritmo ΔK, a través del programa en línea STRUCTURE HARVESTER26. Finalmente, los resultados fueron procesados mediante el programa en línea CLUMPAK27 para interpretar las inferencias obtenidas.
Resultados
Diversidad genética de la población entera
Las estadísticas globales generadas con los nueve microsatélites se muestran en el Cuadro 1. Para la población entera de Pelibuey, se detectaron 99 alelos en 119 animales genotipificados con nueve iniciadores microsatélites. Todos los loci fueron polimórficos en las subpoblaciones analizadas. El número de alelos por locus varió de 9 (OarCP34, OarFCB304 e ILSTS5) a 14 (OarJMP58). Se observó un número promedio de alelos por locus de 11. El NE fue de 5.31 (OarCP34) a 8.68 (OarJMP58).
La HO tuvo valores entre 0.657 (SRCRSP9) y 0.812 (OarFCB304). El promedio de la HO para la población entera fue 0.726. Por otro lado, la HE fue de 0.664 (ILSTS5) a 0.807 (OarJMP29), con un promedio de 0.732. El PIC para cada marcador, varió de 0.79 (OarCP34) a 0.87 (OarJMP58 y DYMS1), con un promedio global de 0.84. El I se encontró entre 1.90 (OarCP34) y 2.35 (OarJMP58), con un promedio global de 2.1.
Los marcadores microsatélites fueron probados para la desviación del HWE. La mayoría de los loci estuvieron en HWE. Sin embargo, OarFCB304, OarJMP29 e ILSTS5 mostraron una desviación significativa del HWE (P<0.05). Los valores globales del FIS, del FIT y del FST fueron 0.007, 0.158 y 0.151, respectivamente y no mostraron diferencias significativas (P>0.05). El valor más alto de FIS (0.136) fue observado en el marcador OarJMP29, mientras que el valor más bajo (-0.176) se observó en el marcador ILSTS5. El valor más alto de FIT (0.255) se observó en el locus SRCRSP9 y el más bajo (0.075) en el locus OarFCB304. Asimismo, el valor más alto de FST (0.228) fue observado en el locus ILSTS5 y el más bajo (0.056) en el locus OarJMP29. Las diferencias entre subpoblaciones evaluadas por medio de los valores multilocus de FST revelaron que, la mayor parte de la variación genética corresponde a diferencias entre individuos dentro de las subpoblaciones (84.9%) y 15.1% resulta de las diferencias entre subpoblaciones.
Diversidad genética entre subpoblaciones
Las medidas de la diversidad genética para cada subpoblación se muestran en el Cuadro 2. La diversidad genética más alta fue observada en la subpoblación UNPA (49 alelos) y la más baja en la subpoblación IN-MOC (28 alelos), con un promedio global de 36.6 alelos por subpoblación. El MNA y NE mostraron promedios de 4.08 y 3.25, respectivamente, con valores más altos en la subpoblación UNPA (5.44 y 4.20, respectivamente) y valores más bajos en la subpoblación IN-MOC (3.11 y 2.46, respectivamente).
El promedio de la HO fue de 0.726. El valor más bajo se observó en la subpoblación IN-MOC (0.666) y el más alto en la subpoblación COL-TEX (0.800). El promedio de la HE fue 0.731. El valor más bajo se observó en la subpoblación IN-MOC (0.627) y el más alto en la subpoblación UNPA (0.840).
Los valores promedio de FIS para cada subpoblación considerando todos los loci, variaron de -0.171 (CEPIPSA) a 0.138 (Rancho Libertad). Nueve de las 24 subpoblaciones analizadas (IN-MOC, ITC, UV, CEIEGT, IN-MAR, Rancho El Paraíso, CEPIPSA, UCOL y Cuba) mostraron valores negativos de FIS, las 15 subpoblaciones restantes tuvieron valores positivos. Seis subpoblaciones mostraron (Cuadro 2) un FIS diferente de cero (P<0.05) con valores positivos (Rancho Garrido, Rancho Libertad y Rancho El Carrizal) y negativos (ITC, IN-MAR y CEPIPSA).
El análisis de alelos (Cuadro 3; final del escrito) reveló la presencia de nueve alelos privados (9.09% de los 99 alelos) distribuidos en siete subpoblaciones (29.1 % de los 24 rebaños). La subpoblación Posta El Cuatro mostró tres alelos privados y las subpoblaciones CEIEGT, COLPOS-CORD, Rancho San Alberto, Rancho Belbesah, Rancho Libertad y Rancho Jalapa un alelo privado. Los loci que contribuyeron con alelos privados fueron OarFCB304 (2), OarJMP29 (2), SRCRSP5 (2), MAF33 (2) y SRCRSP9 (1). Las frecuencias de alelos privados variaron desde 0.083 en la subpoblación Posta El Cuatro (iniciadores OarFCB304 y OarJMP29) hasta 0.400 en la subpoblación CEIEGT (iniciador SRCRSP9). El rebaño Pelibuey cubano no mostró alelos privados.
Cuadro 3 Alelos privados y alelos compartidos en 24 subpoblaciones de oveja Pelibuey caracterizadas genéticamente con nueve marcadores microsatélites
| OarCP34 | OarFCB304 | OarJMP29 | OarJMP58 | DYMS1 | ILSTS5 | SRCRSP5 | SRCRSP9 | MAF33 | |
|---|---|---|---|---|---|---|---|---|---|
| Alelos privados | |||||||||
| Número AP (% de SP) | 0 (0) | 2 (4.33) | 2 (8.33) | 0 (0) | 0 (0) | 0 (0) | 2 (8.33) | 1 (4.17) | 2 (8.33) |
| SP con AP Alelos (frecuencia) | ― | Posta El Cuatro 140 (0.083) 210 (0.083) |
Rancho Libertad 170 (0.100) Posta El C uatro 180 (0.083) |
― | ― | ― | COLPOS- CORD 172 (0.100) Rancho Jalapa 200 (0.125) |
CEIEGT 95 (0.400) |
San Alberto 170 (0.100) Rancho Belbesah 160 (0.100) |
|
Alelos compartidos | |||||||||
| Alelo más compartido Número de SP (%) | 110 23 (95.83) |
164 15 (62.5) |
150 23 (95.83) |
135 16 (67.5) |
175/200 16/16 (66.7/66.7) |
225 19 (79.17) |
152 17 (70.83) |
125 14 (58.3) |
120 18 (75) |
| Alelo menos compartido Número de SP (%) | 100 2 (8.33) |
150 7 (29.17) |
110/140 3/3 (12.5/12.5) |
177/185 2/2 (8.33/8.33) | 225 (4) 16.67 |
180 3 (12.5) |
154 2 (8.33) |
135 5 (20.8) |
155 5 (20.83) |
| Media de SP con AC (%) | 11
(45.8) |
10.3
(42.8) |
9
(37.5) |
7.36
(30.7) |
10.91
(45.45) |
9.89
(44.79) |
7.42
(33.3) |
9.2
(38.3) |
8.18
(34.1) |
AP= alelos privados; SP= subpoblación, AC= alelos compartidos.
A pesar que no se detectaron alelos compartidos por todas las subpoblaciones, se identificaron dos alelos (“110” y “150” de los loci OarCP34 y OarJMP29, respectivamente) que fueron compartidos por 23 subpoblaciones (95.83 %). El análisis de flujo génico (medido a través del Nm) puede apreciarse en el Cuadro 4 (final del escrito). El valor de Nm más alto se observó entre las subpoblaciones CEIEGT y CEPIPSA. De las 276 comparaciones de subpoblaciones posibles, 222 (80.43 %) mostraron un Nm mayor a 1; mientras que en 14 (5.07 %) el Nm fue mayor a 4.
Cuadro 4 Flujo génico medido a través del número de migrantes (Nm) entre pares de subpoblaciones de ovejas Pelibuey en México
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 | 17 | 18 | 19 | 20 | 21 | 22 | 23 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | ― | ||||||||||||||||||||||
| 2 | 3.13 | ― | |||||||||||||||||||||
| 3 | 2.56 | 1.73 | ― | ||||||||||||||||||||
| 4 | 2.62 | 1.83 | 16.39 | ― | |||||||||||||||||||
| 5 | 1.66 | 2.34 | 1.29 | 2.25 | ― | ||||||||||||||||||
| 6 | 2.15 | 1.42 | 1.48 | 1.62 | 2.10 | ― | |||||||||||||||||
| 7 | 0.94 | 1.05 | 0.87 | 0.92 | 1.74 | 5.40 | ― | ||||||||||||||||
| 8 | 0.81 | 0.77 | 0.82 | 1.21 | 1.49 | 1.40 | 1.41 | ― | |||||||||||||||
| 9 | 0.79 | 0.80 | 0.96 | 1.13 | 1.04 | 2.23 | 1.90 | 1.35 | ― | ||||||||||||||
| 10 | 1.12 | 1.07 | 1.18 | 1.22 | 1.21 | 4.17 | 2.08 | 1.07 | 2.20 | ― | |||||||||||||
| 11 | 0.65 | 0.93 | 0.71 | 0.78 | 1.39 | 2.56 | 2.41 | 0.80 | 1.26 | 1.55 | ― | ||||||||||||
| 12 | 1.17 | 1.09 | 1.29 | 2.13 | 1.11 | 2.39 | 1.16 | 0.81 | 1.17 | 1.91 | 1.00 | ― | |||||||||||
| 13 | 1.06 | 0.92 | 1.04 | 1.19 | 1.05 | 2.19 | 1.36 | 0.90 | 1.37 | 2.64 | 1.45 | 1.92 | ― | ||||||||||
| 14 | 1.16 | 1.13 | 1.32 | 1.36 | 1.26 | 3.05 | 1.68 | 1.04 | 1.31 | 2.14 | 1.45 | 1.91 | 3.40 | ― | |||||||||
| 15 | 1.18 | 1.36 | 1.56 | 1.93 | 1.40 | 2.40 | 2.24 | 1.31 | 2.20 | 6.43 | 1.37 | 4.34 | 3.91 | 5.53 | ― | ||||||||
| 16 | 1.04 | 1.09 | 1.98 | 1.79 | 1.07 | 1.73 | 1.17 | 0.82 | 1.08 | 1.49 | 0.84 | 1.87 | 1.90 | 1.92 | 5.41 | ― | |||||||
| 17 | 0.82 | 0.73 | 0.93 | 1.05 | 0.94 | 8.18 | 1.87 | 0.84 | 1.99 | 2.41 | 3.81 | 1.52 | 5.12 | 3.14 | 1.81 | 1.18 | ― | ||||||
| 18 | 0.59 | 0.98 | 0.73 | 0.80 | 1.37 | 1.25 | 1.53 | 0.73 | 0.98 | 1.30 | 1.28 | 0.86 | 1.29 | 1.16 | 1.34 | 0.81 | 1.12 | ― | |||||
| 19 | 0.90 | 0.91 | 1.16 | 1.29 | 0.85 | 1.54 | 0.93 | 0.70 | 1.31 | 1.53 | 0.93 | 2.53 | 2.08 | 1.80 | 2.50 | 2.38 | 1.13 | 0.75 | ― | ||||
| 20 | 0.91 | 0.95 | 1.18 | 1.14 | 1.18 | 1.78 | 1.31 | 1.07 | 2.38 | 1.49 | 1.07 | 1.47 | 2.25 | 1.81 | 2.21 | 1.78 | 1.71 | 0.86 | 2.12 | ― | |||
| 21 | 1.54 | 1.19 | 1.43 | 1.99 | 1.55 | 7.73 | 1.77 | 1.08 | 1.28 | 1.73 | 0.97 | 2.25 | 1.69 | 3.15 | 2.89 | 2.47 | 1.41 | 0.96 | 1.78 | 1.65 | ― | ||
| 22 | 1.13 | 1.06 | 1.28 | 1.66 | 1.53 | 2.59 | 1.48 | 1.87 | 1.77 | 3.37 | 1.34 | 1.53 | 7.85 | 3.07 | 9.46 | 2.05 | 2.14 | 1.05 | 1.42 | 2.73 | 2.52 | ― | |
| 23 | 0.80 | 0.87 | 0.86 | 0.87 | 0.97 | 2.81 | 2.01 | 0.99 | 2.68 | 3.79 | 1.32 | 1.62 | 4.54 | 2.36 | 5.72 | 1.93 | 1.93 | 0.99 | 2.30 | 1.77 | 1.44 | 2.41 | ― |
| 24 | 1.33 | 1.12 | 1.06 | 1.29 | 1.17 | 2.35 | 1.14 | 0.89 | 1.25 | 2.55 | 0.90 | 1.56 | 2.51 | 1.86 | 2.75 | 1.62 | 1.26 | 0.96 | 1.75 | 1.37 | 2.18 | 1.94 | 1.55 |
* La numeración de las subpoblaciones sigue el mismo orden que el Cuadro 2.
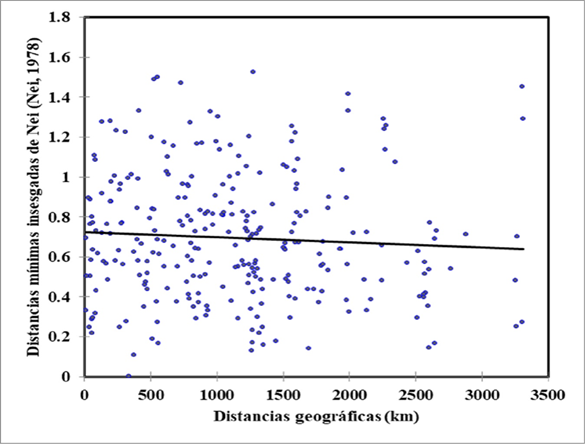
Para tratar de comprobar el aislamiento por distancia en las subpoblaciones, se llevó a cabo la prueba de Mantel (Figura 2). El resultado de este análisis mostró que, la diferenciación genética entre las subpoblaciones de oveja Pelibuey (medida mediante las distancias mínimas insesgadas de Nei) no tiene relación con las distancias geográficas entre las mismas (r= -0.062; P>0.05).
Relaciones genéticas entre subpoblaciones y análisis de estructura poblacional
Se realizó un PCA utilizando las frecuencias de los 99 alelos observados en la población. El PCA global se muestra en la Figura 3. Los primeros dos componentes principales explicaron el 30.21 % de la variación total. El primer eje contribuyó con el 17.04 % de la varianza y separa a los rebaños en dos clústeres, uno de ellos con las subpoblaciones IN-MOC, IN-MAR, CEIEGT, CEPIPSA e ITC. El segundo eje contribuyó con el 13.17 % de la varianza y separó a las subpoblaciones UCOL, COLPOS-CORD, Rancho el Paraíso, Universidad Veracruzana, ITC e IN-MAR.
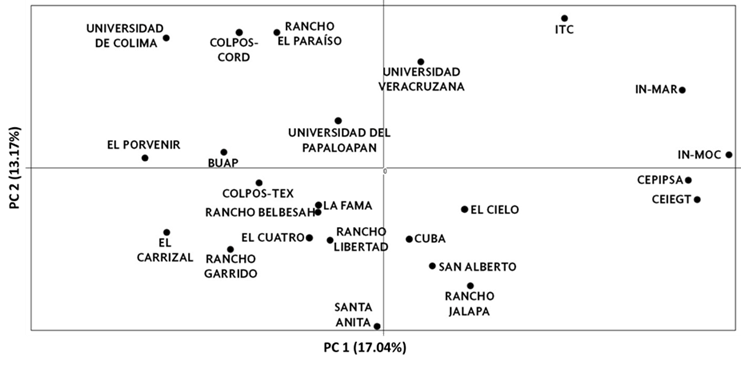
Figura 3 Análisis de componentes principales entre subpoblaciones de oveja Pelibuey de México. Se incluyeron los dos primeros componentes
El análisis con el programa STRUCTURE reveló la presencia de dos poblaciones ancestrales (K), lo cual es consistente con el PCA. El primer grupo estuvo constituido por las subpoblaciones IN-MOC, IN-MAR, CEIEGT, CEPIPSA, e ITC (Figura 4). Si hipotéticamente se considerara una K=3 o K=4, se puede observar la formación de nuevos clústeres, pero las subpoblaciones del primer grupo permanecen constantes.
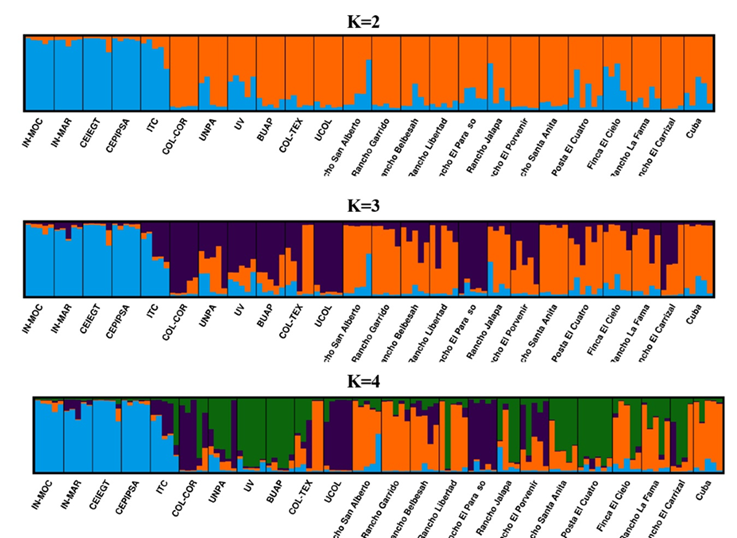
Cada individuo está representado por una barra vertical, la cual generalmente se segmenta en varios colores. Los colores representan las proporciones de las poblaciones ancestrales (K=2, K=3, K=4) que componen el genoma individual. Las subpoblaciones están separadas por líneas negras.
Figura 4 Agrupamiento mediante el programa STRUCTURE de diferentes subpoblaciones de ovejas Pelibuey de México
Discusión
Diversidad genética de la población entera
Este es el primer estudio sobre la caracterización de la oveja Pelibuey usando microsatélites. El promedio del NA fue de 11, un valor más bajo que los 14.27 alelos reportados por en un estudio que se realizó en 13 razas de ovejas colombianas usando 11 marcadores microsatélites28, pero es muy similar al 10.96 reportado en nueve razas españolas, cubanas, mexicanas y africanas usando 26 marcadores microsatélites29. El promedio del NE fue 3.25, el cual es más bajo que el 3.73 encontrado en cinco razas de ovejas chinas30 y el 4.68 observado en razas de ovejas colombianas28. Cuando se contemplaron todos los loci, el NA fue más alto que el NE, lo cual indica que los alelos se distribuyen de manera irregular en cada uno de los loci. Lo anterior puede ser causado por el aislamiento geográfico, selección o flujo de material genético entre subpoblaciones31.
La heterocigosis es un parámetro que refleja la diversidad genética en la población. Los valores promedio de HO y HE (0.726 y 0.732, respectivamente) fueron muy similares en todos los loci. Los valores concuerdan con lo observado en varias razas de ovejas de España, Cuba, México y África, en las que se registró un valor promedio de HE de 0.73129. En otro estudio, se encontraron valores de HO y HE de 0.744 y 0.755, respectivamente, en varias razas de ovejas de Hungría32. En cuatro de los nueve marcadores analizados (OarFCB304, ILSTS5, SRCRSP5 y MAF33) la HO fue más alta que la HE, sugiriendo un exceso de heterocigotos en las subpoblaciones analizadas.
Los valores de PIC obtenidos en este estudio fueron superiores a 0.7, lo cual indica que los marcadores utilizados son apropiados para medir la diversidad genética. El valor promedio del índice de Shannon (2.1) fue similar al valor de 2.2 obtenido en 14 tipos de ovejas de Irán33 y 2.38 reportado en 4 razas de ovejas nigerianas34. Este resultado refleja la alta variabilidad genética de las subpoblaciones analizadas.
Tres de los nueve marcadores mostraron una desviación del HWE: dos debido a una alta heterocigosis (OarFCB304 e ILSTS5) y uno (OarJMP29) debido a baja heterocigosis. Aunque la desviación del HWE puede tener implicaciones biológicas como la selección intensiva, endogamia, migración, mutación o un insuficiente número de muestras35, también es probable que se deba a errores en el genotipificado36. En el presente estudio, únicamente se observó desviación del HWE en 20 de las 216 pruebas exactas de Fisher, por lo que se decidió mantener todos los marcadores en los análisis subsecuentes.
El valor de FIT observado (0.158) indica que hay una deficiencia global de individuos heterocigotos del 15.8 % en la población. Asimismo, a partir del valor de FST (0.151), se deduce que el 15.1 % de la variación genética corresponde a diferencias entre subpoblaciones. Valores similares en ambos índices fueron encontrados en varias razas de ovejas, en donde se observaron valores de FIT y FST de 14.2 y 13.4 %, respectivamente29. Por lo tanto, ambos valores indican que existe una diferenciación genética moderada entre los rebaños estudiados.
Diversidad genética entre subpoblaciones
Los valores más altos de NA, MNA y NE correspondieron a las subpoblaciones UNPA, COL-TEX, Rancho Libertad y Posta El Cuatro. El alto grado de diversidad genética en esas subpoblaciones se puede atribuir a la introducción de nuevos individuos en las mismas, lo cual fue corroborado en los libros de registros de los rebaños. Asimismo, la Posta El Cuatro corresponde a una unidad destinada a la producción de ejemplares de registro de la raza Pelibuey. En los últimos años, los criadores de Pelibuey puro han logrado mejorar sustancialmente el desempeño productivo de los animales de la raza, recurriendo a los cruzamientos entre diferentes líneas de la misma. En el caso del Rancho Libertad, los altos valores de FIS (0.138) no apoyan lo anteriormente expuesto. Sin embargo, este resultado puede atribuirse al reducido tamaño de muestra de la subpoblación. Los valores más bajos de NA, MNA y NE fueron observados en las subpoblaciones IN-MOC, IN-MAR, CEIEGT y CEPIPSA, lo cual podría deberse a que esas subpoblaciones pertenecen a institutos de investigación y universidades, los cuales se han conservado como núcleos cerrados que no han permitido la introducción de nuevos animales. Por lo tanto, es de esperarse que ellos tengan una mejor conservación del material genético original de la raza. Los valores negativos de FIS confirman que en esos rebaños se sigue una política de apareamientos de mínima consanguinidad.
Los valores de HO (0.726) y HE (0.731) obtenidos en este estudio, fueron más altos que los encontrados en un rebaño de Pelibuey en Querétaro, México, en el que se observaron valores de 0.652 y 0.659 para HO y HE, respectivamente29. Asimismo, en un rebaño de Pelibuey colombiana se estimaron valores para HO y HE de 0.72 y 0.71, respectivamente28. Los altos valores globales obtenidos de HO y HE podrían atribuirse a que se analizaron varias subpoblaciones, en contraste con los otros estudios en los cuales sólo un rebaño estuvo involucrado.
El valor total de FIS fue de 0.0072, sin embargo, en nueve subpoblaciones (IN-MOC, UV, Rancho El Paraíso, IN-MAR, ITC, UCOL, CEIEGT, CEPIPSA y Cuba) el valor de FIS fue negativo, sugiriendo un exceso de heterocigotos. En las subpoblaciones restantes, los valores de FIS fueron positivos, lo cual significa que tuvieron una deficiencia de heterocigotos. Lo anterior concuerda con los valores de heterocigosis, ya que, en las mismas subpoblaciones, los valores de HO fueron más altos que los de HE. Estudios previos en ovejas Pelibuey reportaron valores de FIS de 0.02329 y 0.0228, indicando una deficiencia ligera de heterocigotos. La presencia en este estudio de rebaños con valores negativos de FIS es explicada por la ausencia de endogamia, valores de heterocigosis altos y baja presión de selección, como ha sido señalado en otros estudios33. Los altos valores de FIS en el Rancho Garrido, Rancho Libertad y Rancho El Carrizal indican deficiencia de heterocigotos, lo cual es comúnmente atribuido a selección y endogamia37. En las poblaciones de animales domésticos, la endogamia es un hallazgo común debido a las fallas en los programas reproductivos y que se trata de poblaciones relativamente pequeñas. Asimismo, la cantidad tan pequeña de individuos muestreados por subpoblación también pudo contribuir a tal estimación en esas subpoblaciones.
Los alelos privados se definen como aquellos que se presentan en una sola población (en el presente estudio, subpoblación). Nueve alelos privados fueron detectados en seis de las subpoblaciones estudiadas. La subpoblación con mayor número de alelos privados fue Posta El Cuatro, con 3. Las subpoblaciones CEIEGT, COLPOS-CORD, Rancho San Alberto, Rancho Belbesah, Rancho Libertad y Rancho Jalapa mostraron un alelo privado. A excepción del alelo “95” (subpoblación CEIEGT, locus SRCRSP9) que tuvo una frecuencia de 0.400, los otros alelos privados mostraron frecuencias relativamente bajas (0.083-0.125), lo que indica que tienen más riesgo de desaparecer si esto no se toma en cuenta en los programas reproductivos. Los alelos privados con frecuencias más altas indican que una población es única y tiene cierto grado de aislamiento38, lo cual es compatible con la historia reciente del rebaño CEIEGT, que se ha comportado como un núcleo cerrado, evitando la entrada de material genético nuevo. Además, el valor negativo de FIS (-0.064) descarta la posibilidad que la alta frecuencia del citado alelo se deba a un proceso de endogamia.
La presencia de alelos privados con bajas frecuencias dentro de las subpoblaciones analizadas pudiera deberse a eventos de mutación relativamente recientes39. Los microsatélites tienen tasas de mutación altas que van de 10-6 a 1x10-2(40, lo que hace posible la aparición eventual de una mutación en el rango de 1/1,000,000-1/100 individuos/generación. Además, los alelos privados también pueden deberse al alto flujo genético entre subpoblaciones41. Lo anterior, es apoyado por el valor global de FST (0.151) observado en el presente trabajo, que indica la presencia de flujo génico moderado entre las subpoblaciones.
El análisis de alelos privados no permitió diferenciar la subpoblación cubana de los rebaños nacionales. Al respecto, se debe considerar que al introducir la raza Pelibuey a México, se estableció un efecto fundador en el que únicamente se seleccionaron unos cuantos ejemplares de la población total de la raza que en ese momento existía en Cuba. En los ejemplares de Pelibuey introducidos a México, la deriva génica jugó un papel muy importante en la extinción o fijación de los alelos disponibles. Asimismo, las mutaciones pudieron contribuir de manera importante a la aparición de nuevos alelos dentro del rebaño recién formado. Por lo tanto, cabría esperar una diferencia sustancial entre la diversidad genética de los rebaños mexicanos y el rebaño cubano. Sin embargo, en el presente trabajo se observó que el rebaño cubano comparte sus alelos con los rebaños mexicanos. Lo anterior puede atribuirse a que la población cubana analizada estuvo constituida únicamente por cinco individuos, constituyendo una muestra mínima de la diversidad genética actual. Por lo tanto, en el presente trabajo, no se pudo establecer una comparación objetiva entre ambas subpoblaciones y los resultados únicamente deben ser considerados como indicativos.
Al analizar los alelos compartidos por las subpoblaciones, no se logró identificar alelos con presencia en todas ellas. Sin embargo, los alelos “110” y “150”, correspondientes a los loci OarCP34 y OarJMP29 se encontraron en 23 de las 24 subpoblaciones analizadas, estando ausentes únicamente en las subpoblaciones UV y Rancho Belbesah, respectivamente. Por lo tanto, en estudios posteriores, se recomienda enfocar la atención en dichos alelos con el fin de determinar si pueden ser utilizados como marcadores de la raza Pelibuey.
El análisis de flujo génico medido a través del Nm mostró que las subpoblaciones con mayor flujo génico entre ellas son CEIEGT y CEPIPSA (Nm= 16.39), lo cual concuerda con el libro de registros de tales rebaños, ya que pertenecen a la Universidad Nacional Autónoma de México (UNAM) y desde hace algunos años de manera regular, en el CEPIPSA se han introducido sementales provenientes del CEIEGT. Asimismo, el análisis del Nm confirmó que en el 80.43 % de los pares de subpoblaciones, el valor obtenido fue superior a uno, mientras que el 5.07 % de los pares de subpoblaciones, alcanzó valores superiores a cuatro. Los valores de Nm menores a uno, indican que el flujo génico no es suficiente para contrarrestar la diferenciación causada por la deriva génica entre las subpoblaciones, por lo que éstas tienden a diferenciarse, mientras que valores superiores a uno impiden la diferenciación de las mismas. En cambio, si el Nm es mayor a 4, las subpoblaciones se comportan como una población panmíctica42. Lo anterior es apoyado por el valor global de FST (0.151), el cual indica un flujo génico moderado entre las subpoblaciones.
La correlación entre las distancias genéticas y geográficas no resultó significativa, lo que permite deducir que la estructura genética de los rebaños de Pelibuey analizados no se ajusta a un modelo de aislamiento por distancia entre subpoblaciones. Lo anterior puede atribuirse al flujo de ejemplares de la raza Pelibuey a lo largo de todo el territorio de México.
Relaciones genéticas entre subpoblaciones y análisis de estructura poblacional
El PCA mostró que el primer componente separó a las subpoblaciones IN-MOC, IN-MAR, CEIEGT, CEPIPSA e ITC, del resto. Este resultado se esperaba ya que esas subpoblaciones pertenecen a institutos de investigación y universidades, donde el objetivo primario es la investigación y la conservación de la raza. Lo anterior fue comprobado previamente al mostrar valores de FIS negativos, que indican alta heterocigosis. Por el contrario, el segundo grupo estuvo compuesto por rebaños que pertenecen a otras universidades e institutos de investigación (BUAP, COL-TEX, COL-COR, UCOL, y UNPA), todos los productores particulares y el rebaño de Cuba. La divergencia del segundo grupo probablemente se deba al flujo de individuos que actualmente ocurre entre las subpoblaciones de Pelibuey en México, lo cual es apoyado por el valor de FST moderado (0.151) y por la gran proporción de pares de subpoblaciones que tienen un Nm superior a uno. Actualmente, las biotecnologías reproductivas como la transferencia de embriones e inseminación artificial han permitido un mejoramiento genético más rápido8. Tales tecnologías y el flujo de individuos de una unidad productiva a otra (principalmente sementales) probablemente hayan causado que la diversidad genética original de la oveja Pelibuey se haya modificado sustancialmente en los últimos años. Asociado a esto, actualmente ha aumentado de manera importante la mezcla con otras razas, provocando una mayor divergencia, ante lo cual, se tendrían que realizar estudios exhaustivos para tratar de comprobar la erosión genética de la raza.
El segundo componente del PCA, separó a las subpoblaciones UCOL, COLPOS-CORD, Rancho el Paraíso, Universidad Veracruzana, ITC e IN-MAR. Estas subpoblaciones pertenecen tanto a centros de investigación y universidades como a productores particulares. Aunque es difícil explicar, este agrupamiento de subpoblaciones puede deberse a los manejos particulares a los que han sido sometidos los rebaños en años recientes. Por ejemplo, los rebaños UCOL e IN-MAR han reducido dramáticamente su población, en cambio, Rancho El Paraíso ha comenzado a introducir material genético nuevo.
El análisis con el programa STRUCTURE sugiere que todas las subpoblaciones de Pelibuey en México se originaron a partir de dos poblaciones ancestrales, las cuales han divergido como resultado de varios años de adaptación a diferentes ambientes y manejos a los cuales han sido sujetas. Asimismo, cada una de las subpoblaciones muestra una mezcla en mayor o menor grado de ambos clústeres. Las subpoblaciones IN-MOC, IN-MAR, CEIEGT, CEPIPSA e ITC, se agruparon en un clúster, al igual que en el PCA (primer componente). La relación entre las subpoblaciones nacionales de referencia y los otros rebaños se confirmó mediante el análisis de alelos compartidos mencionado previamente. Asimismo, el análisis con STRUCTURE muestra bajos niveles de mezcla entre las subpoblaciones de este clúster con las del otro clúster. También, se confirma que en esas subpoblaciones el objetivo primario ha sido la investigación y la conservación de la raza. Si hipotéticamente se considerara una K=3 o K=4, es posible observar que las subpoblaciones IN-MOC, IN-MAR, CEIEGT, CEPIPSA e ITC se siguen agrupando juntas. Esto permite concluir que esas subpoblaciones tienen el material genético más preservado.
Conclusiones e implicaciones
Las subpoblaciones de oveja Pelibuey aquí estudiadas muestran alta diversidad genética y diferenciación genética entre ellas. El análisis de componentes principales, así como el de estructura de población permiten concluir que las subpoblaciones IN-MOC, IN-MAR, CEIEGT, CEPIPSA e ITC tienen el material genético más preservado. Este resultado hace posible recomendar el uso de animales de esas subpoblaciones para implementar programas nacionales para la conservación de la raza Pelibuey. Durante la realización del estudio, se observó que a pesar del gran valor de la oveja Pelibuey en el desarrollo de la ovinocultura y en la investigación, existen rebaños como IN-MAR y UCOL en los cuales se ha reducido el número de individuos, por lo que están en peligro de desaparecer. Actualmente existen grupos de investigadores y organizaciones en México como la Unidad Nacional de Ovinocultores, los cuales han reconocido la importancia que la oveja Pelibuey representa en la ovinocultura nacional, y mediante esfuerzos individuales han tratado de contribuir a su conservación. Se requiere de un plan integral para la conservación de la raza, que garantice la preservación de su material genético. Asimismo, se recomienda realizar estudios más profundos para determinar si realmente existe erosión genética de la oveja Pelibuey en México.











 texto en
texto en