Introducción
La diversidad genética es el componente fundamental detrás de la diversidad biológica, por lo que su estudio provee importantes pis tas acerca de los mecanismos que moldean la variación genética, sus patrones, niveles e historia evolutiva de un taxón (Conklin et al. 2014; Guillemin et al. 2014; Muangmai et al. 2015; Núñez-Resendiz et al. 2016). En estudios microevolutivos de índole filogeográfica y de genéti ca de poblaciones se han empleado distintos marcadores moleculares para la evaluación de la diversidad genética de especies con poca o alta plasticidad fenotípica (Graur & Li 2000; West-Eberhard 2003). Tales marcadores moleculares incluyen regiones mitocondriales como COI (Yang et al. 2008, 2015; Yow et al. 2011, 2013; Muangmai et al. 2015), la región espaciadora cox2-3 (Destombe et al. 2010; Pareek et al. 2010; García-Rodríguez et al. 2013; Núñez-Resendiz et al. 2015); marcadores cloroplastidiales como la región espaciadora de la RuBisCo (Guillemin et al. 2008; Núñez-Resendiz et al. 2015, 2016; Zuccarello et al. 2005); o marcadores nucleares como la región ITS (Goff et al. 1994). El empleo de estos marcadores, en combinación con estudios de morfología, ha proporcionado evidencias sólidas para la resolución de conflictos de identificación errónea de especies, el hallazgo de nuevas especies o la solución de complejos de especies crípticas (Muangmai et al. 2015, Núñez-Resendiz et al. 2016, 2017).
Por la diversidad de hábitats que contiene, la península de Yucatán representa una región propicia para el establecimiento de diferentes comunidades marinas, tales como arrecifes coralinos, pastos marinos, manglares y macroalgas bentónicas (Dreckmann & Sentíes 2013). Dentro de las comunidades algales destaca la división Rhodophyta, de la cual Solieriaceae es una de las familias más conspicuas y económicamente importantes (Freile-Pelegrin & Robledo 2006, 2008; Freile-Pelegrin et al. 2006a).
Meristotheca cylindrica M. L. Núñez-Resendiz, Dreckmann & Sentíes es un alga roja de la familia Solieriaceae, recientemente descrita a partir de especímenes recolectados a lo largo de la costa de Campeche (Núñez-Resendiz et al., 2017). Se diferencia del resto de las especies del género por poseer un talo cilíndrico, el cual se conforma por múltiples ejes principales, cartilaginosos, de 5-30 cm de longitud y 1-5 mm de grosor. Se han descrito talos altamente ramificados (4, 5 o más veces) así como poco ramificados según lo registrado por Núñez-Resendiz et al. (2017). Al igual que otras especies del género, posee tetrasporangios elipsoidales y zonados que se desarrollan en las capas corticales, y cistocarpos elipsoidales inmersos formados en proliferaciones marginales (Faye et al. 2004, 2005, 2007, 2008). Anatómicamente posee una médula filamentosa y una corteza pesudoparenquimatosa, con células de 50-60 µm de longitud y 18-25 µm en diámetro (Núñez-Resendiz et al. 2017). Como miembro de Solieriaceae, familia que posee el ma yor intervalo de diversidad estructural de carragenanos (Chiovitti et al. 2001; Watt et al. 2003), y dada su abundancia en las costas del estado de Campeche, M. cylindrica constituye un recurso económico potencialmente redituable que no se explota en México (Núñez-Resendiz et al. 2017).
Debido a la particularidad del talo cilíndrico de M. cylindrica, en la península de Yucatán esta especie se registró previamente como Eucheuma isiforme (C. Agardh) J. Agardh (Callejas et al. 2005), otra especie cilíndrica de Solieriaceae fenotípicamente plástica con quien comparte su intervalo de distribución (Núñez-Resendiz et al. 2017, Zuccarello et al. 2006).
Con base en lo anterior, se establecieron cuatro objetivos en el pre sente estudio: 1) determinar la variación genética y morfológica dentro y entre las poblaciones de M. cylindrica; 2) determinar la estructura genética; 3) determinar la distribución haplotípica en las localidades muestreadas en Campeche, y finalmente 4) evaluar si ocurre un evento de especiación entre las poblaciones de M. cylindrica. Se considera relevante la realización del presente estudio por la importancia econó mica y el espectro morfológico presente en M. cylindrica que la hace confundible con otras especies de Solieriaceae.
Material y métodos
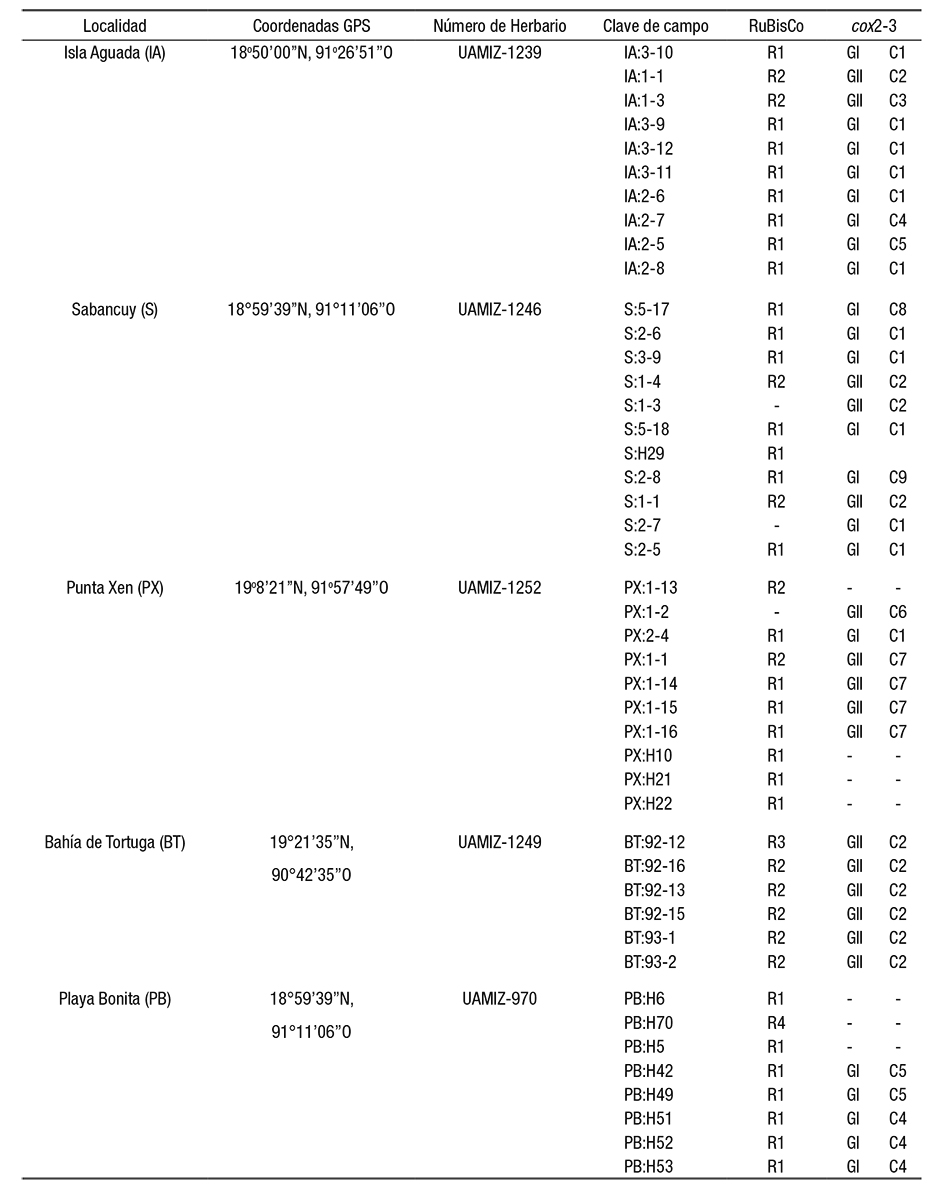
Se recolectaron 45 individuos de Meristotheca cylindrica pertenecientes a cinco poblaciones, en cinco localidades del litoral de Campeche, en la península de Yucatán (Tabla 1), a una profundidad de entre 1 y 1.5 m. Bajo el microscopio estereoscópico, se removieron los epibiontes de cada individuo y un fragmento de las porciones apicales de las ramas (~3 cm) se preservó en sílica gel hasta la posterior extracción de DNA. El resto fue montado en papel de herbario, numerado e integrado a la colección de algas del Herbario Metropolitano (UAMIZ). Todas las muestras fueron designadas con un número de campo y se añadieron a una hoja de herbario con su respectivo número de voucher (Tabla 1).
Tabla 1 Muestras y especímenes de Meristotheca cylindrica procedentes de las costas de Campeche. Se describen los datos de localidad, coordenadas geográficas y números de campo y herbario (UAMIZ= Herbario de la Universidad Autónoma Metropolitana). Se indica el haplotipo al que pertenecen las muestras para ambas regiones genéticas; para el caso de la región espaciadora cox2-3, GI y GII corresponden con la estructura genética revelada por este marcador (ver texto).
La extracción de DNA se realizó a partir de 5-10 mg de tejido seco con la utilización del kit de extracción de Qiagen, DNeasy Plant Mini Kit (Qiagen, Valencia, California USA) según la técnica descrita por el fabricante, con modificaciones menores al añadir una centrifugación por 1 minuto a 8000 rpm después de agregar por segunda vez el buffer AW2, y un aumento en el tiempo de incubación con buffer AE de 5 a 30 minutos.
La amplificación vía PCR se realizó con el kit Taq PCR Core Kit (Qiagen) y cebadores específicos según la región de cada genoma por amplificar. El marcador molecular cloroplástico, la región espaciadora de la RuBisCo (rbc spacer), se amplificó con los cebadores spacer-F y spacer-R descritos en Maggs et al. (1992). Por otro lado, el espaciador de cox2-3 se amplificó utilizando los oligonucleótidos cox2-for y cox3-rev (Zuccarello et al. 1999). El volumen total de PCR por muestra fue de 25 μL con los siguientes reactivos: 2.5 μL de buffer de PCR 10X, 1 μL de primer forward 10μM, 1 μL de primer reverse 10μM, 0.5 μL de solución de dNTPs (10mM por cada uno), 0.5 μL de BSA, 1 μL de MgCl2 25mM, 0.125 μL de Taq polimerasa 5U/μL, 17.375 μL de agua desionizada y 1 μL de DNA genómico. Las condiciones de PCR fueron las descritas por Núñez-Resendiz et al. (2015). La amplificación vía PCR se comprobó mediante una electroforesis en gel de agarosa al 0.8% y los produc tos de PCR se purificaron con el kit QIAquick Purification Kit (Qiagen). Las muestras amplificadas se enviaron a Macrogen Korea (10F, 254 Beotkkot-ro, Geumcheon-gu, Seúl, 08511, República de Corea), donde se utilizó el kit de secuenciación BigDye en un secuenciador ABI PRISM 3100 Genetic Analyzer (Applied Biosystems, Princeton, NJ, EE. UU.).
Las secuencias obtenidas se organizaron en una matriz, se edita ron y alinearon mediante el algoritmo ClustalW (Thompson et al. 1994) con el programa Bioedit (Hall 1999). La búsqueda del modelo óptimo de evolución de DNA se implementó mediante jModelTest v. 2.1.6 con el criterio de información Akaike (AIC) (Darriba et al. 2012, Guindon & Gascuel 2003). El modelo elegido para ambos conjuntos de secuencias fue el GTR G+I (general time reversible + distribución gamma + sitios invariables), el cual se utilizó en los análisis filogenéticos posteriores.
Las siguientes medidas de diversidad genética se estimaron con el programa DnaSP v.5 (Librado & Rozas, 2009): número de sitios segregantes (S), número de haplotipos (H), diversidad haplotípica (Hd), diversidad nucleotídica (π) y sitios segregantes por cada marcador molecu lar, así como los estimados de flujo génico (Nm) e índice de fijación (FST) según Hudson, Slatkin & Maddison (1992). Con la finalidad de observar la diferenciación genética, se realizó un análisis de varianza molecu lar (AMOVA) para cada marcador mediante GenAlEx v.6.503 (Peakall & Smouse 2006, 2012); de igual manera, se obtuvieron los valores de ΦPT según Peakall et al. (1995), análogos de FST.
La evaluación de las relaciones haplotípicas se estimó por medio de la construcción de redes de parsimonia estadística con TCS v.1.21 (Clement et al. 2000). Una secuencia representativa de cada haplotipo, por ambos marcadores moleculares, fue depositada en la base de da tos del GenBank.
Los análisis filogenéticos de inferencia bayesiana (IB) y máxima verosimilitud (ML) se realizaron con el programa TOPALi v. 2.5 (Milne et al. 2004): la inferencia bayesiana mediante Mr. Bayes (Huelsenbeck & Ronquist 2001), con una frecuencia de muestreo de cada 100 ge neraciones durante 1x106 generaciones y burn-in del 25%. Asimismo, la máxima verosimilitud se determinó vía RaxML (Stamatakis 2014); se utilizó un bootstrap con 1000 réplicas para el cálculo de soporte de ramas. El grupo externo seleccionado para ambos marcadores fue Hydropuntia cornea Montagne. Se estimaron distancias genéticas no corregidas (p-distances) en MEGA v. 6 (Tamura et al. 2013).
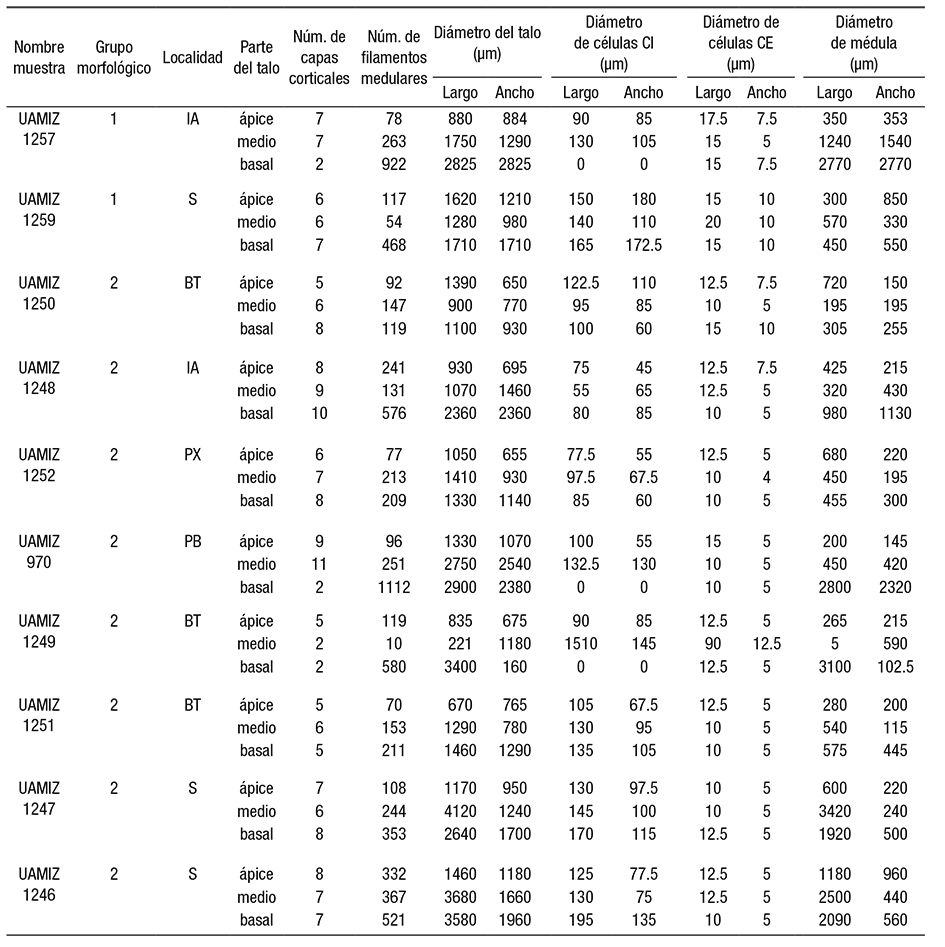
El análisis morfológico se realizó a partir de caracteres anatómicos apreciados en 10 ejemplares de herbario pertenecientes a las poblaciones trabajadas molecularmente. Para ello, se realizaron manual mente cortes transversales observados con la ayuda del microscopio óptico Leica DMLB. Las muestras se dividieron en dos grupos según la ramificación presentada: 1) patrón de ramificación sólo alterna, poco ramificado (2 o 3 veces); 2) patrón de ramificación alterna-opuesta, altamente ramificado (x≥4). Se construyó una matriz con la cuantificación de los siguientes caracteres: número de capas corticales, número de filamentos medulares, diámetro de la médula (largo y ancho), diámetro del talo (largo y ancho) y diámetro de las células de ambas cortezas (interna y externa, largo y ancho de cada una) en tres partes del talo (apical, media y basal), lo que constituye un total de 30 caracteres. Dicha matriz se sometió a un análisis múltiple de varianza en STATIS TICA 8.0.360 (StatSoft Inc. 2007) con el fin de determinar diferencias significativas entre los grupos independientes (1 y 2) a partir de las variables dependientes consideradas (cuantificación de los caracteres en las tres partes del talo).
Resultados
Región espaciadora de RuBisCo. El set de datos consistió en 42 secuencias con 185 pares de bases (pb) de longitud para el análisis poblacional; el análisis filogenético incluyó muestras de especies de la familia Solieriaceae obtenidas de GenBank, con Hydropuntia cornea como grupo externo, por lo que el conjunto de datos abarcó 53 secuencias en total.
Se calcularon las medidas de diversidad y diferenciación genética para este marcador (Tablas 2 y 3), las cuales mostraron en general siete sitios segregantes (Tabla 4), una moderada diversidad haplotípica (Hd= 0.46458) y una diversidad nucleotídica (π = 0.00785). Las poblaciones con la mayor diversidad haplotípica fueron Sabancuy y Punta Xen, ambas con la misma diversidad haplotípica (Hd=0.38889) así como diversidad nucleotídica (π= 0.006344) (Tabla 2). Por el contrario, la población con menor variación fue Playa Bonita con valores de Hd=0.25 y π=0.00272 (Tabla 2).
Tabla 2 Medidas de diversidad genética para las poblaciones de Meristotheca cylindrica, basadas en las regiones espaciadoras de RuBisCo y cox2-3.
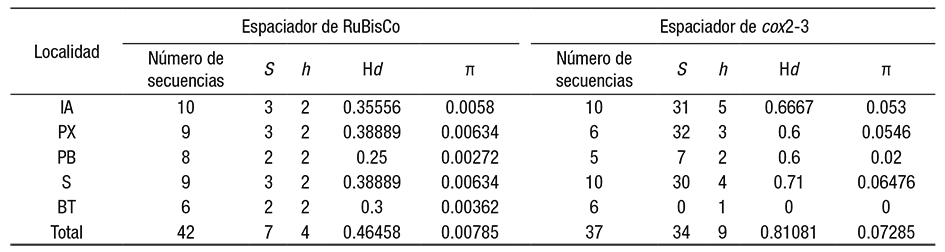
S = sitios segregantes, h = número de haplotipos, Hd = diversidad haplotípica, π = diversidad nucleotídica. IA = Isla Aguada, PX = Punta Xen, PB = Playa Bonita, S = Sabancuy, BT = Bahía de Tortuga, Total = índice general de todas las poblaciones.
Tabla 3 Análisis de varianza molecular (AMOVA) para ambos marcadores en las cinco poblaciones de Meristotheca cylindrica.
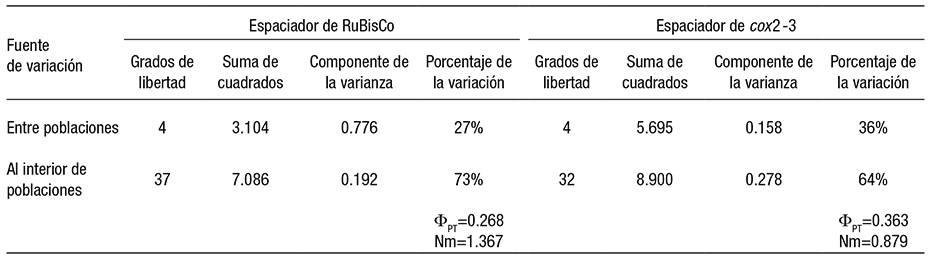
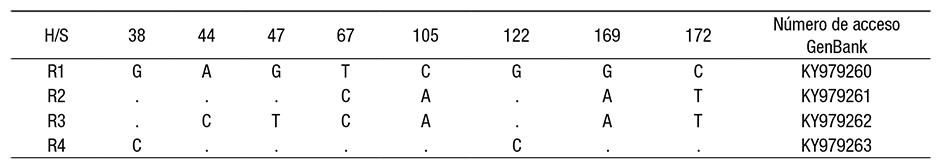
Tabla 4 Sitios variables en el alineamiento de secuencias de DNA de Meristotheca cylindrica para los haplotipos de la región espaciadora de Ru BisCo y sus números de acceso al GenBank.
En cuanto a los índices de diferenciación, se obtuvieron valores de Nm=0.55 y de FST=0.47420. El AMOVA arrojó un índice de ΦPT=0.268 y un valor de Nm=1.367, con un 27% de la varianza explicada por las diferencias del marcador cloroplástico entre las poblaciones, en tanto que un 73% de la varianza explicada por la variación al interior de las mismas (Tabla 3).
Respecto a las relaciones haplotípicas, la red de parsimonia esta dística reveló cuatro haplotipos interconectados: R1, R2, R3 y R4 (Figura 1 y Tabla 1). El haplotipo que agrupó al mayor número de individuos fue R1 (n=29), seguido por el haplotipo R2 (n=11) y finalmente los haplotipos con menor número de individuos, R3 y R4 (ambos con n=1). El haplotipo R1 (designado por el programa como el posible haplotipo ancestral) estuvo separado de R2 por cuatro pasos mutacionales y este, a su vez, estuvo separado de R3 por seis pasos mutacionales; en cuanto a R4, sólo se separó por dos pasos mutacionales (Figura 1).
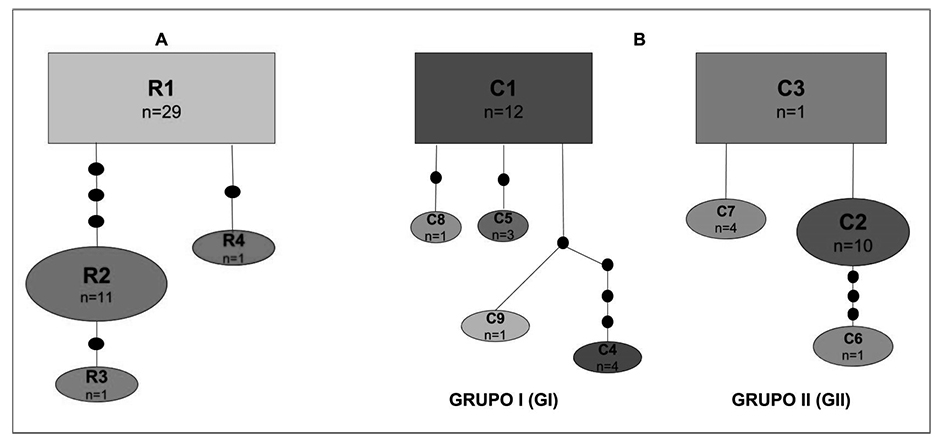
Figura 1 Redes de parsimonia estadística de las regiones espaciadoras de RuBisCo (A) y de cox2-3 (B) de Meristotheca cylindrica. Para la región espaciadora de cox2-3, los dos grupos genéticos encontrados se indican como GI y GII. Para ambas redes, los rectángulos corresponden con el posible haplotipo ancestral; las líneas sencillas indican un paso mutacional; los círculos negros indican haplotipos extintos o no muestreados; n = número de individuos con el mismo haplotipo.
Respecto de su distribución geográfica (Figura 2), el haplotipo R1 estuvo presente en todas las localidades muestreadas, R2 se presentó en todas las localidades exceptuando Playa Bonita y los haplotipos R3 y R4 se restringieron a Bahía de Tortuga y Playa Bonita, respectivamente. En todas las poblaciones se presentó el mismo número de haplotipos (2) (Figura 2).

Figura 2 Distribución haplotípica de las regiones espaciadoras de RuBisCo (derecha) y de cox2-3 (izquierda) para Meristotheca cylindrica de Campeche. Las localidades están indicadas en el mapa con número (1-5) y la clave de localidad se indica entre paréntesis (ver coordenadas GPS en Tabla 1). n= número de individuos pertenecientes a cada haplotipo por población (modificado de Nuñez-Reséndiz et al. 2017).
En cuanto al análisis filogenético, las muestras correspondientes a cuatro haplotipos de M. cylindrica se agruparon en un solo clado soportado por una probabilidad posterior bayesiana y bootstrap de 0.97/90, respectivamente. En su interior, se ubicó un grupo conformado por las muestras de los haplotipos R2 y R3, soportado por valores de 1.0/96 (probabilidad posterior bayesiana y bootstrap, respectivamente) (Figura 3).

Figura 3 Topología bayesiana obtenida con secuencias de la región espaciadora de RuBisCo para las poblaciones de Meristotheca cylindrica. Las secuencias obtenidas en el presente estudio están indicadas en las ramas de color morado y las restantes fueron obtenidas del GenBank, cuyos números de acceso se indican a un lado. Los valores de probabilidad posterior bayesiana (izquierda) y los valores de bootstrap (derecha) están indicados en los nodos de las ramas. Los valores por debajo de 95% no son mostrados.
Los porcentajes de distancia genética al interior de M. cylindrica con el espaciador de RuBisCo fueron: máxima de 4.3% (entre R3-R4) y mínima de 1.1% (entre R3-R2 y R4-R1); el promedio de distancia genética con este marcador fue de 2.5%.
Espaciador de cox2-3. El set de datos consistió en 37 secuencias con 210 pb de longitud para el análisis poblacional; el análisis filogenético incluyó muestras de especies de la familia Solieriaceae obtenidas de GenBank, con Hydropuntia cornea como grupo externo, por lo que el conjunto de datos abarcó 46 secuencias en total.
Las medidas de diversidad genética y diferenciación calculadas (Tablas 2 y 3), mostraron en general 34 sitios segregantes (Tabla 5), una alta diversidad haplotípica general (Hd=0.81081) y una diversidad nucleotídica (π=0.07285). La población con la mayor diversidad haplotípica (Hd=0.71) y diversidad nucleotídica (π=0.06476) fue Sabancuy. Por el contrario, la población con menor variación fue Bahía de Tortuga ya que presentó sólo un haplotipo (Hd=0 y π=0) (Tabla 2).
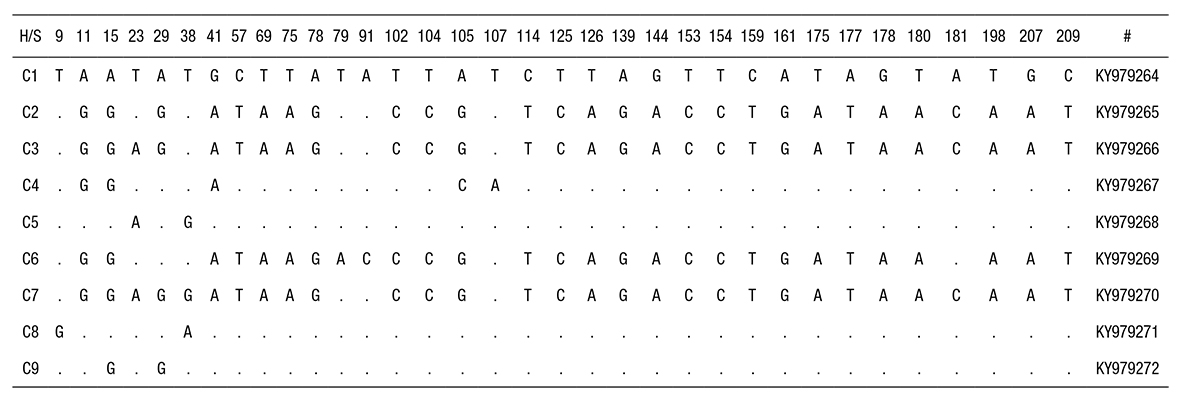
Tabla 5 Sitios variables en el alineamiento de secuencias de DNA de Meristotheca cylindrica para los haplotipos de la región espaciadora de cox2-3 y sus números de acceso al GenBank. En ne grita se resaltan los haplotipos pertenecientes al Grupo I (GI). En tipografía normal se indican los haplotipos del Grupo II (GII). H = haplotipos, S = sitios segregantes, # = no. de acceso al GenBank.
En cuanto a los índices de diferenciación, se obtuvieron valores de Nm=0.45 y de FST=0.52855. El AMOVA arrojó un índice de ΦPT=0.363 y un valor de Nm=0.879, con un 36% de la varianza explicada por las diferencias del marcador mitocondrial entre las poblaciones, en tanto que un 64% de la varianza explicada por la variación al interior de las mismas (Tabla 3).
Respecto al análisis de haplotipos, la red de parsimonia estadística reveló nueve haplotipos agrupados en dos redes, que correspondieron con dos grupos genéticamente diferenciados (GI y GII) con una estruc turación total. La primera red (GI) estuvo conformada por cinco haploti pos: C1 (n=12), C8 (n=1), C5 (n=3), C9 (n=1) y C4 (n=4) (Figura 1 y Tabla 1), mientras que la segunda red (GII) por cuatro haplotipos: C3 (n=1), C2 (n=10), C6 (n=1) y C7 (n=4) (Figura 1 y Tabla 1). En GI, el haplotipo C1 (designado por el programa como el posible haplotipo ancestral) estuvo separado de C8, C5 y C9 por dos pasos mutacionales, y a su vez, estuvo separado de C4 por cinco pasos mutacionales. En GII, el haplotipo C3 (designado como posible haplotipo ancestral incluso con n=1) se separó de C7 y C2 por un solo paso mutacional, mientras que cinco pasos mutacionales lo separaron de C6 (Fig. 1).
Respecto de su distribución geográfica (Fig. 2), el haplotipo C1 se presentó en las localidades de Punta Xen, Sabancuy e Isla Aguada; C2 fue localizado en Sabancuy, Isla Aguada y Bahía de Tortuga; C3 fue exclusivo de Isla Aguada; C4 y C5 se encontraron simultáneamente en Isla Aguada y Playa Bonita; C6 y C7 se delimitaron a Punta Xen y final mente C8 y C9 se restringieron a Sabancuy. El número de haplotipos por población fue el siguiente: Isla Aguada (5), Sabancuy (4), Punta Xen (3), Playa Bonita (2) y Bahía de Tortuga (1) (Fig. 2).
En el análisis filogenético, las muestras correspondientes a los nueve haplotipos de M. cylindrica se agruparon en dos grupos mo nofiléticos (GI, GII), con soporte de probabilidad posterior bayesiana y bootstrap de 0.98 y 0.95, respectivamente (Fig. 4). El grupo I (GI) incluyó las muestras correspondientes a los haplotipos C1, C4, C5, C8 y C9, en tanto que el grupo II (GII) agrupó las muestras correspondientes a C2, C3, C6 y C7 (Figura 4, Tabla 1). Las muestras correspondientes a los haplotipos R2 y R3 del espaciador de RuBisCo se incluyen en GII del espaciador cox2-3 (Tabla 1).

Figura 4 Topología bayesiana obtenida con secuencias de la región espaciadora de cox2-3 para las poblaciones de Meristotheca cylindrica. Las secuencias obtenidas en el presente estudio están indicadas en las ramas de color azul (GII) y rojo (GI); las restantes fueron obtenidas del GenBank, cuyos números de acceso se indican a un lado. Los valores de probabilidad posterior bayesiana (izquierda) y los valores de bootstrap (derecha) están indicados en los nodos. Los valores por debajo de 95% no son mostrados.
Los porcentajes de distancia genética al interior de M. cylindrica (entre ambos grupos) con el espaciador de cox2-3 fueron: máxima de 14.8% (entre C8-C7 y C8-C3) y mínima de 0.5% (entre C3-C7 y C3-C2); la distancia genética promedio fue de 8.3%. Al interior de GI, la distancia mínima se detectó entre C1-C5, C1-C8 y C1-C9 (1%) y la máxima entre C4-C5 y C4-C8 (3.3%); la distancia genética promedio del grupo fue de 2%. Al interior de GII, la distancia genética mínima se detectó entre C2-C3 y C3-C7 (0.5%) y la máxima entre C6-C7 (2.9%); la distancia genética promedio fue de 1.5% para este grupo.
Análisis morfológico. El análisis múltiple de varianzas (MANOVA) a partir de datos cuantitativos asociados a las tres partes del talo (Tabla 6), indicó que no hubo diferencias estadísticamente significativas entre los grupos morfológicos analizados, ya que Wilk’s Λ=0.066267; F (8, 1) =1.76132 y p=0.527258>0.05.
Tabla 6 Cuantificación de caracteres morfológicos de 10 muestras de Meristotheca cylindrica. Los caracteres diagnósticos se establecieron según Ardito et al. (2014) modificado para M. cylindrica, con la adición del número de filamentos medulares y el diámetro de células de las cortezas externa (CE) e interna (CI). IA = Isla Aguada, S = Sabancuy, BT = Bahía de Tortuga, PX = Punta Xen, PB = Playa Bonita.
Discusión
Los resultados obtenidos con las regiones espaciadoras de RuBisCo y cox2-3, así como los análisis morfológicos, revelaron estructura gené tica al interior de M. cylindrica en Campeche.
La red haplotípica obtenida del análisis con la región espaciadora RuBisCo dejó al descubierto moderadas diferencias entre poblaciones con cuatro haplotipos interconectados, mientras que la red haplotípi ca obtenida con la región espaciadora cox2-3 mostró una estructu ración total de nueve haplotipos en dos grupos genéticos (GI y GII), correspondientes con dos grupos o entidades biológicas subyacentes en M. cylindrica. En el grupo GII el haplotipo sugerido por el programa como el ancestro fue C3. Sin embargo, este se restringió a la locali dad de Isla Aguada y sólo estuvo representado por un individuo, por lo que se considera que el haplotipo C2 sería el posible ancestro ya que presentó el mayor número de individuos (10), el mismo número de conexiones que C3 (2) y su localización en tres de cinco localidades. Las poblaciones más ricas en haplotipos y que presentaron el mayor número de haplotipos únicos fueron Isla Aguada, Sabancuy y Punta Xen, mientras que la conformación haplotípica de Bahía de Tortuga y Playa Bonita fue más homogénea. Esta distribución haplotípica sugiere un moderado intercambio genético entre las poblaciones. A diferencia de lo registrado en la región espaciadora de la RuBisCo, con la región espaciadora de cox2-3 se percibió cierta homogeneidad haplotípica en aquellas poblaciones más próximas al estado de Yucatán, mientras que la heterogeneidad haplotípica se observó en aquellas poblaciones más cercanas al estado de Tabasco, tal como describieron Núñez-Resendiz et al. (2016) para el complejo Hydropuntia cornea / Hydropuntia us neoides (C. Agardh) Gurgel & Fredericq.
Para los resultados de esta investigación, la región espaciadora de cox2-3 fue más variable que la región espaciadora de RuBisCo en una proporción de 9:4. Estos resultados son consistentes con lo descrito por Zuccarello et al. (1999), quien menciona a este gen como un marcador altamente recomendable en estudios de variación genética de algas rojas. Yow et al. (2013) mencionan que los marcadores mitocondriales son más variables que los cloroplásticos; a partir de sus resultados enmarca al gen COI como un marcador incluso más variable que la región espaciadora cox2-3. Sin embargo, Núñez-Resendiz et al. (2015) describieron a la región espaciadora de la RuBisCo como un marcador más variable que la región espaciadora cox2-3.
La distribución haplotípica en las poblaciones de M. cylindrica, con la región espaciadora de RuBisCo, evidenció que las poblaciones más parecidas en cuanto a su conformación haplotípica fueron Isla Aguada, Sabancuy y Punta Xen, mismas que a su vez son las poblaciones más cercanas entre sí. Empero, a medida que las poblaciones se acercaron más al estado de Yucatán (Bahía de Tortuga y Playa Bonita), surgieron haplotipos nuevos (R3 y R4) que no se encontraron en las primeras tres localidades (las más cercanas al estado de Tabasco) lo que podría indicar cierto aislamiento reproductivo. La presencia de los haplotipos R1 (ancestro) y R2 en Bahía de Tortuga y Playa Bonita sugiere que aún existe intercambio genético escaso en conjunto con un valor de Nm~1.
A pesar de la poca variación encontrada con la región espaciadora de la RuBisCo, se advirtió que las muestras cuyos haplotipos más le janos genéticamente al ancestro (R2 y R3) pertenecen a la estructura genética equivalente al grupo GII de la región espaciadora cox2-3.
Entre las poblaciones de M. cylindrica se detectaron distintos nive les de diferenciación genética con ambos genes. Altos valores fueron encontrados en Punta Xen y Sabancuy con la región espaciadora de RuBisCo, mientras que en Sabancuy con el espaciador de cox2-3, re sultados que podrían indicar que la poblaciones se encuentran estables o en equilibrio (Grant & Bowen, 1998). No obstante, la forma en estrella de las redes haplotípicas indica un proceso reciente de expansión de mográfica (Slatkin & Hudson, 1991) lo que sugiere que las poblaciones de M. cylindrica no están en equilibrio, similar a lo descrito por Núñez-Resendiz et al. (2016) para las poblaciones del complejo Hydropuntia cornea / Hydropuntia usneoides en Campeche. Los valores generales de FST, para ambos marcadores, indicaron una gran diferenciación ge nética según Hartl &Clark (1997) ya que son mayores a 0.25, lo cual re sulta en una estructuración significativa de las poblaciones estudiadas. Los valores de ΦPT fueron significativamente distintos de cero (0.26 para el marcador cloroplástico y 0.36 para el mitocondrial), lo que re fleja la divergencia ocurrida al interior de M. cylindrica.
Las distancias genéticas no corregidas obtenidas con la región es paciadora de RuBisCo mostraron dos valores entre sus haplotipos: a) entre los conjuntos R1-R4 y R2-R3 (1.1%) y b) entre R3 y R4 (4.3%). En cuanto al espaciador de cox2-3 las distancias fueron aún mayores entre GI y GII, con un máximo de 14.8% y un promedio de 8.3%. Las distancias genéticas interespecíficas reportadas por Núñez-Resendiz et al. (2017) entre M. cylindrica y otras especies del género varían de un mínimo de 2.8% (M. procumbens P. W. Gabrielson & Kraft, de Fiji) a un máximo de 4.4% (Meristotheca sp., de Taiwan), en tanto que las distan cias intraespecíficas se distribuyen en un intervalo de 0.2% a 0.4%. Las distancias genéticas obtenidas sugieren que ambos grupos genéticos corresponden con dos entidades diferenciadas probablemente por en cima del nivel de especie, como lo reportado entre géneros de la familia Gracilariaceae en un promedio de 8.8% así como una divergencia de 12.6% entre Gracilaria chilensis C. J. Bird, McLachlan & E. C. Oliveira y Gracilariopsis sp. (Yang et al., 2008).
Los análisis filogenéticos de inferencia bayesiana y máxima vero similitud fueron congruentes con la estructura genética encontrada con ambos marcadores: en la región espaciadora de RuBisCo se detectó un grupo monofilético con un conjunto constituido por R2 y R3 al interior, lo que coincidió con los haplotipos agrupados en GII de la región espa ciadora de cox2-3; con este marcador se encontró un clado subdividido en los grupos GI y GII con sus respectivos haplotipos. En ambos casos se ubicó sistemáticamente a M. cylindrica como grupo hermano de Eucheuma isiforme.
Anatómicamente, existe homogeneidad entre las muestras de M. cylindrica. Las diferencias se remiten a los caracteres cualitativos ex ternos: el patrón y grado de ramificación en el talo vegetativo, los cua les conforman dos grupos morfológicos distintos.
Recapitulando, es posible concluir que la estructura y diferencia ción genética, así como la variación intra e interpoblacional, revelan la presencia de distintos genotipos utilizando un fenotipo lo suficiente mente similar como para confundir la identificación taxonómica. Esto muestra indicios de un proceso de especiación en curso: se inicia con barreras ecológicas y culmina con barreras reproductivas, dando lu gar a especies crípticas con los grupos genéticos GI y GII como po siblemente ocurre en el presente caso, lo que manifiesta diferencias genéticas incluso a nivel de género exhibidas con la región espacia dora de cox2-3, el cual tuvo mayor variabilidad que el marcador cloro plástico. Posteriormente será necesario un incremento en el intervalo de muestreo a lo largo de la península de Yucatán (o más general, al Golfo de México) así como en el número de individuos por población, para establecer hipótesis más sólidas sobre los procesos que ocurren al interior de los grupos encontrados en M. cylindrica. De igual mane ra, será necesario el análisis de caracteres morfológicos cualitativos para describir la relación entre los grupos genéticos encontrados en este estudio y los grupos morfológicos reportados anteriormente. Por sí sola, la historia geológica de la península de Yucatán sugiere una alta probabilidad de encontrar diversidad críptica en los grupos biológicos reportados a lo largo de su litoral y puede ofrecer explicaciones a los procesos evolutivos en el área, por lo que la necesidad de emprender estudios de ecología evolutiva se hace manifiesta.











 nova página do texto(beta)
nova página do texto(beta)


