Introducción
El inicio de la década de los 20 no fue normal. El año 2020 inició con la noticia que en la ciudad de Wuhan en China, había casos de neumonía viral que provocaba que la gente buscara atención médica y cuya evolución no era igual a las infecciones respiratorias agudas conocidas. Uno de los primeros médicos, el oftalmólogo Li Wenliang notó el alto número de pacientes con síntomas parecidos y en diciembre del 2019 comentó con sus colegas que la enfermedad podría ser causada por un virus nuevo. El 7 de febrero del 2020 el Dr. Wenliang había muerto por la constante exposición a tal virus. Diez días después siguieron las muertes de otros médicos, el Dr. Liu Zhiming y el Dr. Xu Depu quien fuera director del hospital médico de la ciudad Ezhou en Hubei, además de muchos pacientes. Desde entonces, y según la contabilidad llevada por la universidad de Johns Hopkins1, hasta el 30 de marzo hay más de 750,000 infecciones confirmadas y más de 36,000 muertes en todo el mundo, sin que parezca que los contagios comiencen a disminuir.
En este artículo contestamos preguntas que surgen cuando un evento de esta magnitud ocurre, por ejemplo: ¿Qué es lo que ocurre cuando el virus infecta? ¿Por qué un virus antes inofensivo se convierte en un peligro para el hombre? ¿Cómo saber si el nuevo virus fue manipulado intencionalmente o no? ¿Por qué es importante saber si alguien está infectado? ¿En qué consiste la prueba clínica? ¿Qué tipo de vigilancia epidemiológica se lleva a cabo en México?
Historia natural de SARS-CoV-2
En esencia un virus no es un ser vivo, en el sentido de que no es capaz de reproducirse por sí mismo debido a que no tiene metabolismo propio. Los virus son solamente pedacitos de información genética rodeados de una capa protectora. Una vez que un virus logra entrar a una célula utiliza el metabolismo de esta para traducir los mensajes contenidos en su genoma y producir todos los componentes necesarios para ensamblar miles de virus nuevos capaces de repetir el ciclo y propagarse. Esta estrategia es usada aprovechando cualquier célula, y por eso existen virus que infectan bacterias, hongos, plantas y animales -humanos incluidos. Pero no cualquier virus puede infectar cualquier célula debido que estas son impermeables, por lo que para que la infección suceda el virus requiere de un sitio de entrada. Explicaremos este proceso más adelante2.
Dogma central de la biología. La molécula que guarda la receta para construir un individuo -sea este una bacteria microscópica o una ballena azul- es el ADN (ácido desoxirribonucleico). El ADN está muy bien resguardado en el núcleo de las células eucariotes, o protegido por muchas proteínas en las bacterias (y las arqueas), y es solo accesible cuando la información de cierta parte de la molécula necesita activarse (o de manera más técnica, expresarse) para dar lugar a la síntesis de proteínas. Así, cuando se requiere que ciertos genes sean expresados para producir las proteínas correspondientes, las dos cadenas de ADN deben desprotegerse temporalmente y abrirse para que las proteínas encargadas de la expresión puedan leer y copiar la información. Estas copias están formadas por moléculas de ácido ribonucleico o ARN. Los ARN tienen tamaños mucho más pequeños que el ADN por lo que pueden moverse fácilmente y llegar a los ribosomas en donde se comienza la síntesis de proteínas ensamblando uno a uno los aminoácidos indicados en su secuencia2 (Figura 1).
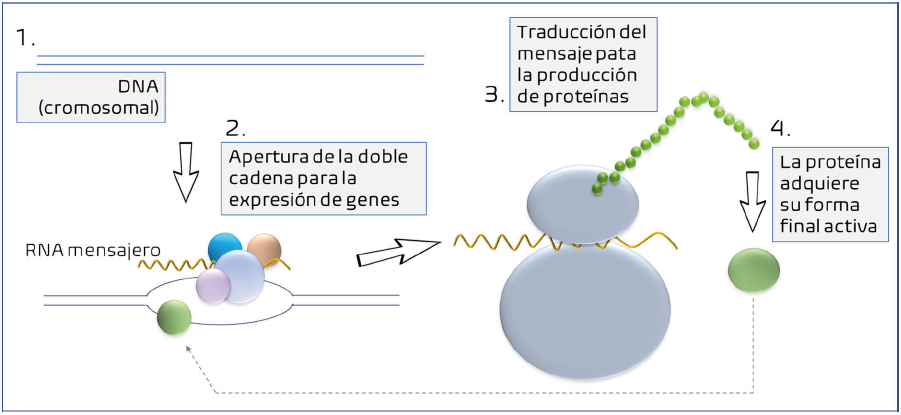
Figura 1 Se describen las etapas de la síntesis de proteína; a partir de la transcripción de un gen. 1. La doble cadena de ADN está representada por las líneas paralelas (DNA cromosomal); 2. cuando un gen deber expresarse, un complejo de múltiples proteínas (representadas por las esferas de diferente color y tamaño) abre las cadenas de DNA y sintetiza una cadena de ARN mensajero (línea dorada); 3. el ARN mensajero se localiza después en los ribosomas (grandes círculos de color gris) que leen los codones que indican el aminoácido a incorporar para sintetizar una proteína; 4. La proteína ya completa se pliega en la forma en la que es funcional (aquí representada como una esfera). La línea punteada indica una posible función de la proteína recién sintetizada en el complejo de transcripción, pero sus funciones son muchísimas dentro de las células.
Genoma viral
Como se mencionó antes, los virus son fragmentos de información genética y su genoma puede ser de dos tipos, de ADN o de ARN. En el caso de los primeros la información contenida en el ADN se expresa, en términos simples, de la manera que acabamos de mencionar: ADN→ARN→proteína. Pero cuando el genoma es de ARN, nuevamente pueden ser de dos tipos: a) los retrovirus, como el HIV que provoca el SIDA, para el cual la información debe primero convertirse a ADN a través de un proceso por el cual la molécula de ARN sirve de molde para la síntesis de ADN y la subsecuente expresión de los genes virales: ARN→ADN→ARN→proteína; y b) los virus de ARN que pueden ser leídos directamente por los ribosomas ARN→proteína. Los coronavirus pertenecen a este último caso, en el que al entrar a la célula que infectan su ARN puede inmediatamente dirigirse a los ribosomas para comenzar la síntesis de proteínas. Además, nuevas moléculas de ARN son replicadas usando como molde el ARN con el que se infectó la célula, con lo que se tienen todos los componentes para ensamblar nuevos virus, como se muestra en la figura 3. Esta es la razón por la que las infecciones por coronavirus ocurren muy rápidamente3.
El virus que actualmente está causando la pandemia se denomina técnicamente 2019-nCov o HCov-19 o SARS-CoV-2 (no confundir con SARS-CoV del que también hablaremos más adelante) y causa la enfermedad a la que se le ha llamado COVID-194. El virus SARS-CoV-2 actual no es el primero en ser descrito, de hecho, existen muchos que forman una gran familia de coronavirus, clasificándose en alfa y betacoronavirus que infectan principalmente mamíferos, mientras que los gama y deltacoronoavirus infectan aves principalmente. De los coronavirus que atacan al hombre, los más conocidos por causar epidemias previas a COVID-19 fueron el SARS-CoV que en 2003 provocó un síndrome agudo respiratorio severo y de allí su nombre (SARS por Severe Acute Respiratory Syndrome), y MERS-CoV que en 2013 afectó la región de Arabia Saudita provocando también un síndrome respiratorio conocido como MERS (por Middle East Respiratory Syndrome)5.
Se ha logrado trazar el origen de muchos tipos de coronavirus a los murciélagos, incluyendo los causantes de SARS y MERS, y uno más que ha causado diarreas y enfermedades respiratorias en cerdos. Y aunque se desconoce el origen del organismo en el que se originó SARS-CoV-2, la evidencia más fuerte indica que al igual que los virus causantes de SARS y MERS este también proviene de un murciélago de la familia Rhinolophidae6. En los murciélagos a diferencia de otros mamíferos y debido a sus hábitos sociales (ya que llegan a formar colonias con millones de individuos viviendo en gran proximidad) los virus son compartidos activamente, y debido a su sistema inmunológico relativamente débil comparado a otros organismos, los murciélagos son capaces de albergar una gran diversidad de virus. Estos dos factores: cercanía entre los individuos y tolerancia a muchos genomas virales, da como resultado una alta tasa de intercambio (o recombinación) de información genética viral entre sí, generando variabilidad7. Además, la rápida replicación de los virus dentro del organismo también permite la acumulación de mutaciones. Todos estos procesos son los que dirigen la evolución natural de los virus, dando lugar a tres escenarios posibles: i) que nuevas secuencias disminuyan la efectividad para infectar del virus o su capacidad para replicarse, por lo que en este caso esos virus desaparecen de las poblaciones; ii) que haya cambios pero que estos no causen ningún efecto y el virus mantenga la capacidad de infectar solo murciélagos, iii) que el cambio permita que ahora el virus pueda infectar las células de otro mamífero además de murciélagos.
El genoma de SARS-CoV-2 consiste en aproximadamente 30,000 nucleótidos lineales de ARN, con marcos abiertos de lectura para 27 proteínas, de las cuales cuatro forman parte de su estructura: la proteína S (por spike en inglés), la E de envoltura, la M de membrana y la N de nucleoproteína, como se observa en la figura 2. Otras 16 proteínas no estructurales tienen la función de replicar (es decir, hacer más copias del RNA viral), mientras que el resto son proteínas con funciones accesorias6,8.
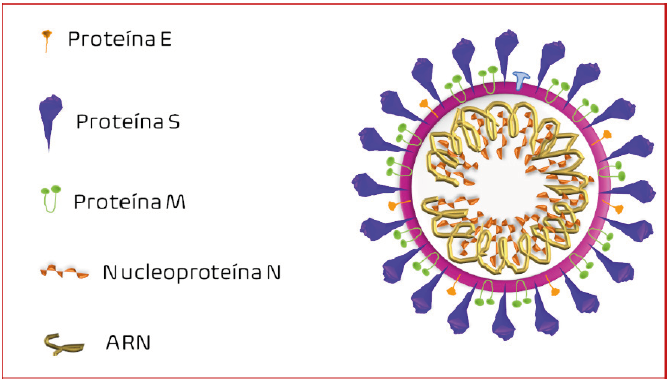
Figura 2 Estructura de SARS-CoV-2. Es un virus de RNA de cadena positiva, es decir que puede ser traducido inmediatamente al entrar a la célula. La membrana viral (morado) está compuesta por una doble capa lipídica en la que se encuentran las proteínas S (en inglés por spike) que es la encargada del reconocimiento del receptor en la célula blanco; proteína E (de envoltura) que funciona en la liberación de los nuevos virus de la célula; proteína M (de membrana) encargada de organizar las nucleoproteínas y la nucleoproteína N une al RNA y lo empaca en forma helicoidal. La membrana del virus es sensible al jabón o el etanol, por lo que se ha insistido en su uso como desinfectantes.
Se sabe que de las proteínas estructurales de SARS-CoV-2, la espiga (o proteína S) es la encargada de interaccionar fuerte y específicamente con otra proteína localizada en célula a la que infecta9. Esta proteína celular que en condiciones donde no hay infección tiene funciones que en muchas ocasiones consisten en interaccionar con otras proteínas, es conocida con el nombre de receptor. Cuando el virus entra en el organismo, solo las células que tienen el receptor adecuado capaz de interactuar con la espiga son susceptibles a ser infectadas y comenzar el ciclo replicativo que se muestra en la figura 3.
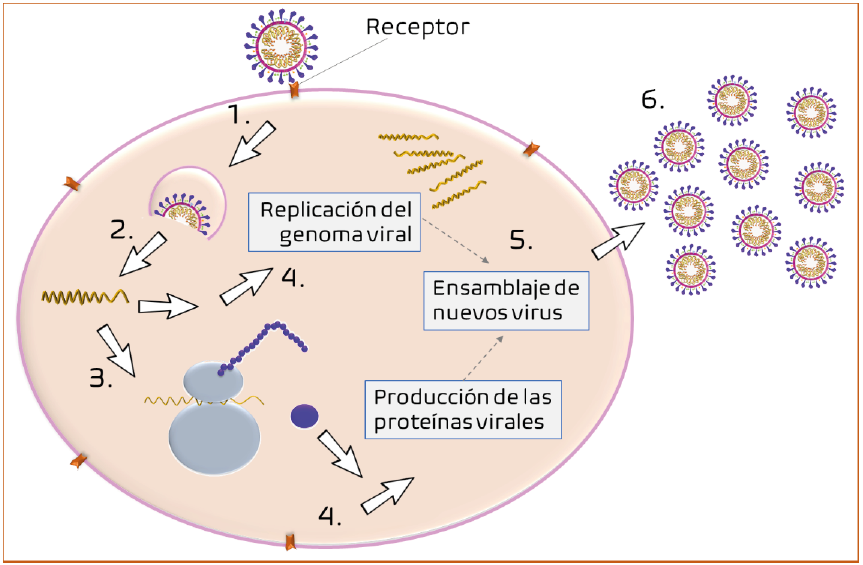
Figura 3 Se esquematiza el proceso de infección de SARS-CoV-2. 1. Hay un reconocimiento específico de la proteína S con un receptor celular (en color naranja); 2. Se internaliza el virus y es liberado el ARN dentro de la célula. 3. El ARN se dirige a los ribosomas que traducen y sintetizan las proteínas virales. 4. El RNA también es replicado por las proteínas virales. 5. Se ensamblan nuevos virus con el material recién sintetizado. 6. Los virus producidos por una sola célula se propagan para comenzar un nuevo ciclo de infección.
Las mutaciones al azar son más comunes en las proteínas espiga de los coronavirus, y específicamente en la región que hace contacto con el receptor10. Estas mutaciones provocan que la proteína espiga tengan formas ligeramente diferentes a la original, resultando en la capacidad de una de estas variables de interactuar con un receptor humano cuando antes no lo hacía, como se ilustra en la figura 4. De esta forma en la naturaleza van estableciéndose los rangos de especies a quienes los virus mutantes pueden infectar. Actualmente se han realizado experimentos de interacción de la espiga de SARS-CoV-2 con receptores conocidos para otros coronavirus, con los que se determinó que la espiga une a un receptor llamado ACE2 de humanos, pero también interacciona con ACE2 de hurones y de gatos6.
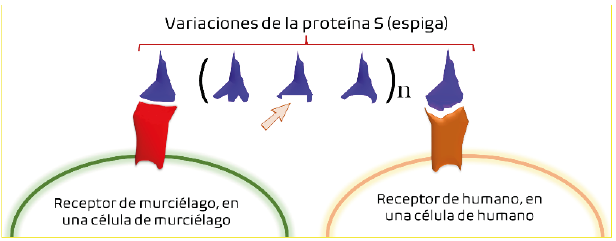
Figura 4 Los genomas de todos los organismos son susceptibles de sufrir mutaciones, lo que genera variabilidad. En el caso de SARS-CoV-2, la proteína S, y más específicamente de la región que une al receptor de la célula blanco (indicada con la flecha), es la que más varía del genoma (indicado aquí con diferentes conformaciones, de las que se pueden lograr un número desconocido n). De todas las variables una de ellas podrá establecer interacciones con un receptor diferente al que unía originalmente.
La comparación de la espiga de SARS-CoV-2 con las de otros coronavirus mostró otra característica interesante además del cambio mencionado arriba. Se determinó que el gen que codifica a la espiga contiene una mutación adicional que le permite ser cortada en una región que ha sido asociada con la patogenicidad en otros virus, en los que se ha observado que el corte de esta proteína es necesario para que ocurra el primer paso en la infección correspondiente a la fusión del virus a la célula blanco. Todavía no es claro si esto es importante para SARS-CoV-2, pero se sabe que virus parecidos al MERS-CoV han adquirido la capacidad de infectar células humanas cuando la espiga es eficientemente cortada9.
¿Es el SARS-CoV-2 un virus fabricado intencionalmente?
Como se mencionó antes, el análisis genómico de diferentes virus SARS-CoV-2 aislados de pacientes muestra un altísimo parecido a otros de la misma familia. La manipulación intencionada de un virus normalmente involucra la combinación de genomas diferentes para introducir en ellos las características deseadas como resultado final, pero en ninguno de los virus aislados de los pacientes de COVID-19 se han encontrado fragmentos de ARN diferentes a los originales. Por estas razones los estudios de filogenética (que es la rama de la genética que se dedica a rastrear a través de la evolución el origen de la información genética actual en los organismos), concluyen que las infecciones son resultado de la evolución natural del virus6,8.
Debido a que los virus aislados de murciélagos difieren un poco de SARS-CoV-2, se cree que existió un hospedero intermedio entre los murciélagos y el hombre, como ocurrió con SARS-CoV cuyo intermediario fue la civeta (que es un mamífero parecido a un gato del que se obtienen granos de café digeridos a partir sus heces, y a los que se les considera como delicatesen), o el dromedario que fue el intermediario entre los murciélagos y los hombre para el MERS-CoV, debido a la cercanía de los dueños con sus animales. Intentando trazar el viaje a partir de murciélagos para SARS-CoV-2, se observó que coronavirus aislados de pangolín (que es un animal parecido a un armadillo y muy apreciado como mascota y alimento en Asia), unen específicamente al receptor ACE2, pero ni estos virus ni los provenientes de murciélagos tienen el sitio de corte de la espiga por lo que hasta la fecha no se puede decir con certeza si el pangolín es el animal intermediario de SARS-CoV-211.
Diagnóstico de COVID-19 en México
Hasta finales de marzo en todo el mundo, se ha aplicado la prueba para detectar SARS-CoV-2 en pacientes con síntomas de fiebre, tos seca y sensación de falta de aire, y en países con suficientes recursos económicos, también en personas asintomáticas para su aislamiento. El diagnóstico se basa en la presencia de secuencias específicas del RNA de SARS-CoV-2 en las muestras, como se detalla más adelante. El protocolo que se usa para la detección del virus en nuestro país es uno de los autorizados por la OMS, y el recomendado por la Organización Panamericana de la Salud (OPS).12,13 En México el método fue validado por el Instituto de Diagnóstico y Referencia Epidemiológicos (INDRE).
Aquí indicamos la manera en la que se realiza el diagnóstico de la enfermedad COVID-19 en el laboratorio de Biología Molecular del Departamento de Infectología del Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán (INCMNSZ).
El INCMNSZ es un instituto con gran experiencia de diagnóstico molecular no solo para COVID-19 ya que desde hace muchos años en este se han implementado técnicas para la detección de diversos patógenos, tanto para la asistencia a pacientes como para el desarrollo de protocolos de investigación. El laboratorio tiene una amplia experiencia en técnicas moleculares para la identificación y la caracterización de virus respiratorios como influenza, adenovirus, virus sincicial respiratorio, rinovirus, parainfluenza, bocavirus, diferentes coronavirus, metaneumovirus, y enterovirus, además de algunas bacterias que afectan el tracto respiratorio como Mycoplasma pneumoniae, Chlamydophila pneumoniae, Legionella pneumophila y Bordetella pertussis, y finalmente HIV, Hepatitis A y C, y algunos otros que son parte de los protocolos de investigación en los que el departamento participa. Una parte importante del trabajo del departamento son los estudios de epidemiología molecular para la vigilancia de influenza, con los que se determinan las mutaciones en las cepas para establecer cuáles serán utilizadas para producir la vacuna para la siguiente temporada. Actualmente, este laboratorio hace el diagnóstico de COVID-19.
Para llevar a cabo la prueba de COVID-19 se necesita un laboratorio que cuente con las medidas de seguridad para la toma de muestras de los pacientes, buscando evitar la contaminación de los trabajadores. Esto implica trabajar en áreas restringidas y con equipo de protección personal como guantes, cubrebocas N95, batas desechables, lentes de protección que sellan y protegen de la entrada de virus por los ojos, y usar campanas de seguridad biológica para llevar a cabo las reacciones de detección.
La prueba diagnóstica identifica secuencias presentes únicamente en el genoma de SARS-CoV-2 mediante la técnica de PCR (reacción en cadena de la polimerasa) basada en la producción de millones de fragmentos de DNA que pueden ser detectados al momento que se sintetizan; a este proceso se le llama amplificación. Debido a que la amplificación de la zona seleccionada es delimitada por pequeñas moléculas con la secuencia complementaria al DNA que se desea analizar, la reacción es altamente específica solo para dicha región. Como se muestra en la figura 5, la muestra clínica contiene ADN y ARN humanos y el ARN viral. Para llevar a cabo el PCR de estas muestras es necesario que el RNA total (viral y humano) sea primero convertido a ADN, por lo que es necesario una reacción previa conocida como retrotranscripción, en la que el ARN sirve como molde para sintetizar en el tubo de ensaye las correspondientes moléculas de ADN complementario (o ADNc). A partir de ADNc se puede hacer la reacción de PCR.
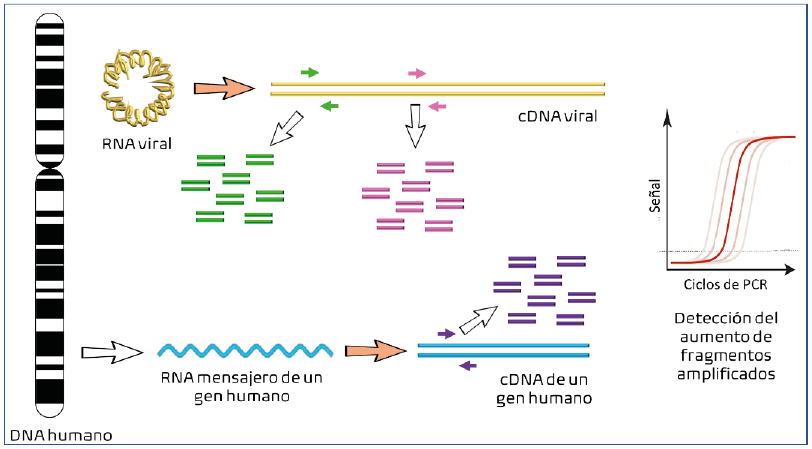
Figura 5 Proceso de detección de SARS-CoV-2 en muestras humanas. Las muestras contienen DNA (indicado con la representación de un cromosoma y moléculas de ARN mensajeros que estaban siendo transcritos en el momento de la toma de la muestra (línea curveada azul), y ARN viral (en color dorado y enrollado) en caso de existir infección. En el laboratorio, el ARN total es convertido a ADNc usando una enzima transcriptasa reversa (flechas naranjas), que sirve de molde para amplificar fragmentos usando oligonucleótidos contra dos regiones específicas de SARS-CoV-2 (flechas verdes y rosas), y una región de un gen humano (flechar moradas). Cuando las muestras son positivas para el virus, se observará la acumulación de señal de los fragmentos de PCR producidos a través de la reacción.
Un juego completo para tener resultados confirmatorios positivos o negativos incluye la detección de dos regiones diferentes del virus y una de humano. Una muestra se considera positiva solamente si hay señal para las tres reacciones, mientras que una muestra es negativa solo si no existió amplificación de ninguna región viral, pero sí la región del gen humano. Reacciones con cualquier combinación intermedia de resultados, deben repetirse. Cada análisis incluye además la amplificación de fragmentos virales sintéticos que permiten saber que los componentes necesarios en el PCR funcionan bien (control positivo), mientras que un control que solamente contenga agua no debería dar ninguna señal (control negativo). Las muestras positivas se preservan en congelación.
Esta prueba diagnóstica es altamente sensible pudiendo detectar hasta cinco copias de ARN viral, pero tiene la desventaja de un alto costo y que toma seis horas en dar el resultado. Por lo que la comunidad científica internacional se ha dado a la tarea de buscar formas más rápidas para la detección del virus como lo muestra la tabla 1.
Tabla 1 Algunas pruebas Comerciales para la detección rápida de SARS-CoV-2, según Sheridan C. (2020)14.
| Desarrollador | Nombre de la Prueba |
Descripción | Estatus |
|---|---|---|---|
| Guangzhou Wondfo Biotech (Guangzhou, China) | Análisis de anticuerpos: Wondfo SARS-CoV-2 | Inmunoensayo de 15 minutos para detector anticuerpos IgM e IgG dirigidos contra SARS-CoV-2 | National Medical Products Administration EUA in China; CE mark in Europe |
| Innovita Biological Technology | Análisis de anticuerpos: SARS-CoV-2 | Inmunoensayo de 15 minutos para detector anticuerpos IgM e IgG dirigidos contra SARS-CoV-2 | National Medical Products Administration EUA in China |
| Jiangsu Medomics Medical Technologies (Nanjing, China) | Juego de anticuerpos combinados (IgM/IgG) para la detección rápida de SARS-CoV-2 | Inmunoensayo de 15 minutos para detector anticuerpos IgM e IgG dirigidos contra SARS-CoV-2 | En tránsito |
| Mammoth Biosciences | SARS-CoV-2 DETECTR | Ensayo de 30 minutos | En estudios de validación |
| Pharmact (Berlin) | Prueba rápida de SARS-CoV-2 | Prueba de 20 minutos para identificar anticuerpos IgG e IGM contra SARS-CoV-2, en el punto de atención al paciente | Aprobado, en tránsito |
| Snibe Diagnostic (Shenzhen, China) | MAGLUMI 2019- nCoV IgM/IgG | Inmunoensayo rápido automatizado que requiere el sistema de quimioluminiscencia MAGLUMI | Usado en Europa |
| Sona Nanotech (Halifax, Nova Scotia) | Prueba rápida de detección de antígeno de SARS-CoV-2 | Prueba que detecta un dominio S1 de la proteína S de SARS-CoV-2 | Desarrollo del ensayo y validación con GE Healthcare Life Sciences, en marcha |
| Sherlock Biosciences, Cepheid | Prueba rápida basada en CRISPR para detectar SARS-CoV-2 y otros patógenos | Combina las enzimas SHERLOCK Cas12 and Cas13 para la detección de ácidos nucleicos con los instrumentos de procesamiento Cepheid’s GeneXpert | Diseñado como prueba de concepto para una amplia alianza de desarrollo de productos en enfermedades infecciosas |
| Zhejiang Orient Gene Biotech (Zhejiang, China) | Prueba rápida de IgG/IgM para COVID-19 | Inmunoensayo en fase sólida | Aytu Bioscience ha sublicenciado los derechos de distribución en Estados Unidos De L.B. Recursos (Hong Kong) y planes para obtener EUA; ya tiene marca CE |
| Biomerica | Prueba rápida de anticuerpos IgM/ IgG | Inmunoensayo con costo en dólares de $10.00 | Aprobado por la FDA y en Europa, en tránsito |
| Caspr Biotech | Detección portátil, ultrasensible y rápida de la secuencia del coronavirus SARS-CoV-2 | Basada en CRISPR-Cas12 | Evaluación de la prueba de concepto |
| Sugentech (Daejeon, Corea del Sur) | SGTi-flex COVID-19 IgM/ IgG | Inmunoensayo de 10 minutos que detecta anticuerpos IgM and IgG dirigidos a SARS-CoV-2 | Marcada como CE en Europa |
| Cepheid | Xpert Xpress SARS-CoV-2 | Prueba rápida de PCR que corre en el Sistema GenXpert benchtop system -rinde resultados en dos horas desde la toma de muestra a la entrega de resultados | Recibió autorización de emergencia por parte de la FDA |
| Xiamen AmonMed Biotechnology (Fujian, China), | Juego para prueba de IgM/IgG contra COVID-19 (oro coloidal) | Inmunoensayo de 10 minutos que detecta anticuerpos IgM and IgG dirigidos a SARS-CoV-2 | Marcado como CE para uso en Europa |
Vigilancia epidemiológica de COVID-19 en México
Cuando un virus nuevo comienza a infectar una población humana, ninguno de nosotros tiene protección contra éste porque a pesar de su parecido con otros virus a los que hemos estado expuestos, nuestras defensas (anticuerpos) no pueden reconocerlo y por lo tanto prevenir la infección. Para el caso del SARS-CoV-2, su capacidad de infección y propagación se debe a que los portadores no presentan síntomas durante un periodo que va de 3 a 5 días, incluso algunas personas nunca llegan a presentar síntomas, en particular los niños. Eso da tiempo a que las personas contagiadas transmitan el virus a sus contactos sin saberlo15.
Para controlar la propagación es necesario contar con un plan de medición del número de personas infectadas. Sin embargo, hacer pruebas diagnósticas a todos los habitantes de un país, contrario a lo que se pueda suponer, no es eficaz, primero por el costo de cada prueba (que en México varía entre dos mil y siete mil pesos), y segundo, porque un resultado negativo un día no garantiza que en las semanas posteriores no ocurra la infección. Entonces es entendible que sea más conveniente tener un estimado del número de casos que existen en el país. A este número estimado se le conoce como carga de enfermedad en la población y durante los próximos meses estaremos oyendo sobre este parámetro casi todos los días que duré la contingencia por COVID-19.
En México, a raíz de la contingencia por influenza H1N1 de 2009, se diseñó un sistema de vigilancia epidemiológica que opera desde el año 2012 para identificar la carga de infecciones respiratorias agudas graves (IRAG) en la población, que corresponden a aquellas que pueden llegar a producir neumonía requiriendo hospitalización de los pacientes16.
En este sistema colaboran laboratorios de diagnóstico a lo largo de todo el país, y en cada ciudad donde existe uno de los laboratorios del Sistema Nacional de Vigilancia Epidemiológica (SINAVE) se seleccionan casos de pacientes que presentan síntomas de IRAGs para identificar al virus que las causa. A estos casos seleccionados y analizados se les conoce como centinelas, por lo cual el sistema también se conoce como Sistema de Vigilancia Centinela, término que también escucharemos con mucha frecuencia en los próximos meses.
Desde 2012, este sistema ha puesto especial énfasis en influenza H1N1 pero desde que la OMS emitió la alerta de pandemia por SARS-CoV-2, el SINAVE se preparó para determinar también la presencia de este nuevo virus en la población mexicana17,18.
El primer día en que el SINAVE reportó los resultados de presencia de SARS-CoV-2 en las muestras centinela fue el 7 de marzo de 2020, diez días antes de que la SEP anunciara la suspensión de clases a lo largo de todo México. Ese día se analizaron muestras de 140 pacientes que presentaban síntomas de enfermedad respiratoria grave distribuidas a lo largo de todo el país, pero enfocadas sobre todo en los estados donde ya se habían identificados casos confirmados y sospechosos de importación de COVID-19 (7 casos de COVID-19 confirmados y 30 sospechosos), y en ninguna de ellas se encontraron infecciones por SARS-CoV-2. El 11 de marzo se volvieron a reportar los resultados de 182 nuevas muestras centinelas y tampoco hubo infecciones por SARS-CoV-2 en ellas (ese día se reportaron 11 casos confirmados y 49 sospechosos de COVID-19 en México). A partir de esta fecha, el SINAVE comenzó una reconfiguración que implicó el cambio del enfoque de particular hacia la vigilancia de influenza para encausar los esfuerzos hacia la vigilancia de COVID-19 en México19.
¿Por qué es importante el sistema centinela? Porque le permite al sistema de salud tener un estimado de la carga de COVID-19 en nuestro país identificando las regiones más afectadas y los posibles brotes. Pensemos que no es lo mismo vivir en una región conectada ampliamente con el resto del mundo, como la Ciudad de México y su zona conurbada, donde existe una concentración muy grande personas viviendo en un espacio reducido, en la que es más probable que surjan más casos y se afecte a mayor cantidad de contactos, a diferencia de regiones no tan conectadas y con menor densidad poblacional o población que vive en regiones más grandes y separadas entre sí.
¿Por qué no se les hace la prueba de SARS-CoV-2 a todos los pacientes? Porque en este momento ya se identificó al nuevo virus como causante de IRAGs y ya hay casos en nuestro país, por lo que todos los casos sospechosos serán tratados y manejados asumiendo que son pacientes de COVID-19. Eso implica que a la mayoría de los pacientes se les pedirá que se queden en casa, y que tomen paracetamol (siempre bajo indicación médica, nunca automedicándose) y líquidos abundantes. Solamente a los pacientes que sufran de fiebre alta, dolor de garganta, tos seca y sensación de falta el aire se les pedirá que acudan -de inmediato- a los hospitales designados para manejar los casos que requieran hospitalización.
Es muy importante mantener la calma y no acudir al hospital si no existen estos síntomas, porque acudir a un hospital aumenta el riesgo contagio de COVID-19, si es que no era este la causa de los síntomas, o de contagiar a un paciente que requiriera de hospitalización por otras causas. También hay que tener muy presente que únicamente a los pacientes que sean seleccionados como muestras Centinela se les realizará la prueba de confirmación de COVID-19, para poder continuar estimando la carga de enfermedad y tomar las medidas que eviten que esta nueva enfermedad salga de control en México.











 nueva página del texto (beta)
nueva página del texto (beta)


