Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista fitotecnia mexicana
versão impressa ISSN 0187-7380
Rev. fitotec. mex vol.36 no.1 Chapingo Jan./Mar. 2013
Artículos Científicos
Caracterización molecular de tres especies de Hylocereus (Cactaceae) presentes en México
Molecular characterization of three species of Hylocereus (Cactaceae) from México
María de los A. García Aguilar1*, Teresa Terrazas3, Obdulia Segura León2, Salvador Arias4, Heike Vibrans1 y Lauro López-Mata1
1 Programa de Botánica, Colegio de Postgraduados, Campus Montecillo. Km 36.5 Carretera México-Texcoco. 56230, Montecillo, Texcoco, Edo. de México. Tel. y Fax. (55)5804 59 47, 595 952 02 47, (595) 952 02 00 Exts. 1300 y 1301. *Autor para correspondencia (gamar@colpos.mx)
2 Programa de Fitosanidad, Colegio de Postgraduados, Campus Montecillo. Km 36.5 Carretera México-Texcoco. 56230, Montecillo, Texcoco, Edo. de México. Tel. y Fax. (55)5804 59 47, 595 952 02 47, (595) 952 02 00 Exts. 1300 y 1301.
3Departamento de Botánica, Instituto de Biología, Universidad Nacional Autónoma de México. 04510, México, D. F.
4Jardín Botánico, Instituto de Biología, Universidad Nacional Autónoma de México. 04510, México, D. F.
Recibido: 11 de Agosto del 2011
Aceptado: 20 de Diciembre del 2012
Resumen
Hylocereus (Berger) Britton y Rose es una cactácea con elevado potencial económico. Se distribuye en climas tropicales, subtropicales y semiáridos. El número de especies reconocidas para México es controvertido, debido a que no existe un consenso en cuanto a su taxonomía, principalmente por la amplia variación morfológica que se observa en las estructuras vegetativas de muchas plantas. Se secuenciaron tres regiones del cloroplasto (matK, rbcL, psbA) y el espaciador intergénico trnL-F para evaluar su conveniencia como código de barras para H. ocamponis (Salm-Dick) Britton y Rose, H. purpusii (Weingart) Britton y Rose e H. undatus (Haworth) Britton y Rose. Las muestras provinieron de 18 localidades, las cuales fueron caracterizadas previamente con base en caracteres morfológicos y anatómicos. Como grupo de comparación se utilizaron dos especies del género Selenicereus. De forma individual, los marcadores rbcL y trnL-F confirmaron la diferenciación de las especies de Hylocereus estudiadas. En el análisis multilocus basado en inferencia Bayesiana, la combinación de rbcL + trnL-F, discriminó a las tres especies con probabilidades > 60 %. Los resultados sugieren que esta combinación de loci puede tener utilidad como código de barras para las tres especies de este género de cactáceas.
Palabras clave: México, código de barras, Hylocereus, variación genética, análisis multilocus matK, rbcL, trnH-psbA, trnL-F.
Abstract
Hylocereus (Berger) Britton and Rose is a cacti with high economic potential. It is distributed in tropical, subtropical and semi-arid environments. The number of species identified in Mexico has been controversial, mainly because of the wide morphological variation observed on their vegetative structures. Three chloroplast regions (matK, rbcL, psbA), and the intergenic spacer trnL-F were sequenced to evaluate their suitability as barcode for H. ocamponis (Salm-Dick) Britton and Rose, H. purpusii (Weingart) Britton and Rose and H. undatus (Hawor-th) Britton and Rose. The samples were collected from 18 localities and previously characterized based on morphological and anatomical traits. Two species of Selenicereus were used as comparison group. Individual markers rbcL and trnL-F confirmed the differentiation of the three species. The multilocus analysis based on Bayesian inference with rbcL and trnL-F combined also discriminated the three species with probabilities > 60 %. These results suggest that this loci combination could be useful as a barcode for Hylocereus species.
Key words: México, barcode, Hylocereus, genetic variation, multilocus analysis, matK, rbcL, trnH-psbA, trnL-F.
INTRODUCCIÓN
El género Hylocereus pertenece a la subfamilia Cactoideae, tribu Hylocereeae, en la que Anderson (2001) reconoció 18 especies. Se distribuye ampliamente en regiones tropicales, subtropicales y semiáridas de México, Centro y Sudamérica (Ortiz, 1999). Sus plantas son trepadoras hemiepífitas, diploides (2n = 2x = 22) o tetraploides (2n = 44), en su mayoría auto-incompatibles (Tel-Zur et al., 2004), aunque previamente se había reportado la autocompatibilidad solamente en algunas de éstas (Raveh et al., 1998). Por la importancia económica nacional e internacional que tiene la comercialización de los frutos de algunas especies de este género (Raveh et al., 1998; Tel-Zur et al., 2004, 2005), y por su excepcional tolerancia a la sequía (Juárez et al., 2009), su cultivo se ha extendido de huertos familiares a una producción intensiva y semi-intensiva en Estados como Hidalgo, Oaxaca, Puebla, Veracruz y Yucatán.
El número de especies de Hylocereus reconocidas para México ha variado de nueve (Calix de Dios, 2004) a tres (García-Aguilar et al., 2009). Actualmente no existe un consenso en el reconocimiento puntual de cada una de las especies debido principalmente a la amplia variación morfológica que presentan las estructuras vegetativas de algunas de éstas (Ortiz, 1999; Grimaldo-Juárez et al., 2007). García-Aguilar et al. (2009) estudiaron la anatomía caulinar de diez poblaciones de Hylocereus en México y señalaron que atributos del tejido primario (grosor de la cutícula) y xilema secundario (diámetro del elemento de vaso, longitud de fibra, alto y ancho de radio) del tallo, permitieron explicar 72.5 % de la variación total, lo cual apoyó el reconocimiento de tres especies: H. ocamponis, H. purpusii e H. undatus.
De estas tres especies, H. undatus es la que ha mostrado mayor variación morfológica (Tel-Zur et al., 2003, 2004). Esta especie tiene una amplia distribución geográfica, ya que se le encuentra en forma silvestre y cultivada en algunas regiones del centro y en las vertientes del Pacífico y Golfo de México (Ortiz, 1999). Presenta un alto nivel de polimorfismo genético, de acuerdo con datos obtenidos mediante la técnica de RAPD (92.5 %) de 50 colectas en nueve estados del país (Legaria et al., 2005). Junqueira et al. (2010) describen también un alto polimorfismo (40.54 %) con 11 iniciadores RAPD en la región del Cerrado, Brasil.
Una de las alternativas más recientes para la identificación taxonómica de especies es el uso del código de barras de ADN, que consiste en el análisis de la variación de secuencias de ADN ortólogas (Hebert et al., 2003). Ésta ha sido particularmente útil en aportar información complementaria a los caracteres morfológicos (Hebert et al., 2003) y también para la identificación directa en algunos grupos de organismos. El Consorcio de Código de Barras para la vida (CBOL, 2009) propuso la combinación de varias regiones del ADN de cloroplasto como posibles candidatos para plantas, entre ellos matK, rbcL, rpoB y rpoC1 que son genes codificantes de proteínas, y de atpF-atpH, trnH-psbA y psbK-psbI que son espaciadores intergénicos no codificantes. De éstos, la combinación de matK + rbcL ha permitido la discriminación entre especies en más de 72 % (CBOL, 2009).
Algunas de las regiones propuestas por la CBOL han sido probadas en diferentes grupos de cactáceas en combinación con otros marcadores y han mostrado ser útiles en el esclarecimiento de relaciones de parentesco; a nivel de familia matK y trnL-F (Hernández et al., 2011), a nivel de tribu trnQ-rps16, rpl32-trnL, psbA-trnH y nuclear ITS (Calvente et al., 2011); a nivel de género rpl16 y psbA-trnH en Mammillaria (Butterworth y Wallace, 2004); rbcL, matK y psbA en Pereskia (Edwards et al., 2005); rpl16 y trnL-F en Peniocereus (Arias et al., 2005); atpB-rbcL, trnK-rps16 y trnL-trnF en Rebutía (Ritz et al., 2007); trnS-G, trnL y rlp16 en Echinopsis (Schlumpberger y Renner, 2012) y seis genes de cloroplasto y dos nucleares en Opuntia (Majure etal., 2012).
El objetivo de este estudio fue evaluar de forma individual y combinada diferentes regiones del ADN de cloroplasto (ADNc) en tres especies del género Hylocereus presentes en México, identificadas con base en caracteres anatómicos y morfológicos, con el propósito de reconocerlas molecularmente. Se emplearon las regiones matK, rbcL (codificantes) y trnH-psbA y trnL-F (espaciadores intergénicos no codificantes) que han sido utilizadas previamente en cactáceas.
MATERIALES Y MÉTODOS
Material vegetal
Las tres especies de Hylocereus se recolectaron en los Estados de Chiapas, Guerrero, Michoacán, Nayarit y Oaxaca, y se obtuvieron cinco individuos para H. ocamponis, seis de H. purpusii y cuatro de H. undatus (Cuadro 1). Las colectas fueron identificadas taxonómicamente con base en caracteres morfológicos y anatómicos propuestos por García-Aguilar et al. (2009). Además, se utilizaron tres individuos de dos especies del género Selenicereus (S. grandiflorus y S. testudo) provenientes del Jardín Botánico de la UNAM, como grupo de comparación.
Extracción de ADN y método de secuenciación
El ADN de las muestras se obtuvo directamente del tejido cortical colectado en campo y deshidratado con sílica gel hasta su utilización, y luego extraido con DNeasy Plant Mini kit de Quiagen® (Austin, USA). Los fragmentos fueron amplificados con los iniciadores reportados en la literatura, matK (Johnson y Soltis, 1994), rbcL (Olmstead et al., 1992), psbA (Sang et al., 1997) y trnL-F (Taberlet et al., 1991) (Cuadro 2). Los productos de la reacción en cadena de la polimerasa (PCR) se corrieron en geles de agarosa a 1.2 % (p/v) con amortiguador TAE (40 mM Tris-acetato, pH 7.6; 1mM Na2 EDTA), durante 30 min a 90 V. Los productos de la PCR fueron teñidos con bromuro de etidio (0.5 mg mL-1) y registrados en un fotodocumentador. Los productos de PCR sin purificar fueron secuenciados en The Genomic Center, Washington University (http://genome.wustl.edu/).
Las secuencias obtenidas fueron revisadas y ensambladas con el programa Sequencher, versión 3.0 (Gene Code, Ann Arbor, Michigan, USA) con refinamiento manual. El alineamiento se realizó con Clustal W (Thompson et al., 1997) implementado en el programa MEGA 4 (Tamura et al., 2007). En este caso se consideró buena lectura bidireccional y tiempo de edición requerido, criterios establecidos por la CBOL (2009). Las secuencias ya alineadas se compararon con las registradas en el Banco de Genes (NCBI) mediante el programa BLASTn (Altschul et al., 1997).
De cada una de las regiones consideradas se obtuvo información básica sobre niveles de variación nucleotídica, (i) sitios conservados, (ii) sitios variables, (iii) sitios informativos en parsimonia y (iv) singletones, implementados en el programa MEGA 4 (Tamura et al., 2007).
Análisis de divergencia de nucleótidos
La discriminación entre las especies se estimó con base en la proporción de sitios segregantes S o polimorfismo (número de sitios nucleotídicos que difieren entre secuencias) (Nei y Kumar, 2000) de cada una de las regiones analizadas. Además, se calculó la divergencia corregida entre las secuencias de las especies con la aplicación del modelo de distancia de Tamura-Nei, implementado en el programa MEGA 4 (Tamura et al., 2007).
Para las regiones matK y rbcL (codificantes) se calcularon varios estimadores de la variabilidad de nucleótidos: (a) π diversidad nucleotídica, dada por el número de nucleótidos diferentes por sitio entre dos secuencias (Nei y Li, 1979); y (b) θ, la proporción de sitios en que los nucleótidos difieren, determinado con base en el número de sitios segregantes (S). También se hicieron análisis de diversidad haplotípica: (i) número de haplotipos (h), (ii) diversidad haplotípica (Hd) (Nei, 1987), (iii) número de posiciones polimórficas (S) y (iv) número medio de diferencias nucleotídicas (k) entre cada par de secuencias (Tajima, 1989).
Prueba de neutralidad
Las secuencias de nucleótidos de las regiones matK y rbcL (codificantes) se transformaron a aminoácidos con el código genético de plastidio. Se estimaron las tasas de substitución sinónimas (dS) y no sinónimas (dN). Seguido de esto, se aplicó la prueba D de Tajima (Tajima, 1989), a fin de detectar desviaciones del modelo neutral, la cual se basa en las diferencias entre diversidad de nucleótidos π (Nei y Li, 1979) y el θ calculado a partir de la expresión θ = 4Ne μ (Kimura, 1968), donde Ne es el tamaño efectivo de la población y μ es la tasa de mutación. Los supuestos de esta prueba son: D = 0 (neutralidad), cuando los estimadores π = θ, - D = (negativa) selección purificadora cuando π < θ; y + D = (positiva), selección balanceadora con π > θ. Estas pruebas se implementaron con el programa DnaSP versión 5.10 (Librado y Rozas, 2009).
Análisis filogenético
La reconstrucción filogenética de las tres especies del género Hylocereus se llevó a cabo de forma independiente para cada región analizada, con base en la matriz de distancias Distancia-P (proporción de diferencias de nucleótidos detectadas entre dos secuencias alineadas), que incluye los espacios como sitios informativos. Se usó el método Neighbor-Joining (NJ) con remuestreo ("bootstrap") y 1000 repeticiones (Saitou y Nei, 1987), y el modelo de sustitución Tamura-Nei (Tamura y Nei, 1993), implementados con el programa MEGA 4 (Tamura et al., 2007).
Análisis multilocus
La combinación de las cuatro regiones se analizó con estadística Bayesiana, las combinaciones fueron de dos y tres marcadores: rbcL + psbA, rbcL + trnL-F, psbA + trnL-F y rbcL + trnL-F + psbA implementadas en MrBayes v. 3.1.2 (Ronquist y Huelsenbeck, 2003). En todos los casos se empleó un modelo de 4 x 4 y el modelo de substitución GTR y 100 000 generaciones de Cadenas de Markov de Montecarlo MCMC, con un muestreo cada 100 generaciones. Para el cálculo de probabilidades posteriores de los árboles, se usó un burn-in (número de árboles no considerados en el análisis) de 5000, equivalente a 25 % de los árboles muestreados. De cada partición, se obtuvo un árbol de consenso con 95 % de probabilidades de estabilidad.
RESULTADOS Y DISCUSIÓN
Las cuatro regiones moleculares seleccionadas se amplificaron en las muestras de los 15 individuos de las tres especies de Hylocereus, y en tres individuos de dos especies de Selenicereus. De éstas, matK fue la región que presentó la más alta calidad en la lectura y alineamiento de las secuencias, seguida por rbcL. Las regiones intergénicas psbA y trnL-F mostraron mayor dificultad en el alineamiento, en comparación con las regiones codificantes, y requirieron un mayor ajuste manual.
Los marcadores utilizados permitieron identificar distintos niveles de variación. Para el marcador matK se obtuvo un fragmento de 813 pb en 18 individuos: cinco correspondientes a H. ocamponis, seis a H. purpusii, cuatro de H. undatus y dos del género Selenicereus. Esta región no presentó variación entre las especies de Hylocereus, pero sí entre las especies de Selenicereus, con cuatro sitios variables (Cuadro 3). Estos resultados coinciden con lo señalado por Yesson et al. (2011), quienes reportaron que matK sólo permitió identificar a 77 % de 528 especies de cactáceas, debido a que esta región no cuenta con iniciadores universales que puedan ser usados en todos los grupos de plantas. Otros autores han señalado la eficacia de este marcador en la resolución taxonómica en niveles supraespecíficos, como diversas familias de angiospermas (Hilu et al., 2003).
Los bajos niveles de variación de nucleótidos de la región matK a nivel especie pueden ser explicados por la expresión génica del mismo. Es considerado un gen putativo del grupo de intrones tipo II que codifica para la proteína madurasa, la cual participa en la regulación del desarrollo de las plantas y funciona como un "regulador genético" que limita su evolución. Ello explica la relativamente baja variación intra-específica pero alta a nivel inter-específico (La-haye et al., 2008). En el futuro sería deseable explorar las regiones no conservadas del intrón a fin de buscar variación inter-específica para las especies de Hylocereus. La región matK fue excluida del análisis de diversidad de nucleótidos e inferencia filogenética, debido a que no detectó variación entre las especies de Hylocereus.
El fragmento amplificado con el marcador rbcL se obtuvo en los 18 individuos estudiados, con una longitud de 605 pb, de los cuales 544 fueron útiles y codificaron para 181 aminoácidos. Esta región presentó variación intra-específica en una de las tres especies de Hylocereus. A nivel inter-genérico, mostró seis mutaciones, lo que permitió diferenciar a Selenicereus de Hylocereus (Cuadro 3).
De las tres especies de Hylocereus, H. ocamponis es la más divergente con base en el modelo de distancia de Tamura-Nei, cuyo valor fue de 0.002, mientras que en H. purpusii e H. undatus no se detectaron diferencias importantes. Esto fue apoyado por la reconstrucción filogenética en la cual se muestran dos grandes grupos. En uno se ubica a H. ocamponis con un soporte interno "bootstrap" de 56 y en otro a H. purpusii e H. undatus con un soporte bajo de 32, del cual se deriva un subgrupo conformado por dos individuos de H. purpusii y uno de H. undatus, provenientes de las localidades de Ixtlán del Río, Nay. (15), El Nayar, Nay. (6) y Ocozocoautla, Chis. (18) (Figura 1A). Este resultado apoya la diferenciación clara de H. ocamponis realizada por García-Aguilar et al. (2009) con base en atributos anatómicos asociados a la epidermis.
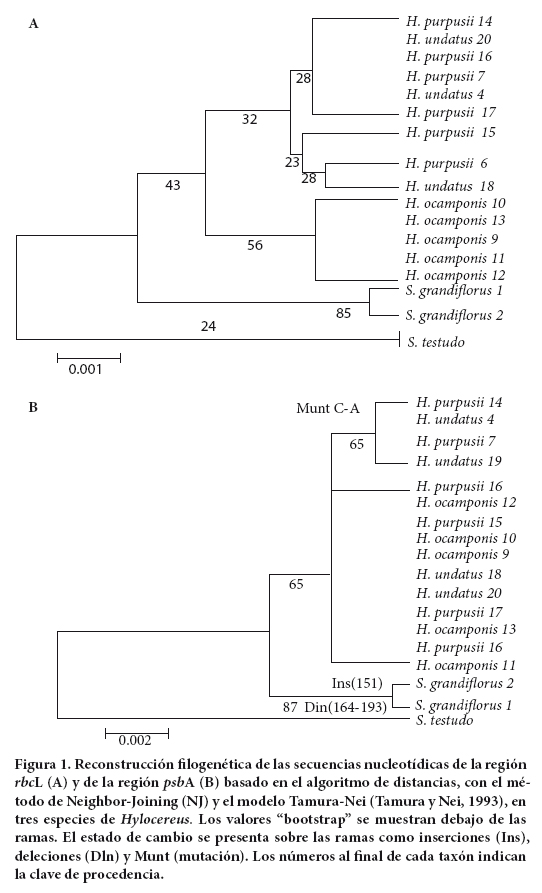
Sólo H. purpusii mostró diversidad genética para rbcL, mientras que las otras dos especies de Hylocereus no mostraron variación de nucleótidos a nivel intra-específico. La diversidad haplotípica para esta especie fue Hd = 0.500, mientras que la diversidad nucleotídica fue π = 0.00092 en la cual π < θ, valores apenas por debajo de los obtenidos para Selenicereus (Cuadro 4). La tasa de sustitución dN/ dS fue R = 0.08 y la prueba de neutralidad de Tajima D = -0.61237 no fue significativa (α = 0.01), para así rechazar que los cambios detectados son de tipo neutral.
Sin embargo, el valor negativo de esta prueba señala un proceso de selección purificadora (Simmonsen et al., 1995), lo cual puede estar relacionado al tipo de propagación vegetativa muy frecuente en las especies de este género. Aunque estos resultados no revelan un proceso de selección positiva en H. purpusii, que permitan reconocerla como independiente, sí reflejan un probable proceso de adaptación, ya que en todos los individuos de esta especie fue posible detectar una mutación de tipo sinónima en la tercera posición del codón (CCA-CCT) (379, 380, 381), que codifica para el aminoácido prolina. Esto muestra un uso preferencial de codón en la síntesis de este aminoácido (Nei y Kumar, 2000), el cual no implica cambios a nivel de aminoácido. Algunos estudios han relacionado a este fenómeno con cambios en la eficiencia de la transcripción del codón, inducido probablemente por una presión de selección (Sharp et al., 2010). Gran parte de la variación encontrada en algunas especies parece estar relacionada con este tipo de patrón (Hershberg y Petrov, 2008).
La prolina es un aminoácido libre que se encuentra en hojas, tallos y raíces de las plantas de manera natural. Se tiene evidencia de que aumenta su concentración como respuesta y adaptación al estrés hídrico (Claussen, 2005), y por efectos a la alta salinidad, deficiencia de nitrógeno, alta intensidad de luz y en respuesta a temperaturas extremas (Xu et al., 2002). Las plantas expuestas a sequía pueden llegar a acumular en las vacuolas osmolitos orgánicos y solutos como la prolina para el ajuste osmótico (Handa et al., 1986). Los resultados sugieren que H. purpusii presenta un proceso de selección impuesto por las condiciones ambientales en las cuales se distribuye.
El fragmento amplificado para psbA presentó una longitud de 474 pb, y fue secuenciado en todos los individuos estudiados. Esta región presentó variación intra-específica en dos de las tres especies de Hylocereus, dos sitios variables en dos individuos de H. purpusii provenientes de la localidad de Xaltianguis, Gro. (14) y Nochistlán, Oax. (7), y un sitio variable en dos individuos de H. undatus, uno de Tierra Colorada, Gro. (19) y otro de La Trinitaria, Chis. (4) (Cuadro 3). En la reconstrucción filogenética basada en la matriz de Distancias-P estos individuos formaron un grupo independiente con un "bootstrap" de 65. El resto de los individuos de las tres especies de Hylocereus se agruparon juntos en una politomía (Figura 1B).
Aunque no fue posible con este marcador el reconocimiento de las especies de Hylocereus, sí se observó afinidad de H. purpusii con H. undatus, especies que presentan una distribución geográfica continua desde Xaltianguis, Gro. a La Trinitaria, Chis. (Cuadro 1). Selenicereus vuelve a diferenciarse claramente con 11 sitios variables (Cuadro 5). Aunque este espaciador intergénico ha sido útil en la discriminación de algunas especies (Goremykin et al., 2005) y varios géneros como Gastrolobium (Chandler et al., 2001) y Cerastium (Scheen et al., 2004), no siempre permite detectar variación entre especies muy cercanas (Kress y Erickson, 2007), como ocurrió en el presente estudio en el cual sólo fue posible diferenciar a los individuos a nivel genérico.
El fragmento amplificado para el marcador trnL-F mostró una longitud de 1087 pb y fue secuenciado en muestras de todos los individuos estudiados. Esta región permitió detectar variación intra-específica en dos de las tres especies de Hylocereus (Cuadro 3). En la reconstrucción filogenética H. ocamponis e H. purpusii se ubicaron en grupos definidos, caracterizados por una deleción en las posiciones 265-335 y un soporte "bootstrap" de 95 y 54, respectivamente. Dos individuos de H. purpusii correspondientes a las poblaciones de Nochistlán, Oax. (7) y Xaltianguis Gro. (14), mostraron afinidad con el grupo conformado por los individuos de H. undatus, el cual se caracterizó por una inserción en las posiciones 100-101 (Figura 2A). El análisis de divergencia con base en el modelo de distancia de Tamura-Nei mostró que de las tres especies estudiadas, H. undatus es la más divergente con un valor de 0.002.
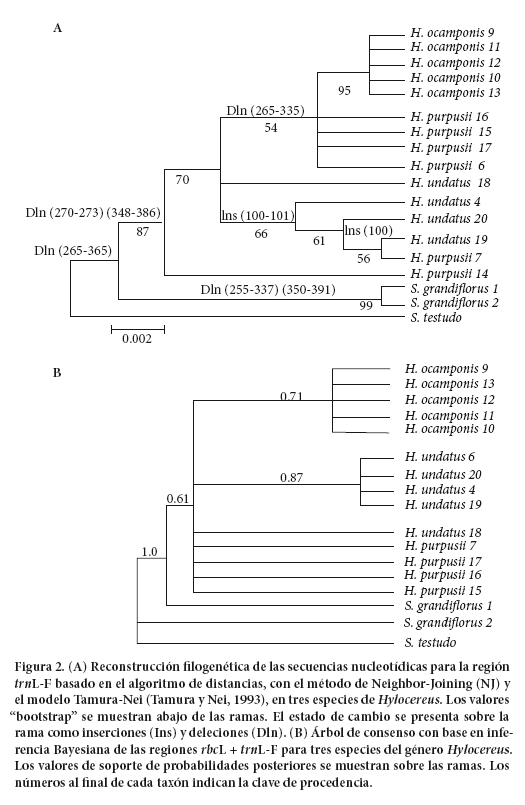
Selenicereus se volvió a diferenciar con trnL-F, ahora con 41 sitios variables. Esta región mostró el mayor número de diferencias de las cuatro regiones analizadas en este estudio. Estos resultados concuerdan con lo reportado por Calvente et al. (2011), quienes señalaron a esta región como particularmente útil en la resolución de relaciones entre géneros cercanos. En combinación con matK, esta región ha permitido resolver relaciones filogenéticas a nivel subfamilia de Cactaceae (Hernandez et al., 2011), pero no a nivel de especie cuando se tienen más de dos muestras (Bárcenas et al., 2011).
Análisis multilocus. Los resultados del análisis multilocus obtenidos con base en el Teorema de Bayes, mostraron que la combinación que permitió discriminar a las especies de Hylocereus con la mayor probabilidad fue rbcL + trnL-F (Figura 2B). En la reconstrucción filogenética, las tres especies de Hylocereus formaron grupos definidos. El grupo formado por H. undatus mostró una probabilidad de 87 %, con excepción de un individuo proveniente de la localidad de Ocozocoautla, Chis. (18) el cual formó una politomía junto con los individuos de H. purpusii. Este resultado fue similar a lo encontrado en el análisis individual de rbcL, donde este individuo (18) mostró afinidad con un individuo de H. purpusii, proveniente de la localidad del Nayar, Nay. (6). Estos resultados sugieren una probable zona de transición entre estas dos especies, apoyado por la reconstrucción filogenética individual del espaciador intergénico psbA, en el cual se observó un agrupamiento similar entre un individuo de H. purpusii proveniente de la localidad de Xaltianguis, Gro. (14) que mostró afinidad con tres individuos de H. undatus provenientes de las localidades de Tierra Colorada, Gro. (19), Nochistlán, Oax. (7), y La Trinitaria, Chis. (4). Lo anterior tendrá que confirmarse con un mayor número de colectas.
En estudios previos se ha aceptado como código de barras la combinación de regiones codificantes como rbcL con regiones intergénicas no codificantes, que evolucionan rápidamente en algunos grupos (Kress et al., 2009). La combinación de rbcL y trnL-F se ha usado con éxito en la discriminación de especies en pteridofitas (Hollingsworth et al., 2009). Recientemente Groot et al. (2011) analizaron de forma individual y combinada los marcadores rbcL y trnL-F, y señalaron que la combinación de estos loci proporciona una resolución de 100 % en especies de helechos. Los resultados obtenidos en el presente estudio sugieren que esta combinación de loci podría ser útil como código de barras para las especies del género Hylocereus.
CONCLUSIONES
La combinación de las regiones rbcL+ trnL-F permitió reconocer a las tres especies de Hylocereus. Por consiguiente, la diferenciación de las tres especies de Hylocereus reconocidas con base en caracteres morfo-anatómicos queda confirmada molecularmente. En H. ocamponis no se detectó variación intra-específica para ninguna de las cuatro regiones analizadas, por lo que se sugiere hacer una exploración de genes nucleares, así como un muestreo exhaustivo para esta especie. Los resultados sostienen a esta combinación de loci como candidata para emplearse como el código de barras para las especies de Hylocereus.
AGRADECIMIENTOS
Al CONACYT por la beca para realizar estudios de doctorado a María de los Ángeles García Aguilar (161921). Al Laboratorio de Sistemática Molecular del Instituto de Biología (UNAM), por las facilidades para la extracción y amplificación de ADN. A la Dra. Monserrat Vázquez-Sánchez, por su asesoría en dicho laboratorio, y al Colegio de Postgraduados por las facilidades para realizar el trabajo de campo.
BIBLIOGRAFÍA
Altschul S, T Madden, A Schaffer, J Zhang, Z Zhang, W Miller, D Lipman (1997) Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Res. 25:389-402. [ Links ]
Anderson E (2001) The Cactus Family. Timber Press. Portland, USA. 776 p. [ Links ]
Arias S, T Terrazas, H J Arreola-Nava, M Vázquez-Sánchez, K Cameron (2005) Phylogenetic relationships in Peniocereus (Cacta-ceae) inferred from plastid DNA sequence data. J. Plant Res. 118:317-328. [ Links ]
Bárcenas T R, C Jesson, J A Hawkins (2011) Molecular systematic of the Cactaceae. Cladistics 27:470-489. [ Links ]
Butterworth C A, R S Wallace (2004) Phylogenetic studies of Mammillaria (Cactaceae) insights from chloroplast sequence variation and hypothesis testing using the parametric bootstrap. Amer. J. Bot. 91:1086-1098. [ Links ]
Cálix de Dios H (2004) Distribución geográfica de la pitahayas (Hylocereus) en la República Mexicana. Cact. Suc. Mex. 49:4-21. [ Links ]
Calvente A, D C Zappi, F Forest, L G Lohmann (2011) Molecular phylogeny of tribe Rhipsalideae (Cactaceae) and taxonomic implications for Schlumbergera and Hatiora. Mol. Phylogen. Evol. 58:456-468. [ Links ]
CBOL Plant Working Group (2009) A DNA barcode for land plants. PNAS USA 31:12794-12797. [ Links ]
Chandler G T, R J Bayer, M D Crisp (2001) A molecular phylogeny of the endemic Australian genus Gastrolobium (Fabaceae: Mirbelieae) and allied genera using chloroplast and nuclear markers. Amer. J. Bot. 88:1675-1687. [ Links ]
Claussen W (2005) Proline as a measure of stress in tomato plants. Plant Sci. 168:241-248. [ Links ]
Edwards E, R Nyffeller, J Donoghue (2005) Basal cactus phylogeny: implications of Pereskia (Cactaceae) paraphyly for the transition to the cactus life form. Amer. J. Bot. 92:1177-1188. [ Links ]
García-Aguilar M A, T Terrazas, S Arias (2009) Anatomía caulinar de tres especies del género Hylocereus (Berger) Britton y Rose (Cactaceae) en México. Rev. Fitotec. Mex. 32:201-208. [ Links ]
Goremykin V V, B Holland, K I Hirsch-Ernst, F H Hellwig (2005) Analysis of Acorus calamus chloroplast genome and its phylogenetic implications. Mol. Biol. Evol. 22:1813-1822. [ Links ]
Grimaldo-Juárez O, T Terrazas, A García-Velásquez, M Cruz-Villagas, J F Ponce-Medina (2007) Morphometric analysis of 21 pitahaya (Hylocereus undatus) genotypes. JPACD 9:99-117. [ Links ]
Groot A, H During, J Mass, H Schneider J Vogel, R Erkens (2011) Use of rbcL and trnL-F as a two-locus DNA Barcode for identification of NW-European ferns: A ecological perspective. PLoS One 1:1-10 [ Links ]
Handa S, A K Handa, P M Hasegawa, R A Bressan (1986) Proline accumulation and the adaptation of cultured plant cells to water stress. Plant Physiol. 80:935-945. [ Links ]
Hebert P D N, A Cywinska, S L Ball, J R de Waard (2003) Biological identifications through DNA barcodes. Proc. Royal Soc. Biol. Sci. 270:313-321. [ Links ]
Hernández T, H Hernández, A De nova, R Puente, L Eguiarte, S Magallón (2011) Phylogenetic relationships and evolution of growth from in Cactaceae (Caryophyllales, Eudicotyledoneae). Amer. J. Bot. 98:44-61. [ Links ]
Hershberg R, D A Petrov (2008) Selection on codon bias. Annu. Rev. Genet. 42:287-299. [ Links ]
Hilu K W, T Borsch, K Müller, D E Soltis, P S Soltis, V Savolainen, M W Chase, M P Powell, L A Alice, R Evans, H Sauquet, C Neinhuis, T A Slotta, J Rohwer, C S Campbell, L W Chatrou (2003) Angiosperm phylogeny based on matK sequence information. Amer. J. Bot. 90:1758-1776. [ Links ]
Hollingsworth ML, A A Clark, L L Forrest, J Richardson, R T Pennington (2009) Selecting barcoding loci for plants: evaluation of seven candidate loci with species-level sampling in three divergent groups of land plants. Mol. Ecol. Resour. 9:439-457. [ Links ]
Johnson L A, D E Soltis (1994) matK DNA sequences and phylogenetic reconstruction in Saxifragaceae. Syst. Bot. 19:143-156. [ Links ]
Juárez A I, F J M Ramírez, T Cruz-Hérnandez (2009) Caracterización de dos clones de pitahaya roja (Hylocereus purpusii) de Jalisco, México. Rev. Chapingo S. Zonas Áridas 82:115-122. [ Links ]
Junqueira K P, F G Faleiro, G Bellon, N T V Junqueira, K G Fonseca, C A Lima, E C Santos (2010) Variabilidade genética de acessos de pitaya com diferentes níveis de producao por meio de marcadores RAPD. Rev. Brasil. Frutic. 32:840-846. [ Links ]
Kimura M (1968) Evolutionary rate at the molecular level. Nature 217:624-626. [ Links ]
Kress W J, D L Erickson (2007) A two-locus global DNA Barcode for Land Plants: The Coding rbcL gene complements the non-coding trnH-psbA spacer region. PLoS One 6 508:1-10. [ Links ]
Kress WJ, D L Erickson, F A Jones, N G Swenson, R Perez (2009) Plant barcodes and a community phylogeny of a tropical forest dynamics plot in Panama. PNAS USA 106:18627-18632. [ Links ]
Lahaye R, M van der Bank, D Bogarin, J Warner, F Pupullin, G Gigot, O Maurin, S Duthoit, T G Barraclough, V Savoloinen (2008) DNA barcoding the floras of biodiversity Hotspots. PNAS USA 105:2761-2762. [ Links ]
Legaria S J P, M E Alvarado, R Gaspar (2005) Diversidad genética en pitahaya (Hylocereus undatus (Haworth) Britton y Rose). Rev. Fitotec. Mex. 28:179-185. [ Links ]
Librado P, J Rozas (2009) DnaSP v5: A software for comprehensive analysis of DNA polymorphism data. Bioinformatics 25:1451-1452. [ Links ]
Majure L C, R Puente, M P Griffith, W S Judd, P S Soltis, D E Soltis (2012) Phylogeny of Opuntia s.s (Cactaceae): Clade delineation geographic origins, and reticulate evolution. Amer. J. Bot. 99:847-864. [ Links ]
Nei M (1987) Molecular Evolutionary Genetics. Columbia University Press. New York, USA. 331 p. [ Links ]
Nei M, S Kumar (2000) Molecular Evolution and Phylogenetics. Oxford University Press. New York, USA. 329 p. [ Links ]
Nei M, W H Li (1979) Mathematical model for studying genetic variation in terms of restriction endonucleases. PNAS USA 76:5269-5273. [ Links ]
Olmstead R G, H J Michaels, K M Scott, J D Palmer (1992) Monophyly of the Asteridae and identification of their major lineages inferred from DNA sequences of rbcL. Ann. Miss. Bot. Garden 79:249-265. [ Links ]
Ortiz Y (1999) Pitahaya a New Crop for Mexico. Ed. LIMUSA. México, D.F. 75 p. [ Links ]
Raveh E, J Weiss, A Nerd, Y Mizrahi (1998) Pitayas (genus Hylocereus): A New Fruit Crop for the Negev Desert of Israel: In: New Crops. J Janick, J E Simon (eds). Wiley. New York, USA. 491p. [ Links ]
Ritz C, L Martins, R Mecklenburg, V Goremykin, F H Hellwing (2007) The molecular phylogeny of Rebutia (Cactaceae) and its allies demonstrates the influence of paleogeography on the evolution of South American mountain cacti. Amer. J. Bot. 94:1321-1332. [ Links ]
Ronquist F, J P Huelsenbeck (2003) MRBAYES 3: Bayesian phylogenetic reference under mixed models. Bioinformatics 19:1572-1574. [ Links ]
Saitou N, M Nei (1987) The neighbor-joining method: A new method for reconstructing phylogenetic trees. Mol. Biol. Evol. 4:406-425. [ Links ]
Sang T, D Crawford, T Stuessy (1997) Chloroplast DNA phylogeny, reticulate evolution and biogeography of Paeonia (Paeoniaceae). Amer. J. Bot. 84:1120-1136. [ Links ]
Scheen A C, C Brochmann, A K Brysting, R Eleven, A Morris, D E Soltis (2004) Northern hemisphere biogeography of Cerastium (Caryophyllaceae): insights from phylogenetic analysis of non-coding plastid nucleotide sequences. Amer. J. Bot. 91:943-952. [ Links ]
Schlumpberger B O, S Renner (2012) Molecular phylogenetics of Echinopsis (Cactaceae): Polyphyly at all levels and convergent evolution of pollination modes and growth forms. Amer. J. Bot. 99:1335-1349. [ Links ]
Sequencher, version 3.0 Gene Code. Ann Arbor, Michigan, USA. Disponible en: http://genecodes.com/Links ]com/" target="_blank">.
Sharp P M, L R Emery, K Zeng (2010) Forces that influence the evolution of codon bias. Phil. Trans. Royal Soc. London. Ser. B Biol. Sci. 365:1203-1212. [ Links ]
Simmonsen K L, G A Churchill, C F Aquadro (1995) Properties of statistical tests of neutrality for ADN polymorphism data. Genetics 141:413-429. [ Links ]
Taberlet P, L Gielly, G Patou, J Bouvet (1991) Universal primers for amplification of three non-coding regions of chloroplast DNA. Plant Mol. Biol. 17:1105-1109. [ Links ]
Tajima F (1989) Statistical method for testing the neutral mutation hypothesis by DNA polymorphism. Genetics 123:585-595. [ Links ]
Tamura K, M Nei (1993) Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Mol. Biol. Evol. 10:512-526. [ Links ]
Tamura K, J Dudley, M Nei, S Kumar (2007) MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software versión 4.0. Mol. Biol. Evol. 24:1596-1599. [ Links ]
Tate J A, B B Simpson (2003) Paraphyly of Tarasca (Malvaceae) and diverse origins of the polyploid species. Syst. Bot. 28:723-737. [ Links ]
Tel-Zur N, S Abbo, D Bar-Zvi, Y Mizrahi (2003) Chromosome doubling in vine cacti hybrids. J. Heredity 94:329-333. [ Links ]
Tel-Zur N, S Abbo, D Bar-Zvi, Y Mizrahi (2004) Genetic relationships among Hylocereus and Selenicereus vine cacti (Cactaceae): evidence from hybridization and cytological studies. Ann. Bot. 94:527-534. [ Links ]
Tel-Zur A, S Abbo, Y Mizrahi (2005) Cytogenetics of semi-fertile triploid and aneuploid intergeneric vine cacti hybrids. J. Heredity 6:124-131. [ Links ]
Thompson J D, T J Gibson, F Plewniak, F Jeanmougin, D G Higgins (1997) The Clustal X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucl. Acids Res. 25:4876-4882. [ Links ]
Xu S, L An, H Feng, X Wang, X Li (2002) The seasonal effects of water stress on Ammopiptanthus mongolicus in a desert environment. J. Arid Environ. 51:437-447. [ Links ]
Yesson C, R Bárcenas, H Hernández, M Ruíz-Maqueda, A Prado, V Rodríguez, J Hawkins (2011) DNA barcodes for Mexican Cactaceae, plants under pressure from wild collecting. Mol. Ecol. Resour. 11:775-783. [ Links ]














