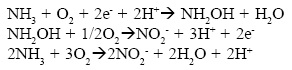Introducción
La constante transformación de los elementos químicos más importantes para la vida, por medio de fenómenos físicos y biológicos, se ve reflejada en los ciclos biogeoquímicos de la naturaleza. Estos ciclos son un claro ejemplo de las interacciones entre factores bióticos y abióticos que se dan en los sistemas, con fenómenos de sinergia y antagonismo, así como otras formas de interacción de los seres vivos. El equilibrio entre los integrantes de los ecosistemas y la participación particular de cada organismo en su hábitat lleva a la preservación de dichos ecosistemas; el papel de los microorganismos es fundamental en muchos de estos ciclos. Uno de los ciclos biogeoquímicos en el cual es mayor la participación de microorganismos ambientales, es el del nitrógeno, que incluye procesos que son realizados de forma exclusiva por estos, en algunos casos, en asociaciones simbióticas con plantas.
La evolución social de la humanidad tuvo como uno de sus puntos cruciales el desarrollo de la agricultura, porque la producción de alimentos de manera controlada fue uno de los elementos cardinales para una sociedad sedentaria. Aún con el paso del tiempo, la presencia de nitrógeno de forma asimilable para las plantas en el suelo es esencial para el crecimiento de éstas, por lo que los procesos integrados en el ciclo del nitrógeno son de particular importancia en la agricultura. El estudio de los procesos microbianos de nitrificación y desnitrificación incorpora conocimientos útiles para la productividad agrícola que permiten, por una parte, mejorar la calidad de la productividad alimentaria y, por otra, reconocer la importancia del cuidado ambiental, con la finalidad de mantener el equilibrio de los elementos fundamentales para la vida (Wrage et al., 2001; Tourna et al., 2008; Turrini et al., 2015).
Proceso de Nitrificación
El ciclo global del nitrógeno describe la transformación de gases de nitrógeno y compuestos que contienen nitrógeno en la tierra. Se compone, principalmente, de procesos impulsados por distintos microorganismos, que incluyen asimilación, amonificación, nitrificación, desnitrificación, fijación de nitrógeno, y oxidación anaerobia del amoníaco. En los sistemas de ingeniería, tales como plantas de tratamiento de aguas residuales y los humedales construidos, la nitrificación y desnitrificación acoplada se considera el principal mecanismo de eliminación de nitrógeno. La oxidación del amoníaco anaeróbico por comunidades bacterianas de Planctomycetes (Strous et al., 1999), se denomina Anammox y, en algunos casos, es de importancia en el ambiente (Sliekers et al., 2002). La nitrificación, la conversión biológica de amoníaco (NH3) a nitrato (NO3) vía nitrito (NO2), es un componente central del ciclo natural del nitrógeno (Prosser, 1989; Kowalchuk y Stephen, 2001). Se trata de un proceso aeróbico de dos pasos que está mediado por procesos biológicos; el primer paso se centra en la oxidación del amoníaco a nitritos ( NH3 → NO2- ), y posteriormente, los nitritos se oxidan a nitrato (Caffrey et al., 2007). Los microorganismos que llevan el primer paso se conocen como microorganismos oxidantes de amoníaco, mientras que los encargados de llevar el segundo paso del proceso son conocidos como microorganismos nitrito-oxidantes.
El primer paso es considerado como el paso limitante de la velocidad en el ciclo global del nitrógeno (Caffrey et al., 2007) y, por tanto, es fundamental para la eliminación de nitrógeno en aguas residuales y en el reciclaje mundial de nitrógeno (Kowalchuk et al., 2000; Kaynak et al., 2008). En este paso se asegura la conversión de amoníaco (derivado de nitrógeno orgánico durante la descomposición y la mineralización de la biomasa) a la forma oxidada y más soluble de nitrato, y proporciona el sustrato para la desnitrificación, lo que devuelve el nitrógeno de nuevo a la atmósfera. El nitrato es el sustrato preferido de plantas y microorganismos aeróbicos. Sin embargo, las consecuencias del aumento en la nitrificación debido al desequilibrio ecológico por actividades humanas también son considerables. El proceso se utiliza en las plantas de tratamiento de aguas residuales para eliminar la urea y el amoníaco de éstas (Kaynak et al., 2008). En los suelos agrícolas, la oxidación de amoníaco a nitrato no sólo aumenta la disponibilidad de nitrógeno para las plantas, también tiene consecuencias negativas, porque da lugar a la pérdida de grandes cantidades de fertilizantes por lixiviación del nitrato, que es más soluble, con la consecuente contaminación de aguas subterráneas (Wrage et al., 2001). Los microorganismos oxidantes de amoníaco tienen un impacto ambiental importante como contribuyentes a las emisiones de gases de efecto invernadero a través de la oxidación del amoníaco directamente, pero también a partir de mecanismos de nitrificación-desnitrificación (Wrage et al., 2001). Se ha demostrado que la fertilización con nitrógeno aumenta significativamente la abundancia en el suelo de las bacterias oxidantes del amoníaco (AOB), pero no de las arqueas oxidantes de amoníaco (AOA) (Di et al., 2009).
Las reacciones implicadas en el proceso de nitrificación se enlistan a continuación:
Bacterias Oxidantes del Amoníaco (AOB)
En las últimas décadas, el número de ambientes ricos en amonio ha aumentado drásticamente debido a las actividades humanas; por lo tanto, la nitrificación se ha convertido en un proceso que afecta el ciclo global del nitrógeno. En este proceso, las AOB son un componente esencial, responsable de la oxidación del amoníaco a nitrito, con hidroxilamina como producto intermedio (Ward, 1986). Estos organismos pueden degradar numerosos compuestos orgánicos (Hyman y Arp, 1989), y se considera que participan en la primera etapa de nitrificación en grandes áreas de los océanos (Olson, 1981). También están implicados en la transformación de metano y monóxido de carbono (Jones y Morita, 1983), y contribuyen al ciclo global de los gases que se encuentran en concentraciones bajas en el ambiente, tales como los óxidos de nitrógeno y nitroso (Goreau et al., 2000). Debido a su crecimiento extremadamente lento, las AOB se encuentran, por lo general, en pequeñas cantidades en ambientes naturales, lo que hace que sean difíciles de detectar por los métodos de cultivo microbiológico tradicional (Ward, 1986). En las últimas décadas, se han desarrollado herramientas moleculares independientes de cultivo basado en la caracterización del 16S ARNr para identificar bacterias oxidantes de amoníaco, y existen varios reportes sobre la diversidad de las AOB a partir de hábitats terrestres y marinos (Bothe et al., 2000; Kowalchuk et al., 2000). En estos estudios, se han observado similitudes elevadas de secuencias parciales de 16S ARNr de AOB en muestras ambientales, lo que sugiere que la fisiología y el fenotipo también se deben considerar para distinguir entre todas las cepas oxidantes del amoníaco (Stephen et al., 1996; Purkhold et al., 2000). Las AOB son quimiolitótrofas obligadas, que obtienen energía y poder reductor de la oxidación del amoníaco a partir de la conversión de amoníaco a hidroxilamina (Bock et al., 1986). Dicha etapa está catalizada por la enzima amoníaco monooxigenasa (AMO) (Hyman et al., 1992), que es exclusiva del grupo de microorganismos integrados en las bacterias oxidantes del amoníaco. La identificación de genes para AMO se ha utilizado recientemente como marcador en el análisis molecular de bacterias oxidantes de amoníaco y su presencia en ambiente microbianos particulares (Norton et al., 1996; Rotthauwe et al., 1997). La enzima AMO se compone de tres subunidades (Figura 1): amoA, de 27- a 30- kDa, proteína unida a la membrana que contiene el sitio activo de AMO; amoB, de 38- a 43- kDa, y amoC (Bergmann y Hooper, 1994). Todas las AOB pertenecientes al grupo de proteobacterias estudiadas hasta el momento parecen contener hasta tres copias casi idénticas del operónamo (Norton et al., 2002; Norton et al., 1996), mientras que sólo se detectó una copia en las bacterias oxidantes de amoníaco del grupo gamma-proteobacterial Nitrosococcus oceani y Nitrosococcus ssp. C-113 (Alzerreca et al., 1996). La enzima AMO en bacterias oxidantes de amoníaco está relacionada evolutivamente con la enzima metano monooxigenasa de partículas (pMMO), que se encuentra en bacterias oxidantes de metano (Hommes et al., 1995; Klotz y Norton, 1998). Ambas enzimas están codificadas por un mismo operón (Zahn y DiSpirito, 1996; Stolyar et al., 1999; Murrell et al., 2000). Los genes del operónamo, como el gen amoA, están presentes en todos los oxidantes del amoníaco (Sayavedra-Soto et al., 1998; Holmes et al., 1999). Por lo tanto, el estudio de los genes del operónamo permite la identificación del hábitat del grupo fisiológico de las AOB. Por el contrario, el estudio de 16S ARNr con primers específicos sólo es posible para ciertos grupos filogenéticos que pertenecen a las proteobacterias, con identificación de algunos subgrupos de AOB (Calvó et al., 2004). En este caso, el gen amoA parece ser menos resolutivo de 16S ARNr cuando se utiliza para estudios de filogenia (Purkhold et al., 2003).
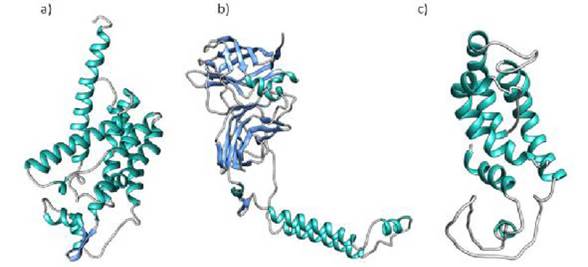
Figura 1 Modelado molecular de las subunidades de la enzima amoníaco monooxigenasa (AMO) bacteriana construidas por el servidos Swiss-Model (Kiefer et al., 2009). a) Subunidad A de Nitrosococcus oceani (ATCC 19707) reportada por Norton et al. (2002) (Genbank: AAB57809.1), se utilizó la subunidad A de la metano monooxigenasa de partículas (pMMO) (PDB: 3RGB) reportada por Smith et al. (2011) como templete. b) Subunidad B de Nitrosococcus oceani (ATCC 19707) reportada por Norton et al. (2002) (Genbank: AAC25092.1), se utilizó la subunidad B2 de la metano monooxigenasa de partículas (pMMO)(PDB: 3RGB) reportada por Smith et al. (2011) como templete. c) Subunidad C de Nitrosomonas eutropha (C91) reportada por Stein et al. (2007) (Genbank: ABI59765.1), se utilizó la subunidad C2 de la metano monooxigenasa de partículas (pMMO) (PDB: 1YEW) reportada por Lieberman y Rosenzweig (2005) como templete.
Las AOB utilizan nitrógeno reducido (por ejemplo, amoníaco) como fuente de energía, dióxido de carbono como fuente de carbono, y oxígeno molecular como aceptor de electrones. Estas AOB quimiolitotróficas comúnmente pertenecen a la Beta- y Gamma-proteobacterias, incluyendo Nitrosomonas (Beta), Nitrosospira (Beta), y Nitrosococcus (Gamma) (Nold et al., 2000; Purkhold et al., 2000). A partir de estudios de 16S ARNr y secuencias de genes amoA, se ha descrito que las especies de Nitrosomonas/Nitrosospira dominan los sistemas naturales y artificiales, tanto a partir de cultivos como de clones ambientales (Ward y O'Mullan, 2002; Park et al., 2002). Por lo tanto, se han utilizado las AOB de la subclase Beta de proteobacteria como organismos modelo en estudios ecológicos microbianos (Kowalchuk y Stephen, 2001).
Algunas AOB tienen un metabolismo anaeróbico (Bock et al., 1995; Mulder et al., 1995), organismos relacionados con Planctomycetales en el uso nitrito (en lugar de O2) como aceptor de electrones, lo que se traduce en producción de N2 (Mulder et al., 1995; Strous et al., 1999). Desde el reconocimiento de las bacterias de amoníaco y nitrito-oxidante, por Percy Faraday y Sergei Winogradsky, hace más de 100 años, sólo las proteobacterias de las subdivisiones Beta y Gamma se consideraron capaces de realizar la oxidación del amoníaco en aerobiosis (Purkhold et al., 2000). A continuación, se resumen los conocimientos actuales sobre las arqueas oxidantes del amoníaco (AOA), su fisiología, recientemente descubierta, el potencial genómico y la distribución y su actividad en varios ambientes.
Arqueas Oxidantes del Amoníaco (AOA)
Hasta hace poco, el papel de las arqueas en los ciclos biogeoquímicos, así como su diversidad fisiológica, se habían subestimado; sólo se había descrito la distribución de arqueas metanogénicas (García et al., 2000). El resto de las arqueas se consideraban extremófilos, con adaptaciones específicas que les permiten vivir en ambientes inhóspitos para la mayoría de otros organismos, como los lagos de sal saturadas, fuentes terrestres de alta temperatura y los respiraderos de aguas profundas (Woese, 1987). Sin embargo, con la ayuda de técnicas moleculares como la amplificación de genes 16S ARNr directamente de muestras ambientales, se ha demostrado que las arqueas no se limitan a hábitats extremos (Schleper et al., 2005). Por el contrario, tienen una distribución ubicua en el planeta y se encuentran en cantidades importantes en entornos comunes, como en el suelo, en el plancton marino y en los sedimentos marinos, así como en el subsuelo profundo (Schleper et al., 2005). El reciente descubrimiento de los genes homólogos de la amoníaco monooxigenasa en arqueas (Venter et al., 2004; Treusch et al., 2005; Hallam et al., 2006a) y el cultivo de arqueas autótrofas oxidantes del amoníaco (AOA) (Kónneke et al., 2005; De la Torre et al., 2008; Hatzenpichler et al., 2008; Blainey et al., 2011) revelaron un grupo adicional de microorganismos capaz de catalizar el proceso de nitrificación. La amplia distribución en estudios ambientales de los genes relacionados con la monooxigenasa de amoníaco de arqueas (genes amo) y su dominio numérico sobre sus homólogos bacterianos en ambientes marinos y terrestres sugieren que las AOA juegan un papel importante en la nitrificación a nivel mundial (Wuchter et al., 2006; Francis et al., 2008; Zhang et al., 2008). Sin embargo, desde su descubrimiento inicial, tomó más de una década que se describiera cualquier aspecto de la fisiología o su papel ecológico (Zhang et al., 2008). Sólo muy recientemente, los estudios de metagenómica y cultivo han proporcionado pruebas de que las arqueas de ambientes terrestres y marinos son capaces de oxidar el amoníaco y actúan de forma importante en el ciclo global del nitrógeno (Cao et al., 2011).
Mientras que las bacterias oxidantes del amoníaco (AOB) se consideran críticas en la nitrificación, los estudios biológicos moleculares recientemente demuestran que los miembros del reino Crenarchaeota, dentro del dominio Archaea, tienen también un papel importante en la nitrificación de suelos y en sistemas acuáticos (Kónneke et al., 2005; Hansel et al., 2008; Tourna et al., 2008). Kónneke y colaboradores aislaron una nueva cepa de arquea amoníaco-oxidante (AOA), Candidatus N. maritimus, que contiene genes para las tres subunidades, amoA, amoB y amoC de la amoníaco-monooxigenasa (Figura 2), la enzima responsable de la oxidación del amoníaco (Kónneke et al., 2005). La arquea Candidatus Cenarchaeum symbiosum también contiene los genes para la oxidación de amoníaco, aún cuando este microorganismo no se ha aislado en cultivo puro (Hallam et al., 2006b), se han realizado cultivos enriquecidos para caracterizar su hábitat (Xu et al., 2012).

Figura 2 Modelado molecular de las subunidades de la enzima amoníaco monooxigenasa (AMO) arqueal construidas por el servidos Swiss-Model (Kiefer et al., 2009). a) Subunidad A de una arquea crenarchaeote incultivable reportada por Li et al. (2011) (Genbank: ACT85495.1), se utilizó la subunidad A de la metano monooxigenasa de partículas (pMMO) (PDB: 1YEW) reportada por Lieberman y Rosenzweig (2005) como templete. b) Subunidad B de una arquea crenarchaeote incultivable reportada por Mincer et al. (2007) (Genbank: ABL11446.1), se utilizó la subunidad B de una amoníaco monooxigenasa (PDB: 4O65) reportada por Lawton et al. (2014), como templete. c) Subunidad C de una arquea crenarchaeote incultivable reportada por Konstantinidisy DeLong (2008) (Genbank: ABZ07092.1), se utilizó la subunidad C2 de la metano monooxigenasa de partículas (pMMO) (PDB: 1YEW) reportada por Lieberman y Rosenzweig (2005) como templete.
Durante la década de 1990, se reportó del descubrimiento de un nuevo subtipo de 16S ARNr perteneciente al dominio Archaea a partir de muestras de aguas superficiales del océano, sugiriendo la existencia de un grupo de organismos mesófilos cercanos a los Crenarchaeota hipertermófilos (DeLong, 1992; Fuhrman y Davis, 1997). Cuando se hizo evidente que este nuevo grupo contenía AOA, estos organismos fueron clasificados como mesófilos Crenarchaeota (Beman et al., 2008). Esta clasificación fue cuestionada a partir del análisis filogenético de la primera secuencia del genoma disponible de una AOA. Cuando Brochier-Armanet y sus colaboradores analizaron los datos de un conjunto de 53 proteínas ribosomales comunes a Archaea y Eukarya, se observó que C. Symbiosum se bifurca antes de la separación de Crenarchaeota y Euryarchaeota (Brochier-Armanet et al., 2008). Basado en este análisis filogenético, en los datos de presencia-ausencia de genes, y en la diversidad y la amplia distribución de AOA, se propuso que estos organismos pertenecen al filo Thaumarchaeota (Brochier-Armanet et al., 2008). Recientemente, este análisis se extiende también a la arquea oxidante del amoníaco Candidatus Nitrosopumilus maritimus (Schouten et al., 2008).
Casi todos los estudios que investigan a las AOA Thaumarchaeota utilizan el gen amoA para explorar su diversidad y abundancia, con la suposición implícita de que la presencia de amoA implica que la arquea es capaz de oxidar el amoníaco (Santoro et al., 2008). Sin embargo, aunque las enzimas se encuentran estrechamente relacionadas filogenéticamente, a menudo se encuentran otras monooxigenasas que realizan funciones distintas: la familia de las enzimas cobre-monooxigenasas de membrana (Cummo), por ejemplo, se relaciona filogenéticamente de manera muy cercana con el grupo al que pertenecen las arqueas amoníaco monooxigenasas (Semrau et al., 2010). En el grupo, además de la amoníaco monooxigenasa (AMO) en Beta-proteobacteria, Gamma-proteobacteria y Thaumarchaeota (Schleper y Nicol, 2010), también se incluye la metano monooxigenasa de partículas (pMMO) en Alfa-proteobacteria, Gamma-proteobacteria, Verrucomicrobia y Candidatus Methylomirabilis oxyfera (Ettwig et al., 2010), y monooxigenasa de alcanos de cadena corta (monooxigenasa de butano de partículas, pBMO) en Nocardioides Gram-positivos (Sayavedra-Soto et al., 2011). En algunos miembros de esta familia de enzimas, se ha observado que la actividad catabólica no es substrato- específico, tal como se presenta en la oxidación de etenos clorados y los hidrocarburos aromáticos (Chang et al., 2002; Yoon et al., 2011). Por ello, se ha sugerido que la maquinaria enzimática presente en un organismo define el metabolismo de obtención de energía del mismo (Tavormina et al., 2011); por ejemplo, la Gamma-proteobacteria AOB Nitrosococcus oceani puede oxidar metano, pero carece de todas las enzimas posteriores para obtener energía a partir de dicha oxidación (Klotz et al., 2006). Del mismo modo, la co-oxidación de amoníaco por las bacterias oxidantes de metano no es compatible con su crecimiento (Trotsenko y Murrell, 2008). Por otra parte, es interesante observar que las enzimas AMO de las Gamma-proteobacterias están relacionadas más estrechamente con las enzimas pMMO de las Gamma-proteobacterias que con las AMO de las Beta-proteobacterias y tienen igual especificidad de sustrato por amoníaco y metano (Lontoh et al., 2000). Por consiguiente, la sola presencia de un gen amoA, su ARNm transcrito a la proteína, no es suficiente para inferir que el microorganismo respectivo es oxidante de amoníaco.
Para la oxidación del amoníaco por el grupo I.1a Thaumarchaeote N. maritimus, es suficiente contar con un umbral extremadamente bajo de sustrato (<10 nM total de NH4+ + NH3, que representa el límite de detección del método utilizado) y un valor de Km aparente de 133 nM, estos valores son muy similares a las mediciones in situ de la nitrificación realizadas en los océanos oligotróficos (Martens-Habbena et al., 2009). La adaptación a bajas concentraciones de amonio también se ha reportado para el grupo termófilo I.1b Thaumarchaeote N. gargensis (Hatzenpichler et al., 2008), lo que indica una distribución generalizada de la oxidación del amoníaco oligotrófica dentro del filo Thaumarchaeota (Pester et al., 2011). En comparación, las concentraciones mínimas de amonio total necesarias para el crecimiento de AOB cultivadas son 100 veces más altas (> 1 mM cerca de pH neutro) con valores de Km que van desde 1.780 hasta 46 mM de amonio total (Hatzenpichler et al., 2008; Martens-Habbena et al., 2009) . Por lo tanto, es predominante la actividad de AOA en las grandes extensiones de agua de los océanos oligotróficos en comparación con la actividad de las AOB, posiblemente, por la restricción de partículas ricas en materia orgánica y ambientes costeros con altas cargas de nutrientes (Koper et al., 2010). En suelos, la actividad de AOA se observó cuando las concentraciones totales de amoníaco estaban por debajo de 15 g N-NH4+ g suelo seco-1 (Pratscher et al., 2011) mientras que AOB respondieron a altas concentraciones de amoníaco > 100 g N-NH4+ g suelo seco-1 (Jia y Conrad, 2009; Di et al., 2010). Además, la forma de nitrógeno suministrado también podría desempeñar un papel crítico: la actividad AOA se observó cuando se suministró nitrógeno mineralizado o nitrógeno orgánico derivado de composta de estiércol o materia orgánica del suelo, y la actividad AOB se presentó con amoníaco a partir de fertilizante inorgánico (Schleper y Nicol, 2010) . Otros estudios han reportado que las AOA crecieron tanto en bajas como en altas concentraciones de amoníaco, mientras que las AOB sólo crecen en altas concentraciones del compuesto. Esto sugiere que las AOA y las AOB ocupan nichos ecológicos distintos (Verhamme et al., 2011).
La primera idea de un metabolismo energético específico del Grupo I de las arqueas se derivó del estudio de una clona obtenida de una biblioteca metagenómica a partir de suelo (Treusch et al., 2004; Treusch et al., 2005), que contenía un inserto de aproximadamente 43 kb de genes 16S y 23S ARNr. Contenía además, genes homólogos a los genes bacterianos implicados en el ciclo del nitrógeno, específicamente dos marcos de lectura abiertos (ORF) que codifican para subunidades alfa y beta (amoA y amoB, respectivamente) de una amoníaco monooxigenasa (AMO), así como un gen cuyo producto era altamente similar a la nitrito reductasa dependiente de cobre (NIRK) (Treusch et al., 2005). Estudios in silico, comparando secuencias ambientales depositadas en bases de datos públicas, demostraron que la amoA y los genes amoB de la arquea de suelos eran muy similares a las arqueas asociadas a mares (Proyecto de secuenciación del Mar de los Sargazos) (Schleper et al., 2005; Leininger et al., 2006). Además, los fragmentos genómicos de arqueas marinas del proyecto del Mar de los Sargazos contenían genes que codifican para la subunidad C, aparentemente, organizado en un grupo junto con amoA y amoB, contrastando que la disposición C, A, B se encuentra en bacterias oxidantes de amoníaco (AOB). Además, se realizaron algunos experimentos en suelos fangosos para estudiar la transcripción de los genes amoA de arqueas en estos ambientes. Tras la incubación con NH4+ se observó un aumento significativo en la actividad transcripcional en el gen amoA de las arqueas, lo que sugiere que los genes amo codifican, en efecto, para la síntesis de la proteína monooxigenasa implicada en la oxidación de amoníaco (Treusch et al., 2005). Estos hallazgos permiten sugerir la presencia y actividad biológica de arqueas oxidantes de amoníaco en muchos entornos terrestres y marinos, debido a la presencia de fragmentos de 16S ARNr y secuencias de genes amoA relacionados con los de N. Maritimus en muestras de diversos ambientes (Schleper et al., 2005).
Herramientas Analíticas en el Estudio de AOB y AOA en Ambientes Naturales
Las técnicas moleculares han logrado sustituir las limitaciones asociadas con el aislamiento de los microorganismos oxidantes del amonio de sistemas naturales y artificiales. Estas técnicas, además, permiten conocer la diversidad biológica implicada en el ciclo del nitrógeno, así como reconocer a los genes que codifican a las enzimas implicadas en las transformaciones del nitrógeno (Leininger et al., 2006).
Por ejemplo, se estudió la heterogeneidad genética natural de poblaciones bacterianas autótrofas oxidantes del amoníaco que pertenecen a la subclase de Beta-proteobacteria, usando una prueba basada en la técnica de reacción en cadena de la polimerasa (PCR) (Talbot et al., 2008). Se han utilizado, además, los genes amoA y la presencia y cantidad de lípidos tetraéter, para cuantificar las AOA presentes (Leininger et al., 2006). Los isoprenoides glicerol diacilglicerol tetraéteres (GDGT por sus siglas en inglés) son lípidos de membrana que se encuentran únicamente en arqueas. Se ha estimado que existe una buena correlación entre la cantidad de GDGT y la abundancia de genes amoA arqueales. Por lo tanto, estos lípidos pueden ser biomarcadores útiles para estudiar la presencia, distribución y las actividades específicas de AOA. Park et al. (2006) extrajeron el ADN genómico a partir de lodos activados y sedimentos, utilizando un kit de extracción de ADN para muestras de suelo; a partir de éste, se obtuvo un fragmento de 635 pb del gen amoA arqueal. Este fragmento es similar a las del clon de suelo metagenómico y a las secuencias marinas reportadas por otros autores (Kónneke et al., 2005; Leininger et al., 2006).
Como con el estudio de otros grupos microbianos en relación a la abundancia y composición de las comunidades de AOB y AOA en diferentes microambientes, se utilizan a menudo ensayos de PCR en tiempo real y electroforesis en gel de gradiente de desnaturalización (DGGE). El número de copias de genes determinados a partir de q-PCR se convierten en el número de células con base en una relación entre los recuentos totales y la cantidad total de ADN extraído (Herrmann et al., 2008; Urakawa et al., 2008). En un reporte, se correlacionó positivamente la cuantificación de las arqueas por el número de copias por q-PCR de amoA, con el recuento de células Crenarchaeota por el método de TargetFish, lo que sugiere que cada célula Crenarchaeota tiene dos o tres copias del gen amoA (Wuchter et al., 2006).
El DGGE es un método que permite distinguir la composición de una comunidad microbiana particular, con base en las diferencias en secuencia genética de fragmentos de ADN generados por PCR cuando se sujetan a un gradiente desnaturalizante, lo que conlleva la separación de los fragmentos con base en su composición nucleotídica. Durante el corrimiento del ADN en el gel de DGGE, los productos de PCR se enfrentan a concentraciones cada vez mayores de desnaturalizante químico a medida que migran a través de un gel de poliacrilamida. Las diferentes secuencias de ADN (de diversos microbios) se desnaturalizan a distintas concentraciones del desnaturalizante, lo que resulta en un patrón de bandas específico. Cada banda representa, teóricamente, una población microbiana específica presente en la comunidad. Por ejemplo, los patrones de DGGE revelan que la fertilización de nitrógeno, fosfato, potasio y la concentración de materia orgánica producen cambios significativos en la composición de la comunidad de AOB, pero no en la comunidad de AOA en suelos arenosos alcalinos (Shen et al., 2008). El DGGE, junto con PCR en tiempo real (q-PCR), no sólo determina la abundancia de AOB y AOA bajo diferentes prácticas de fertilización, sino que también caracteriza a la diversidad microbiana, su comunidad, su composición y los cambios en la población por modificaciones en las condiciones ambientales.
Inspirados por los rápidos avances en las técnicas de genómica aplicada a microorganismos cultivados, Stein et al. (1996) utilizaron un vector BAC para hacer una biblioteca de gran inserción de las aguas marinas del Pacífico Noreste para caracterizar arqueas marinas. Empleando sondas de genes 16S rARN, se generó un fragmento genómico de 38.5 kb y, a partir de éste, se identificaron 3552 clonas de Crenarchaeota mesófilos. Este estudio fue el comienzo del campo de la genómica microbiana ambiental o metagenómica (Riesenfeld et al., 2004). Posteriormente, se han clonado fragmentos de gran tamaño del genoma en cromosomas bacterianos artificiales (BACs) o, más comúnmente, en los llamados fósmidos (un híbrido de un cósmido y BAC), que se archivan en bibliotecas de clonas de Escherichia coli (Schleper et al., 1998; Herrmann et al., 2008). Por otro lado, la secuenciación a gran escala con el enfoque de secuenciación del genoma total permite la generación de una pequeña secuencia, que se determina en muestras ambientales y se puede estudiar in silico (Venter et al., 2004). Estas técnicas se utilizan ahora cada vez más para la caracterización de las comunidades microbianas, particularmente, desde que nuevas tecnologías de secuenciación permiten el análisis cada vez más barato y de alto rendimiento (Riesenfeld et al., 2004). El primer genoma completo de una potencial AOA (C. symbiosum) también se obtuvo a partir de una biblioteca metagenómica, y ahora, con los organismos cultivados o enriquecidos disponibles, es posible referenciar genomas completos (Schleper et al., 1998; Riesenfeld et al., 2004; Schouten et al., 2008). Estas secuencias del genoma completo son de gran valor para la reconstrucción de las vías metabólicas; los datos de metagenómica de AOA son de gran importancia para el estudio de la distribución y el potencial genómico de los organismos en los distintos ambientes (Schleper et al., 1998).
Se han producido varias bibliotecas metagenómicas de microorganismos asociados al plancton marino o al suelo, que han permitido caracterizar el contenido genómico de arqueas no cultivables del Grupo I (Ochsenreiter et al., 2003; Martin-Cuadrado et al., 2008). Un análisis comparativo de las clonas del metagenoma del plancton marino indica la conservación de los genes en todo el gen 16S ARNr, lo que confirma la estrecha relación de las arqueas planctónicas, incluso en muestras provenientes de diferentes provincias oceánicas (Bejá et al., 2000; Briée et al., 2007). Dada la abundancia y ubicuidad de las arqueas planctónicas marinas, es posible que un gran número de genes de arqueas se detecten en los estudios globales de secuenciación aleatoria de ADN obtenidos de las aguas filtradas (Venter et al., 2004). Las enormes bases de datos de BAC (Leininger et al., 2006; Martin-Cuadrado et al., 2008; Luo et al., 2011), así como los esfuerzos de secuenciación a gran escala, son recursos valiosos para dilucidar la diversidad y distribución de la AOA, sobre todo las técnicas para la obtención de genomas de cepas que no son posibles de cultivar. Éstas permiten poner a prueba hipótesis sobre las funcionalidad de los genes y su metabolismo (Bejá et al., 2000; Briée et al., 2007; Luo et al., 2011).
Los estudios ecológicos que utilizan diferentes herramientas de análisis permiten valorar de mejor manera el efecto de las condiciones ambientales en la prevalencia de las comunidades de AOA y AOB. Se ha estudiado, por ejemplo, el efecto del cultivo de maíz genéticamente modificado en la composición de las comunidades de microorganismos oxidantes del amoníaco. Se demostró un cambio en la abundancia de la comunidades de AOA y AOB, más que en la composición de los microorganismos presentes (Turrini et al., 2015). La presencia de condicionantes abióticos, como el incremento en la materia orgánica debido a la presencia de contaminantes, también influyen en la composición de las comunidades de microorganismos oxidantes del nitrógeno. Un reporte que evalúa la influencia de contaminantes de una papelera sobre la comunidad microbiana de un estuario demostró que la composición de la comunidad de oxidantes del nitrógeno no se modificó, pero disminuyó la abundancia de AOB en los sitios impactados (Abell et al., 2014).
El uso de DGGE como herramienta de análisis también ha permitido evaluar el efecto de los cambios estacionales que se presentan en la abundancia de microorganismos oxidantes del amoníaco en suelos áridos y semiáridos, en donde la disponibilidad de agua juega un papel importante en la actividad biológica del sitio. La actividad oxidación del amoníaco fue mayor en el invierno y en el suelo árido. La proporción AOB fue similar, sin diferencias entre suelos o temporadas estacionales. En contraste, se presentaron diferencias en las AOA dependiendo del suelo y fue mayor su abundancia en el invierno (Sher et al., 2013).
Por otro lado, el uso de pirosecuenciación permite el análisis de un número grande de muestras, con la posibilidad identificar factores ambientales relacionados con la abundancia de comunidades de microorganismos oxidantes del amoníaco. En un estudio de 27 suelos provenientes de bosques en Alemania, utilizando amplificados del gen amoA, se determinó que el pH del suelo tenía un fuerte impacto en la composición de la comunidad de AOA y AOB. El análisis de las unidades taxonómicas operacionales (OTU), que se derivan del análisis de secuencias obtenidas por pirosecuenciación, sugiere que la composición de las AOA en los diferentes suelos depende, principalmente, del pH del mismo, más que de su composición o de otros factores abióticos presentes (Stempfhuber et al., 2015).
Conclusiones
Desde el descubrimiento de Candidatus N. maritimus, se ha hecho evidente que las arqueas oxidantes de amoníaco (AOA) juegan un papel importante en la oxidación de amoníaco y el ciclo del nitrógeno global. Aún no está claro el proceso; sin embargo, si las AOA oxidan más amoníaco en los océanos que las bacterias normales en los sistemas naturales y artificiales, esto puede significar que las AOA tienen un alto impacto ecológico mundial. Por tanto, varias preguntas deben abordarse antes de definir explícitamente el papel de las AOA en la eliminación de nitrógeno. El predominio de AOA sobre las bacterias capaces de oxidar el amoníaco (AOB) en muchos ambientes terrestres no se puede explicar tan fácil. Esto podría apuntar a una contribución de algunos ecotipos de AOA a la nitrificación, especialmente, en condiciones de baja disponibilidad de amoníaco. Por otro lado, tanto AOA como AOB pueden utilizar otros sustratos para la generación de energía o activar la oxidación del amoníaco sólo bajo condiciones ambientales particulares.
Por todo lo anterior, es necesaria investigación adicional para determinar el grado en que este nicho de separación entre las AOA y las AOB resulta de diferentes afinidades para el amoníaco, la inhibición por la alta concentración de amonio o de otros factores ambientales. Es además interesante lograr entender la interacción entre la ecofisiología de estos dos grupos de microorganismos oxidantes de amoníaco.











 nueva página del texto (beta)
nueva página del texto (beta)