Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de micología
versión impresa ISSN 0187-3180
Rev. Mex. Mic vol.42 Xalapa dic. 2015
Originales
Técnicas moleculares clásicas para la diferenciación de formas especiales de Fusarium oxysporum
Classical molecular techniques for differentiation of special forms of Fusarium oxysporium
Iobana Alanis-Martínez1, Carmen Medina-Mendoza2, Nahum Marbán-Mendoza1 y Ernestina Valadez-Moctezuma2*
1 Departamento de Parasitología Agrícola.
2 Departamento de Fitotecnia de la Universidad Autónoma Chapingo. Chapingo, Estado de México.
*Autor para correspondencia
Ernestina Valadez-Moctezuma evaladez@correo.chapingo.mx
Recibido: 27/02/ 2014.
Aceptado: 28/10/2015.
Resumen
La caracterización y diferenciación de formas especiales de Fusarium oxysporum mediante análisis de polimorfismos genéticos es más apropiado que solo considerar caracteres morfológicos o fisiológicos. Actualmente se han desarrollado técnicas moleculares sofisticadas para estimar variación genética, diferenciar o identificar microorganismos, tales como las basadas en secuenciación de segunda generación, pero éstas aún tienen altos costos y no son accesibles para muchos laboratorios que requieren diagnosticar o diferenciar a un determinado fitopatógeno. En este trabajo se compararon tres técnicas moleculares clásicas: RAPD, MP-PCR y RAMPnr con la finalidad de diferenciar aislamientos de F. oxysporum f. sp. ciceris, F. oxysporum f. sp. lycopersici, F. oxysporum f. sp. radicis-lycopersici y F. oxysporum f. sp coffeae. Estas técnicas detectaron polimorfismos entre las formas especiales de Fusarium e incluso entre los genotipos de F. o. f. sp. lycopersici. Los agrupamientos representados en los dendrogramas, indican que F. oxysporum es muy diverso y que las formas especiales analizadas son genéticamente diferentes. Estas técnicas proporcionan un procedimiento simple, rápido y de bajo costo para la caracterización y posterior identificación rutinaria de patógenos.
Palabras clave: MP-PCR, RAMPnr, RAPD, patógeno, polimorfismo.
Abstract
The characterization and differentiation of special forms of Fusarium oxysporum by analysis of DNA polymorphisms are more appropriate than just considering morphological or physiological characteristics. Currently, they have developed sophisticated molecular techniques to estimate genetic variation, differentiate or identify microorganisms, such as those based on second-generation sequencing, but they still have high costs and are not accessible to many laboratories that require diagnose or differentiate a particular plant pathogen. In this paper, three classical molecular techniques were compared: RAPD, MP-PCR and RAMPnr in order to differentiate isolates of F. oxysporum f. sp. ciceris, F. oxysporum f. sp. lycopersici, F. oxysporum f. sp. radicis-lycopersici and F. oxysporum f. sp coffeae. These techniques detected polymorphisms between the special forms of Fusarium and even between genotypes of F. o f. sp. lycopersici. The groups represented in dendrograms indicate that F. oxysporum is very diverse and that special forms analyzed are genetically different. These techniques provide a simple, fast and inexpensive routine procedure for the characterization and subsequent pathogen's identification.
Keywords: MP-PCR, RAMPnr, RAPD, pathogen, polymorphism.
Introducción
Fusarium oxysporum Schlechtend.:Fr es un complejo de especies económicamente importante, porque causa daños a una amplia variedad de cultivos agrícolas (Baayen et al., 2000). Se han descrito más de 150 formas especiales que se caracterizan por presentar propiedades fisiológicas que le otorgan la especialización patogénica a determinadas especies, géneros o grupos de plantas (Arbeláez, 2000; Baayen et al., 2000); por lo que se ha asumido que están altamente relacionadas y presentan morfología similar aún cuando pertenecen a diferentes grupos biológicos (Kistler, 1997).
Los marcadores moleculares han sido eficientes en el estudio de la diversidad genética de fitopatógenos (Baysal, 2013) y en la identificación de formas especiales (Baysal, 2009). Algunas técnicas utilizadas para determinar la diversidad genética del género Fusarium son RAPD (Random Amplified Polymorphyc DNA), RFLP (Restriction Fragment Length Polymorphism) (Bentley, 1998), AFLP (Amplified Fragment Length Polymorphism), SSR (Simple Sequence Repeats) y actualmente la secuenciación de porciones o genes completos (Wulff et al., 2010; Geiser et al., 2004; Kristensen et al., 2005). La técnica RAPD ha sido empleada para el estudio de formas especiales, razas y aislamientos con diferentes patotipos de Fusarium oxysporum (Kelly et al., 1998, Luna et al., 2004), ya que reduce el tiempo necesario para la caracterización y proveé información genética. La tecnica MP-PCR (Microsatellite Primed-PCR) y RAMPnr (non radioactive Random Amplified Microsatellite Polymorphism) fueron utilizadas por Valadez et al. (2005) como una opción para la detección de perfiles de ADN más abundantes e informativos, ofreciendo mayor estabilidad y cantidad de huellas. Actualmente existen técnicas sofisticadas basadas en secuenciación de segunda generación para el estudio de la diversidad genética de Fusarium y de otros organismos, pero éstas aún tienen altos costos. Es por ello que este trabajo tuvo como objetivo utilizar técnicas moleculares clásicas para diferenciar de forma rápida cuatro formas especiales de Fusarium oxysporum: f. sp. ciceris, f. sp. radicis-lycopersici, f. sp. lycopersici y f. sp. coffeae, los cuales pertenecen a un género de hongos fitopatógenos que morfológicamente no es posible distinguir. El conocimiento de las diferencias entre este tipo de patógenos es importante para su adecuado control, además de que estas técnicas consumen poco tiempo, proporcionan resultados confiables y son de bajo costo.
Materiales y métodos
Aislamientos
Se emplearon cuatro aislamientos por duplicado de formas especiales de Fusarium oxysporum: dos cepas (f. sp. lycopersici y f. sp. radicis-lycopersici.) procedentes de la colección de hongos fitopatógenos del Colegio de Postgraduados (Montecillos), Estado de México. La f. sp. ciceris se aisló de plantas de garbanzo con síntomas de marchitez de la zona del Bajío y la f. sp. coffeae se aisló de plantas de café con corchosis en la raíz, procedentes del estado de Veracruz. Los hongos se cultivaron en medio PDA (150g de papa, 10g de dextrosa, 18g de agar y 4 gotas de ácido láctico (25%, MERCK); se desarrollaron cultivos monósporicos, los cuales fueron cultivados en medio líquido Czapeck-Dox (30g de sacarosa, 2g NaNO3, 0.1g K2HPO4, 0.5g MgS04 7H2O, 0.5g KCL, 0.01g FeSO4 7H2O) para extracción de ADN.
Extracción de ADN
Se utilizó el método reportado por Kelly et al. (1994). La calidad de la molécula se determinó mediante electroforesis en gel de agarosa 0.8% y se cuantificó mediante espectrofotometría. Por cada forma especial se emplearon dos genotipos procedentes del mismo aislamiento, pero de conidio diferente: Fusarium oxysporum f. sp. coffeae A, coffeae B, ciceris A, ciceris B, radicislyco A, radicis-lyco B, lycopersici A, lycopersici B y una planta de garbanzo como grupo comparativo.
RAPD
Para el análisis de huellas aleatorias de ADN, se seleccionaron siete iniciadores de la serie C (05, 08, 09, 14, 16, 18 y 19) de la Casa comercial Carl Roth GMBH. La reacción de PCR se llevó a cabo en un volumen final de 25 μL con 80 ng de DNA, 20 pmoles de iniciador, amortiguador de reacción 1X, 200 μM de dNTPs, 1.5 mM de MgCl2 y 2U Taq ADN polimerasa. La amplificación se llevó a cabo con las siguientes condiciones: 94 °C, 1min, 38 ciclos [94 °C, 30 s; 35 °C, 30 s; 72 °C , 1.5 min] y 72 °C, 2.5 min de extensión final en un termociclador Perkin Elmer 480.
MP-PCR
Para esta técnica se emplearon cinco iniciadores complementarios a microsatélite: [GGAT]4, [GACA]4, [GATA]4, [CT]8 y [CA]8 de Gibco-BRL. La mezcla de reacción de 25 μL contenía 50 ng de ADN; 20 pg del iniciador, amortiguador de reacción 1X, 200 μM dNTPs, 3 mM MgCl y 1.5 U Taq ADN polimerasa. Las condiciones de termociclaje fueron las siguientes: 93 °C, 1 min; 40 ciclos [93 °C, 2 s; 40 °C, 1 min para [GATA]4 y 48 °C 1 min para [GGAT]4, [GACA]4, [CT]8 y [CA]8); 72 °C, 20 s] y 72 °C por 6 min de extensión final.
RAMPnr
Esta técnica consiste en la combinación de dos tipos de iniciadores en cada reacción, uno para microsatélites y otro aleatorio, ambos sin marcaje (Valadez et al., 2005). Se utilizaron seis pares de iniciadores: [GACA]4+C05; [GACA]4+C19; [GATA]4+C05; [GATA]4+C19; [CT]8+C05 y (CT)8+C19. El volumen final de reacción también fue de 25 μL y contenía 80 ng de ADN, 10 pg de cada iniciador, amortiguador de reacción 1X, 200 μM dNTPs, 3 mM MgCl2 y 1.5 U Taq ADN polimerasa. Los productos de amplificación para las tres técnicas utilizadas se separaron en geles de agarosa Ultra PureTM de GIBCO BRL 1.2, 1.4 y 2 % respectivamente, se tiñeron con bromuro de etidio 10 min y se documentaron con la cámara Kodak Digital Science 1D 2.0.
Análisis estadístico
Las huellas de ADN obtenidas se codificaron en una matriz binaria, a partir de la cual se construyó una matriz de similitud con el programa NTSyS pc.2.0 y el coeficiente de similitud Dice. Para el análisis de conglomerados se utilizó el método del promedio aritmético de pares no ponderados (UPGMA). Se obtuvieron los valores de correlación cofenética y por último se utilizó el programa WinBoot para obtener los valores del Boos-traping con 5000 replicaciones. En el análisis se incluyó al garbanzo con el fin de contrastar los agrupamientos.
Resultados
Características morfológicas
Las formas especiales de F. oxysporum cultivadas en medio PDA desarrollaron abundante micelio de distintas tonalidades que van desde violeta tenue hasta rojo intenso, lo cual ha sido reportado previamente por Seifert (2001). La forma especial lycopersici presentó micelio escaso a diferencia de las formas especiales restantes.
Análisis molecular
RAPD
El ADN obtenido con la técnica empleada fue de buena calidad y de cantidad adecuada para el análisis molecular propuesto. Las huellas genómicas obtenidas con los distintos iniciadores RAPDs presentaron polimorfismos entre las formas especiales y entre los genotipos de la f. sp. lycopersici (Figura 1). En el dendograma se conformaron seis grupos con un coeficiente de similitud de 0.7 (Figura 5). La técnica considera idénticos a los genotipos de la f. sp. coffeae con una confiabilidad del 100% al igual que a la f. sp. ciceris. Para en el caso de la f. sp. radicis-lycopersici, los dos aislamientos no se mantienen juntos como se esperaba; lo que sugiere contaminación en los aislamientos iniciales. El genotipo lycopersici A se relaciona con los genotipos de la f. sp. radicis-lycopersici a una distancia aproximadamente de 0.57 y confiabilidad del 99.9%. El genotipo lycopersici B se mantiene fuera del agrupamiento, relacionándose con las otras formas especiales a una distancia de 0.05 y una confiabilidad del 98.4%, lo que indica que este genotipo presenta un nivel de asociación muy bajo. El garbanzo se mantiene fuera del agrupamiento de Fusarium. El valor del coeficiente de correlación cofenética fue de r = 0.98, lo que indica muy buen ajuste.
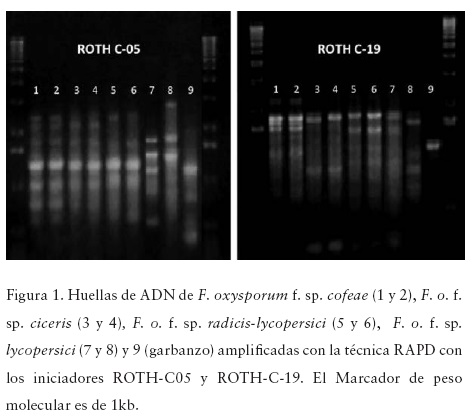
MP-PCR
Con esta técnica se detectaron pocos datos y también pocos polimorfismos; pero abundantes barridos (caracterísitica propia de la técnica) entre las cuatro formas especiales. El iniciador [GGAT]4 detectó cambios a nivel de ADN entre los genotipos de la f. sp. radicis-lycopersici (Figura 2) y [GACA]4 mostró diferencias entre los genotipos de la f. sp. Coffeae (datos no mostrados). Con el dendrograma generado a partir de la codificación de 35 datos, se identificaron cinco grupos a una distancia de 0.67 (Figura 6). La técnica consideró como idénticos a los genotipos de la f. sp. ciceris con una confiabilidad del 94.4%. Los genotipos de la f. sp. coffeae se relacionaron con una distancia de 0.77 y confiabilidad de 98.5%. El genotipo lycopersici A se agrupó con los dos genotipos de la f. sp. radicis-lycopersici. El genotipo lycopersici B se relaciona con el resto de las formas especiales a una distancia de 0.04 y confiabilidad del 52.2%. El garbanzo se asocia con el resto de los genotipos a una distancia de 0.06 y confiabilidad de 46.5%. El valor de correlación cofenética fue de r = 0.95, valor que indica muy buen ajuste.
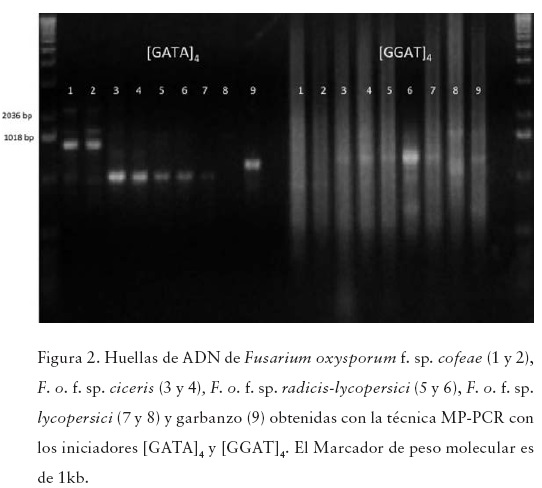
RAMPnr
Con esta técnica las huellas de ADN obtenidas fueron más abundantes y definidas con respecto a las dos técnicas anteriormente descritas. También detectaron polimorfismos entre las cuatro formas especiales y entre los genotipos de f. sp. lycopersici con los seis pares de iniciadores utilizados (Figuras 3 y 4). El dendograma se construyó con 150 datos, identificando a seis grupos con un coeficiente de 0.99 (Figura 7). Esta técnica consideró a los genotipos coffeae A-coffeae B, así como a ciceris A-ciceris B como idénticos con una confiabilidad del 100%. A los dos genotipos de la f. sp. radicis-lycopersici los relaciona a una distancia de 0.99 y confiabilidad del 100% y a su vez se relacionan con el genotipo lycopersici A a una distancia 0.66 con una confiabilidad del 82.4%. El genotipo lycopersici B se relaciona con el resto de los genotipos a una distancia de 0.08 con una confiabilidad de 97.2% lo que indica, una menor relación con el resto de las formas especiales (Figura 9). El garbanzo se mantiene fuera del agrupamiento. El valor de correlación cofenética fue de r=0.99198 que representa un buen ajuste.
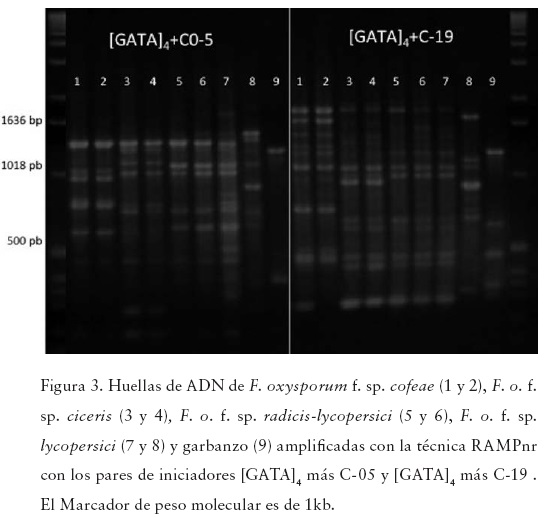
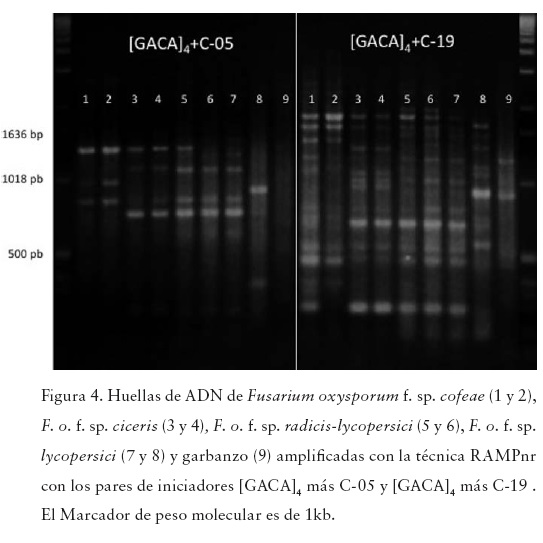
Discusión
Con las técnicas moleculares utilizadas y comparadas RAPD, MP-PCR y RAMPnr, se observaron diferencias claras en los patrones de bandas de las cuatro formas especiales: F. oxysporum f. sp. coffeae, F. oxysporum f. sp. ciceris, F. oxysporum f. sp. lycopersici y F. oxysporum f. sp. radicis-lycopersici, lo cual indica que existen diferencias genéticas. Estos resultados concuerdan con lo reportado por Vakalounakis y Fragkiadakis (1999) quienes establecieron diferencias genéticas por medio de RAPD entre las formas especiales: cucumerinum y radicis-cucumerinum.
Al comparar los agrupamientos de las tres técnicas, los resultados se muestran similares, pero los genotipos lycopersici se mantienen separados entre si, lo que sugiere que existe variabilidad genética entre la misma forma especial, o que posiblemente está contaminada desde su aislamiento original, que es lo más probable. También se observa la separación entre el genotipo lycopersici B y los dos genotipos de radicis-lycopersici, lo cual puede deberse a variación molecular a nivel de ADN entre estas formas especiales (Kistler et al. 1987, Kuninaga y Yokosawa 1989). Los polimorfismos detectados en este tipo de hongos indican que existen fuentes de variabilidad genética entre sí, debido a que tienen un origen polifilético (O'Donell, et al., 1998; Baayen et al., 2000). Por otro lado, el genotipo lycopersici A se agrupa con los dos genotipos de radicis-lycopersici, lo que sugiere que existen similitudes genéticas entre estas dos formas especiales, tal como lo han reportado Van Putten et al. (2003).
Los análisis con marcadores moleculares permiten identificar diferencias a nivel de ADN y en este estudio las tres técnicas lo evidenciaron sobre las cuatro formas especiales utilizadas. RAPD detectó polimorfismos entre los genotipos: coffeae, ciceris, radicis-lycopersici y lycopersici, e incluso variación entre los genotipos lycopersici; esta técnica ha sido ampliamente usada para estimación de la diversidad genética de especies y razas de Fusarium (Chandra et al., 2008). Por otra parte Paavanen et al. (1999) detectaron alta variabilidad genética en aislamientos de F. oxysporum con RAPD, además de que la resolución fue mejor comparada con isoenzimas; por otro lado, Manulis et al. (1994) indicaron que RAPD es un procedimiento simple, rápido y reproducible comparado con otras técnicas. MP-PCR también detectó polimorfismos entre las cuatro forma especiales, sin embargo, éstos no fueron lo suficientemente claros en este estudio debido a que hay presencia de barridos originados por una gran cantidad de fragmentos amplificados por la secuencia de los iniciadores utilizados, característica propia de la técnica que indica la gran cantidad de amplicones; pero que debido a su cercanía en pesos moleculares, no se aprecian en esta clase de geles. Barve et al. (2001) identificaron diferencias con este tipo de marcadores en cuatro razas de F. oxysporum f. sp. ciceris y Bogale et al. (2005) mostraron que los polimorfismos detectados son suficientes para el estudio de la diversidad genética en aislamientos de F. oxysporum.
La técnica RAMPnr detectó alta variabilidad genética a través de perfiles abundantes y mayor número de polimorfismos claramente nítidos. Según Barve et al. (2001) señalan que los marcadores basados en microsatélites son los mejores para el estudio de F. oxysporum, debido a que son marcadores versátiles particularmente útiles para el análisis de poblaciones. Por otro lado, Hancock y Simon (2005) mencionan que este tipo de marcadores son capaces de diferenciar especies altamente relacionadas y de acuerdo a Esselman et al. (1999) también detectan mayor cantidad de polimorfismos, por lo que se han utilizado ampliamente para el análisis de poblaciones de hongos (Chandra et al. 2008). El análisis filogenético hace posible la identificación de formas especiales y razas de patógenos, este tipo de comparaciones es necesario para la identificación de estos organismos, en donde las medidas de control pueden resultar poco efectivas si no se identifica correctamente al organismo causal, sobre todo cuando no existe diferencia a nivel morfológico. Las tres técnicas utilizadas fueron capaces de diferenciar de manera rápida e inequívoca a cada forma especial de Fusarium oxysporum, además de proporcionar datos con los cuales es posible diferenciar organismos de una misma especie tan compleja como es el caso de F. oxysporum.
Literatura citada
Arbelaéz, T.G., 2000. Algunos aspectos de los hongos del género Fusarium y de la especie Fusarium oxysporum. Agronomía Colombiana 17: 11-22. [ Links ]
Baayen, R.P., K. O'Donnell, P.J.M. Bonants, E. Cigelnik, L.P.N.M. Kroon, E.J.A. Roebroeck, C. Waalwijk, 2000. Gene genealogies and AFLP analyses in Fusarium oxysporum complex identify mononphyletic and nonmonophyletic formae speciales causing wilt and rot disease. Phytopathology 90: 891-900. [ Links ]
Barve, M.P., M.P. Haware, M.N. Sainani, P.K. Ranjekar, V.S. Gupta, 2001. Genetic diversity in Indian isolates of Fusarium oxysporum f. sp. ciceris, chickpea wilt pathogen. Theoretical and Applied Genetics 102: 138-147. [ Links ]
Baysal, Ö., Ç. Karaaslan, M. Siragusa, R. Alessandro, F. Carimi, F. De Pasquale, J.A. Teixeira da Silva, 2013. Molecular markers reflect differentiation of Fusarium oxysporum forma speciales on tomato and forma on eggplant. Biochemical Systematics and Ecology 47: 139-147. [ Links ]
Baysal, Ö., M. Siragusa, H. Ikten, I. Polat, E. GÜmrkcÜ, F. Yigit, F. Carimi, J. A. Teixeira da Silva, 2009. Fusarium oxysporum f. sp. lycopersici races and their genetic discrimination by molecular markers in West Mediterranean region of Turkey. Physiological and Molecular Plant Pathology 74: 68-75. [ Links ]
Bentley, S., K.G. Pegg, N.Y. Moore, R.D. Davis, I.W. Buddenhagen, 1998. Genetic variation among vegetative compatibility groups of Fusarium oxysporum f. sp. cubense analyzed by DNA fingerprinting. Phytopathology 88: 1283-1293. [ Links ]
Bogale, M.B., D. Wingfield, M.J. Wingfield, E.J. Steenkamp, 2005. Characterization of Fusarium oxysporum isolates from Ethiopia using AFLP, SSR and DNA sequence analyses. Fungal Diversity 23: 51-66. [ Links ]
Chandra, N.S., S.A.C. Udaya, S.R. Niranjana, H.S. Prakash, 2008. Molecular detection and characterisation of Fusarium verticillioides in maize ( Zea mays L.) grown in southern India. Annals of Microbiology 58: 359-367. [ Links ]
Esselman, E.J., L. Jianqiang, D.J. Crawford, J.L. Windus, A.D. Wolfe, 1999. Protocols for the inter simple sequence approach. Molecular Ecolology 8: 443-451. [ Links ]
Geiser, D.M., G.M.M. Jiménez, S. Kang, I. Makalowska, N. Veeraraghavan, T.J. Ward, N. Zhang, G.A. Kuldau, K. O'Donnell, 2004. FUSARIUM-ID v. 1.0: a DNA sequence database for identifying Fusarium. European Journal of Plant Pathology 110: 473-479. [ Links ]
Hancock, J.M., M. Simon, 2005. Simple sequence repeats in proteins and their significance for network evolution. Gene 345: 113-118. [ Links ]
Kelly, A.G., B.W. Bainbridge, J.B. Heale, A.E. Pérez, D.R.M. Jiménez, 1998. In plant-polymerase chain reaction detections of the wilt indicing pathotypes of Fusarium oxysporum f. sp. ciceris in chickpea (Cicer arietinum). Physiological and molecular Plant Pathology 52: 397-409. [ Links ]
Kelly, A., J.A.R. Alcalá, B.W. Bainbridge, J.B. Heale, A.E. Pérez, D.R.M. Jiménez, 1994. Use of genetic fingerprinting and random amplified polymorphic DNA to characterize pathotypes of Fusarium oxysporum f. sp. ciceris infecting chickpea. Phytopathology 84: 1293-1298. [ Links ]
Kistler, H.C., 1997. Genetic diversity in the plant-pathogenic fungus Fusarium oxysporum. Phytopathology 87: 474-479. [ Links ]
Kistler, H.C., P.W. Bosland, U. Benny, S. Leong, P.H. Williams, 1987. Relatedness of strains of Fusarium oxysporum from crucifer measured by examination of mitochondrial and ribosomal DNA. Phytopathology 77: 1289-1293. [ Links ]
Kristensen, R., M. Torp, B. Kosiak, J.A. Holst, 2005. Phylogeny and toxigenic potential is correlated in Fusarium species as revealed by partial translation elongation factor 1 alpha gene sequences. Mycological Research 109: 173-186. [ Links ]
Kuninaga, S., R. Yokosawa, 1989. Genetic relatedness within and between formae speciales of Fusarium oxysporum measured by DNA-DNA reassociation kinetics. Annals of the Phytopathological Society of Japan 55: 216-223. [ Links ]
Luna, P.A., R.H.V. Silva, M.N. Marbán, M.E. Valadez, 2004. Variabilidad genética de Fusarium oxysporum Schlechtend.:FR. f. sp. ciceris (Padwick) Matuo y К. Sato mediante PCR-RAPD's en el Bajío México. Revista Mexicana de Fitopatología 22: 44-51. [ Links ]
Manulis, S., R.M. Kogan, Y. Benyephet, 1994. Use of the RAPD technique for identification of Fusarium oxysporum f. sp. dianthi from carnation. Journal of Phytopathology 84: 98-101. [ Links ]
O'Donnell, К., H.C. Kistler, P.R.C. Cigelnik, 1998. Multiple evolutionary origins of the fungus causing Panama disease of banana: concordant evidence from nuclear and mitochondrial gene genealogies. Proceedings of the National Academy of Sciences of the USA 95: 2044-2049. [ Links ]
Paavanen, H.S., N.J. Hyvo, S.A. Bulat, M.T. Yli, 1999. RAPD-PCR, isozyme, rDNA RFLP and rDNA sequence analyses in identification of Finnish Fusarium oxysporum isolates. Mycological Research 103: 625-634. [ Links ]
Seifert K.A., 2001. Fusarium and anamorph generic concepts. In: Summerell B.A., J.F. Leslie, D. Backhouse, W.L. Bryden, L.W. Burgess (eds.), Fusarium. APS Press. St. Paul, Minnesota. pp. 15-28. [ Links ]
Vakalounakis, D.J., G.A. Fragkiadakis, 1999. Genetic diversity of Fusarium oxysporum isolates from Cucumber: Differentiation by pathogenicity, vegetative compatibility and RAPD fingerprinting. Phytopathology 89: 161-168. [ Links ]
Valadez, M.E., G. Kahl., I.A. Rubluo, R. Arreguín-Espinoza de los Monteros, 2005. Optimización de las huellas de DNA obtenidas con RAPDs y MP-PCR mediante la técnica RAMPnr. Revista Chapingo Serie Horticultura XI: 351-356. [ Links ]
Van Putten, W.F., A. Biere, J.M.M. Van Damme, 2003. Intraspecific competition and mating between fungal strains of the anther smut Microbotryum violaceum from the host plants Silene latifolia and S. dioica. Evolution 57: 766-776. [ Links ]
Wulff, E.G., J.S. S ørensen, M. Lübeck, K.F. Nielsen, U. Thrane, J. Torp, 2010. Fusarium spp. associated with rice Bakanae: ecology, genetic diversity, pathogenicity and toxigenicity. Environmental Microbiology 12: 649-657. [ Links ]














