INTRODUCCIÓN
El síndrome de Down es la alteración cromosómica más frecuente y la causa principal de discapacidad intelectual en todo el mundo. En la mayoría de los casos su causa es una copia extra del cromosoma 21 (human chromosome 21 Hsa21). Abarca un conjunto complejo de patologías que involucran prácticamente todos los órganos y sistemas. Las alteraciones más prevalentes y distintivas son la dificultad para el aprendizaje, dismorfias craneofaciales, hipotiroidismo, cardiopatías congénitas, alteraciones gastrointestinales y leucemias. Se estima que es la causa de 1 de cada 150 abortos del primer trimestre y de 8% de las anomalías congénitas en Europa.1,2
Fue descrito por John Langdon Down en 1866, dentro de su propuesta de clasificación de pacientes con discapacidad intelectual.3 Se asoció por primera vez con una alteración cromosómica en 1959, cuando Lejeune, Gautier y Turpin describieron 5 niños y 4 niñas con discapacidad intelectual y 47 cromosomas en el cultivo de fibroblastos, siendo un acrocéntrico pequeño el cromosoma extra. Los autores propusieron que el origen de este cromosoma extra se debía probablemente a una falta de disyunción, que por lo tanto ésta era la razón por la que la frecuencia del padecimiento aumentaba con la edad materna.4
El síndrome de Down se debe a una trisomía completa Hsa21 o una trisomía parcial que incluye la región crítica 21q22.3. El 95% de los casos se debe a una trisomía completa o regular; alrededor de 3% se debe a mosaicismo, una alteración en la que los pacientes tienen conjuntamente células normales y células con un Hsa21 extra; menos de 2% se origina por una traslocación no equilibrada; es decir, un cariotipo con 46 cromosomas, pero uno de ellos, usualmente el cromosoma 14, contiene material cromosómico extra del Hsa21.
La Organización Mundial de la Salud estima una prevalencia mundial de 1 en cada 1,000 recién nacidos vivos; sin embargo, estas cifras varían, lo que refleja que la prevalencia depende de variantes socioculturales, como el acceso al diagnóstico prenatal y la interrupción legal del embarazo.5 En México, la Secretaría de Salud estima una prevalencia de 1 en 650 recién nacidos vivos;6 pero el informe de 2010 del Registro y Vigilancia Epidemiológica de Malformaciones Congénitas Externas (RYVEMCE) estimó una tasa de 14.32 por 10,000 recién nacidos vivos: 1 en 698.7
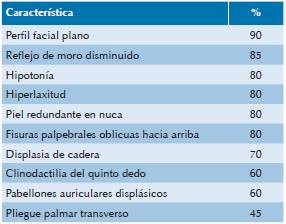
El diagnóstico es clínico y se confirma por citogenética. El patrón de características físicas observables (Gestalt) es altamente sugestivo, así como las alteraciones sistémicas. Sin embargo, no todas las alteraciones están presentes en cada individuo afectado. En recién nacidos el diagnóstico puede dificultarse; no obstante, diez características son altamente prevalentes. Hall, en 1966, analizó 48 recién nacidos afectados y encontró que 100% tuvieron 4 o más características y 89% tuvieron 6 o más. Desde entonces, estas características se utilizan para evaluar a todo recién nacido vivo, conocidas como criterios de Hall8 (Cuadro 1).
En la década de los 50 del siglo pasado sólo 47% de los recién nacidos vivos con síndrome de Down sobrevivían un año; esta cifra se incrementó a más de 90% en los 80 y de 1983 a 1997 la esperanza de vida aumentó de 25 a 49 años. Los factores de riesgo que influyen en la sobrevida son: madre de raza negra, cardiopatía congénita, defectos mayores no cardiacos y prematurez. Los predictores de sobrevida no difieren en pacientes con síndrome de Down comparados con pacientes con discapacidad intelectual en general.9
La morbilidad de los pacientes con síndrome de Down implica costos médicos 12 a 13 veces mayores en comparación con la población general durante los primeros cuatro años de vida, especialmente los pacientes con cardiopatía congénita que tienen la mayor mortalidad y en quienes se estima que requieren de 5 a 7 veces más atención médica que los pacientes con síndrome de Down sin cardiopatía congénita.10 Otras causas frecuentes de hospitalización son complicaciones de leucemia, respiratorias, hipotiroidismo y demencia; las respiratorias causan incluso mayor mortalidad.11
El antecedente de un hijo con translocación de novo no representa un riesgo incrementado y la recurrencia tiene que relación solamente con la edad materna. Sin embargo, si el padre es portador de una translocación robertsoniana el riesgo es de 3 a 5%; si la madre es la portadora el riesgo aumenta 10 a 15%. La situación es diferente cuando alguno de los padres es portador de una translocación 21:21, ya que el riesgo de recurrencia es de 100%.
MANIFESTACIONES CLÍNICAS Y SU ASOCIACIÓN CON LA GENÓMICA
Aunque algunas características de los pacientes con síndrome de Down son muy constantes existe gran variabilidad fenotípica. Diversos estudios en ratones y humanos han intentado identificar genes sensibles a dosis que expliquen de forma individual cada uno de los datos clínicos. Inicialmente se delimitó una región del Hsa21 llamada región crítica del síndrome de Down; sin embargo, otros estudios han determinado diferentes regionesque también contribuyen al fenotipo.12
Manifestaciones neurológicas
Desarrollo
Los pacientes adquieren los hitos del desarrollo de forma tardía tanto en el área motora como en el lenguaje. El coeficiente intelectual promedio en pacientes con síndrome de Down es de 35 a 70 puntos.13 Estudios en ratones han sugerido que los defectos en la neurogénesis, transmisión sináptica y vías de señalización celular podrían contribuir al problema del desarrollo a través de una inhibición excesiva de la neurotransmisión.14 Estudios en individuos con trisomía parcial de Hsa21 han sugerido diversas regiones del Hsa21 que contribuyen con esta discapacidad intelectual;15 sin embargo, estudios en ratones no confirmaron estos hallazgos.16
Existen diversos genes en la región crítica del síndrome de Down. El gen DYRK1A (21q22.13) que se expresa en el sistema nervioso en desarrollo y del adulto, su función es la inhibición de la proliferación celular y promoción de la diferenciación neuronal prematura. Estudios en ratón que sobreexpresa Dyrk1a, mostraron problemas de aprendizaje graves, así como defectos de memoria espacial.17 De igual forma, el gen SIM2 (21q22.13) ortólogo al gen Drosophila single minded, es un factor de transcripción y principal regulador del desarrollo; también se expresa en el cerebro humano en desarrollo y en ratones transgénicos que sobreexpresan Sim2, han demostrado problemas de aprendizaje leve y problemas de memoria.18 La molécula de adhesión del síndrome de Down (DSCAM, 21q22.2) se expresa en dendritas neuronales y contribuye a la plasticidad sináptica; sin embargo, inhibe la ramificación de las dendritas cuando se sobreexpresa en las neuronas del hipocampo in vitro y en el ratón con tres copias de Dscam.19 Otro gen asociado con la discapacidad intelectual es el Kcnj6 (GIRK2, 21q22.1), el cual se ha visto sobreexpresado en el hipocampo en ratón.20
También existen genes fuera de la región crítica del síndrome de Down que se han asociado al fenotipo neurológico de los pacientes con síndrome de Down. La synaptojanin1 (SYNJ1, 21q22.2) es una proteína, formadora de vesículas en la sinapsis neuronal, que juega un papel importante en la neurotransmisión desfosforilando el fosfatidilinositol bifosfato alterado en un modelo ratón con síndrome de Down que tenía problemas de aprendizaje y de memoria, que se normalizaron al reducir la dosis génica de Synj1 de tres a dos.21 Finalmente, el análisis de trisomías segmentarias confirma el papel importante de la proteína precursora amiloide (APP, 21q21.3) ya que los inhibidores de los metabolitos de esta proteína, en un modelo ratón, mejoraron su aprendizaje y memoria, sugiriendo que la triple dosis del gen APP podría ser causante del fenotipo neurológico en pacientes con síndrome de Down.22
Control motor e hipotonía
Los neonatos con síndrome de Down comúnmente presentan hipotonía y la mayoría alteraciones motoras. Los hallazgos en humanos y en modelos ratón han mostrado un número reducido de neuronas granulares en el cerebelo.23 Esta neurogénesis cerebelar reducida podría deberse a un defecto de la señalización de sonic hedgehog (SHH) en neuronas precursoras, causado por niveles elevados de APP.24 Otro gen ya mencionado es el DYRK1A, que también se propone como gen candidato para el déficit motor en estos pacientes.
Otra teoría son los defectos en la morfología de la sinapsis y en la formación de vesículas sinápticas en la unión neuromuscular. Fortaleciendo esta teoría, la sobreexpresión de los genes ITSN1 (21q22.1), SYNJ1 (21q22.2) y DSCR1 (21q22.12) en moscas transgénicas, homólogos en Drosophila, causaron defectos locomotores y falla en el reciclaje de las vesículas en la unión neuromuscular, sugiriendo que estos tres genes, junto con DYRK1A y APP, son genes candidato dosis-sensibles causantes de los defectos motores en pacientes con síndrome de Down.25
Enfermedad de Alzheimer
Los pacientes con síndrome de Down se caracterizan por presentar Alzheimer a edades tempranas y el eventual inicio de demencia. Un gen candidato importante es el ya mencionado APP, ya que su proteólisis genera amiloide β (A-β), el principal componente de las placas de amiloides en cerebros de pacientes con enfermedad de Alzheimer y cuyas mutaciones de tipo duplicaciones se han relacionado con inicio temprano de dicha enfermedad.26 Otro gen implicado, también ya mencionado, es el DYRK1A, cuyo producto ha mostrado que fosforila a la proteína APP. Un modelo ratón que sobreexpresa Dyrk1a muestra niveles elevados de fosfo-APP y A-β.27
Características craneofaciales
Microcefalia, occipital plano, braquicefalia, cara pequeña y maxilares de tamaño reducido son características del síndrome de Down. Estudios en modelos ratón han revelado un patrón craneofacial de anormalidades similares a éstas, e incluso se delimitó una región responsable de dichas características.28 Esta región contiene el gen Ets2 (ETS2, 21q22.2) cuya sobreexpresión mostró una asociación con las alteraciones esqueléticas observadas en pacientes con síndrome de Down.29
Alteraciones hemato-oncológicas
Hematológicas
Los pacientes con síndrome de Down tienen mayor riesgo de padecer leucemia (riesgo relativo de 18) y de forma particular la leucemia megacarioblástica tiene un riesgo relativo mucho mayor (de 500).30
Cabe mencionar que 10 a 20% de los pacientes con síndrome de Down desarrollan una llamada leucemia transitoria, también conocida como trastorno mieloproliferativo transitorio o mielopoyesis anormal transitoria. Ésta es una forma de leucemia casi exclusiva de los recién nacidos con síndrome de Down, la cual suele acompañarse de mutaciones en el gen del factor de transcripción hematopoyético GATA1 (Xp11.23) y aunque suele resolverse espontáneamente a los 3 meses de edad 20% de los pacientes recuperados de una leucemia transitoria desarrollan leucemia megacarioblástica en los primeros 4 años de vida y ésta siempre se acompaña de mutaciones somáticas en GATA1, lo cual indica que las mutaciones en este gen podrían ser un evento in utero y que los blastos de leucemia megacarioblástica podrían derivar de subclonas persistentes de células de leucemia transitoria como resultado de mutaciones adicionales.31 Además, estudios en humanos con distintas trisomías 21 parciales identificaron una región de 8.35 Mb en Hsa21, que involucra a los genes RUNX1 (21q22.3), ERG (21q22.2) y ETS2 (21q22.3) como candidatos para el desarrollo de leucemia megacarioblástica en síndrome de Down. Particularmente, el factor de transcripción RUNX1 está involucrado en la megacariopoyesis y mantenimiento de células troncales hematopoyéticas.12
Tumores sólidos
El riesgo para tumores sólidos es menor en pacientes con síndrome de Down. Ratones heterocigotos para mutaciones en el gen Apc (APC, 5q22.2) cruzados con modelos trisómicos para 33 genes con homología a genes humanos en el Hsa21, mostraron una clara reducción en la frecuencia de tumores en la descendencia trisómica comparada con la descendencia euploide. El gen Ets2 (ETS2, 21q22.3), incluido en esta región, inhibió el crecimiento de tumores cuando se expresaba en tres copias, a la inversa, cuando se reduce a una copia, resultó en incremento en la tasa de tumores.32
Recientemente se ha postulado que una disminución en la angiogénesis podría impedir el crecimiento de tumores en el síndrome de Down. En este contexto, DSCR1 ha mostrado inhibir la angiogénesis inducida por el factor de crecimiento vasculoendotelial. Otros genes que han disminuido la angiogénesis en modelos ratones incluyen a Adamts1, Erg, Jam2 y Pttg1ip.33
Cardiopatías congénitas
Cerca de 50% de los pacientes con síndrome de Down tiene una cardiopatía congénita.8 En uno de los estudios poblacionales más grandes las malformaciones más frecuentes fueron canal atrioventricular completo, comunicación interventricular, comunicación interatrial, tetralogía de Fallot y persistencia del conducto arterioso.34 Datos en población mexicana indican una prevalencia de cardiopatías congénitas (en el síndrome de Down) de 58%; sin embargo, en contraste con los informes a nivel mundial el canal atrioventricular completo muestra una frecuencia mucho menor que las comunicaciones interventricular e interatrial (Cuadro 2).35 En 2008, el National Down Syndrome Project of United States of America encontró que ser descendiente de hispanos confiere una oportunidad relativa de 0.48 para canal atrioventricular completo.36
Cuadro 2 Prevalencia de cardiopatías congénitas reportadas a nivel mundial y en México
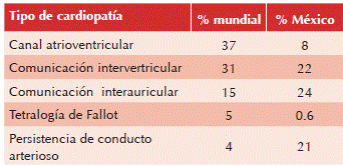
Irving C et al. Arch Dis Child.2012 Apr;97(4):326-30; de Rubens J et al. Rev Esp Cardiol.2003 Sep.56(9):894-9.
Los mecanismos genómicos implicados en esta variabilidad siguen siendo ampliamente discutidos. Debido a su alta frecuencia, comparada con individuos sin síndrome de Down y su espectro clínico variable, las cardiopatías congénitas se han utilizado como modelo en la búsqueda de loci de riesgo, mutaciones patogénicas, variación de dosis génica y variantes en número de copias (CNVs por sus siglas en ingles) que expliquen esta compleja etiopatogenia del fenotipo del síndrome de Down.37
Análisis en humanos con diferentes trisomías, 21 parciales, llevó a la identificación de una región de 1.77 Mb que contiene 10 genes. De estos, DSCAM (21q22.2) es el único que se expresa en el corazón en desarrollo, por lo que se planteó como un gen candidato; este análisis descarta genes previamente identificados como candidatos, entre ellos DIRK1A y COL6A1 (21q22.3), genes altamente expresados en las almohadillas endocárdicas a nivel atrioventricular en fetos con síndrome de Down. (12) Desafortunadamente, no se han encontrado defectos cardiacos en modelos ratones trisómicos para 33 genes, incluyendo DSCAM.
Fuera del Hsa21, mutaciones en CRELD1 (3p25.3) se han relacionado con canal atrioventricular completo en población general y se ha postulado su asociación con dicho defecto en pacientes con síndrome de Down. Otros estudios han mostrado que polimorfismos en los genes SLC19A1 (21q22.3) y MTHFR (1p36.3) involucrados en la vía del folato, están asociados con canal atrioventricular completo en pacientes con síndrome de Down.38
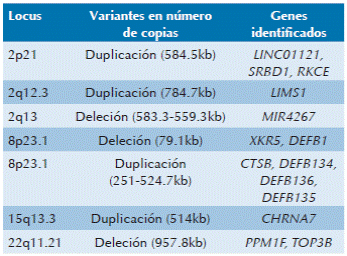
Dentro de las variantes en número de copias del cromosoma 21 asociadas con cardiopatía están algunas duplicaciones que involucran a regiones reguladoras del gen RIPK4 (21q22.2), el cual se ha asociado con morfogénesis epitelial (RR: 2.29) y del gen ZBTB21 que participa en la regulación de la vía de señalización de WNT/beta-catenina, vía requerida para la diferenciación cardiaca de células embrionarias (RR: 1.84). Además, se han identificado variantes en número de copias fuera del cromosoma 21, sobre todo regiones previamente asociadas con cardiopatías congénitas no sindromáticas. Este grupo afecta a genes del cilioma, el cual es un componente crítico de la septación atrioventricular39 (Cuadro 3).
Cuadro 3 Variantes en número de copias raras asociadas con cardiopatía congénita en síndrome de Down
Finalmente, es importante resaltar que a pesar de todos estos avances existen aún diversas manifestaciones clínicas frecuentes en paciente con síndrome de Down que aún no tienen una correlación con genes específicos, como alteraciones oftalmológicas, audiológicas, hipotiroidismo, alteraciones dermatológicas, genitourinarias como criptorquidia, hipospadias, malformaciones renales, así como la desregulación inmunológica que se asocia con infecciones recurrentes.
CONCLUSIONES
El estudio de los genes implicados en el síndrome de Down continúa siendo un tema central, ofreciendo grandes retos para su análisis. La información obtenida hasta el momento no ha permitido aclarar con exactitud los genes sensibles a dosis implicados en el fenotipo de estos pacientes, lo cual podría explicarse por el efecto de múltiples genes implicados y la compleja interacción entre ellos. Por otro lado, muchas de las manifestaciones clínicas de los pacientes con síndrome de Down se comportan como entidades multifactoriales, donde no existe un solo gen causal involucrado, sino un grupo de genes que interactúan entre sí y con el ambiente, lo cual confiere una alta complejidad a la fisiopatología subyacente en esta enfermedad.











 nueva página del texto (beta)
nueva página del texto (beta)