La soya (Glycine max) representa una de las leguminosas más importantes en el mundo, donde destacan como países productores Estados Unidos de Norteamérica, Brasil y Argentina, con producciones de 125 millones de toneladas y Argentina con 50 millones de t (FAO, 2020). La importancia de la soya en México radica en la aplicación de subproductos que tienen demanda en el ámbito alimenticio para la elaboración de harinas y aceites comestibles. En México se sembraron 155,000 ha en 2019, con una producción de 233, 000 t y un rendimiento promedio de 1.6 t ha-1 (SIAP, 2020). Sinaloa, ocupa el primer lugar en cuanto a soya cultivada en superficie de riego con 10,331 ha, con una producción de 24,718 t y un rendimiento de 2.4 t ha-1 durante el ciclo primavera-verano (SIAP, 2020). La producción y calidad de soya en el mundo se ve limitada por agentes abióticos y bióticos en estos últimos sobresalen las enfermedades.
En Estados Unidos de Norteamérica las pérdidas anuales por enfermedades de la soya se estiman en 4.5 billones de dólares (Bandara et al., 2020). Las enfermedades foliares incluyen a la mancha marrón (Septoria glycines), mancha ojo de rana (Cercospora sojina), tizón de la hoja y mancha púrpura (Cercospora kikuchii), antracnosis (Colletotrichum truncatum), tizón de la vaina y tallo (Phomopsis sojae), mildiú (Peronospora manshurica), mancha foliar por Alternaria (Alternaria spp.), mancha anillada (Corynespora cassiicola), pústula bacteriana (Xanthomonas campestris pv. glycines) y tizón bacteriano (Pseudomonas syringae pv. glycinea) (Carmona et al., 2010). A estas enfermedades se les conoce como de fin de ciclo, pues ocurren en plantas en etapa de madurez, lo cual coincide con un ambiente lluvioso y cálido (Carmona et al., 2015).
En Sinaloa se han consignado a C. sojina, C. cassiicola, C. kikuchii y P. manshurica como agentes causales de la mancha foliar de la soya (Rodríguez-Cota et al., 2019). En virtud de la alta incidencia de la enfermedad en ciclos agrícolas recientes, se realizó el presente trabajo con los siguientes objetivos: a) identificar al agente causal mediante estudios morfométricos y moleculares y b) determinar la patogenicidad de los hongos asociados a la enfermedad en lotes comerciales de esta fabácea durante el verano de 2018.
Materiales y métodos
Colecta de muestras y obtención de aislamientos monospóricos. La colecta de foliolos de plantas de soya con síntomas en el Valle del Carrizo y Valle del Fuerte, se llevó a cabo en los meses de septiembre y octubre de 2018 (Cuadro 1). Se obtuvieron muestras de ocho lotes comerciales de soya var. Nainari. Los predios variaron de 10 a 30 ha de cultivo, cinco en etapa de llenado de grano (R6) y tres en etapa de inicio de la madurez (R7) (Fehr y Caviness, 1977); en cada lote se utilizó el método de muestreo de cinco de oros para seleccionar cinco plantas de las cuales se tomaron cinco foliolos con manchas foliares de un color café claro a obscuro rodeadas de un halo amarillento. Los aislamientos de hongos asociados a foliolos sintomáticos se realizaron en agua agar (AA; Bioxon; Cuautitlán Izcalli, Estado de México, México) siguiendo procedimientos descritos por Maraite et al. (1997). Las siembras se incubaron a 25 ± 2 °C por 72 h, hasta observar crecimiento micelial. Para la purificación de los aislados, se transfirieron puntas de hifas a papa-dextrosa-agar (PDA; Bioxon; Cuautitlán Izcalli, Estado de México, México).
Cuadro 1. Sitios de colecta de aislados fúngicos de foliolos de plantas de soya var. Nainari con síntomas en el Valle del Carrizo y Valle del Fuerte.
| Muestra | |||||
|---|---|---|---|---|---|
| Aislado | Localidad | Fecha | Latitud Norte | Longitud Oeste | |
| Cv-3 | Ejido Chihuahuita, Ahome | 01/sept/2018 | 26° 9’ 56” | 109° 2’ 57” | |
| Cv-4 | Ejido Chihuahuita, Ahome | 01/sept/2018 | 26° 9’ 46” | 109° 2’ 26” | |
| Cv-5 | Poblado 5, Ahome | 01/sept/2018 | 26° 11’ 26” | 109° 3’ 24” | |
| Cv-8 | Poblado 5, Ahome | 01/sept/2018 | 26° 11’ 26” | 109° 3´43” | |
| Cv-11 | Ejido Chihuahuita, Ahome | 01/sept/2018 | 29° 9’ 12” | 109° 2’ 57” | |
| Cv-19 | El Carrizo, Ahome | 15/sept/2018 | 26° 18’ 44.43” | 108° 59’ 30.27” | |
| Cv-21 | El Carrizo, Ahome | 15/sept/2018 | 26° 18’ 1.01” | 109° 2’ 3.81” | |
| Cv-29 | Bachoco, Guasave | 02/oct/2018 | 25° 43’ 55.83” | 108° 46’ 33.98” | |
Los aislados monospóricos se obtuvieron mediante diluciones seriales, que se sembraron en AA al 1.6% y se incubaron durante 4 h; de ahí un conidio germinado se transfirió a medio PDA y las siembras se incubaron a 25 °C. Los aislados monospóricos se conservaron a 15 °C en tubos de ensayo con PDA inclinado, adicionados con aceite mineral (Flores-Córdova et al., 2013).
Características fenotípicas de colonias miceliales. Las características fenotípicas de las colonias se determinaron en PDA. Los cultivos se incubaron por 11 días a 25 °C, con un régimen de 12 h de luz y 12 h de oscuridad, y se determinó la tasa de crecimiento micelial (Kim et al., 2005) y el color de la colonia, usando colores estándares de Kelly y Judd (1976).
Morfometría de los conidios de los aislados. Fragmentos de 5 mm de diámetro se cortaron del margen de las colonias de cuatro días de edad en PDA y se transfirieron al mismo medio. La forma, número de septos, así como la longitud y ancho de 40 conidios de cada aislado se determinó a los siete días después de la siembra en el mismo medio e incubados a 25 °C. Posteriormente, se hicieron preparaciones con azul de lactofenol para medirse mediante un micrómetro instalado en un microscopio compuesto (Labmed; Lab America, Inc. Los Angeles, CA, USA).
Extracción de ADN, PCR y secuenciación. La extracción de ADN genómico se realizó a partir de 50-100 mg de micelio en cultivos en PDA de siete días, utilizando el reactivo DNAzol (Molecular Research Center Inc., Car. No. DN127, Cincinnati, OH, USA), siguiendo las instrucciones del fabricante. La calidad y concentración del ADN extraído se determinó en un espectrofotómetro NanoDrop 2000c (Thermo Scientific, Wilmington, DE). Para confirmar la identidad de los aislados, se amplificó el gen de la gliceraldehído-3-fosfato deshidrogenasa (gapdh), utilizando los iniciadores gpd1 (5’-CAA CGG CTT CGG TCG CAT TG-3’) y gpd2 (5’-GCC AAG CAG TTG GTT GTG C-3’), que amplifican un fragmento de aproximadamente 580 pares de bases (Berbee et al., 1999). La reacción de PCR se realizó en un volumen final de 50 mL, los cuales contenían 1 mL de ADN genómico, Buffer GoTaq Flexi 1X, 1.25 mM de MgCl2, 0.2 mM de cada iniciador, 0.2 mM de dNTP´s y 1 U de DNA polimerasa GoTaq Flexi (Promega, Madison, WI, USA). Las condiciones de amplificación fueron las siguientes: desnaturalización inicial a 95 °C por 2 min, 35 ciclos de desnaturalización a 95 °C por 1 min, alineamiento a 60 °C por 1 min, extensión a 72 °C por 45 s y una extensión final a 72 °C por 10 min. Los fragmentos amplificados se visualizaron en un gel de agarosa al 2%, teñido con bromuro de etidio, en un Chemidoc (BIO-RAD) y las bandas de interés fueron escindidas para su posterior purificación utilizando el kit QIAquick Gel Extraction (Qiagen, Hilden, Germany). La cuantificación de los productos de PCR purificados se realizó con un espectrofotómetro NanoDrop 2000c. Los fragmentos se secuenciaron unidireccionalmente con el iniciador gpd1 en un equipo AB3730 (Applied Biosystems, Foster City, CA, USA), en el laboratorio de servicios genómicos del Laboratorio Nacional de Genómica para la Biodiversidad (LANGEBIO).
Análisis filogenético. Las secuencias se editaron en el software BioEdit versión 7.0.5.3 (Hall, 1999) y fueron comparadas contra otras secuencias depositadas en la base de datos del GenBank, utilizando el algoritmo BlastN. Las secuencias se alinearon junto con un set de 68 secuencias de referencia del género Curvularia y una secuencia de Bipolaris maydis como grupo externo (Madrid et al., 2014; Manamgoda et al., 2015; Tomaso-Peterson et al., 2016; Mehrabi-Koushki et al., 2018; Tan et al., 2018; Kiss et al., 2019), utilizando el alineador MUSCLE (Edgar, 2004) implementado en MegaX (Kumar et al., 2018). El análisis filogenético se realizó en MegaX, empleando el método de Máxima Verosimilitud y el modelo de Hasegawa-Kishino-Yano (HKY), con distribución gamma (cuatro categorías) y sitios invariables. Todos los “gaps” fueron considerados en el análisis y la robustez de la topología se evaluó con 1000 réplicas bootstrap. Finalmente, el filograma se editó en el software FigTree versión 1.4.0 (Rambaut, 2014).
Pruebas de patogenicidad en invernadero. Para determinar la patogenicidad de los ocho aislados asociados a la enfermedad, se utilizaron plantas de soya var. Nainari, las cuales se sembraron en macetas de plástico de 5 L de capacidad con sustrato pasteurizado (arena 48.0, limo 19.4 y arcilla 32.5%, respectivamente y pH 7.0); se colocó una semilla por maceta. Las plantas se regaron según sus requerimientos hídricos y se fertilizaron semanalmente con Miracle-Gro (The Scotts Company LLC; Marysville, Ohio, USA) de acuerdo a las recomendaciones del fabricante. Se emplearon cuatro repeticiones por aislamiento (cuatro macetas con una planta).
Preparación de inóculo. Los aislados monoconidiales se desarrollaron en PDA a 25 °C, con un régimen de 12 h luz y 12 h de oscuridad durante 10 días. Los conidios se obtuvieron agregando 10 mL de agua destilada estéril a cada cultivo y raspando con una espátula estéril para desprenderlos. Las suspensiones de conidios se filtraron a través de doble malla quesera para remover el micelio (Prieto et al., 2016) y las concentraciones de inóculo de cada aislado se ajustaron de 5 x 104 a 7 x 104 conidios mL-1 mediante un hematocitómetro.
Las suspensiones conidiales se asperjaron en plantas (en etapa R6) hasta punto de escurrimiento. Las plantas testigo se asperjaron con agua destilada estéril. Posteriormente, las plantas se cubrieron durante 24 h con bolsas plásticas de 90 x 20 cm con papel absorbente saturado con agua destilada para lograr humedad relativa de 85-90% en su interior y se mantuvieron de 22 a 28 °C; posteriormente se sometieron siete días consecutivos a las mismas condiciones de humedad y temperatura durante 12 h diarias. Después, las plantas permanecieron 21 días sobre los estantes del invernadero donde la temperatura varió de 20 a 24 °C con rangos de humedad relativa de 50 a 75%. Las plantas inoculadas y testigo se distribuyeron en un arreglo completamente al azar. La patogenicidad de los aislados se determinó a los 30 días después de la inoculación. Se colectaron 10 foliolos al azar de cada planta y se determinó el porcentaje de área foliar afectada (AFA) de acuerdo a procedimientos descritos por Soares et al. (2009). Para cumplir con los postulados de Koch, al final de los experimentos se efectuaron aislamientos a partir de foliolos sintomáticos de las plantas inoculadas. La identidad de los aislados se confirmó con base en la morfología de las colonias y la morfometría de veinte conidios de cada aislado.
Análisis estadístico. Los porcentajes de AFA se transformaron usando la función arcoseno (Gómez y Gómez, 1984), previo al ANOVA. La comparación de medias se realizó mediante el procedimiento de Tukey (Little y Hills, 1973), utilizando el paquete estadístico SAS® versión 9.4 (1999). Los experimentos se condujeron en dos ocasiones. En virtud de que los análisis estadísticos indicaron interacción significativa entre los aislados y los experimentos, se presentan los resultados de los dos experimentos.
Resultados y discusión
Características culturales y morfométricas de los aislados fúngicos. Las características fenotípicas de las colonias, los conidióforos y conidios de ocho aislados obtenidos durante el periodo de muestreo (Cuadro 1) se determinaron en PDA después de ocho días de incubación. Esto permitió la ubicación de los aislados en el género Curvularia (Manamgoda et al., 2015). No se encontró el teleomorfo del hongo durante el período de muestreo, aun cuando el género Cochliobolus se ha consignado como el teleomorfo de algunas especies de Curvularia (Marin-Felix et al., 2020). La fenología de las colonias, las características de los conidióforos y morfometría de conidios de las especies de Curvularia se describen a continuación.
Aislados Cv-3, Cv-5 y Cv-8. Colonias con tasa de crecimiento micelial de 3.7 - 4.4 mm/día en PDA, de aspecto aterciopelado, gris oliváceo, con anillos concéntricos tenues (Figura 1A); reverso de las colonias con pigmentación café-olivo (Figura 1B). Conidióforos café oscuro rectos, septados, geniculados en la parte terminal (Figura 1C); conidios de 15.0 - 27.5 μm de largo por 7.5 - 15.0 μm de ancho, con 2 a 4 septos transversales, con el septo medio más voluminoso y con mayor pigmentación, curvatura pronunciada e hilio oscuro (Cuadro 2; Figura 1D). Las características morfométricas de estos aislados son similares a las consignadas para C. muehlenbeckiae (Berbee et al., 1999; Madrid et al., 2014).
Aislado Cv-4. Colonias de apariencia algodonosa a gamuza suave, con tasa de crecimiento micelial de 4.0 mm/día en PDA, de color gris obscuro con tonalidades no uniformes de café a gris claro (Figura 1E) y anverso de color café obscuro dorado (Figura 1F). Conidióforos de color café claro y geniculados en la parte superior. Conidios cilíndricos, con tres a cuatro distoseptas, elipsoides, color marrón dorado (Figura 1G y H), de 10.0 - 22.5 µm de largo, por 7.5 - 10.0 µm de ancho (Cuadro 2). Las características culturales y morfométricas son similares a las de C. spicifera (Ayoubi et al., 2017; Qostal et al., 2019).
Aislado Cv-11. Colonias color gris oliváceo, poco densas, con aspecto aterciopelada, con anillos concéntricos muy marcados (Figura 1I) y tasa de crecimiento micelial de 3.9 mm/día en PDA. Reverso de las colonias verde olivo a oscuro (Figura 1J). Conidióforos solitarios y geniculados en la parte terminal, sin ramificación (Figura 1K). Conidios de 16.2 a 27.5 µm de largo, por 6.2 a 10.0 µm de ancho (Cuadro 2), con pared lisa, elipsoides a cilíndricos, extremos redondeados, con 2 a 4 distoseptas transversales (Figura 1L), hilio protuberante (Flecha en figura 1L), coincidiendo con las características descritas para C. ahvazensis (Mehrabi-Koushki et al., 2018).

Figura 1 Morfología de especies de Curvularia en PDA asociadas a la mancha foliar de la soya. A-D) Morfología representativa de los aislados Cv-3, Cv-5 y Cv-8 (C. muehlenbeckiae); A) Anverso de colonia; B) Reverso de colonia; C-D) Conidióforos y conidios. E-H) Aislado Cv-4 (C. spicifera); E) Anverso de colonia; F) Reverso de colonia; G y H) Conidióforos y conidios. I-L) Aislado Cv-11 (C. ahvazensis); I) Anverso de colonia; J) Reverso de colonia; K-L) Conidióforos y conidios; L) Conidios. M-Q) Morfología representativa de los aislados Cv-19, Cv-21, Cv-29 (C. hawaiensis); M) Anverso de colonia; N) Reverso de colonia; O-P-Q) Conidióforos y conidios. Barra de escala = 20 µm.
Cuadro 2 Morfometría de conidios de hongos asociados a la mancha foliar de la soya var. Nainari.
| Especie/Aislado | Longitud (µm) | Ancho (µm) | Relación (L/A) (µm) | Septos/Distoseptas |
|---|---|---|---|---|
| C. muehlenbeckiae Cv-3 | 15.0-27.5x | 7.5-12.5 | 2.0 | 2.0-4.0 |
| 21.75y | 11.25 | 3.6 | ||
| 1.0z | 0.6 | 0.7 | ||
| C. spicifera Cv-4 | 10.0-22.5 | 7.5-10.0 | 2.1 | 3.0-4.0 |
| 18.75 | 9.0 | 3.9 | ||
| 1.0 | 0.5 | 0.4 | ||
| C muehlenbeckiae Cv-5 | 20.0-27.5 | 10.0-15.0 | 1.9 | 2.0-4.0 |
| 23.25 | 12.0 | 3.6 | ||
| 0.9 | 0.5 | 0.7 | ||
| C. muehlenbeckia Cv-8 | 17.5-25.0 | 7.5-15.0 | 2.0 | 3.0-4.0 |
| 22.0 | 11.0 | 3.6 | ||
| 0.7 | 0.6 | 0.5 | ||
| C. ahvazensis Cv-11 | 16.2-27.5 | 6.2-10.0 | 2.9 | 2.0-4.0 |
| 22.3 | 7.6 | 4.2 | ||
| 2.7 | 0.5 | 1.1 | ||
| Curvularia sp. Cv-19 | 10.0-22.5 | 7.5-10.0 | 2.1 | 3.0-4.0 |
| 18.75 | 9.0 | 3.9 | ||
| 1.0 | 0.5 | 0.4 | ||
| Curvularia sp. Cv-21 | 10.0-35.0 | 5.0-7.5 | 3.7 | 2.0-5.0 |
| 20 | 5.5 | 3.8 | ||
| 1.5 | 0.4 | 0.6 | ||
| Curvularia Cv-29 | 17.5-27.5 | 7.5-12.5 | 2.4 | 2.0-5.00 |
| 21.75 | 9.0 | 3.8 | ||
| 1.1 | 4.0 | 0.7 |
x El primer renglón representa la longitud máxima y mínima de las estructuras; yel segundo renglón indica la media de las 40 estructuras en cada muestra; zel tercer renglón se refiere a la desviación estándar de los datos.
Aislados Cv-19, Cv-21 y Cv-29. Colonias con tasa de crecimiento micelial de 2.8 - 4.3 mm/día en PDA, con apariencia de gamuza suave, con anillos concéntricos poco definidos y penachos blanquecinos (Figura 1M), reverso marrón obscuro a negro (Figura 1N). Los conidióforos son erectos a flexuosos, septados, a menudo geniculados (Figura 1O). Conidios de 10 a 35 µm de largo por 5 a 12.5 µm de ancho, elipsoidales o redondeados en los extremos, marrón pálido a marrón oscuro, 2 a 5 distoseptas (generalmente 4), pared conidial lisa (Cuadro 2; Figura 1P y Q). Las características fenotípicas de las colonias, así como la morfometría de los conidios son similares a las de especies filogenéticamente relacionadas a C. hawaiensis (Manamgoda et al., 2015).
Identificación molecular. Las secuencias parciales del gen gapdh fueron depositadas en la base de datos GenBank (acceso MT680146-MT680150 y MT680152-MT680154). Los análisis confirmaron la identidad de los aislados a nivel género. A su vez, la comparación de las secuencias en la base de datos del GenBank mostró altos porcentajes de similitud con diversas especies de Curvularia; los aislados Cv-3, Cv-5 y Cv-8 fueron similares a C. muehlenbeckiae cepa UTHSC 08-2905 (No. Acc. LT715807; 99% de identidad) y CBS 144.63 (No. Acc. LT715806, 100% de identidad); el aislado Cv-4 fue similar a C. spicifera cepa CBS 198.31 (No. Acc. LT715814.1, 99% de identidad); el aislados Cv-11 registró altos porcentajes de similitud con las especies C. hawaiiensis cepa CBS 173.57 (No. Acc. LT715812, 99% de identidad), C. nodosa cepa CPC 28812 (No. Acc. MF490840, 98.5% de identidad), C. perotidis cepa CBS 350.90 (No. Acc. LT715813, 97.9% de identidad) y C. dactyloctenii cepa BRIP 12846 (No. Acc. KJ415401, 98.5% de identidad); mientras que los aislados Cv-19, Cv-21 y Cv-29 compartieron altos porcentajes de similitud con las especies C. beasleyi cepa BRIP 15854 (No. Acc. MH433639, 99.1% de identidad), C. dactyloctenii cepa BRIP 12846 (No. Acc. KJ415401, 98.9% de identidad), C. hawaiiensis cepa CBS 173.57 (No. Acc. LT715812, 98.7% de identidad), C. nodosa cepa CPC 28812 (No. Acc. MF490840, 98.5% de identidad) y C. perotidis cepa CBS 350.90 (No. Acc. LT715813, 97.9% de identidad).
La Figura 2 muestra el filograma inferido a partir de la secuencia parcial del gen gapdh. Los aislados Cv-3, Cv-5 y Cv-8 se agruparon junto con la secuencia tipo de C. muehlenbeckiae, con alto soporte bootstrap (90.9%), lo cual concuerda con la descripción morfológica de estos aislados para la especie en cuestión (Figura 1A-D). El aislado Cv-4 fue identificado como C. spicifera ya que se agrupó con la secuencia tipo de esta especie (bootstrap 94.1%) y sus características morfológicas coinciden con las ya reportadas. El aislado Cv-11 se agrupa con la especie C. ahvazensis (bootstrap de 52.1%), y sus características morfológicas coinciden con las reportadas para esta especie. El filograma muestra que los aislados Cv-19, Cv-21 y Cv-29 son diferentes y no corresponden a ninguna de las especies de Curvularia utilizadas como referencia en el análisis, por lo que se designaron como Curvularia sp. Los tres aislados son filogenéticamente cercanos a C. beasleyi especie con las que comparten porcentajes de similitud de 99 - 99.2%.
Este grupo de hongos posee una alta complejidad morfológica y diversidad de especies crípticas, por lo que su taxonomía no está completamente resuelta (Manamgoda et al., 2015). Actualmente, el género comprende poco más de 100 especies descritas, de las cuales solo ~80 han sido aceptadas y se ha establecido su ubicación taxonómica mediante análisis filogenético multigen (Manamgoda et al., 2015; Tan et al., 2018; Kiss et al., 2019); sin embargo, el gen gapdh por si solo puede resolver las especies de Curvularia (Manamgoda et al., 2015), y es el locus con mayor información filogenética entre los loci analizados para este género fúngico (Ferdinandez et al., 2019). En el caso de los aislados Cv-19, Cv-21 y Cv-29, cuya identidad no se pudo determinar, se recomienda realizar una caracterización molecular más detallada, para corroborarlos como un nuevo linaje dentro del género y asignarles un nombre. Para ello, se deberían incluir la secuenciación de la región ITS y/o el gen del factor de elongación de la traducción (TEF), como se ha realizado previamente en el registro de nuevas especies de Curvularia (Heidari et al., 2018; Mehrabi-Koushki et al., 2018).
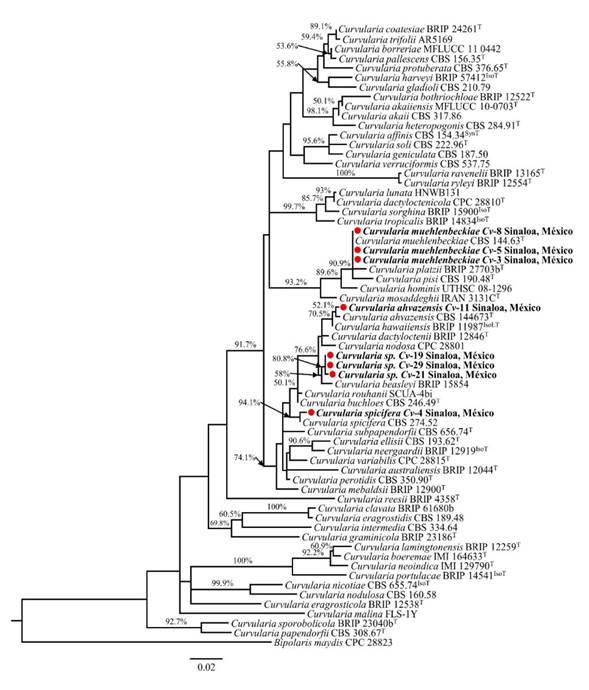
Figura 2 Árbol filogenético del gen de gliceraldehído-3-fosfato deshidrogenasa (gapdh), inferido por Máxima Verosimilitud (lnL -4286.90) en MEGA X. La escala representa el promedio de sustituciones por sitio. Los valores Bootstrap ≥70% se presentan sobre las ramas, cerca de los internodos. Las especies encontradas en este estudio se muestran en negritas, precedidas de un círculo rojo. ATCC, American Type Culture Collection, Virginia, USA; BRIP, Queensland Plant Pathology Herbarium, Brisbane, Australia; CBS, Westerdijk Fungal Biodiversity Institute, Utrecht, the Netherlands; MFLUCC, Mae Fah Luang University Culture Collection, Chiang Ria, Thailand; UTHSC, Fungus Testing Laboratory, Department of Pathology at the University of Texas Health Science Center, San Antonio, Texas, USA; CPC, Culture Collection of Pedro Crous, Westerdijk Fungal Biodiversity Institute; IMI, International Mycological Institute, CABI-Bioscience, Egham, Bakeham Lane, UK. ET , IsoT , IsoLT , SynT y T indican cepas ex-epitipo, ex-isotipo, ex-isolectotipo, ex-sintipo y ex-tipo, respectivamente.
Prueba de patogenicidad. Todos los aislados obtenidos de foliolos de soya sintomáticos, resultaron patogénicos en soya var. Nainari y causaron síntomas similares a los observados en campo (Figura 3). Los primeros síntomas ocurrieron en el tercio basal y parte media de las plantas seis días después de la inoculación, mientras que la defoliación inició 15 días después de la inoculación; a su vez, los síntomas causados por las distintas especies de Curvularia fueron similares, pero con variación en el grado de virulencia. En el experimento 1, a los treinta días después de la inoculación, las plantas inoculadas con los aislados de C. ahvazensis (Cv-11) y Curvularia sp. (Cv-21), presentaron un AFA de 30%, sin diferencias (F=73.3; P<0.0001) entre estas especies, pero si las hubo con respecto a los aislados Cv-3 y Cv-8 de C. muehlenbeckiae y los aislados Cv-19 y Cv-29 de Curvularia sp., que causaron un AFA de 10%. Los aislados de C. spicifera (Cv-4) y C. muehlenbeckiae (Cv-3 y Cv-5), fueron los menos virulentos pues causaron un AFA de 3 a 9% (Cuadro 3). En el experimento 2 el porcentaje de AFA causado por los aislados de C. ahvazensis (Cv-11) y Curvularia sp. (Cv-21) fue de 24.0 y 19.0%, respectivamente, con diferencias (F=27.0; P<0.0001) entre estos aislados y el resto de ellos pertenecientes a diversas especies de Curvularia, las cuales causaron de 5.0 a 15.0% de AFA. En los dos experimentos las plantas testigo asperjadas con agua destilada permanecieron asintomáticas durante el desarrollo del trabajo (Figura 1B).

Figura 3 A) Síntomas de mancha foliar en foliolo de planta de soya a partir de infecciones naturales en campo; B) Foliolos de planta testigo sin inocular; C) Foliolo con síntomas, característicos de la mancha foliar de la soya, causados por especies de Curvularia (aislado Cv-11) en inoculaciones en el invernadero.
Cuadro 3 Porcentaje de área foliar afectada en plantas de soya variedad Nainari, inoculadas con aislados de diferentes especies de Curvularia.
| Especie/Aisladox | Experimento 1 | Experimento 2 |
|---|---|---|
| C. ahvazensis Cv-11 | 30.0 az | 24.0 az |
| Curvularia sp. Cv-21 | 30.0 a | 19.0 ab |
| C. muehlenbeckiae Cv-8 | 21.0 b | 15.0 bc |
| Curvularia sp. Cv-19 | 10.0 c | 8.0 d |
| Curvularia sp. Cv-29 | 10.0 c | 11.0 cd |
| C. muehlenbeckiae Cv-3 | 9.0 c | 9.0 cd |
| C. spicifera Cv-4 | 9.0 c | 10.0 cd |
| C. muehlenbeckiae Cv-5 | 3.0 d | 5.0 de |
| Testigo sin inoculación | 0.0 d | 0.0 e |
| CV | 29.8 | 39.2 |
xLa patogenicidad de los aislados se evaluó a los 30 días después de la inoculación.
zMedias con la misma letra en cada columna no son diferentes en la prueba de Tukey (p≤0.05).
Para cumplir con los postulados de Koch, las especies de Curvularia inoculadas se re-aislaron de las plantas sintomáticas y su identidad se confirmó mediante la comparación de las características fenotípicas de las colonias y morfometría de 20 conidios con los aislados originales.
Se demostró que las especies de Curvularia antes señaladas causan la mancha foliar de la soya, lo cual contrasta con reportes en Sinaloa, donde se mencionan a Corynespora casiicola, Cercospora sojina y Cercospora kikushii como agentes causales de la enfermedad (Rodríguez-Cota et al., 2019), al igual que en Brasil (Teramoto et al., 2017), Estados Unidos de Norteamérica (Sumabat et al., 2018) y Argentina (Reznikov et al., 2018). Aun cuando los síntomas causados por las especies de Curvularia en el presente estudio son similares a los causados por otros hongos (Stewart y Rodríguez, 2016; Mwase y Kapooria, 2000), éstos no se encontraron durante el periodo de muestreo en el presente trabajo. A las enfermedades foliares de origen fungoso en la soya, que ocurren en la etapa de llenado de grano se les denomina enfermedades de fin de ciclo (EFC), pues se presentan en estadios intermedios a avanzados del cultivo (De Lisi et al., 2015). En virtud de que la mancha foliar de la soya causada por C. spicifera, C. muehlenbeckiae, C. ahavenzis y Curvularia spp. en Sinaloa se presenta a partir del llenado de grano, también deben ubicarse como EFC. Esta enfermedad ocurrió durante septiembre y octubre, cuando la precipitación varió de 321 a 373 mm en la región y la temperatura osciló entre 26 y 39 °C (Estación Meteorológica, Junta Local del Valle del Carrizo, Sinaloa, 2019). En otras regiones productoras de soya se han registrado como agentes causales de EFC a Colletotrichum truncatum, Septoria glycines, Phomopsis sojae, Peronospora manshurica y Alternaria spp. (Carmona et al., 2015), pero estos patógenos no se detectaron durante el desarrollo del presente trabajo. Estudios en Argentina indican que las EFC reducen de 8 a 10% el rendimiento de la cosecha, con un máximo de hasta 30% (Carmona et al., 2004); en este sentido, se justifica el desarrollo de investigación en Sinaloa para determinar el impacto de la enfermedad en el rendimiento y calidad de la cosecha, y de ser necesario implementar estrategias para su control.
Conclusiones
Los hongos Curvularia spicifera, C. muehlenbeckiae, C. ahvazensis y Curvularia spp., se encontraron asociados a la mancha foliar de la soya la cual ocurrió a partir de la etapa de llenado de grano (R6). La identidad de las especies se realizó mediante el análisis de sus características morfométricas y reconstrucción filogenética basada en un fragmento del gen gapdh. Las especies de Curvularia resultaron patogénicas en soya var. Nainari y mostraron diferentes grados de virulencia. Este es el primer reporte en México implicando especies de Curvularia en la etiología de la mancha foliar de la soya.











 texto en
texto en 


