Los fitoplasmas son bacterias no cultivables carentes de pared celular pertenecientes a la clase Mollicutes (Weisburg et al., 1989), son responsables de cientos de enfermedades en plantas cultivadas y arvenses en todo el mundo (Lee y Gundersen-Rindal, 2000). Estos patógenos están restringidos al floema, se transmiten por injerto y mediante insectos vectores (Weintraub y Beanland, 2006) y semilla (Rojas-Matínez et al., 2009). Los fitoplasmas infectan sistémicamente a sus hospedantes al moverse a través de los poros de las placas del floema distribuyéndose por todo su sistema vascular (Lee y Gundersen-Rindal, 2000). A la fecha, estos microorganismos no se cultivan en un medio libre de células, lo cual sugiere que tienen un metabolismo más reducido que otros mollicutes como se ha evidenciado en los genomas de fitoplasmas que a la fecha se han secuenciado (Oshima et al., 2004).
Los fitoplasmas inducen una variedad de síntomas entre los que se incluyen, amarillamientos, retraso del crecimiento, esterilidad de las flores, necrosis, escoba de bruja, filodia y virescencia entre otros (Rojas et al., 2013). Estos tres últimos síntomas sugieren que los fitoplasmas interfieren con el metabolismo de las hormonas vegetales (Weintraub y Bealand, 2006). La severidad de los síntomas depende del aislamiento, la edad de la planta y el momento en que ocurre la infección.
La interacción de los fitoplasmas con sus insectos vectores es compleja e implica su replicación intra y extracelular en el intestino, glándulas salivales, tejidos epiteliales, musculares y otros órganos. La infección sistémica de los fitoplasmas dentro del insecto puede tomar diez días o más dependiendo del grupo, la especie de insecto y la temperatura (Sugio y Hogenhout, 2012). Considerando que hay evidencia de que algunos fitoplasmas se transmiten verticalmente en sus insectos vectores (Weintraub y Bealand, 2006), los medios predominantes de supervivencia más efectiva es por esta vía. Los insectos que pueden ser vectores de estos patógenos pertenecen principalmente a las familias Cicadellidae, Fulgoridae y Psilidae en menor grado (Weintraub y Bealand, 2006).
A la fecha se tienen totalmente secuenciados cuatro genomas de fitoplasmas entre ellos el causante del amarillamiento del áster (Candidatus Phytoplasma asteris) quien tiene una amplia gama de hospedantes y son transmitidos por diferentes insectos polífagos. Diversos factores contribuyen a la reducción del genoma de fitoplasmas, incluidos tamaños poblacionales pequeños, asexualidad, un sesgo mutacional que favorece las eliminaciones frente a las inserciones, un entorno de crecimiento metabólicamente rico y, por último, la ausencia de flujo del genoma de otras fuentes debido tanto el ambiente intracelular restringido como la incapacidad de incorporar ADN extraño por recombinación (Bai et al., 2006). A pesar de que los fitoplasmas están sujetos a la mayoría de estos factores, no están restringidos a un único insecto y las poblaciones pueden consistir en múltiples variantes de fitoplasma (Weintraub y Bealand 2006). En particular, los fitoplasmas que colonizan muchas plantas e insectos son más propensos a encontrar otros fitoplasmas y organismos que constituyen fuentes para la adquisición de elementos genéticos.
Debido a la importancia económica de estos patógenos y al poco conocimiento que se tiene de las especies de insectos vectores en México, es necesario conocer las plantas arvenses y/u ornamentales con potencial para ser reservorios de estos patógenos y contribuir a su conocimiento. Por lo que la presente investigación tuvo como objetivo, identificar el fitoplasma asociado a la filodia en margarita (Dimorphotheca sinuata).
Extracción de ADN. Se realizó a partir de hojas en cinco plantas con síntomas de filodia, (Figura 1), la toma de muestras fue dirigida a plantas que presentaban este síntoma; así como de dos plantas asintomáticas como testigo. El método que se utilizó para la extracción de ADN fue el reportado por Dellaporta et al. (1983) con algunas modificaciones.
Detección de fitoplasmas por PCR. Se realizó PCR anidada con los iniciadores universales P1 (Deng y Hiruki, 1991) y P7 (Kirkpatrick et al., 1994) para fitoplasmas que amplifican un fragmento de 1800 pb (primera amplificación) en un volumen final de 25 µL que contenía: 1 X de amortiguador para PCR (10 X, 100 mM tris-HCl, 500 mM KCl, pH 8.3) (Invitrogen®), 0.2 mM de cada dNTP, 1.5 mM de MgCl2 (Invitrogen®), 10 pmol de cada iniciador (Sigma-Aldrich®), 1 U de ADN polimerasa (Invitrogen®) y 100 ng de ADN molde. La amplificación fue realizada en un termociclador Techne®. TC-300 con el siguiente programa: desnaturalización a 94 °C por 5 min, seguido por 35 ciclos de 94 °C por 1 min, 55 °C por 2 min, 72 °C por 3 min con una extensión final de 72 °C por 5 min. La segunda amplificación (anidada) se realizó con los iniciadores R16F2n / R16R2 (Gundersen y Lee, 1996) que amplifican un fragmento de 1200 pb de la región 16S rADN de los fitoplasmas. Como ADN molde se utilizó el producto amplificado de la primera reacción de PCR diluido en agua estéril libre de nucleasas (1:20) usando las mismas concentraciones y reactivos que en la amplificación directa, con el siguiente programa: desnaturalización a 94 °C por 2 min, seguido de 35 ciclos de 94 °C por 1 min, 58 °C por 2 min, 72 °C por 3 min y una extensión final a 72 °C por 10 min.

Figura 1 A-C. Plantas de margarita exhibiendo síntomas de filodia y proliferación de brotes, (D) planta asintomática.
Secuenciación y análisis filogenético. El producto obtenido de PCR se purificó y secuenció (Macrogen Inc. Corea) en ambas direcciones. Las secuencias obtenidas se analizaron, depositaron y compararon en la base de datos del Centro Nacional para la Información Biotecnológica (NCBI, 2016) (http://blast.ncbi.nlm.nih.gov/Blast.cgi) utilizando la herramienta BLAST. Con la secuencia obtenida se realizó un análisis filogenético con otras especies de fitoplasmas (Figura 3).
En todas las muestras con síntomas se obtuvo el fragmento esperado de 1200 pb para fitoplasmas (Figura 2A). Las secuencias obtenidas (No. acceso MK278895 y MK278896) tuvieron 99% de similitud con Candidatus Phytoplasma asteris grupo 16srI-B obtenido de Vitis vinifera en Irán (MH638316.1). El análisis de RFLP in vitro (Figura 2B) indicó que el fitoplasma de este estudio pertenece al grupo del amarillamiento del áster, reconocido ahora como Candidatus Phytoplasma asteris.
El uso de enzimas de restricción para identificar grupos de fitoplasmas siguen siendo útiles ya que los perfiles electroforéticos que genera cada una de las enzimas son un indicativo de la variación presente en su genoma por lo que estos patrones se convierten en una huella genética. En la mayoría de las investigaciones, las enzimas que más se utilizan con este propósito son MseI (Tru 91), AluI, RsaI, Hha, HpaII y HpaIII (De Oliveira et al., 2011). Los fitoplasmas del amarillamiento del áster constituyen un grupo cosmopolita que se encuentra en la mayoría de las plantas en condiciones naturales, debido a su plasticidad para adaptarse a muchos hospedantes, por lo que pueden llegar a constituirse eventualmente en un serio problema (Lee et al., 2000). Se conoce que la mayoría de los síntomas que inducen los fitoplasmas son los mismos en diferentes especies y que en un cultivo o en una planta se puede encontrar más de un fitoplasma. Debido a que la sintomatología es la misma, se puede hacer caso omiso del potencial infectivo que puedan tener algunos de ellos y diseminarse a cultivos de interés agronómico y, por lo tanto, causar un problema económico. En México la mayoría de los estudios están enfocados en la detección de estos patógenos y, en pocos casos como el amarillamiento letal del cocotero, se ha trabajado con mejoramiento genético y reemplazo de material en las zonas seriamente afectadas debido al impacto de la enfermedad. Otra enfermedad de impacto económico en un cultivo ocasionada por un fitoplasma es el engrosamiento del cladodio del nopal al reducir de manera considerable la producción de tuna en todas las zonas productoras (Suaste et al., 2012). En los últimos años se ha observado la presencia de fitoplasmas en plantas de ornato que se introducen a nuestro país (Rojas et al., 2017) debido a que exhiben abundantes proliferaciones y, en algunas ocasiones, flores verdes que resultan muy llamativas para los consumidores; esta situación puede favorecer la aparición de enfermedades emergentes. Por otro lado, la literatura indica que los síntomas pueden aparecer una semana después de la inoculación del fitoplasma; sin embargo, esto depende de las condiciones de temperatura y de la especie vegetal, la severidad de los síntomas varía según la edad de la planta, aislamiento de fitoplasma y el tiempo de infección (Weintraub y Bealand 2006). Asimismo, en ocasiones se detecta al fitoplasma en plantas con síntomas que en su periodo de desarrollo no exhibieron ninguna alteración (Rojas et al., 1999), algunos fitoplasmas son adquiridos y transmitidos exclusivamente por una especie de insectos; sin embargo existen otros que son trasmitidos por distintas especies. Desde el punto de vista epidemiológico la transmisión de fitoplasmas por insectos es particularmente importante si éstos son polífagos como es el caso de los cicadélidos quienes constituyen además un importante reservorio de estos patógenos (Weintraub y Bealand 2006).
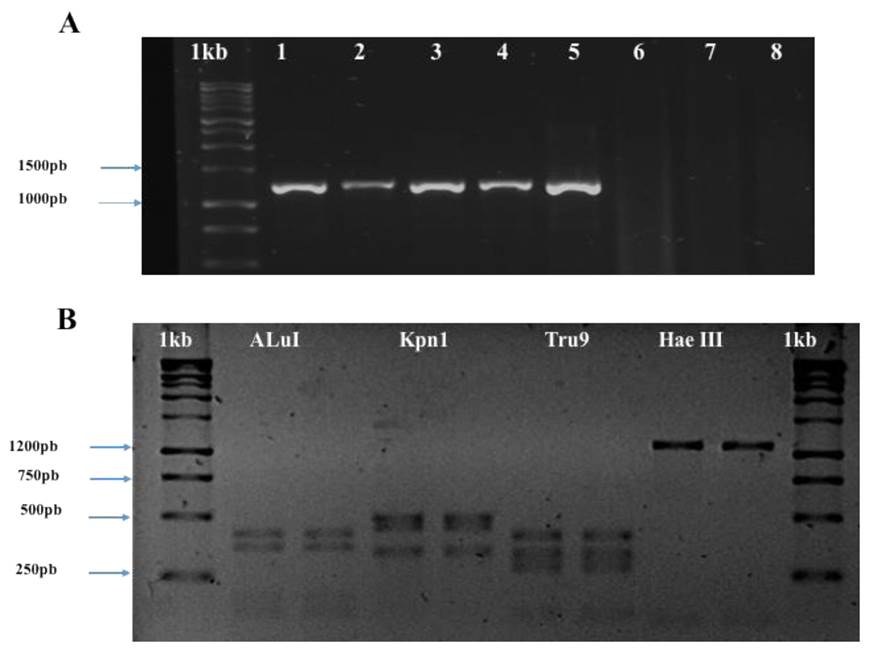
Figura 2 (A) Productos de PCR obtenidos con los iniciadores R16F2n/R16R2. Gel de agarosa al 1%. Carril 1-5, muestras de margarita con síntomas de filodia; Carril 6 y 7, muestras asintomáticas; Carril 8, Control negativo (agua); Carril 1kb, Marcador molecular 1kb (Promega®). (B) Digestión de los productos de PCR anidada con enzimas de restricción. Carril 1 y 10 Marcador 1kb (Promega®), Carril 2 y 3 producto de digestión de la enzima AluI; Carril 4 y 5 digestión con la enzima Kpn1; Carril 6 y 7 producto de la digestión con la enzima Tru9I; Carril 8 y 9 productos de digestión con la enzima Hae III.
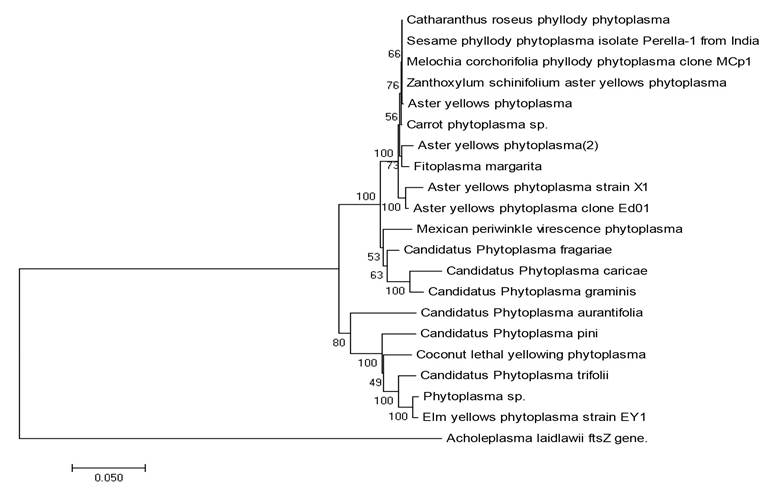
Figure 3 Phylogenetic tree based on ribosomal DNA 16S of phytoplasma sequences available in the NCBI. Acholeplasma laidlawii, a that can be cultivated in vitro and that is not genetically related to phytoplasmas data base, was consid ered as a root. The tree was created using p CLUSTAL_X with the neighbor-joining method with 500 repetitions.
Los fitoplasmas tienen un genoma metabólicamente limitado con elementos móviles que contienen información para codificar diferentes efectores que son factores de transcripción que modulan diversas respuestas en la planta (Bai et al., 2006). Resultado de lo anterior, se originan alteraciones como la escoba de bruja; así como cambios en la estructura y color de las hojas o la supresión de la respuesta de defensa de la planta a los insectos vectores de estos patógenos (Cettul y Fierrao, 2011). Por último, es conveniente realizar investigación relacionada con los insectos que colonizan esta planta de ornato ya que se encuentra ampliamente distribuida en el país, particularmente aledaña a huertas de durazno, ciruela, nopal, haba, frijol y calabaza entre otros.
Con la detección de Dimorphotheca sinuata como un nuevo hospedante de Candidatus Phytoplasma asteris se amplía el conocimiento de la gama de hospedante alternos para este grupo de fitoplasmas y lo convierte en una fuente de inóculo potencial para otros cultivos de interés económico hospedantes de este fitoplasma, como lo son la avena donde el subgrupo 16SrI-B induce serios daños a este cultivo como, proliferación anormal y amarillameinto de espigas, estériles, retraso de crecimiento y producción de semillas estériles (Urbanavičienė et al., 2006). En esta especie ornamental (Dimorphotheca sinuata), solo se tiene el reporte de un fitoplasma perteneciente al grupo 16SrIX, causando retraso de crecimiento, filodia y virescencia en Italia (Marcone et al., 2001).
Los síntomas de filodia y proliferación de margarita (Dimorphotheca sinuata) se asociaron a la infección de Candidatus Phytoplasma asteris, relacionado filogenéticamente al grupo 16Sr1-B. Se reporta por vez primera a Dimorphotheca sinuata como un nuevo hospedante de Candidatus Phytoplasma asteris.











 texto en
texto en 


