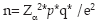La salmonelosis transmitida por alimentos es una de las principales preocupaciones de salud pública a escala global.1-3 La carne de diferentes especies, entre ellas la de bovino, es un importante vehículo de transmisión de Salmonella,4-6 de la cual se han identificado más de 2 500 serotipos,7 algunos de los cuales tienen predominancia en ciertas especies animales o zonas geográficas. Además, algunas cepas de este patógeno han mostrado una creciente resistencia a antimicrobianos, lo que constituye un gigantesco desafío para las autoridades de salud. De ahí que la tipificación de aislamientos sea de gran utilidad para entender la epidemiología de este agente infeccioso y, por tanto, desarrollar medidas preventivas para minimizar los riesgos asociados con el mismo.
Desafortunadamente, la frecuencia con la que se presentan determinados serotipos de Salmonella spp., así como su perfil de resistencia a antibióticos en distintas especies animales, suele variar espacial y temporalmente.1-8 En México, las investigaciones más recientes sobre carne de res fueron realizadas entre cinco y 13 años atrás.9-13 Por tanto, resulta necesario generar información actualizada que contribuya a evaluar el riesgo a la salud pública derivado de la presencia de Salmonella spp. en carne de bovino, que es el segundo tipo de carne más consumido en el país. Por ello, el presente trabajo tiene por objetivo generar información sobre los principales serotipos y perfiles de resistencia a antibióticos de las cepas de Salmonella spp. asociadas con la carne de res que se expende en la capital mexicana.
Material y métodos
Este trabajo no requirió de la aprobación del Comité Institucional para el Cuidado y Uso de Animales de Experimentación (CICUAE), de la Facultad de Medicina Veterinaria y Zootecnia de la Universidad Nacional Autónoma de México. Lo anterior en virtud de que en el estudio no se emplearon animales de experimentación.
Diseño del experimento
En este experimento se realizó un muestreo por conveniencia en la Ciudad de México, por ser el principal centro de consumo del país y donde llega carne de varios estados de la República. La unidad muestral consistió en 100-200 gramos de carne molida de res, expedida en supermercados, carnicerías, mercados públicos y mercados sobre ruedas (tianguis).
Muestreo
Para determinar el número de lugares a muestrear, se utilizó la fórmula para determinar el tamaño de muestra para una proporción de una población, cuando no conocemos el número de elementos en esa población:14
donde:
n= tamaño de la muestra
Zα 2= 1.962 (ya que el nivel de confianza es de 95%).
p= proporción esperada (como no tenemos idea de dicha proporción, utilizamos el valor p=0.5 (50%) que maximiza el tamaño de muestra)
q= 1-p
e= precisión (en este caso se fijó en 10%)
Con base en la fórmula anterior, el tamaño de muestra fue de 96, el cual se redondeó a 100. De éstas, la mitad se tomó en supermercados (n=50) e igual número en carnicerías, mercados públicos y tianguis. Los supermercados pertenecen a las principales cadenas de tiendas que conforman los sistemas de distribución formal de carne refrigerada.15 A su vez, los mercados públicos, mercados sobre ruedas (tianguis) y carnicerías forman parte de los sistemas de distribución informal de carne caliente.15
Los lugares para la toma de muestras se seleccionaron al azar, utilizando números aleatorios generados en Microsoft Excel. En el caso de los supermercados, se seleccionaron cuatro cadenas comerciales pertenecientes a la Asociación Nacional de Tiendas de Autoservicio y Departamentales (ANTAD). El número de puntos de venta a muestrear por cadena comercial se determinó con base en la probabilidad proporcional al tamaño (número de tiendas) en cada una de éstas. Los lugares para la toma de muestras también fueron escogidos al azar. El muestreo se llevó a cabo entre abril y noviembre de 2013. Las muestras se colocaron en hieleras con geles refrigerantes para su transporte al laboratorio (2-4 °C) y fueron procesadas dentro de las siguientes cuatro horas.
Análisis microbiológico
Para la preparación de las muestras, se siguió el procedimiento descrito en la Norma Oficial Mexicana NOM-110-SSA1-1994,16 que describe la preparación y dilución de muestras de alimentos para su análisis microbiológico. Asimismo, el aislamiento microbiológico se realizó de acuerdo con la Norma Oficial Mexicana NOM-114-SSA1-1994,17 según el cual se deben seguir los pasos de preenriquecimiento, enriquecimiento selectivo, aislamiento selectivo diferencial e identificación bioquímica.
Confirmación molecular
Para la confirmación molecular de la Salmonella, se determinó la presencia del gen invA (284 pb), característico de Salmonella spp. y relacionado con su capacidad invasiva. Se usaron iniciadores específicos para Salmonella, utilizados anteriormente por Rahn y colaboradores.18 La reacción con estos iniciadores fue llevada a cabo en 2.5 μL de buffer 10X para PCR (500 mM KCL, 200 Mm Tris HCL), 1.25 μL de dNTPs (10Mm), 1.6 μL de MgCl2, 0.5 μL de Taq DNA polimerasa (Fermentas) y 1.5 μL de la extracción de cada aislado. La PCR se corrió bajo las siguientes condiciones: incubación inicial a 94°C por 60 segundos; seguida por 35 ciclos de desnaturalización a 94°C por 60 segundos; alineación a 64°C por 30 segundos; finalmente una amplificación a 72°C por 30 segundos, seguida de un periodo de extensión final de 7 minutos a 72°C. La amplificación de los productos de PCR fue analizada usando geles con 1.2% de agarosa, teñidos con bromuro de etidio y visualizados con un transiluminador.
Serotipificación
La serotipificación se realizó de acuerdo con el esquema de Kauffmann-White,7 para lo cual los aislamientos fueron enviados al Instituto de Diagnóstico y Referencia Epidemiológicos (InDRE) de la Secretaría de Salud.
Resistencia a antibióticos
Para determinar la sensibilidad de los aislamientos a antibióticos, se utilizó la prueba de difusión en discos (sensidiscos individuales BIO-RAD) mediante el Método de Kirby-Bauer.19 Los resultados se reportaron de acuerdo con los criterios de interpretación propuestos por el Clinical & Laboratory Standards Institute (CLSI).20 Se probaron 14 antibióticos: ampicilina (Am; 10 μg), amoxicilina-ácido clavulánico (Ax; 30 μg), amikacina (Ak; 30 μg), ceftriazona (Cro; 30 μg), cefalotina (Cf; 30 μg), cefotaxima (Ctx; 30 μg), cloranfenicol (Chl; 30 μg), pefloxacina (Pef; 5 μg), gentamicina (Ge; 10 μg), nitrofurantoína (Nf; 30 μg), netilmicina (Net; 30 μg), trimetoprim sulfametazol (Stx; 250 μg), carbenicilina (Cb; 100 μg) y tetraciclina (Tet; 30 μg). Se utilizaron como organismos de control de calidad: Escherichia coli ATCC 8739, Enterococcus faecalis ATCC 29212, Staphylococcus aureus ATCC 25923 y Pseudomonas aeruginosa ATCC 9027. Éstos fueron donados por el laboratorio de Microbiología General de la Escuela Nacional de Ciencias Biológicas (ENCB) del Instituto Politécnico Nacional de México. Las cepas fueron consideradas multirresistentes cuando fueron resistentes a tres grupos de antibióticos o más. Se utilizaron las herramientas de análisis de datos de Microsoft Excel para elaborar la estadística descriptiva. Para el análisis de la relación entre de la frecuencia de muestras positivas a Salmonella y los distintos puntos de venta, se utilizó la prueba de ji cuadrada (χ2), con un nivel de significancia de 95%.
Resultados
Se encontró una frecuencia relativa promedio de Salmonella spp. en carne de res molida de 16% en la muestra estudiada. No obstante, en los supermercados, el porcentaje de muestras positivas fue del 6% (3/50); mientras que en el sector informal fue del 32% (16/50): 2 de tianguis, 4 de carnicerías y 10 de mercados públicos (Cuadro I). La prueba de ji cuadrada evidenció que el porcentaje de muestras positivas a Salmonella spp. estuvo asociado significativamente (P<0,05) con el tipo de mercado (formal o informal) en el que se expende la carne.
Cuadro I Frecuencia relativa de Salmonella spp. y serotipos identificados en muestras de carne de res molida de la Ciudad de México. Abril-noviembre 2013

* Serotipo no determinado en aislamientos con resultados no concluyentes
Los serotipos predominantes fueron Lomita y Derby, aunque también se encontraron cepas de Senftenberg, Javiana y Cannstatt. Cinco de los aislamientos tuvieron resultados no concluyentes, por lo que se reportan como no serotipificados.
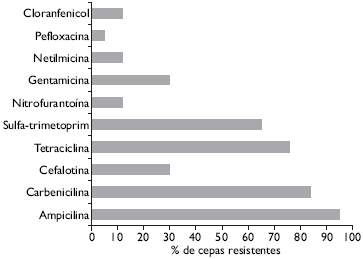
En materia de susceptibilidad a antibióticos (Cuadro II), las resistencias más frecuentes se presentaron en β-lactámicos; mientras que 73.7% (14/19) de los aislamientos resultaron multirresistentes. Como se muestra en la Figura 1, entre 70 y 95% de las cepas presentaron resistencia a ampicilina (18/19), carbenicilina (16/19), tetraciclina (13/19) y trimetoprim-sulfametoxasol (13/19). En los demás antibióticos, el porcentaje de cepas resistentes fue mucho más bajo (entre 5 y 30%).
Cuadro II Perfil de resistencia a antibióticos de los serotipos de Salmonella spp. aislados de carne molida de res en la Ciudad de México. Abril-noviembre 2013
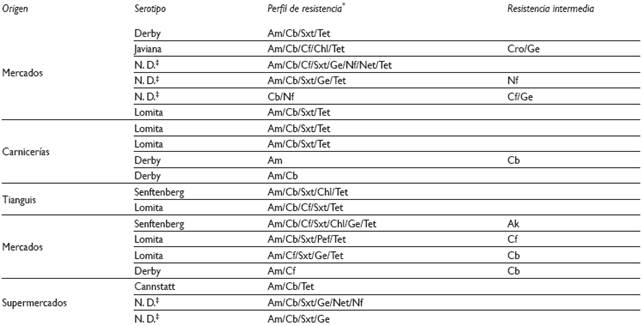
* Am: Ampicilina; Cb: Carbenicilina; Tet: Tetraciclina; Sxt: Sulfa-trimetoprim; Cf: Cefalotina; Nf: Nitrofurantoína; Ge: Gentamicina; Pef: Pefloxacina; Chl: Cloranfenicol; Net: Netilmicina; Cro: Ceftriaxona; Ak: Amikacina
‡ ND: No determinado
Discusión
Estudios previos en cortes de carne cruda9 y molida21 reportan porcentajes de positividad a Salmonella spp. mucho mayores a los aquí observados. Sin embargo, estas diferencias no son necesariamente el reflejo de un mejor control del patógeno en fechas más recientes, pues se ha demostrado que su prevalencia en el ganado puede variar con la zona geográfica y con la época del año.22 En cualquier caso, la frecuencia relativa observada en este estudio dista mucho de ser baja, lo que indica que la carne bovina se mantiene como un vehículo potencial para la transmisión de Salmonella spp.
Se ha sugerido que la presencia de nódulos linfáticos en los recortes y carne molida de res podría ser una de las principales fuentes de contaminación con Salmonella.23 Lo anterior a partir de la clonalidad observada entre cepas presentes en nódulos linfáticos y carne cruda. Además, la alta prevalencia del patógeno (55-91%) que suele observarse en los nódulos linfáticos,22,24 los cuales son muy difíciles de eliminar por completo de la carne durante el faenado en los rastros, explica de manera plausible el hallazgo persistente de tasas de positividad a Salmonella spp., de moderadas a altas, en muestras de carne cruda en los puntos de venta a lo largo de décadas.
Al parecer, para un mejor control de la diseminación de Salmonella a través de la carne de res, será necesario estudiar los factores que expliquen la colonización de los nódulos linfáticos por esta bacteria en bovinos aparentemente sanos. Asimismo, el desarrollo de estrategias para reducir la contaminación de carne con el contenido de los nódulos linfáticos, desde los rastros, podría ser un factor clave para la mitigación de este riesgo.
En adición a lo anterior, se requiere una mejor aplicación de la ley, específicamente en lo relativo al expendio de carne caliente. A pesar de que esta práctica está prohibida en la normatividad vigente,25 es tolerada en todo el país. La prueba de ji cuadrada evidenció una asociación significativa de la frecuencia de positividad a Salmonella con el mercado informal, donde el porcentaje de muestras positivas fue casi seis veces superior en relación con los supermercados. Por tanto, es evidente el potencial para reducir el riesgo de diseminación del patógeno en la cadena de abasto si tan solo se aplicaran, de manera efectiva, las medidas ya contempladas en la legislación vigente.
Con respecto a los serotipos, afortunadamente no se detectó ninguno de los de más alta virulencia (ej. Enteritidis y Typhimurium DT104). Sin embargo, todos los que se identificaron se han encontrado anteriormente en muestras de carne cruda y se han reconocido como causantes de infecciones transmitidas por alimentos en humanos en México y en otros países.6,26-28
Aunque la distribución de serotipos de Salmonella tanto en alimentos como en casos clínicos suele ser muy variable,1,2,8,29 la predominancia de S. Lomita no coincide con estudios anteriores realizados en México,6,9,12,26,30 en los que se reportan frecuencias nulas de esta serovariedad tanto en muestras clínicas como en alimentos. No obstante se ha sugerido que el hallazgo de cepas que podrían considerarse "exóticas" en determinadas regiones podría deberse a la globalización comercial y a la intensificación del turismo, los cuales favorecen la introducción e intercambio de distintos serotipos de Salmonella entre países.31,32
Desafortunadamente, las muestras incluidas en el presente trabajo fueron tomadas sólo en punto de venta y en un corto periodo de tiempo (aproximadamente ocho meses). Estas limitaciones imposibilitan establecer las razones que condicionan el predominio de determinados serotipos. Tampoco se encontraron reportes previos de estudios similares en la ciudad de México que permitieran establecer comparaciones cronológicas. No obstante, resulta interesante analizar por qué S. Lomita, un serotipo asociado principalmente con las palomas,27,28 fue aislado sólo de muestras obtenidas en el mercado informal. Los expendios informales tienen en común el pobre manejo sanitario de la carne y la falta de refrigeración, además de que en estos locales el control de plagas es, por lo general, muy limitado o inexistente. Sin embargo, esto no explica el predominio de S. Lomita, una cepa que no había sido reportada anteriormente en muestras de alimentos en México. Aun así, aparece como serotipo predominante en carne molida y además, exhibe un perfil de multirresistencia a antimicrobianos. Por ello, sería conveniente realizar otros estudios para identificar un origen más probable, así como los factores que condicionan su diseminación en la cadena de abasto hasta llegar al punto de venta.
En materia de susceptibilidad a antibióticos, la elevada tasa de resistencia a β-lactámicos, tetraciclina y trimetoprim-sulfametoxasol, así como el hecho de que casi las tres cuartas partes de los aislamientos resultaron multirresistentes, evidencia que existe una considerable presión de selección de cepas en diferentes segmentos de la cadena productiva.
En este trabajo se observó resistencia a medicamentos empleados exclusivamente en terapéutica humana, tales como el cloranfenicol, cuyo uso en producción animal está prohibido desde hace décadas.33 Este hallazgo coincide con lo observado en un estudio que se realizó en 2009 en el estado de Hidalgo,10 en donde el porcentaje de cepas resistentes a cloranfenicol fue ligeramente superior (23.1) al reportado en este trabajo. Ello reafirma la importancia de evaluar los riesgos relacionados con el empleo de antibióticos en medicina humana y veterinaria.
Por otra parte, se observó que casi 60% de las cepas de Salmonella mostraron resistencia a sulfa-trimetoprim, utilizado para tratar infecciones tanto en humanos como en animales. Cabe mencionar que el porcentaje de resistencia aquí encontrado es tres veces superior al reportado hace seis años.10 Adicionalmente, un número considerable de aislamientos resistieron antibióticos de amplio espectro, tales como la gentamicina, usada en medicina veterinaria y humana en el tratamiento de múltiples infecciones como septicemias, meningitis, peste, neumonías, entre otras. Asimismo, la resistencia a pefloxacina, si bien se presentó sólo en una cepa, es de las más preocupantes, pues se trata del antibiótico de elección para tratar casos de salmonelosis entérica en adultos.34
Aunque los resultados de este trabajo no permiten vincular las cepas aisladas de la carne con casos clínicos en humanos, la alta proporción de aislamientos que resistieron entre seis y ocho antibióticos sugiere que la exposición del consumidor mexicano a cepas de Salmonella multirresistentes constituye un riesgo latente. Por ejemplo, serotipos como Javiana suelen presentar una combinación de alta virulencia (la infección provoca complicaciones como meningitis, abscesos hepáticos y colecistitis)35 y un perfil de multirresistencia26,29 semejante al reportado para dicho serotipo en este trabajo (Am/Cb/ Cf/Chl/Tet). El tratamiento de infecciones causadas por cepas con estas características puede ser muy complicado. De ahí la importancia de identificar medidas que contribuyan a prevenir la presencia del patógeno en la carne que se expende en los puntos de venta.
Si bien los resultados de este estudio no son necesariamente representativos de la situación nacional, el muestreo en la principal urbe del país constituye una buena aproximación a lo que puede estar sucediendo en el resto de la república. Además, contribuye a fundamentar la necesidad de migrar hacia un sistema activo de vigilancia epidemiológica, como el que se aplica en países desarrollados.29 Esto es, que se identifique y se tipifique de manera sistemática a los patógenos causantes de brotes/casos, así como al alimento involucrado en los mismos. De esta forma, se puede realizar una mejor gestión de los riesgos asociados con el consumo de alimentos.
La presencia de serotipos de Salmonella multirresistentes a antibióticos en carne de res molida de venta al público en México evidencia la necesidad de investigar la epidemiología de la contaminación por Salmonella durante toda la cadena productiva de carne bovina. Estudios futuros sobre diversidad genética, presencia y mecanismos de transmisión de genes de resistencia y de virulencia, e incluso de secuenciación completa del genoma de las cepas circulantes, podrían ofrecer información vital para mejorar la gestión de este riesgo microbiológico.











 nueva página del texto (beta)
nueva página del texto (beta)