Introducción
Los enterococos son bacterias ácido lácticas, Gram-positivas, catalasa-negativas, anaerobias facultativas, quimio-organotótrofas, no esporuladas, en general inmóviles que se distinguen fácilmente de otras bacterias como los Streptococcus y los Lactococcus por su capacidad para crecer en una concentración hasta del 6.5% de NaCl, en presencia del 40% de sales biliares y con un pH de hasta 9.6 (Ferchichi et al., 2021). Estas bacterias son auxótrofas a varios aminoácidos, por lo que su crecimiento no suele ser abundante (Dapkevicius, Sgardioli, Câmara, Poeta & Malcata, 2021).
En un inicio, debido a sus características fenotípicas, se les clasificó como Streptococcus spp. del serotipo D. Es hasta el año 1984 cuando, con la aplicación de métodos moleculares de hibridación de ADN - ADN y ADN - ARNr, se determinó que Enterococcus era filogenéticamente distante a Streptococcus y se encontraba más cercano a otros géneros de la familia Enterococcaceae, como Bavariicoccus, Catellicoccus, Melissococcus, Pilibacter, Tetragenococcus y Vagococcus (Lebreton, Willems & Gilmore, 2014). Dentro del género existen aproximadamente 74 especies oficialmente reconocidas, pero las dos más conocidas son E. faecium y E. faecalis, seguido de otras especies como E. durans, E. gallinarum, E. casseliflavus y E. mundtii (García-Solache & Rice, 2019; Ferchichi et al., 2021). Los enterococos se encuentran distribuidos en una gran variedad de hábitats y no se limitan al tracto gastrointestinal de animales, pues también se encuentran presentes de forma natural en las plantas, el agua y el suelo (Lebreton et al., 2014). Como consecuencia, las características fenotípicas y genotípicas de estas bacterias son altamente dependientes del hábitat que colonizan. La recopilación de información vertida en este artículo expone esas características no discutidas comúnmente. Considerando que su presencia en los alimentos fermentados de manera natural es frecuente, se abordará su papel en el proceso fermentativo y como probióticos, ya que no necesariamente forman parte del grupo de patógenos oportunistas. Estos últimos han generado interés en la salud pública debido a su resistoma, lo que recientemente ha propiciado una mayor investigación sobre este género y los mecanismos de adquisición de factores de virulencia. Asimismo, exponemos algunos avances en los análisis genómicos que permiten diferenciar las cepas de origen alimentario de las patógenas oportunistas.
Importancia de Enterococcus en la salud pública
Aun siendo parte de la microbiota nativa del tracto gastrointestinal, el papel epidemiológico de Enterococcus en entornos intrahospitalarios, en especial en las cepas que presentan múltiples resistencias a los antibióticos, no debe ser subestimado. Estos microorganismos suelen estar asociados al desarrollo de enfermedades como las infecciones en el tracto urinario, la peritonitis, la endocarditis bacteriana y la septicemia, e incluso la muerte. Se encuentra reportado que la tasa de mortalidad de pacientes en estado inmunocomprometido, infectados con Enterococcus multirresistente es del 32 - 66.7% (Hemapanpairoa, Changpradub, Thunyaharn & Santimaleeworagun, 2021). Dentro de los factores de mayor importancia asociados a esta cifra, es de principal preocupación, su resistencia a la vancomicina. Por esto, la Organización Mundial de la Salud (OMS) considera a los enterococos resistentes a vancomicina (VRE) de alta prioridad en cuanto al desarrollo de nuevos antibióticos (Organización Mundial de la Salud, 2017). Al problema de multirresistencia a antibióticos también se adiciona su plasticidad genómica.
Virulencia: colonización
El primer paso para que un microorganismo lleve a cabo un proceso de infección exitoso es la colonización del hospedero. Lo que no necesariamente involucra una patogénesis, pues tanto microorganismos probióticos, como comensales y patógenos requieren colonizar al hospedero. En Enterococcus, los factores de virulencia que participan en la colonización del hospedero son la proteína de superficie enterocóccica (Esp), la sustancia de agregación (AS), la proteína de unión al colágeno y la adhesina de pared celular (EfaA) (Chajęcka-Wierzchowska, Zadernowska & Łaniewska-Trokenheim, 2017).
Dentro de los factores antes citados, la presencia de la proteína Esp (codificada por el gen esp) es el principal signo de alerta en los enterococos de importancia en salud pública y el de mayor preocupación hospitalaria por la formación de biopelículas, que le permiten al microorganismo unirse irreversiblemente a una superficie tanto biótica como abiótica y es de especial relevancia en infecciones en el tracto urinario y en pacientes intubados (Hashem, Abdelrahman & Aziz, 2021). La proteína Esp está codificada dentro de una isla de patogenicidad comúnmente asociada con bombas de eflujo, y su presencia en el genoma suele acompañarse de resistencia a la vancomicina, el cual es uno de los fenotipos de mayor importancia en entornos intrahospitalarios (Dapkevicius et al., 2021). Esp también está asociada a una mayor respuesta inflamatoria e infiltración de neutrófilos durante un proceso infeccioso (Zou & Shankar, 2015). Aunque se ha observado que la prevalencia de Esp es mayor en las cepas provenientes de aislados clínicos, se debe mencionar que algunos Enterococcus provenientes de alimentos también poseen el gen esp, lo que podría dar razón al motivo de no otorgarle al género la clasificación GRAS por la FDA o QPS (por sus siglas en inglés, Qualified Presumption of Safety) por la Unión Europea (Dapkevicius et al., 2021). A pesar de poseer ese factor asociado con los entornos nosocomiales, se ha observado que la presencia de ciertos marcadores genéticos relacionados con virulencia no siempre está correlacionada con su expresión (Deekshit & Srikumar, 2022).
La síntesis de biopelículas también contribuye a la capacidad de resistencia a los antibióticos en el género, no solo actuando como una barrera física, sino que su producción se encuentra estrechamente relacionada con la transferencia horizontal de genes mediante quorum-sensing, en particular de elementos de resistencia a diversos compuestos antimicrobianos (Conwell, Dooley & Naughton, 2022). Esta transferencia se encuentra en parte mediada por la sustancia de agregación (AS), la cual forma agregados conjugativos al recibir señales de las feromonas sexuales. Este fenómeno está asociado con la transferencia de plásmidos dentro de la especie, que pueden contener (además de genes de resistencia) otros factores de virulencia como el principal factor de invasión en enterococos, la citolisina (Chajęcka-Wierzchowska et al., 2017). La AS no solamente promueve el fenómeno de transferencia horizontal de genes, sino también la adhesión a las células del tubo renal y, de igual forma, a las células del epitelio intestinal (Dapkevicius et al., 2021), es importante tanto en los procesos de patogénesis como en los de colonización natural del tracto gastrointestinal.
El conjunto denominado factor accesorio de colonización, también conocido como conjunto de proteínas de unión al colágeno, es otro elemento asociado con la adhesión y formación de biopelículas. El colágeno es un componente esencial de la matriz extracelular de las células animales. La capacidad de las variantes de Enterococcus de reconocer al colágeno tipo I y IV y su alta prevalencia en aislados clínicos provenientes de pacientes con endocarditis infecciosa, sugieren que este componente es relevante en el proceso de colonización del hospedero (Arora, Gordon & Hook, 2021). Sin embargo, su presencia no es indicativa de la patogenicidad de la cepa, pues contrario a lo que se observa con Esp, las proteínas de unión al colágeno suelen encontrarse distribuidas de forma indistinta entre los aislados clínicos y los alimentarios (Medeiros et al., 2014).
Enterococcus es intrínsecamente resistente a varios grupos de antibióticos, como los aminoglucósidos y los macrólidos, pero puede adquirir resistencia a múltiples grupos como los glucopéptidos, las tetraciclinas y las penicilinas, entre otros; sin embargo, destaca la resistencia a la vancomicina como la de mayor relevancia en los entornos intrahospitalarios (Sattari-Maraji, Jabalameli, Node Farahani, Beigverdi & Emaneini, 2019). Estas resistencias pueden transferirse dentro y entre especies mediante elementos móviles como los plásmidos y los transposones. Por ejemplo, en los transposones Tn917 y Tn1546, se encuentran las resistencias a la vancomicina, y a los macrólidos, las lincosamidas y las estreptograminas, respectivamente (Ramos, Dapkevicius, Igrejas & Poeta, 2020). La adquisición de elementos móviles con genes de resistencia y/o virulencia se relaciona de forma inversa a la capacidad del microorganismo para degradar el ADN exógeno. Se ha observado que cepas incapaces de degradar el material genético exógeno mediante un sistema CRISPR-Cas funcional, suelen tener una mayor cantidad de genes de resistencia a antibióticos y de genes codificantes para factores de virulencia, y viceversa (Palmer & Gilmore, 2010). De igual forma, se ha observado que la ausencia de elementos CRISPR-Cas en cepas nosocomiales se correlaciona con una mayor presencia del gen esp (Tao, Chen, Li, Fang, Xu & Liang, 2022). Esto contrasta con el comportamiento observado en las cepas probióticas y las provenientes de alimentos (como los cárnicos y los lácteos), donde se aprecia una predominancia de las que tienen sistemas CRISPR-Cas funcionales y, por lo tanto, una menor presencia de genes de resistencia no intrínsecos (Bonacina, Suárez, Hormigo, Fadda, Lechner & Saavedra, 2016). Por lo antes expuesto, se observa que a pesar de que Enterococcus tiene un genoma flexible, los aislados clínicos presentan una mayor capacidad de adquirir elementos genómicos exógenos, lo que nos hace pensar que esta flexibilidad se encuentra relacionada con el nicho en el que habitan.
Virulencia: invasión
Una vez establecido dentro del hospedero, el microorganismo produce sustancias que le permiten diseminarse y colonizar otros tejidos, lo que se denomina proceso de invasión, y en el género Enterococcus se debe a tres proteínas principales: la citolisina (Cyl), la gelatinasa (GelE) y la hialuronidasa (Hyl) (Chajęcka-Wierzchowska et al., 2017).
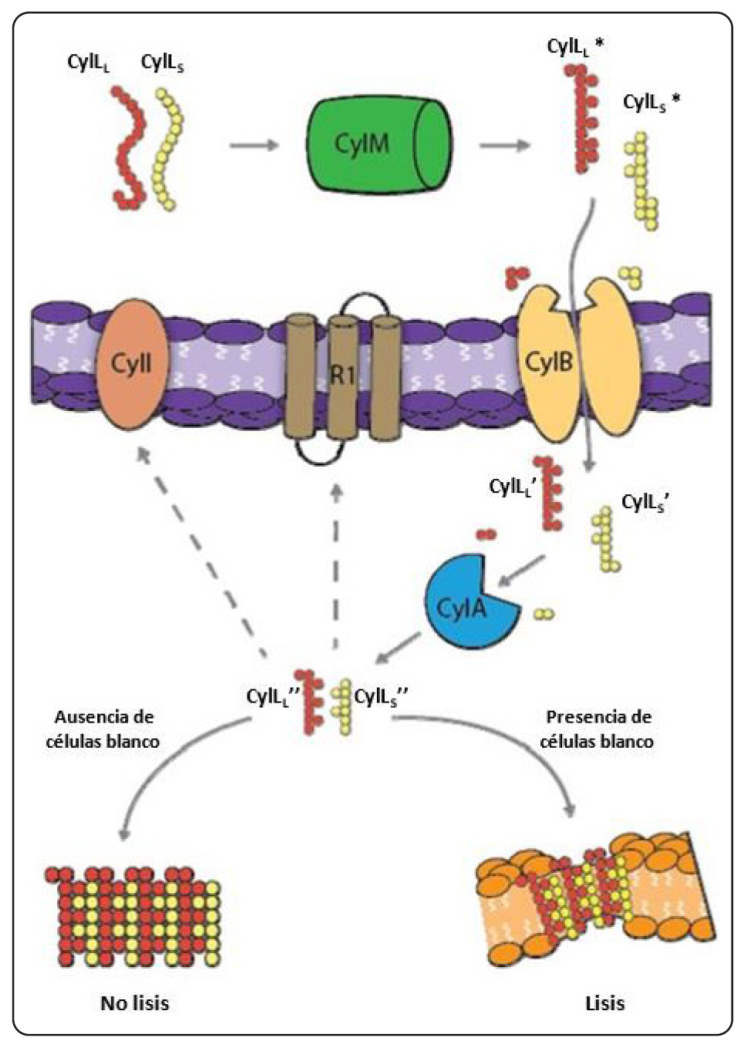
La citolisina es una bacteriocina tipo lantibiótico, conformada por dos subunidades: CylL y CylS, con actividad lítica en proporción equimolar, que actúan tanto en contra de células bacterianas como en contra de células eucariontes (Rahman, Sanchez, Tang & van der Donk, 2021). Aunque esta bacteriocina se encuentra descrita en varias especies de Enterococcus, es más común su presencia en E. faecalis. La citolisina está codificada en un operón dentro de una isla de patogenicidad móvil, y es el factor de virulencia mejor descrito del género: su papel en el proceso de patogénesis se correlaciona directamente con el desarrollo de una condición intratable en modelos de endocarditis y endoftalmitis, además de un aumento de la letalidad de hasta 5 veces por bacteriemia en seres humanos (Van Tyne, Martin & Gilmore, 2013). Se ha observado que, al igual que con esp, cylA (el gen codificante para la proteína responsable de procesar y activar a los precursores de la citolisina madura, CylLL y CylLS) predomina en muestras provenientes de hospitales. Estas muestras no siempre presentan actividad β-hemolítica aun con la presencia del gen cylA (Medeiros et al., 2014). Este comportamiento se debe a la complejidad del sistema de procesamiento de CylLL y CylLS hasta los productos maduros CylLL’’ y CylLS’’. La ausencia de uno solo de los elementos codificados en el operón de citolisina (CylLL, CylLS, CylM, CylI, CylB y CylA) desencadena en un procesamiento incompleto de dichos péptidos y la ausencia de la actividad lítica (Figura 1) (Van Tyne et al., 2013).
GelE es una metalopeptidasa que hidroliza diversos componentes celulares como la elastina, la gelatina, el colágeno y la hemoglobina, y es otro factor de virulencia importante en el proceso de invasión del hospedero (Chajęcka-Wierzchowska. et al., 2017). GelE tiene una actividad directa sobre el sistema inmune, debido a su capacidad de hidrolizar al componente C3b del complemento, inhibiendo la formación del complejo de ataque a membrana y el proceso de opsonización, relacionado con la neutralización del agente extraño (Wang, Yang & Huycke, 2020). Se ha reportado que hasta un 70% de aislados provenientes de matrices alimentarias, particularmente cárnicos, contienen el gen gelE, probablemente debido a la alta abundancia de gelatina y colágeno en éstas (Ferchichi et al., 2021). A pesar de esto, muchos aislados de Enterococcus positivos para gelE no presentan actividad gelatinasa (Medeiros et al., 2014).
Virulencia: otros problemas a considerar
Cada factor de virulencia involucrado en los procesos de colonización e invasión tiene un papel en la patogenicidad del microorganismo. Sin embargo, es la forma en la que interaccionan estos factores entre ellos y con el entorno lo que determina la letalidad de la infección. Se ha reportado que la expresión de la citolisina junto con la sustancia de agregación puede volver letal un episodio de endocarditis que bajo otras condiciones no lo sería (Van Tyne et al., 2013). Lo que puede ser mediante la regulación positiva de la citolisina en el sistema de quorum-sensing (Chajęcka-Wierzchowska et al., 2017). La regulación del quorum-sensing es dependiente de las feromonas enterocóccicas que, además de propiciar la transferencia de elementos móviles, actúan como moléculas quimiotácticas capaces de reclutar leucocitos humanos (Șchiopu et al., 2023). Se ha demostrado que cuando Enterococcus infecta, es capaz de sobrevivir el proceso de fagocitosis y producir altos niveles de estrés oxidativo, por lo que también se le ha asociado con procesos genotóxicos y cancerígenos (Wang et al., 2020).
Adicionalmente, el microorganismo presenta sinergismo con miembros de otros géneros bacterianos. Se ha reportado que infecciones con la presencia simultánea de E. faecalis y Escherichia coli presentan una mayor mortalidad que cuando actúan por separado (Lavigne, Nicolas-Chanoine, Bourg, Moreau & Sotto, 2008).
Enterococcus como reservorios y el resistoma ambiental
Uno de los factores por el que Enterococcus es considerado un patógeno oportunista de importancia es su alta plasticidad genómica y, por lo tanto, su capacidad de ser reservorio de múltiples determinantes genéticos de relevancia epidemiológica. Adicionalmente, su capacidad para formar y vivir en biopelículas facilita la adquisición de genes de resistencia a antibióticos en contraste con su forma planctónica (Michaelis & Grohmann, 2023).
Se conoce como resistoma al conjunto de genes de resistencia a antibióticos de una población determinada (conformada por microorganismos patógenos y no patógenos) en un entorno particular. El tracto gastrointestinal posee un resistoma con elementos que, con el tiempo, pueden volverse de importancia clínica. Sin embargo, y contrario a lo que se esperaría, se ha observado que eventos de transferencia de genes de resistencia entre microorganismos patógenos con comensales son escasos (Kim & Cha, 2021) . En particular, los Enterococcus de naturaleza probiótica, presentan una baja receptividad al ADN exógeno debido a la presencia de elementos como el sistema CRISPR-Cas, comportamiento opuesto a lo observado en las cepas de origen intrahospitalario (Bonacina et al., 2016; Palmer & Gilmore, 2010). Se ha reportado una correlación entre la resistencia al ciprofloxacino, a la eritromicina y a la tetraciclina con la presencia del gen esp en aislados de Enterococcus spp. provenientes de perros y gatos (Yuan et al., 2023).
Además de los genes de resistencia que sí se expresan, existe un conjunto de ellos que no lo hacen, llamados genes de resistencia crípticos. Estos están silenciados debido a la presencia de promotores no funcionales río arriba del gen, a mutaciones en el propio gen, a la acción de proteínas represoras y/o a modificaciones epigenéticas, con la finalidad de evitar el uso de recursos en su expresión cuando ésta no es necesaria (Deekshit & Srikumar, 2022). Este fenómeno también da sentido a la falta de expresión de ciertos factores de virulencia a pesar de estar presentes en el genoma, como gelE y cylA (Medeiros et al., 2014).
MLST y WGS como herramientas en la evaluación de la patogenicidad de Enterococcus
La expresión génica de Enterococcus es variable y la sola presencia de ciertos componentes genómicos no permite emitir una certeza sobre la naturaleza comensal o patogénica de una cepa. A pesar de ello, las herramientas de secuenciación del genoma completo (WGS) y tipificación por secuencias multi-locus (MLST) son utilizadas para la evaluación del potencial patogénico de alguna cepa.
La técnica de MLST consiste en una amplificación y posterior secuenciación de las regiones internas altamente conservadas de un conjunto de genes estandarizado para cada microorganismo y las diferencias en las secuencias representan alelos diferentes de cada región. Según la combinación de alelos, se le asigna a la cepa en cuestión una secuencia tipo (ST) y los resultados de estos experimentos suelen ser añadidos a las bases de datos públicas (como PubMLST: https://pubmlst.org/) con la finalidad de construir una biblioteca que permita determinar tendencias de origen, patogenicidad y comportamiento de las ST (Jolley, Bray & Maiden, 2018). Dentro del género Enterococcus se han identificado cepas provenientes de entornos nosocomiales que comparten entre sí muchas características de relevancia clínica. En el caso de E. faecium, a este grupo de cepas se le denomina complejo clonal 17 (CC17) y está compuesto por las ST 17, 18, 78, 80 y 117.
Este complejo clonal (CC) se caracteriza por tener el alelo purK 1, al gen esp y presentar resistencia a la ampicilina y a la vancomicina (Carmen Fariñas & Torres, 2007). El equivalente para E. faecalis son los complejos clonales CC2 y CC9 que se caracterizan por su resistencia al ciprofloxacino y por la presencia del gen esp (Muruzábal-Lecumberri, Girbau, Canut, Alonso & Fernández-Astorga, 2015). La estandarización de la metodología MLST para E. faecium fue llevada a cabo en el año 2002, con la finalidad de dar lugar a una técnica de vigilancia epidemiológica de bajo costo y alta capacidad de rastreo de brotes infecciosos (Homan et al., 2002). Desde entonces, se han propuesto mejoras a la metodología MLST con la finalidad de volverla aún más discriminatoria y eficiente. En el caso de E. faecium, se ha sugerido la expansión de la tipificación de 7 a 8 genes, para obtener un poder discriminatorio y número de secuencias tipo mayor a las observadas en el método tradicional (Bezdicek et al., 2023). Esta metodología no solamente tiene utilidad entre brotes intrahospitalarios, también permite ubicar a las ST de muestras de distintos orígenes con respecto a los CC de preocupación.
Los análisis de MLST suelen complementarse con estudios del genoma completo, debido a que el MLST solamente utiliza el 1% del núcleo genómico para el análisis (O’Toole, Leong, Cumming & Van Hal, 2023). Los análisis de WGS son ahora más que una herramienta analítica, y se han vuelto parte esencial en la vigilancia epidemiológica, lo que permite encontrar nuevas ST asociadas con genes de resistencia a antibióticos y de virulencia (Rogers et al., 2021). Este tipo de análisis permite establecer relaciones filogenéticas entre cepas de la misma especie y determinar diferencias entre aislados obtenidos a partir de distintos orígenes, como el clínico y el alimentario (Olvera-García, Sanchez-Flores & Quirasco Baruch, 2018). La combinación de ambas metodologías ha permitido el desarrollo de herramientas en línea que pueden llevarlos a cabo a partir de datos de WGS, lo que facilita el análisis de Enterococcus de relevancia epidemiológica (Marbjerg et al., 2021).
Sin embargo, y como se mencionó, la presencia de un gen de virulencia o resistencia en el genoma de una cepa no permite asegurar que éste se expresará, pues los genes crípticos pueden estar en un estado activo o inactivo, dependiendo de las presiones ambientales en las que se encuentre el microorganismo (Deekshit & Srikumar, 2022).
Importancia de Enterococcus en los alimentos
A pesar de ser un género relacionado con brotes infecciosos en los hospitales, Enterococcus se encuentra distribuido de forma natural en una gran variedad de alimentos fermentados y su presencia no es debida a su uso como cultivo iniciador, sino a la ubicuidad del microorganismo. El género es de especial importancia en la elaboración y conservación de alimentos fermentados de forma natural debido a las características metabólicas que posee y a su correlación con las propiedades sensoriales del alimento en el que se encuentra.
Una gran variedad de bebidas tradicionales mexicanas contiene a este microorganismo, como el pozol, el tepache, la tuba y el tejuino (Díaz-Ruiz, Guyot, Ruiz-Teran, Morlon-Guyot & Wacher, 2003; de la Fuente-Salcido, Castañeda-Ramírez, García-Almendárez, Bideshi, Salcedo-Hernández & Barboza-Corona, 2015; Rubio-Castillo et al., 2021). En lo que respecta a los productos lácteos, algunos quesos en los que se ha encontrado Enterococcus incluyen, pero no se limitan a: Roquefort, Cheddar, Manchego, Cabrales, Cotija, Beaufort, Serra da Estrela y Mozzarella, entre otros (Tabla I) (Gelsomino et al., 2004, Olvera-García et al., 2018).
Tabla I Ejemplos de alimentos fermentados de forma natural que contienen a Enterococcus spp.
| Alimento | Especie | Referencias |
|---|---|---|
| Sauerkraut | E. faecium | Yang & Pei, 2020 |
| Pozol | E. sulfureus | Díaz-Ruiz et al., 2003 |
| Kimchi | E. faecium | Rho et al., 2017 |
| Tuba | E. faecium | de la Fuente-Salcido et al., 2015 |
| Tepache | E. faecium | de la Fuente-Salcido et al., 2015 |
| Pulque | E. faecium, E. faecalis | Huezo-Sánchez, Ortega-Rodríguez, Pérez- Armendáriz & El-Kassis, 2023 |
| Aguamiel | E. casseliflavus | Huezo-Sánchez et al., 2023 |
| Atole agrio | Enterococcus spp. | Väkeväinen et al., 2018 |
| Queso Manchego | E. faecium, E. faecalis | Nieto-Arribas, Seseña, Poveda, Chicón, Cabezas & Palop, 2011 |
| Queso Cotija | E. faecium, E. faecalis | García-Cano et al., 2014, Escobar-Zepeda, Sanchez-Flores & Quirasco Baruch, 2016 |
| Queso Pecorino | Enterococcus spp. | Grispoldi et al., 2022 |
| Pepinillos | E. casseliflavus | Satomi et al., 2022 |
| Aceitunas | E. olivae | Lucena-Padrós, González, Caballero-Guerrero, Ruiz-Barba & Maldonado-Barragán, 2014 |
| Mezcal | E. faecium | Hernández-Delgado et al., 2021 |
Maduración: olores, sabores y texturas
Se suele dividir a los sistemas de generación de olores, sabores y texturas de Enterococcus en 3 grupos: sistema proteolítico, lipolítico y de metabolismo de citrato (Terzić-Vidojević, Veljović, Popović, Tolinački & Golić, 2021).
En los alimentos provenientes de matrices lácteas, el sistema proteolítico es de especial importancia metabólica por la alta disponibilidad de proteínas en el medio. La hidrólisis de la caseína hasta péptidos la realiza el sistema Clp y el posterior metabolismo de estos últimos se efectúa mediante el sistema Pep (Olvera-García et al., 2018; Dapkevicius et al., 2021). El proceso de proteólisis produce precursores de varios compuestos involucrados en el desarrollo de sabores agradables, como ésteres y aldehídos, principalmente derivados de los péptidos ricos en metionina, fenilalanina, treonina, leucina, isoleucina y valina (Smit, Smit & Engels, 2005). Dentro de la proteólisis, la peptidasa PepP se encarga del metabolismo de los péptidos de prolina tipo Xaa-Pro-Xaa, responsables de un sabor amargo, y es esencial en el balance entre la producción de compuestos agradables y no deseables (Dapkevicius et al., 2021). Se ha propuesto el uso de cultivos de naturaleza enterocóccica en la producción de salchichas y otras carnes fermentadas para mejorar sus características sensoriales (Carvalho, de Santos, Gomes & Hoffmann, 2017). En el caso de los quesos bajos en grasa, se ha propuesto el uso del exopolisacárido (EPS) de Enterococcus para mejorar su textura debido a sus propiedades higroscópicas (Dapkevicius et al., 2021). Enterococcus también tiene un papel importante en la industria panificadora, pues se encuentra presente en la masa del pan y es parte del primer grupo de BAL dominantes en el microbioma de este alimento. Este grupo inhibe el crecimiento de las bacterias no fermentativas y no deseables, como Bacillaceae y Enterobacteriaceae; para posteriormente favorecer el crecimiento de BAL como Lactobacillus (Oshiro, Zendo & Nakayama, 2021).
El segundo sistema de importancia en el desarrollo de características deseables en los alimentos fermentados es el sistema lipolítico. Mediante la lipólisis de los triglicéridos se producen ácidos grasos libres, precursores de compuestos con sabores agradables, principalmente lactonas, ésteres, alcoholes y metilcetonas (Smit et al., 2005). A pesar de que se han identificado cientos de compuestos con olores producto de la lipólisis, solamente unos cuantos son responsables de la mayor parte de los aromas característicos de los alimentos fermentados, entre los que se encuentran el ácido acético, el ácido butírico, el ácido hexanóico, el ácido decanóico, el ácido dodecanóico y el ácido isovalérico, entre otros (Wolf, Meinardi & Zalazar, 2009). Las enzimas de Enterococcus involucradas en el proceso de la lipólisis son las lipasas y las esterasas. En particular, la enzima EstA se ha identificado como clave en el desarrollo de los sabores y los olores en los alimentos lácteos madurados; ésta junto con la peptidasa PepP son responsables de la producción de compuestos característicos del sabor de los alimentos lácteos madurados, como el diacetilo, acetona y 2,3-pentadiona (Engels, Siu, van Schalkwijk, Wesselink, Jacobs & Bachmann, 2022). Se ha observado que la especie E. faecium tiene una mejor actividad lipolítica que E. faecalis, y esta actividad es dependiente de la cepa (Terzić-Vidojević et al., 2021).
Otro sistema de desarrollo de aromas en alimentos fermentados donde participa Enterococcus es el metabolismo del citrato. La degradación de este ácido tricarboxílico produce moléculas como el diacetilo, que se encuentra relacionado con notas mantequillosas y cremosas, además de acetoína y 2,3-butanodiol (Foulquié Moreno, Sarantinopoulos, Tsakalidou & De Vuyst, 2006). Adicionalmente, el metabolismo del citrato también es responsable de la formación de las burbujas de aire características de quesos como el Gouda (Dapkevicius et al., 2021).
Con respecto a los clusters del metabolismo del citrato en Enterococcus, se ha propuesto una división según el tipo de regulador (CitI o CitO), transportador (CitP o CitH) y localización de la oxalacetato descarboxilasa (citoplásmica o membranal) presente en la cepa (Martino, Quintana, Espariz, Blancato & Magni, 2016). Se ha observado que la producción de compuestos específicos a partir del citrato es altamente dependiente de otras fuentes de energía y microorganismos presentes en el medio. En el caso de E. faecium FAIR-E 198 experimentos in vitro demostraron una mayor producción de acetato en presencia de lactosa, sacrificando la producción de acetoína, que tampoco se produce en cocultivo con Leuconostoc mesenteroides (De Vuyst, Vaningelgem, Ghijsels, Tsakalidou & Leroy, 2011).
Conservación: bacteriocinas y peptidoglucano hidrolasas
Enterococcus es una bacteria tolerante al estrés producido por el pH, con la capacidad de acidificar el medio en el que se encuentra, aunque en un grado menor que otros géneros de BAL como Lactobacillus. Esto se debe a la producción de ácidos orgánicos como el láctico, propiónico, fórmico y succínico, que, como consecuencia, pueden inhibir el crecimiento de microorganismos que no son capaces de sobrevivir en bajos niveles de pH (Wang et al., 2021). Aunado a esto, durante el proceso de fermentación del alimento Enterococcus produce otros compuestos antimicrobianos de naturaleza proteínica, como las bacteriocinas y las peptidoglucano hidrolasas (PGHs); que en conjunto con procesos como la producción de peróxido de hidrógeno y los ácidos orgánicos mencionados, y aunado a cambios fisicoquímicos del alimento (como la diminución de la actividad acuosa durante su maduración) aportan a la inocuidad del alimento, sin la necesidad de llevar a cabo procesos como la pasteurización de la materia prima. Esto es de especial importancia en alimentos cuya calidad sensorial es altamente dependiente de su ecología microbiana, pues un proceso de pasteurización sobre la materia prima sería deletéreo para su microbiota y, por consiguiente, a la generación de propiedades organolépticas complejas.
Las bacteriocinas son péptidos ribosomales que generalmente presentan actividad antimicrobiana contra microorganismos filogenéticamente cercanos a la bacteria productora (espectro reducido), pero que también pueden actuar contra organismos de otros filos e inclusive reinos (espectro amplio) (Simons, Alhanout & Duval, 2020). Estas biomoléculas se clasifican según su estructura y, a la fecha, se reconocen 4 clases: clase I (menores a 10 kDa y con modificaciones post-traduccionales extensas), clase II (menores a 10 kDa y donde la única modificación post-traduccional es la remoción del péptido líder), clase III (péptidos termolábiles, mayores a 10 kDa) y clase IV (proteínas mayores a 30 kDa) (Antoshina, Balandin & Ovchinnikova, 2022). Algunas bacteriocinas de Enterococcus son de importancia epidemiológica para el ser humano, como la citolisina ya mencionada, que es una bacteriocina clase I con actividad hemolítica (Rahman et al., 2021). Sin embargo, en el proceso de conservación alimentaria las bacteriocinas de mayor interés son las de la clase II, por su actividad contra Listeria monocytogenes, un microorganismo gram-positivo, presente en alimentos contaminados y es el agente etiológico de la listeriosis, una infección con la capacidad de provocar meningitis y abortos espontáneos (Organización Mundial de la Salud, 2018). Se han propuesto diversos mecanismos por los que las bacteriocinas de la clase II producidas por BAL pueden llevar a cabo la inhibición del crecimiento de Listeria spp. y generalmente se dividen en: a) la formación de poros en la membrana citoplasmática y b) la inhibición de la síntesis del peptidoglucano mediante la unión a sus precursores (lípido A y undecaprenilpirofosfato fosfatasa, UppP) (Figura 2), (Antoshina et al., 2022).
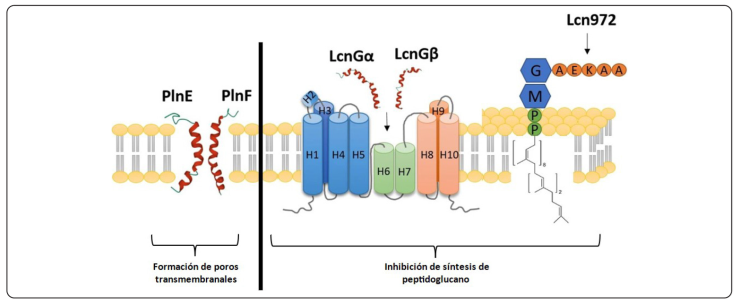
Adaptado de Antoshina et al., 2022.
Figura 2 Principales mecanismos de acción de las bacteriocinas clase II plantaricina (PlnE y PlnF), lactoccina G (LcnGα y LcnGβ) y lactoccocina 972 (Lcn972); H1 a H10 (UppP).
La mayoría de las bacteriocinas de interés alimentario producidas por Enterococcus pertenecen a la clase II y se denominan enterocinas (Yusuf, 2018). La enterocina A (EntA), producida por una cepa de E. faecium proviene de una salchicha española fermentada y fue la primera en ser estudiada y caracterizada (Aymerich, Holo, Håvarstein, Hugas, Garriga & Nes, 1996). Esta molécula con actividad antilisteral en productos lácteos, también inhibe a otros microorganismos como Salmonella enterica y Staphylococcus aureus (Wu, Pang, Wu, Liu & Zhang, 2022). Desde entonces, se han descubierto más enterocinas de cepas de Enterococcus aisladas de varias fuentes, de diversas clases y con diversos mecanismos de acción (Tabla II).
Tabla II Enterocinas obtenidas de diversas fuentes
| Enterocina | Fuente | Referencias |
|---|---|---|
| Enterocina A | Salchicha española | Aymerich et al., 1996 |
| Enterocina B | Salchicha española | Casaus, Nilsen, Cintas, Nes, Hernández & Holo, 1997 |
| Enterocina X | Noi-Na tailandés | Hu, Malaphan, Zendo, Nakayama & Sonomoto, 2010 |
| Enterocina 1071 | Heces de cerdo | Balla, Dicks, Du Toit, Van Der Merwe & Holzapfel, 2010 |
| Enterocina P | Salchicha española | Cintas et al., 2000 |
| Enterocina HF | Heces de Gyps fulvus subsp. fulvus | Arbulu et al., 2015 |
| Enterocina L50A/B | Salchicha española | Cintas et al., 2000 |
| Enterocina Q | Salchicha española | Cintas et al., 2000 |
| Mundticina K | Forraje fermentado | Kawamoto et al., 2002 |
| Enterocina SE-K4 | Forraje fermentado | Eguchi et al., 2001 |
| Enterocina RJ-11 | Arroz | Yamamoto, Togawa, Shimosaka & Okazaki, 2003 |
| Enterocina EJ97 | Agua residual | Gálvez et al., 1998 |
| Enterocina AS-48 | Exudado de herida humana | Gálvez et al., 1989 |
| Enterocina AS-48RJ | Queso de cabra | Abriouel et al., 2005 |
| Bacteriocina T8 | Secreciones vaginales | De Kwaadsteniet, Fraser, Van Reenen & Dicks, 2006 |
| Enterocina 96 | Queso Munster | Izquierdo, Wagner, Marchioni, Aoude-Werner & Ennahar, 2009 |
| Enterocina E-760 | Intestino de pollo | Line et al., 2008 |
Las bacteriocinas son posiblemente los productos derivados de Enterococcus más estudiados en el ámbito biotecnológico. Las enterocinas A y AS-48 han sido útiles en la conservación del jamón ahumado y el queso cottage, y se ha propuesto usarlas en conjunto con empaques compuestos de polímeros biodegradables (Robles Camacho, 2019; Gumienna & Górna, 2021). Las bacteriocinas A, B y AS-48 son activas contra L. monocytogenes y otros microorganismos de interés alimentario como Salmonella spp., E. coli y Bacillus coagulans (Yusuf, 2018). Adicionalmente, son capaces de resistir un tratamiento térmico (100 °C por 30 min), conservan su actividad en valores de pH entre 2 y 8, y son útiles en la mejora del aspecto de la carne roja mediante la reducción de la metamioglobina (Kasimin, Shamsuddin, Molujin, Sabullah, Gansau & Jawan, 2022). Algunas bacteriocinas enterocóccicas presentan una actividad anticancerígena, al inhibir líneas celulares de cáncer de pulmón, cervical y de osteosarcoma, cuya eficacia depende de la dosis (Sharma, Kaur , Chadha, Kaur, Kaur & Kaur, 2021).
Otro conjunto de moléculas producidas por Enterococcus con la capacidad de inhibir el crecimiento de algunas bacterias son las peptidoglucano hidrolasas (PGHs), que son proteínas importantes en el proceso del remodelado de la pared celular bacteriana, y debido a la complejidad del peptidoglucano, las PGHs pueden tener diferentes especificidades según el sitio que reconocen (Figura 3) (Layec, Decaris & Leblond-Bourget, 2008).
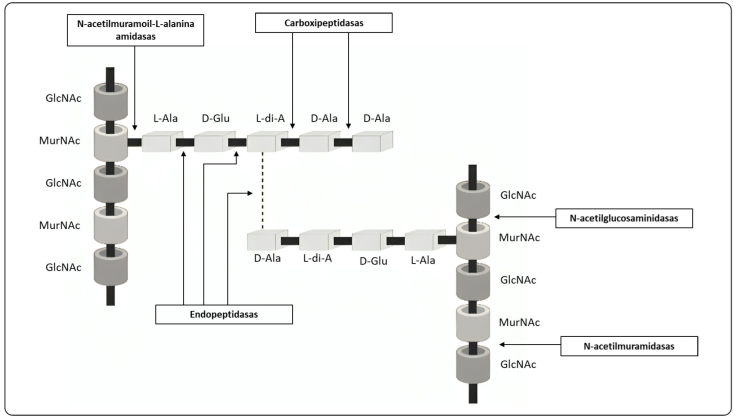
Adaptado de Layec et al., 2008; Serrano Maldonado, 2013.
Figura 3 Mecanismos de acción de distintas peptidoglucano hidrolasas.
Dentro del género Enterococcus se han encontrado diversas PGHs con actividad antibacteriana (García-Cano et al., 2014). Una de ellas es AtlA, la principal PGH de tipo N-acetilmuraminidasa de E. faecalis. Esta proteína ha demostrado ser esencial en la formación del septo al momento de la división celular, además de tener actividad antimicrobiana contra Micrococcus lysodeikticus (Roig-Zamboni et al., 2022). Una N-acetilmuraminidasa novedosa descrita por nuestro equipo de investigación, AtlD, demostró tener actividad contra patógenos de importancia alimentaria como L. monocytogenes, S. aureus, E. faecium y E. faecalis de origen clínico (Serrano-Maldonado, García-Cano, González-Canto, Ruiz- May, Elizalde-Contreras & Quirasco, 2018). Por otro lado, la PGH de tipo endopeptidasa EnpA CD ha mostrado la capacidad de lisar 107 células de S. aureus a una concentración de 500 nM a temperatura ambiente, especialmente en contra de especies cuyo peptidoglucano contiene puentes peptídicos compuestos principalmente de glicina, serina y alanina (Małecki, Mitkowski, Jagielska, Trochimiak, Mesnage & Sabała, 2021). Se ha reportado que las PGHs también actúan en contra de los patógenos que afectan a otros animales como el camarón, en concreto contra Vibrio spp., agente etiológico del cólera (Chino de la Cruz, Cornejo-Granados, Gallardo-Becerra, Rodríguez-Alegría, Ochoa-Leyva & López Munguía, 2023).
Ventajas de su consumo: Enterococcus como probiótico y postbiótico
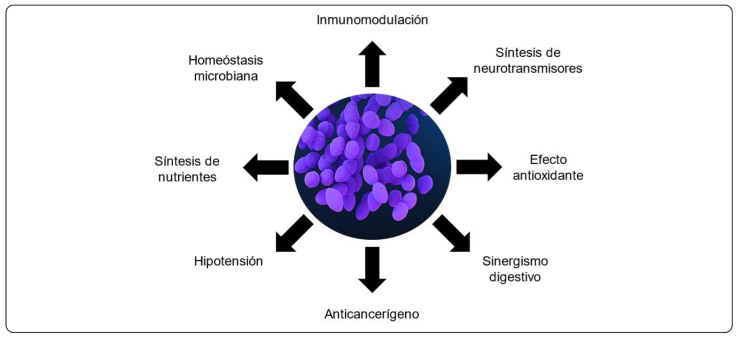
La función de Enterococcus en la maduración y conservación de los alimentos fermentados no puede ser subestimada; la comunidad científica ha reconocido su importancia en estos procesos. Sin embargo, la propuesta de que Enterococcus se desempeñe como probiótico sigue siendo un tema controversial debido a la prevalencia que tiene en entornos nosocomiales y su correlación con afecciones como la endocarditis e infecciones del tracto urinario (Hemapanpairoa et al., 2021). Se ha observado que Enterococcus tiene varias propiedades que lo convierten en un microorganismo con potencial probiótico como: la resistencia al paso por el tracto gastrointestinal hasta llegar al colon, la adhesión a la mucosa intestinal, la inmunomodulación, la síntesis de compuestos bioactivos y el metabolismo de compuestos no digeribles. Por consiguiente, sus preparados postbióticos han comenzado a ser estudiados de igual forma (Sakoui et al., 2024; Sun, Wang & Jiang, 2010; Popović et al., 2023; Olvera-García et al., 2018). Se define como postbiótico a una preparación de microorganismos no viables, o de sus restos celulares, obtenidos posterior a una inactivación térmica o enzimática de ellos, que son benéficos para la salud humana (Vinderola, Sanders & Salminen, 2022). Su principal cualidad es el hecho de que permite explorar las propiedades benéficas de los metabolitos, de los componentes celulares o de las proteínas no termolábiles o resistentes al tratamiento enzimático, sin la necesidad de contar con el microorganismo viable, lo que ofrece ventajas tecnológicas importantes en su aplicación industrial. Hoy en día contamos con escasos probióticos y postbióticos comerciales enterocóccicos, como el producto Symbioflor 1, compuesto de células vivas y lisados de 10 aislados distintos de E. faecalis (Fritzenwanker, Chakraborty, Hain, Zimmermann & Domann, 2016).
Como probiótico, se conoce que ciertas cepas de E. faecalis estimulan la producción de un perfil de citocinas que conllevan a un fenotipo de actividad inmune intracelular, además de activar células dendríticas mediante la inducción de la producción de interleucinas tanto proinflamatorias como antiinflamatorias (Molina et al., 2015). Estudios en Caenorhabditis elegans han demostrado la capacidad de E. faecalis de inducir la transcripción de genes codificantes para lisozima y lectinas en eventos de infección con Staphylococcus aureus (Ford, Drew & King, 2022). La producción y actividad anticancerígena de ciertas bacteriocinas de Enterococcus thailandicus han sugerido la posibilidad de utilizar al microorganismo como apoyo en el tratamiento del cáncer hepático (Al-Madboly, El-Deeb, Kabbash, Nael, Kenawy & Ragab, 2020). Este efecto anticancerígeno también ha sido observado en otras especies como E. muntdii, E. faecalis, E. durans, E. hirae, E. casseliflavus y E. faecium, con varios mecanismos, que incluyen la producción de enterocinas y metabolitos con actividad apoptótica y antiproliferativa en células cancerígenas de mama, colon y tejido cervicouterino (Grenda, Grenda, Domaradzki & Kwiatek, 2022). La capacidad proteolítica de Enterococcus no solamente ha demostrado ser de utilidad en la síntesis de compuestos con aroma y sabor, sino que este proceso también tiene la capacidad de producir péptidos bioactivos. Se ha reportado la síntesis de péptidos enterocóccicos con actividad antihipertensiva que actúan mediante la inhibición de la enzima convertidora de angiotensina (ACE), con el potencial de ser usados como agentes antihipertensivos sin los efectos secundarios de compuestos como el captopril (Graham, Stack & Rea, 2020). Los productos de fermentación de múltiples cepas de E. faecalis han demostrado ser antioxidantes debido a la producción de compuestos fenólicos y a una actividad antihiperglucemiante al inhibirse la enzima α-glucosidasa (Graham, Rea, Simpson & Stack, 2019). Otros metabolitos benéficos para la salud humana y que son sintetizados por bacterias comensales intestinales, incluyendo a Enterococcus, son las vitaminas. Éstas sintetizan in situ vitamina K y algunas vitaminas B de novo, como el folato y la cobalamina (Hadadi, Berweiler, Wang & Trajkovski, 2021; Engevik et al., 2019). Adicionalmente, se ha demostrado que, junto con otras bacterias de la microbiota intestinal, produce ácido γ-aminobutírico (GABA), un neurotransmisor asociado con efectos antihipertensivos y diuréticos que forma parte del eje intestino-cerebro (Rutsch, Kantsjö & Ronchi, 2020).
Por otro lado, Enterococcus lleva a cabo un sinergismo con el metabolismo digestivo del hospedero. Es capaz de degradar polisacáridos no digeribles, volviéndolos más accesibles para el hospedero (Wang et al., 2021). Se ha reportado que, en presencia de sales biliares, la cepa E. faecium 96B4 disminuye los niveles de colesterol mediante posibles mecanismos como la conversión a coprostanol, su incorporación a la membrana bacteriana o su adsorción a la superficie celular (Abedini, Zaghari, Jabbari, Salekdeh & Hashemi, 2023).
En conjunto, estas características probióticas han demostrado la utilidad de Enterococcus como suplemento alimenticio, al mejorar la absorción de nutrientes y nitrógeno, y disminuir la cantidad de coliformes totales sin afectar la presencia de otros microorganismos comensales probióticos como Lactobacillus (Park, Jeong, Lee & Kim, 2016). Como parte de la microbiota intestinal, Enterococcus forma parte del eje intestino-cerebro, actuando directamente sobre la salud mental del hospedero al ser capaz de producir neurotransmisores como serotonina, GABA y acetilcolina (Rutsch et al., 2020). Se ha reportado la actividad antidepresiva de una cepa de E. faecalis gracias a la disminución de la producción de citocinas inflamatorias en el hipocampo y el tracto gastrointestinal, y el aumento de la síntesis de citocinas antiinflamatorias como la IL-10, por lo que disminuye el proceso inflamatorio asociado con afecciones como la depresión y el trastorno obsesivo-compulsivo (Takahashi et al., 2019). Algunos preparados probióticos y postbióticos de Enterococcus ya se comercializan, y se ha demostrado su eficacia en la reducción de episodios de diarrea infecciosa tras tratamiento con antibióticos, y para atender el síndrome de colon irritable y mantenimiento general de la homeóstasis microbiana intestinal (Figura 4), (Popović et al., 2023; Graham et al., 2020).
Otras aplicaciones biotecnológicas
Las propiedades proteolíticas de Enterococcus han sido exploradas más allá de la maduración de alimentos fermentados y se ha propuesto su uso en la proteólisis de proteínas de la leche, para dar lugar a péptidos bioactivos como producto final, con actividades antihipertensivas, anticariogénicas, inmunomoduladoras y antimicrobianas (Graham et al., 2020). Sus proteasas y lipasas también tienen aplicaciones en el procesamiento de residuos de la industria alimentaria y, en conjunto con detergentes, en la industria textil. Se ha reportado la producción de proteasas y lipasas de interés industrial en cepas de E. faecium, E. durans y E. hirae y se ha propuesto su uso en la producción de lácteos (Ramakrishnan, Balakrishnan, Rai, Narayan & Halami, 2012).
Enterococcus forma una gran variedad de exopolisacáridos. Estos compuestos son emulsificantes y floculantes, por lo que pueden ser utilizados en la modificación de las propiedades reológicas de diversos productos (Kavitake, Devi, Delattre, Reddy & Shetty, 2023). Estudios mencionan que estos compuestos mejoran la textura de quesos madurados gracias a su alta capacidad higroscópica (Dapkevicius et al., 2021). Se ha observado que algunos expolisacáridos enterocóccicos tienen propiedades inmunomoduladoras, antioxidantes, antimicrobianas, citotóxicas y antitumorales (Kavitake et al., 2023). Adicionalmente, el exopolisacárido de Enterococcus sp. MG6 tiene actividad inmunomoduladora y anticancerígena, al inducir la producción de TNF-α, NO, IL-6 e IL-β, citocinas involucradas en el proceso inflamatorio relacionado con la respuesta inmune al cáncer (Sharma & Gosh, 2021).
Además de los diversos metabolitos de Enterococcus, los bacteriófagos que infectan a este género también han demostrado utilidad en el área biotecnológica. Un aspecto negativo del desarrollo de algunas cepas de Enterococcus en alimentos fermentados es la producción de aminas biógenas. Éstas son producto del metabolismo de desaminación de ciertos aminoácidos, que tienen efectos deletéreos en el ser humano como náuseas, diarrea y taquicardia (Doeun, Davaatseren & Chung, 2017). El bacteriófago 156 de E. faecalis controla la producción de las aminas biógenas tiramina y putrescina en los alimentos fermentados, mediante la regulación del crecimiento de Enterococcus spp. (del Rio et al., 2019). Otra aplicación de los bacteriófagos es su uso en el tratamiento de infecciones dentales provocadas por E. faecalis (Rodríguez-Lucas & Ladero, 2023).
Enterococcus: una perspectiva equilibrada
El género Enterococcus lleva consigo un estigma producto tanto de su plasticidad genómica y su correlación con entornos nosocomiales, como de su mismo nombre (p. ej. E. faecalis). Sin embargo, y como se ha expuesto en este artículo, el papel de Enterococcus en los alimentos y sus potenciales aplicaciones en el área de la biotecnología son muy amplias como para reducirlo a patógeno oportunista. Enterococcus no es el primer género de BAL (ni será el último) que preocupe desde el punto de vista clínico. Géneros GRAS como Lactobacillus pueden comportarse como patógenos en pacientes con diabetes mellitus, de la misma forma en que Enterococcus puede comportarse como patógeno oportunista en pacientes inmunocomprometidos (Rossi, Amadoro & Colavita, 2019). Se ha reportado que en Lactobacillus spp. existen numerosos genes de resistencia a antibióticos (tetraciclina, macrólidos, clindamicina y aminoglucósidos) y de virulencia (citolisina, sustancia de agregación, antígeno específico de endocarditis y gelatinasa) algunos de los que también se encuentran en Enterococcus spp. (Colautti, Arnoldi, Comi & Iacumin, 2022). Existen algunas cepas de Lactobacillus que, de igual forma que algunas cepas de Enterococcus, presentan resistencia a la vancomicina (Campedelli et al., 2019). Esto no se menciona con la finalidad de desacreditar la clasificación GRAS que ostenta Lactobacillus, sino para incitar una discusión acerca del enfoque que se le da a Enterococcus y resaltar que, así como tiene características de preocupación, también las tiene positivas y de utilidad para la tecnología alimentaria y la biotecnología, tal como sucede con otros microorganismos. Adicionalmente, es de resaltar que el género Enterococcus está presente en prácticamente cualquier alimento de fermentación láctica de manera natural, es decir, que se ha elaborado sin la adición de cultivos iniciadores, cuya fermentación se debe a la microbiota presente de manera nativa en las materias primas, sin que esto conlleve a considerar riesgoso el consumo del alimento por la presencia de este tipo de bacterias (Suvorov, 2020).
Conclusiones
El papel del género Enterococcus en la salud no está en discusión: existen infecciones nosocomiales que pueden llevar a la muerte, causadas por cepas de este microorganismo virulentas y resistentes a antibióticos. Sin embargo, pensar que todos los miembros de este género son patógenos oportunistas se ha convertido en una limitante en cuanto a la percepción de peligro cuando se encuentra como microbiota nativa de un alimento o respecto a su potencial aplicación biotecnológica. Con la finalidad de no caer en prejuicios sobre este género bacteriano, el estudio de los enterococos debe abordarse desde una perspectiva objetiva. Se necesita hacer análisis genotípicos y fenotípicos de cada cepa en particular para conocer las ventajas o las desventajas de su presencia en distintos entornos, ya que la profundización en el conocimiento científico de los microorganismos lleva a una mejor evaluación del papel que desempeñan en cada hábitat.











 nueva página del texto (beta)
nueva página del texto (beta)