Introducción
Fusarium spp. es un hongo filamentoso que produce hifas hialinas y septadas. Las diversas especies están ampliamente distribuidas en la naturaleza, son saprófitas del suelo y material en descomposición. Este hongo está fuertemente asociado como agente causal de enfermedades en plantas, tanto silvestres como de agricultura. No obstante, algunas especies pueden ocasionar una variedad de manifestaciones clínicas en los seres humanos inmunocompetentes o inmunocomprometidos; estas enfermedades son llamadas hialohifomicosis, por las características del hongo.1-4 Fusariosis o fusariomicosis son otros términos con los que se le conoce a las infecciones producidas por Fusarium spp. Actualmente, se conocen alrededor de 300 especies de este género de microorganismos.5 Se han descrito 10 complejos de importancia clínica, en los cuales se agrupan alrededor de 70 especies, algunas de ellas aún sin nombre asignado debido a que no se pueden distinguir fenotípicamente (Tabla 1).6-8 Es precisamente en este género que se empezó a usar la denominación de complejo de especies, integrado por microorganismos crípticos tan cercanos entre sí que aún falta por definir si se trata de nuevas especies.9
Tabla 1 Complejo de especies del género Fusarium de importancia clínica
| Complejo | Especies |
|---|---|
| Solani | F. falciforme |
| F. keratoplasticum | |
| F. lichenicola | |
| F. petroliphilum | |
| F. pseuddensiforme | |
| F. solani | |
| 18 especies sin nombre | |
| Oxysporum | F. oxysporum |
| 3 especies sin nombre | |
| Incarnatum-equiseti | F. equiseti |
| F. incarnatum | |
| 18 especies sin nombre | |
| Fujikuroi | F. acutatum |
| F. ananatum | |
| F. andyazi | |
| F. fujikuroi | |
| F. globosum | |
| F. guttiforme | |
| F. musae | |
| F. napiforme | |
| F. nygamai | |
| F. proliferatum | |
| F. ramigenum | |
| F. sacchari | |
| F. subglutinans | |
| F. temperatum | |
| F. thapsinum | |
| F. verticillioides | |
| Dimerum | F. delphinoides |
| F. dimerum | |
| F. penzigii | |
| 2 especies sin nombre | |
| Tricinctum | F. acuminatum |
| F. flocciferum | |
| 2 especies sin nombre | |
| Chlamydosporum | F. chlamydosporum |
| 3 especies sin nombre | |
| Concolor | F. anguioides |
| F austroafricanum | |
| F. concolor | |
| Sambucinum | F. armeniacum |
| F. brachygibbosum | |
| F. langsethiae | |
| F. sporotrichioides | |
| Lateritium | F. cassiae |
| F. lateritium | |
| F. magnoilae-champaca | |
| F. sarcochroum | |
| F. stilboides |
Las muestras biológicas donde puede aislarse Fusarium spp. son muy diversas e incluyen tejidos obtenidos por biopsia, líquido pleural, abdominal, sinovial y peritoneal, ojos, sangre, líquido cefalorraquídeo, especímenes respiratorios, lesiones ulcerativas, etcétera. La obtención de una muestra apropiada es esencial, así como en cantidad suficiente para realizar diversas pruebas (exámenes directos mediante microscopio y cultivos); además, el procesamiento de las muestras dentro de dos a cuatro horas después de recibirse en el laboratorio es crucial para la recuperación de los microorganismos. Estos criterios están establecidos para el procesamiento de estudios microbiológicos en general.
Los cultivos de hongos por lo general se llevan a cabo en agar dextrosa de Sabouraud con o sin antibióticos y con una temperatura de incubación entre 25 y 30 °C. En este momento, los procedimientos considerados como el estándar de oro en la identificación de Fusarium spp. son los fenotípicos, que incluyen las características de las colonias en ciertos medios de cultivo y las características microscópicas de las estructuras producidas por este microorganismo en el cultivo: fiálides, macroconidios, microconidios, mesoconidios y clamidosporas, entre las más importantes.10 El gran número de especies y la existencia de complejos de especies ha complicado la identificación de numerosos aislamientos, ya que se requiere personal con entrenamiento y experiencia en este campo de la microbiología, del cual con frecuencia se carece en los laboratorios convencionales; además, son necesarios microscopios más poderosos que los disponibles en la mayoría de los laboratorios clínicos. Por esta razón, una identificación precisa a nivel de especie debe involucrar procedimientos alternativos, los cuales raramente se llevan a cabo en forma rutinaria en los laboratorios de diagnóstico. Procedimientos como la reacción en cadena de la polimerasa (PCR) y la secuenciación de genes han resuelto obstáculos que presenta la identificación fenotípica de este hongo.11-13 Existen varios genes y regiones que se han propuesto para la identificación o confirmación molecular de este microorganismo, tales como el ITS (internal transcribed spacer), el gen de TEF1-α (translation elongation factor 1α), la región RPB2 (RNA polymerase II subunit B) y el gen de calmodulina.6,11-13
El Instituto de Estándares Clínicos y de Laboratorio ha propuesto los métodos que rigen las pruebas de susceptibilidad con antifúngicos, tanto para levaduras como hongos filamentosos.14 En el caso de Fusarium spp. no se han establecido puntos de corte que permitan categorizar a las cepas como susceptibles, intermedias o resistentes, aunque se han establecido valores de corte epidemiológico al menos para las especies de Fusarium más frecuentemente aisladas de patología humana: F. solani, F. oxysporum y F. verticillioides.15-17
En el mundo, son escasos los reportes de identificación con cepas clínicas o ambientales de Fusarium spp.,18-23 razón por la cual en este trabajo se propuso llevar a cabo la caracterización fenotípica y molecular de una colección de 116 cepas de Fusarium spp. en el Laboratorio de Micología del Departamento de Microbiología, Facultad de Medicina, Universidad Autónoma de Nuevo León, Monterrey, Nuevo León, México.
Material y métodos
Cepas de Fusarium spp.
Se utilizaron 116 cepas clínicas y ambientales de Fusarium spp. recolectadas en el Centro Regional de Control de Enfermedades Infecciosas, anexo al Departamento de Microbiología. Las cepas de origen clínico fueron 84 (ojos, uñas, esputo y otros especímenes respiratorios, sangre y piel) y se obtuvieron entre 1996 y 2022, en diferentes instituciones de salud en Nuevo León, Ciudad de México, San Luis Potosí y Jalisco; así como 32 de origen ambiental (muestras de suelo y raíces de plantas en los estados de Nuevo León, Ciudad de México, Guanajuato, San Luis Potosí y Jalisco). Se almacenaron por duplicado en crioviales de 2 mL con agua destilada estéril y glicerol a 10 %; se guardaron tanto a temperatura ambiente como a ´20 °C hasta su uso.
Identificación fenotípica
Las cepas almacenadas fueron subcultivadas en agar dextrosa de Sabouraud (BD Bioxon, México), agar papa dextrosa (BD Bioxon, México) y agar avena (agar papa dextrosa suplementado con 6 % peso/volumen [p/v] de avena) e incubadas a 30 °C por tres a cinco días, para después llevar a cabo la observación de las características de macro y micromorfología. La estimulación de la producción de conidios se llevó a cabo mediante crecimiento sobre agar Spezieller Nährstoffarmer (0.1 % p/v de fosfato monopotásico, 0.1 % p/v de nitrato de potasio, 0.05 % p/v de sulfato de magnesio heptahidrato, 0.05 % p/v de cloruro de potasio, 0.02 % p/v de glucosa, 0.02 % p/v de sacarosa y 2 % p/v de agar) y en agar agua (2 % de agar). Las placas Petri se incubaron a 30 °C durante siete a 14 días. Los medios de cultivo agar dextrosa de Sabouraud y agar papa dextrosa son comúnmente utilizados en laboratorios clínicos; el resto de los medios de cultivo, en investigación. Las observaciones al microscopio se llevaron a cabo mediante los procedimientos estándar de cinta adhesiva y microcultivo. La identificación de las características coloniales y microscópicas se llevaron a cabo conforme los lineamientos del Manual de Laboratorio de Fusarium.24
Identificación molecular
Las cepas se subcultivaron en agar dextrosa de Sabouraud por cinco días a 30 °C, después de lo cual su ADN genómico se extrajo conforme el método fenol-cloroformo,25 con adición de 0.1 UI de quitinasa. El ADN se utilizó para la amplificación de la región ribosomal de ITS1-5.8S-ITS2 (ITS) con los cebadores (primers) ITS5 (5´-GGA AGT AAA AGT CGT AAC AAGG-3´) e ITS4 (5´-TCC TCC GCT TAT TGA TAT GC-3´), mediante el siguiente proceso: exposición a 95 °C por cuatro minutos, después 30 ciclos de exposición a 94 °C por un minuto, 55 °C por 90 segundos y 72 °C por 90 segundos, con un período de extensión final de 72 °C por cinco minutos. Algunas cepas fueron posteriormente tipificadas por amplificación del gen de TEF1-α, RPB y/o el gen de la calmodulina. TEF1-α fue amplificado con los primers Fsp-EF1 (5´-ATG GGT AAG GAR GAC AAG AC-3´) y Fsp-EF2 (5´- GGA RGT ACC AGT SAT CAT GTT-3´) y conforme el siguiente programa: exposición por 95 °C por cuatro minutos, seguida por 35 ciclos de 94 °C por un minuto, 50.8 °C por 90 segundos y 72 °C por 90 segundos, con un período de extensión final de 72 °C por cinco minutos.26,27
La amplificación de la RPB se llevó a cabo con los primers RPB2-5f2 (5´-GGG GWG AYC AGA AGA AAG C-3´) y RP2-7cR (5´-CCC ATR GCT TGY TTR CCC AT-3´): exposición 94 °C por cinco minutos, seguida por 35 ciclos de 94 °C por un minuto, 58 °C por 30 segundos y 72 °C por dos minutos, con un período de extensión de 72 °C por cinco minutos.
El gen de la calmodulina se amplificó con los primers CL1 (5´-GAR TWC AAG GAG GCC TTC TC-3´) y CL2a (5´-TTT TTG CAT CAT GAG TTG GAC-3´): exposición a 94 °C por cinco minutos, seguida por 35 ciclos de 94 °C por un minuto, 55 °C por 45 segundos, 72 °C por un minuto y un período de extensión final de 72 °C por 10 minutos.
Se añadieron soluciones-Q (Qiagen, Hilden, Alemania) en todas las reacciones de PCR.
La amplificación de los productos fue secuenciada con el método Sanger en un secuenciador automático 3130 Genetic Analyzer (Applied Biosystems, Foster City, California, Estados Unidos). Las lecturas de secuenciación fueron depuradas con la eliminación de los extremos del cromatograma de mala calidad mediante el programa BioEdit28 y la identificación se llevó a cabo por comparación de secuencias de cepas previamente reportadas en ISHAM ITA Barcoding Database (http://its.mycologylab.org/) y MycoBank Database (https://www.mycobank.org/).
Resultados
En la Tabla 2 se presentan los resultados de la identificación fenotípica y molecular de las 116 cepas de Fusarium spp. que se estudiaron. Con base en las características macroscópicas y microscópicas distintivas, solo 39 cepas pudieron agruparse en cinco complejos de especies de Fusarium; no se logró llegar al nivel de especie: F. solani complejo de especies (n = 15), F. oxysporum complejo de especies (n = 6), F. fujikuroi complejo de especies (n = 11), F. incarnatum-equiseti complejo de especies (n = 3) y F. dimerum complejo de especies (n = 4). Las restantes 77 cepas no pudieron ser identificadas debido básicamente a la nula o escasa producción en los cultivos de estructuras como fiálides, conidios, clamidosporas y otras que son importantes para la tipificación fenotípica. En la figura 1 y en la tabla 3 se muestran algunas imágenes representativas y características fenotípicas de los complejos identificados. Mediante procedimientos moleculares se reconocieron las especies de las 77 cepas (y se buscó la confirmación de las 39 cepas mencionadas); no obstante, aun con los procedimientos de secuenciación en dos cepas solamente se llegó a la determinación como sensu lato (s. l.). En la tabla 2, ocho especies de Fusarium se indican con un asterisco, lo que significa que se agruparon en algún complejo en forma incorrecta cuando se utilizaron solamente los parámetros fenotípicos, lo que evidencia la poca precisión que brindan estos para la identificación de una especie de este género de hongo.
Tabla 2 Identificación fenotípica y molecular de 116 cepas de Fusarium spp.
| Identificación fenotípica (n = 116) | Identificación molecular (n = 116) |
|---|---|
| F. solani complejo de especies (n = 15) | F. falciforme (n = 6) |
| F. solani (n = 4) | |
| F. keratoplasticum (n = 3) | |
| F. coffeatum (n = 1)* | |
| F. oxysporum (n = 1)* | |
| F. oxysporum complejo de especies (n = 6) | F. oxysporum (n = 5) |
| F. keratoplasticum (n = 1) | |
| F. fujikuroi complejo de especies (n = 11) | F. verticillioides (n = 6) |
| F. proliferatum (n = 1) | |
| F. oxysporum (n = 3)* | |
| F. falciforme (n = 1)* | |
| F. incarnatum-equiseti complejo de especies (n = 3) | F. laceratum (n = 1) |
| F. longuifundum (n = 1) | |
| F. flocciferum (n = 1)* | |
| F. dimerum complejo de especies (n = 4) | F. delphinoides (n = 2) |
| F. Penigar (n = 2) | |
| Fusarium spp. (n = 77) | F. falciforme (n = 32) |
| F. verticillioides (n = 7) | |
| F. oxysporum (n = 7) | |
| F. keratoplasticum (n = 6) | |
| F. solani (n = 6) | |
| F. petroliphilum (n = 2) | |
| F. proliferatum (n = 4) | |
| F. annulatum (n = 1) | |
| F. nelsonii (n = 1) | |
| F. nygamai (n = 1) | |
| F. adiyazi (n = 1) | |
| F. concolor (n = 1) | |
| F. dimerum (n = 1) | |
| F. nanum (n = 1) | |
| F. mirum (n = 1) | |
| F. mucidum (n = 1) | |
| F. terrícola (n = 1) | |
| F. nirenbergiae (n = 1) | |
| F. equiseti s. l. (n = 1) | |
| F. fujikuroi s. l. (n = 1) |
*No corresponde al complejo. s. l.: sensu lato (en sentido amplio").
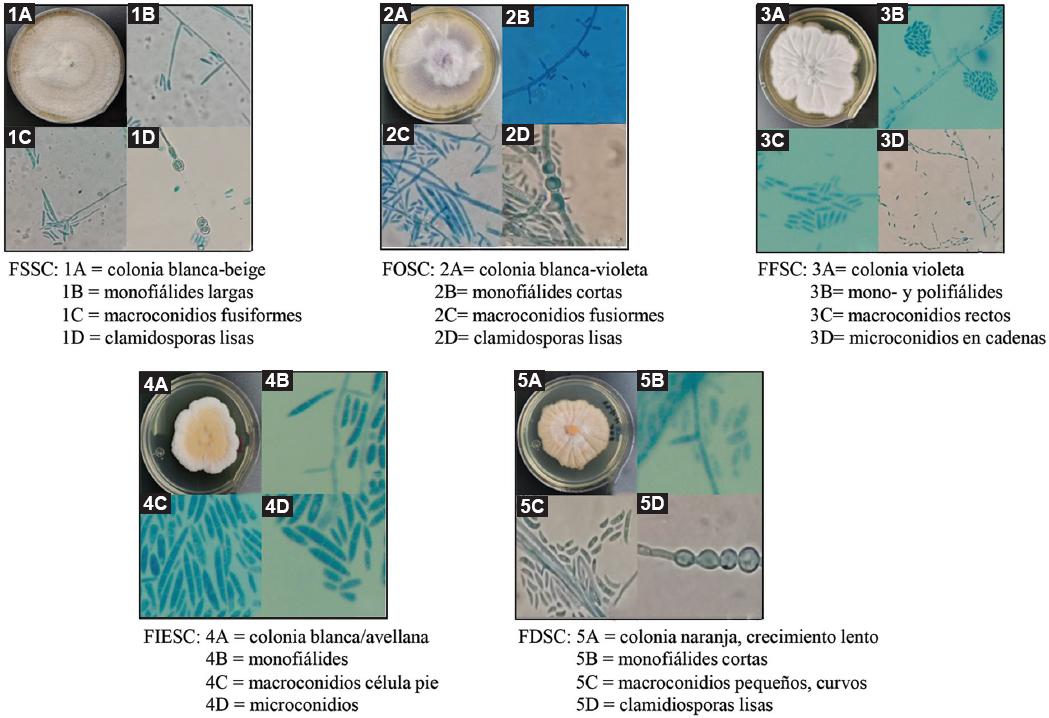
Figura 1 Características fenotípicas de los complejos de Fusarium identificadas en este estudio. Aumento de 400×.
Tabla 3 Características fenotípicas distintivas de los complejos identificados
| Complejo | Colonia | Fiálides | Macroconidios | Clamidosporas | Caraterísticas extras |
|---|---|---|---|---|---|
| Fusarium solani complejo especies | Color blanco a crema, crecimiento rápido | Monofiálides largas | Fusiformes, cilíndricos, ligeramente curvos, 3-5 septos | Lisas, rugosas, se agrupan en pares o solas entre las hifas o laterales a ella. Abundantes |
Microconidios abundantes con 0-2 septos, pigmentación marrón azulado cuando hay esporodoquios |
| Fusarium oxysporum complejo especies | Color blanco con el tiempo se vuelve morado, crecimiento rápido | Monofiálides cortas o medianas | Fusiformes, ligeramente curvos con célula apical y pie puntiaguda Normalmente 3 septos |
Lisas, rugosas, entre las hifas o terminales. Abundantes |
Microconidios abundantes, curvos y en su mayoría no septados |
| Fusarium fujikuroi complejo especies | Color vino a violeta, crecimiento rápido | Monofiálides y polifiálides, longitud variable | Fusiformes, lanceolados, delgados, ligera curvatura, célula pie trunca o recta. Normalmente 3-5 septos |
Ausentes | Microconidios abundantes, agrupados en cadenas o falsas cabezas |
| Fusarium incarnatum-equiseti complejo especies | Color blanco con el tiempo se vuelve avellana, café o melocotón, crecimiento rápido | Monofiálides cortas | Fusiformes a falcados, normalmente se producen en esporodoquio. Normalmente 3-7 septos | Lisas, rugosas, agrupadas en cadenas o solas entre las hifas. Escasas |
Microconidios escasos o ausentes |
| Fusarium dimerum complejo especies | Color naranja o albaricoque, crecimiento lento | Monofiálides cortas, abultadas | Pequeños, muy curvos con célula apical puntiaguda. Normalmente 1-3 septos | Lisas, agrupadas en cadenas o solas entre hifas o terminales. Escasas |
Microconidios, 0-2 septos |
Mediante la distinción molecular de las 116 cepas (Tabla 2), se identificaron 26 especies de Fusarium, que se agruparon en nueve complejos: F. solani en 52.6 %, seguido por F. oxysporum y F. fujikuroi, en 14.6 y 20 %, respectivamente. Si bien la especie más reconocida fue F. falciforme, solo algunas como F. falciforme (6/39; 15 %), F. keratoplasticum (3/10; 30 %) y F. solani s.s. (4/10; 40 %) fueron exitosamente agrupadas en F. solani complejo de especies mediante identificación fenotípica. Los resultados de identificación fenotípica fueron similares en otras especies; menos de la mitad de F. oxysporum s.s. (5/16; 31 %) y F. verticillioides (6/13, 46 %) fueron agrupadas en sus respectivos complejos. Cuatro especies fueron inconfundiblemente identificadas por las características morfológicas: F. delphinoides y F. penzigii (en F. dimerum complejo de especies) y F. lacertarum y F. lonfigundum (en F. incarnatum-equiseti complejo de especies).
En la tabla 4 se muestran los resultados de la identificación molecular según el origen clínico de las cepas. Se obtuvieron tipificaciones de las cepas en 22 especies agrupadas en siete complejos de especie, de los cuales F. solani, F. oxysporum y F. fujikuroi fueron los más frecuentes. Por su parte, en las cepas ambientales se observó un ligero cambio en la frecuencia en el orden de las caracterizaciones de los complejos de especie: F. solani, F. oxysporum y F. fujikuroi. En las cepas ambientales se identificaron nueve especies diferentes agrupadas en seis complejos (Tabla 5).
Tabla 4 Identificación molecular de 84 cepas de Fusarium de origen clínico
| Complejo | Frecuencia (%) | Especie |
|---|---|---|
| Solani | 51.2 | F. falciforme (n = 29) |
| F. keratoplasticum (n = 9) | ||
| F. solani (n = 9) | ||
| F. petroliphilum (n = 2) | ||
| Fujikuroi | 22.6 | F. verticillioides (n = 13) |
| F. proliferatum (n = 2) | ||
| F. nygamai (n = 1) | ||
| F. andiyazi (n = 1) | ||
| F. mirum (n = 1) | ||
| F. fujikuroi s. l. (n = 1) | ||
| Oxysporum | 12 | F. oxysporum (n = 9) |
| F. nirenbirgiae (n = 1) | ||
| Dimerum | 6 | F. delphinoides (n = 2) |
| F. penzigii (n = 2) | ||
| F. dimerum (n = 1) | ||
| Incarnatum-equiseti | 6 | F. coffeatum (n = 1) |
| F. mucidum (n = 1) | ||
| F. lancertarum (n = 1) | ||
| F. longifundum (n = 1) | ||
| F. equiseti s. l. (n = 1) | ||
| Concolor | 1.1 | F. concolor (n = 1) |
| Tricinctum | 1.1 | F. flocciferum (n = 1) |
s. l.: sensu lato (en sentido amplio).
Tabla 5 Identificación molecular de 32 cepas de Fusarium de origen ambiental
| Complejo | Frecuencia (%) | Especie |
|---|---|---|
| Solani | 56.2 | F. falciforme (n = 16) |
| F. keratoplasticum (n = 1) | ||
| F. solani (n = 1) | ||
| Oxysporum | 22 | F. oxysporum (n = 7) |
| Fujikuroi | 12.4 | F. proliferatum (n = 3) |
| F. annulatum (n = 1) | ||
| Incarnatum-equiseti | 3.1 | F. nanum (n = 1) |
| Chlamidosporum | 3.1 | F. nelsonii (n = 1) |
| Staphyleae | 3.1 | F. terrícola (n = 1) |
Discusión
Las especies que forman parte del género Fusarium son reconocidas como microorganismos fitopatógenos. Sin embargo, existen diversas especies que han sido documentadas como patógenos oportunistas en los seres humanos, siendo el principal grupo de riesgo las personas que presentan algún trastorno hematológico maligno, que se han sometido a algún trasplante o que padecen síndrome de inmunodeficiencia adquirida.22
En el presente trabajo, se identificaron fenotípica y molecularmente 116 cepas de Fusarium spp., 84 clínicas y 32 ambientales, todas se agruparon en 26 del género Fusarium pertenecientes a nueve complejos: solani, oxysporum, fujikuroi, dimerum, incarnatum-equiseti, chlamydosporum, concolor, staphyleae y tricinctum. Solani fue el complejo más frecuente, tanto en cepas ambientales como clínicas, y dentro de ese complejo la especie más aislada fue F. falciforme; le siguieron el complejo fujikuroi y oxysporum en las cepas clínicas y oxysporum y fujikuroi, en las ambientales.
Estos resultados concuerdan con lo reportado en la bibliografía, ya que las especies de Fusarium que se han reportado como patógenas oportunistas de humanos están agrupadas en los siguientes 10 complejos: solani, oxysporum, incarnatum-equiseti, fujikuroi, dimerum, tricinctum, chlamydosporum, concolor, sambucinum y lateritium. De todos ellos, se identificaron ocho (excepto sambucinum y lateritium). Las especies de Fusarium que se encuentran con mayor frecuencia en infecciones humanas son las que comprenden los complejos solani, oxysporum y la especie F. verticillioides del complejo fujikuroi.22,29,30
Este estudio es concluyente respecto a que las características morfológicas macroscópicas y microscópicas son de valor limitado para la identificación precisa de las especies Fusarium. Los cultivos han sido muy criticados y quedan claras sus desventajas: la lentitud del crecimiento fúngico, la falta de estandarización, las características morfológicas inestables, la dependencia de condiciones ambientales y el alto grado de subjetividad. Lo más que se puede lograr es la tipificación de un complejo y llegar a una especie con los parámetros fenotípicos es prácticamente imposible. Por lo anterior, en el escenario clínico, en los laboratorios de microbiología es imperante la implementación de procedimientos moleculares y el entrenamiento del personal para llegar a identificaciones precisas y en un tiempo más corto (uno o dos días), en particular en microorganismos en los cuales se han descrito complejos de especies. Solo recientemente se está logrado ese objetivo, pero desafortunadamente queda confinado prácticamente a laboratorios de referencia, ya que la infraestructura requerida y el personal con experiencia se reserva a esos sitios.
Por ahora, el cultivo sigue siendo muy importante porque a partir de conidios de Fusarium spp. se realizan las pruebas de susceptibilidad in vitro que pueden ayudar en la selección de un antifúngico para el tratamiento de los pacientes. En la micología moderna, se enfrentan obstáculos para la caracterización precisa por procedimientos convencionales. La columna vertebral del diagnóstico de las enfermedades fúngicas invasivas es la microscopia, el cultivo y la histopatología. Un buen microscopio, una buena preparación y personal entrenado son esenciales, y aunque estos recursos constituyen una excelente propuesta para el trabajo en los laboratorios clínicos, la incorporación de métodos moleculares es imperante en los laboratorios de diagnóstico, ya que permitirá con mayor confianza la identificación precisa de numerosos hongos patógenos.
Se han descrito otros hongos que también están agrupados en secciones o complejos de especies: Aspergillus, en cuyas secciones están incluidas numerosas especies que son crípticas,31-33 Scedosporium,34-36 Candida parapsilosis, Candida glabrata,37 Sporothrix schenckii,38-40 por mencionar solo algunos, y en todos ellos solo las herramientas de biología molecular ayudan a una identificación a nivel de especie. El conocimiento y aplicación de dichos avances en el laboratorio clínico sin duda permitirán una sustancial mejora del tratamiento de numerosas enfermedades fúngicas, ya que fundamentalmente depende de la correcta identificación de las especies implicadas.











 nueva página del texto (beta)
nueva página del texto (beta)


