Introducción
La enfermedad de Stargardt o distrofia macular de Stargardt (STGD) (Online Mendelian Inheritance in Man [OMIM]: 248200) es una forma autosómica recesiva de distrofia macular de inicio juvenil descrita por primera vez en 1909 por el oftalmólogo alemán Karl Stargardt1. Es el tipo más común de distrofia macular hereditaria recesiva, con una incidencia estimada de 1 en cada 10,000 nacidos vivos. Representa aproximadamente el 7% de todas las enfermedades de la retina, y se presenta típicamente en niños y adultos jóvenes2,3. El diagnóstico de STGD se basa clásicamente en los antecedentes familiares, la agudeza visual, el examen del fondo de ojo, la angiografía con fluoresceína, la autofluorescencia del fondo y la electrorretinografía4.
La forma típica de STGD (STGD1, OMIM #248200) es causada por mutaciones en el gen del transportador de casete de unión a ATP 4 (ABCA4) ubicado en 1p22.1, con una transmisión autosómica recesiva homocigótica o heterocigótica compuesta. Las mutaciones del gen de la proteína 1 similar a la prominina (PROM1), ubicado en 4p15.32, causan una forma autosómica dominante (STGD4, OMIM #603786). Las mutaciones dominantes del gen de la elongasa 4 de ácidos grasos (ELOVL4) (6q14.1) causan distrofias maculares similares a Stargardt (STDG3, OMIM #600110)5.
La patogénesis de la enfermedad de Stargardt asociada con mutaciones del gen ABCA4 ha sido bien establecida. ABCA4 participa en el ciclo retinoide como una «flipasa» que transporta el subproducto N-retinilideno-fosfatidiletanolamina (N-retinilideno-PE) a través de las membranas del disco. Mediante este mecanismo, ABCA4 previene la acumulación de retinoides en el espacio subcelular después del fotoblanqueo de rodopsina5. La ausencia del transporte de N-retinilideno-PE a través de la membrana del disco conduce a su atrapamiento y reacción con todo-trans-retinal (atRAL) para formar di-retinoide-piridinio-fosfatidiletanolamina (A2PE). Cuando los fotorreceptores se renuevan, el A2PE se convierte en di-retinoide-piridinio-etanolamina (A2E), un dímero de la vitamina A, que queda atrapado permanentemente en el epitelio pigmentario de la retina (EPR) porque no puede hidrolizarse. El A2E es un componente importante de la lipofuscina, un sello distintivo de la degeneración celular5. El atRAL acumulado puede volver a asociarse con la opsina, y el complejo resultante es menos efectivo para activar la cascada visual que la rodopsina fotoactivada; esto puede explicar la prolongada adaptación a la oscuridad encontrada en pacientes con STGD5,6. La lipofuscina puede acumularse hasta 5 veces por encima de los valores normales, y el exceso de A2E en el EPR afecta su función y supervivencia porque actúa como un detergente que compromete la membrana celular y las funciones metabólicas. Además, en presencia de oxígeno y luz de longitud de onda azul, la lipofuscina forma radicales libres de epóxido que inducen la muerte de células del EPR6. La muerte de las células del EPR conduce a la degeneración secundaria de fotorreceptores7.
La activación microglial por proteínas retinianas endógenas que surgen de fotorreceptores lesionados juega un papel importante en la patogénesis de la degeneración retiniana. En un estudio exhaustivo en un modelo de ratón de STGD (causado por la eliminación retardada de atRAL de la retina), se demuestra una contribución importante de la activación microglial mediada por el receptor Toll de tipo 4 (TLR4) en la inflamación de la retina, que agrava la muerte de las células de la retina8.
El gen del TLR4 ha sido asociado en varios informes con apoptosis de células retinianas. Por ejemplo, el TLR4 y su vía de señalización se han relacionado con la muerte de las células ganglionares de la retina (RGC)9-11 y con la apoptosis de las RGC y las células endoteliales de la retina en modelos con alto contenido de glucosa que imitan el microambiente de la retinopatía diabética12,13.
El TLR4 es un miembro de la familia de receptores de reconocimiento de patrones que son una parte clave del sistema inmune innato. Se ha identificado en el sistema nervioso central y en la retina9. El gen del TLR4 está ubicado en el locus 9q33.1 que abarca una región genómica de ~13.3 kb con cuatro exones (Ensamblado; GRCh38.p2, RefSeqGene; NG_011475.1, ID de gen; TLR4 7099)14 que codifica un receptor de reconocimiento de patógenos transmembrana involucrado en la detección de lipopolisacáridos (LPS) de bacterias Gram negativas y otros ligandos exógenos o endógenos15. Este gen muestra varios polimorfismos de un solo nucleótido (SNP) que presumiblemente influyen en la actividad y la función de su producto16. Se ha demostrado que los polimorfismos de TLR4 en humanos pueden contribuir a la gravedad de las enfermedades degenerativas oculares como el glaucoma17,18 o la degeneración macular relacionada con la edad19.
Con base en estudios que sugieren la participación del TLR4 en la patogénesis de la enfermedad de Stargardt y la asociación de polimorfismos del TLR con trastornos degenerativos de la retina, decidimos evaluar la asociación de SNP en regiones codificantes y no codificantes del gen TLR4 (locus 9q32- q33) con la enfermedad de Stargardt macular en una familia con diagnóstico clínico de enfermedad de Stargardt. Sorprendentemente, esta marca genética ha sido descrita como proapoptótica en otros estudios.
Pacientes y métodos
Pacientes
Se obtuvo ADN de 4 pacientes afectados (4 mujeres) y 12 familiares no afectados de una familia mexicana multigeneracional. El pedigrí de la familia afectada se muestra en la figura 1. Se incluyeron un total de 109 (40 hombres y 69 mujeres; edad 63.28 ± 7.93 años) muestras de control de una población mexicana emparejada. Los pacientes diagnosticados con STGD se sometieron a un examen ocular completo, angiografía con fluoresceína y tomografía de coherencia óptica de alta definición (HD-OCT). Además, se realizó una electrorretinografía para corroborar el diagnóstico de STGD. La fotografía del fondo de ojo, la fotografía de fondo libre de rojo y las imágenes de HD-OCT de pacientes con STGD se presentan en la figura 2. Los sujetos control tenían una historia clínica completa, ninguno de ellos tenía antecedentes familiares de enfermedad macular, y fueron examinados cuidadosamente para descartar cualquier enfermedad ocular. Se obtuvo el consentimiento informado de todas las personas que participaron en el estudio. El estudio fue aprobado por el comité de ética local y cumplió las directrices de la declaración de Helsinki. Todos los detalles del estudio se explicaron a los pacientes y a los sujetos control antes de obtener el consentimiento informado para el cribado genético.

Figura 1 Pedigrí de la familia afectada por distrofia macular de Stargardt (STGD). Las personas afectadas se muestran en sombreado y los sujetos sanos se muestran como figuras abiertas. Los círculos representan mujeres y los cuadrados representan hombres. Cuatro hermanos en la segunda generación (II: 8, II: 9, II: 10 y II: 22) están afectados. Este patrón es compatible con la transmisión autosómica recesiva observada en la STGD.
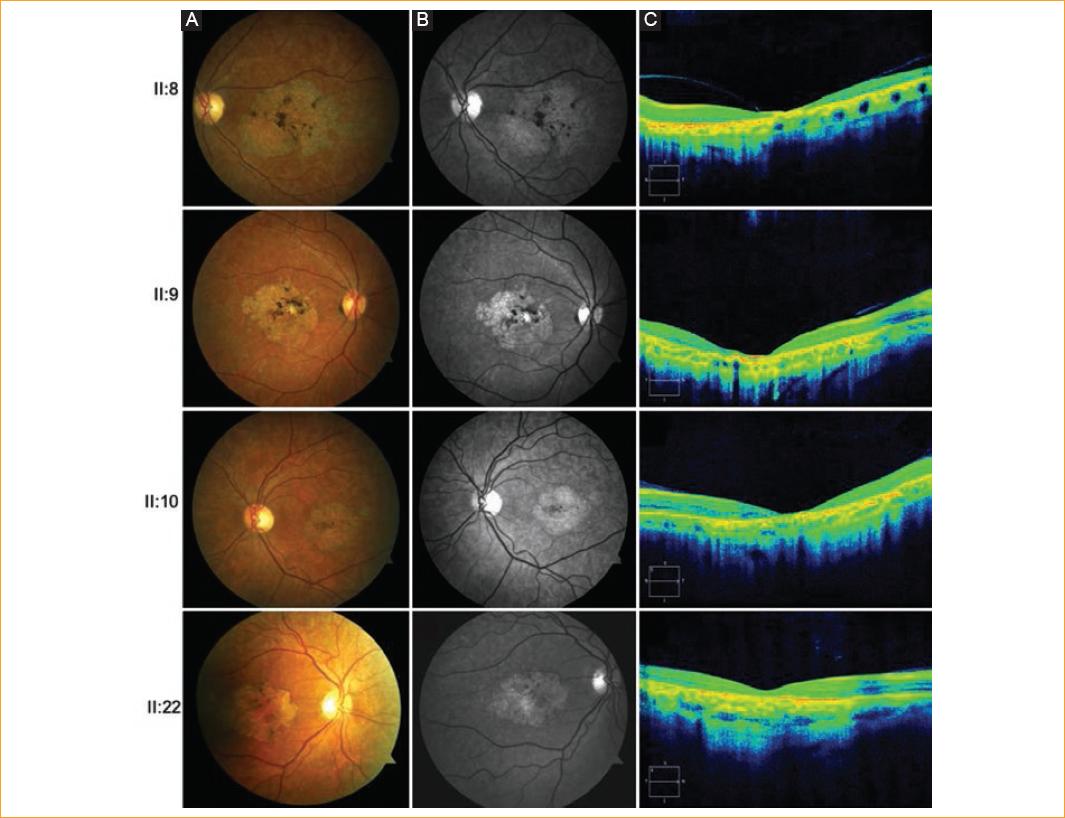
Figura 2 Imágenes representativas de los estudios diagnósticos de sujetos afectados. Fotografía de fondo (A), fotografía de fondo libre de rojo (B) e imágenes de tomografía de coherencia óptica macular de alta definición (C) de pacientes con enfermedad de Stargardt (STGD). Las filas 1, 2, 3 y 4 corresponden a imágenes de diagnóstico de II: 8, II: 9, II: 10 y II: 22, pacientes con STGD.
Preparación de muestras y genotipado
Los SNP objetivo se encuentran en regiones no codificantes del gen TLR4 (locus 9q32-q33); rs1927911 G/A, rs12377632 T/C y rs2149356 G/T dentro de los intrones, rs11536889 G/C dentro de la región 3 no traducida y rs4986790 A/G (Asp299Gly) ubicado en el tercer exón (variante de transcripción 1; RefSeq: NM_138554.3). La posición de los polimorfismos es 8595, 11271, 12740, 16672 y 13843, respectivamente (Ensamblaje; GRCh38.p2, RefSeqGene; NG_011475.1, ID de gen; TLR4 7099).
El ADN genómico se extrajo de leucocitos de sangre periférica y se purificó con el kit para sangre de Qiagen QIAmp DNA (Qiagen, Valencia, CA, EE.UU.). El genotipado de los SNP objetivo se realizó utilizando ensayos de genotipado de SNP con TaqMan validados (TaqMan: Applied Biosystems, Inc. [ABI], Foster City, CA). Los SNP se amplificaron utilizando reacción en cadena de la polimerasa (PCR) en tiempo real con 0.5 μM de cebadores y Master Mix para genotipado de TaqMan (TaqMan: Applied Biosystems, Inc. [ABI], Foster City, CA) con 30 ng de ADN templado en la mezcla de amplificación (25 μl). La señal de fluorescencia de la sonda se detectó con el sistema de PCR en tiempo real StepOnePlus (Applied Biosystems, Inc. [ABI], Carlsbad, CA).
Análisis estadístico
Se utilizó el software estadístico IBM SPSS (versión 21.0, SPSS, Inc., Chicago, IL) para los análisis estadísticos. El equilibrio de Hardy-Weinberg se analizó utilizando frecuencias de genes obtenidas mediante un simple conteo de genes y la prueba Chi-cuadrada (X2)bcon la corrección de Yates para comparar los valores observados y esperados. Las diferencias en las frecuencias de genotipo entre los casos y los controles se analizaron mediante la prueba exacta de Fisher o X2 dependiendo de los recuentos celulares. Los haplotipos inferidos y el LD (desequilibrio del ligamiento) cuantificados entre los loci bialélicos se estimaron utilizando el software en línea SNPStats (http://bioinfo.iconologia.net/SNPStats). La importancia de una asociación se determinó mediante análisis de tablas de contingencia utilizando pruebas de X2 o prueba exacta de Fisher. La significancia estadística se definió como una p < 0.05.
Resultados
Frecuencias de alelos y genotipos
Las frecuencias alélicas de SNP en pacientes con STGD, sus familiares y los sujetos control se muestran en la tabla 1. Los SNP rs1927911 G/A, rs12377632 T/C, rs2149356 G/T, rs11536889 G/C y rs4986790 A/G se genotipificaron en todos los sujetos y se encontraban en equilibrio de Hardy-Weinberg. La frecuencia del alelo menor de rs4986790 A/G (G) fue significativamente mayor en pacientes con STGD en comparación con los sujetos control (25 frente a 1%; p = 0.0012).
Tabla 1 Frecuencia de alelos SNP en los grupos de STGD y control
| SNP/alelos | STGD | Familiares | Control | p | p | |
|---|---|---|---|---|---|---|
| n = 8* | n = 24 | n = 218* | caso vs. control | general | ||
| rs4986790 | A/G | 2 (0.25) | 1 (0.04) | 3 (0.01) | 0.00120 | 0.00772 |
| rs1927911 | G/A | 2 (0.25) | 6 (0.25) | 56 (0.26) | 0.71229 | 0.99651 |
| rs12377632 | T/C | 3 (0.375) | 5 (0.21) | 105 (0.48) | 0.81624 | 0.06467 |
| rs2149356 | G/T | 4 (0.5) | 7 (0.29) | 60 (0.28) | 0.32393 | 0.38194 |
| rs11536889 | G/C | 0 (0) | 0 (0) | 28 (0.13) | 0.59151 | 0.26700 |
*Frecuencia del alelo menor seguida de su proporción.
SNP: polimorfismos de un solo nucleótido; STGD: distrofia macular de Stargardt.
Las frecuencias de genotipo de los SNP analizados se muestran en la tabla 2. El genotipo que porta el alelo menor de rs4986790 (AG) fue más frecuente en los pacientes con STGD (50%) en comparación con sus familiares y los sujetos control no relacionados (8 y 2.75%, respectivamente), con significación estadística (p = 0.0048).
Tabla 2 Frecuencia de genotipos en polimorfismos de un solo nucleótido de TLR4 objetivo
| Posición del gen | SNP | Posición del SNP* | Ubicación del SNP | Genotipos | STGD | Familiares | Control | p |
|---|---|---|---|---|---|---|---|---|
| n = 4 | n = 12 | n = 109 | ||||||
| 9q32-q33 | rs4986790 | 13843 | Exón | A/A | 2 (0.5) | 11 (0.92) | 106 (0.9724) | 0.0048 |
| A/G | 2 (0.5) | 1 (0.08) | 3 (0.0275) | |||||
| G/G | 0 (0) | 0 (0) | 0 (0) | |||||
| rs1927911 | 8595 | Intrón | G/G | 2 (0.5) | 6 (0.5) | 64 (0.587) | 0.55 | |
| A/G | 2 (0.5) | 6 (0.5) | 34 (0.312) | |||||
| A/A | 0 | 0 (0) | 11 (0.101) | |||||
| rs12377632 | 11271 | Intrón | T/T | 2 (0.5) | 7 (0.58) | 31 (0.284) | 0.29 | |
| C/T | 2 (0.5) | 5 (0.42) | 51 (0.468) | |||||
| C/C | 0 | 0 (0) | 27 (0.248) | |||||
| rs2149356 | 12740 | Intrón | G/G | 1 (0.25) | 6 (0.5) | 61 (0.56) | 0.44 | |
| T/G | 2 (0.5) | 5 (0.42) | 36 (0.33) | |||||
| T/T | 1 (0.25) | 1 (0.08) | 12 (0.11) | |||||
| rs11536889 | 16672 | 3 × UTR | G/G | 4 (1) | 12 (1) | 84 (0.771) | 0.36 | |
| C/G | 0 (0) | 0 (0) | 22 (0.202) | |||||
| C/C | 0 (0) | 0 (0) | 3 (0.028) |
*RefSeqGene, NG_011475.1; ID del gen, TLR4 7099.
SNP: polimorfismo de un solo nucleótido; STGD: distrofia macular de Stargardt; UTR: región no traducida.
Las frecuencias de alelos y genotipos de los SNP restantes no fueron significativas entre los casos y los sujetos control (p > 0.05). Los familiares no afectados de los pacientes con STGD mostraron frecuencias de alelos y genotipos similares a las observadas en los sujetos control.
Análisis de haplotipos
Los SNP etiqueta se ubicaron en un bloque de haplotipos, y la magnitud del LD entre los loci bialélicos fue muy alta, con una D > 0.98 pareada para todos los casos al comparar pacientes con STGD con los sujetos control. Los haplotipos con una frecuencia superior al 1% se muestran en la tabla 3. Encontramos un haplotipo de riesgo significativo. Como se muestra en la tabla 3, el haplotipo GGTTG alcanzó el cociente de probabilidad (OR) más alto (p = 0.011; OR: 33.84; intervalo de confianza del 95%: 2.38-481.17). Curiosamente, el alelo menor de rs4986790 AG (G) está presente en el haplotipo de riesgo.
Tabla 3 Frecuencias de haplotipos de SNP etiqueta en el gen TLR4 entre los casos y los sujetos control
| SNP etiqueta | Control | STGD | Familiares | Asociación d haplotipos con Stargardt* | p | ||||
|---|---|---|---|---|---|---|---|---|---|
| rs4986790 | rs1927911 | rs2149352 | rs12377632 | rs11536889 | n = 109 | n = 4 | n = 12 | OR (IC 95%) | |
| A | G | G | C | G | 0.3532 | 0.25 | 0.2083 | 1 | |
| A | A | T | T | G | 0.2523 | 0.25 | 0.25 | 1.57 (0.21-11.71) | 0.66 |
| A | G | G | T | G | 0.2385 | 0.25 | 0.5 | 1.32 (0.15-11.30) | 0.8 |
| G | G | T | T | G | 0.0138 | 0.25 | 0.0417 | 33.84 (2.38-481.17) | 0.011 |
*Los valores de OR se calcularon utilizando tablas de frecuencia de dos vías con los grupos control y con Stargardt.
IC: intervalo de confianza; OR: cociente de probabilidades; SNP: polimorfismos de un solo nucleótido.
Discusión
Los TLR son proteínas transmembrana capaces de activar vías de señalización de respuestas inflamatorias e inmunes. Se expresan en el sistema inmune innato, así como en el sistema nervioso central, incluida la retina. Hasta el momento se han identificado 13 TLR (TLR1-TLR13) en humanos9. El TLR4 es un receptor transmembrana involucrado en la detección de LPS de bacterias Gram negativas y muchos otros ligandos exógenos o endógenos. Tiene un dominio extracelular (rico en leucina) responsable del reconocimiento, un dominio transmembrana y un dominio intracelular (similar al dominio interleucina [IL]-1)15. Después de la activación, varias proteínas están involucradas en la señalización intracelular: Molécula adaptadora que induce interferón y contiene al dominio TIR (TRIF) y gen 88 de respuesta primaria de diferenciación mieloide (MYD88)20. El TLR4, a través del factor nuclear kappa B (NF-B), es capaz de iniciar la producción de citocinas proinflamatorias como IL-1, IL-6 y el factor de necrosis tumoral-α (TNF-α). Además, al activar el factor regulador 3 de interferón (IFN) (IRF3), inicia la expresión de genes inducibles por IFN e IFN-β21. Todas las vías mencionadas controlan la supervivencia celular y la apoptosis22.
El gen del TLR4 humano muestra varios SNP que se han asociado con una amplia variedad de enfermedades infecciosas y no infecciosas, incluyendo artritis reumatoide23,24, asma bronquial25, periodontitis crónica22, susceptibilidad a aspergilosis pulmonar y tuberculosis26,27, cáncer de pulmón28 y riesgo de úlcera péptica29.
Los SNP del gen TLR4 presumiblemente influyen en la actividad y la función de su producto16. Por ejemplo, las variaciones del gen TLR4 rs4986790 y rs4986791 conducen a variantes sin sentido (Asp299Gly y Thr399Ile, respectivamente) que alteran el dominio extracelular de la proteína, y afectan la función del TLR4 al disminuir su capacidad de respuesta30,31. El mecanismo por el cual estos SNP inducen hiporreactividad aún no se ha aclarado, pero actualmente se sabe que estas mutaciones inducen cambios conformacionales que afectan el plegamiento y la estabilidad de las proteínas, las interacciones con las proteínas mensajeras y la expresión en la superficie celular32. Además, se ha demostrado que Asp299Gly induce un reclutamiento deficiente de MYD88 y TRIF33.
Como se mencionó anteriormente, el gen del TLR4 se ha asociado con la apoptosis de las células de la retina, incluidas las RGC y las células endoteliales de la retina9-13, pero de manera intrigante, los polimorfismos del TLR4 rs4986790 y rs4986791 pueden inducir la apoptosis de las células estrelladas hepáticas activadas al reducir a fosfo-ERK y Bcl-234.
La apoptosis del EPR6 y la degeneración de fotorreceptores7 son fenómenos característicos de la STGD. Aunque la fisiopatología de estos procesos se ha establecido con base en las mutaciones del gen ABCA4 y sus efectos en la STGD5-7, se debe explorar la contribución de otros genes para descubrir otros mecanismos de lesión en la STGD.
De forma interesante, rs4986790 se ha descrito como un promotor de la apoptosis34. Se debe evaluar la importancia de este efecto, pero para la STGD, podría estar relacionado con la activación microglial mediada por el TLR4, como se sugirió en estudios previos8.
Conclusión
El alelo menor de los SNP rs4986790 y los genotipos que lo portaban se relacionaron con la STGD clínica en una familia. Somos conscientes de que la principal limitación de nuestro estudio es el bajo número de pacientes incluidos, pero consideramos que la evidencia en estos y otros informes respalda que el gen TLR4 y sus polimorfismos deberían considerarse en futuros estudios para determinar si son marcadores moleculares útiles para el diagnóstico, pronóstico o tratamiento de la STGD.











 nova página do texto(beta)
nova página do texto(beta)


