Introducción
El tomate es una de las hortalizas ampliamente producidas y consumidas a nivel mundial como producto fresco y para industrialización (Kimura y Sinha, 2008). El rendimiento promedio del Perú fue de 41.5 t ha-1 en el año 2017, siendo las principales provincias productoras: Ica, Lima y Arequipa (SIEA, 2017; INEI,2018). Sin embargo, la rentabilidad se ha visto mermada por diferentes factores y entre ellos problemas patológicos. Tal es el caso de Fusarium oxysporum f. sp. lycopersici (Sacc.) Snyder y Hans, agente causal de la marchitez vascular en el cultivo de tomate (Blancard, 2011). Este patógeno es responsable de numerosas pérdidas económicas y actualmente se encuentra distribuido por todos los continentes (González et al., 2012). Se conoce que hasta el momento F. oxysporum f. sp. lycopersici (Fol) ha sido agrupado en tres razas fisiológicas (Raza 1, 2 y 3) de acuerdo con Hirano y Arie (2006) y Arie (2010). Fol ingresa por el sistema radicular y se desplaza a través del tejido vascular para habitar y obstruir los vasos del xilema, lo que conlleva al estrés hídrico severo en la planta, cuya sintomatología se manifiesta en el amarillamiento de las hojas, iniciando por la parte del tercio inferior de un solo lado en los foliolos, acompañado de una decoloración vascular, marchitez y finalmente la muerte de la planta (Srinivas et al., 2019). Durante la infección Fol secreta algunas enzimas que degradan la pared celular de la planta, tal es el caso de las poligalacturonasas (PG) a partir de las cuales se han diseñado oligonucleótidos basadas sobre las diferencias nucleotídicas de los genes de la exopoligalacturonas (pgx4) y endopoligalacturonas (pg1) que poseen los tipos patogénicos (Hirano y Arie, 2006; Balogun et al., 2008; Çolak y Bicici, 2013). Así mismo, otros oligo-nucleótidos fueron diseñados a partir de proteínas únicas secretadas a nivel del xilema, llamadas proteínas SIX (Secreted In Xylem, por sus siglas en inglés), tres de las cuales, AVR1 (SIX4), AVR2 (SIX3) y AVR3 (SIX1) promueven la virulencia y colonización en el cultivo de tomate (Lievens et al., 2009). Por la supresión de mecanismos de resistencia basales, el conjunto de estas proteínas determina la virulencia del patógeno hacia un hospedante en particular (Hirano y Arie, 2006; González et al., 2012; Inami et al., 2012). Las tres razas fisiológicas de Fusarium oxysporum f. sp. lycopersici solo pueden ser detectados mediante métodos basados en la PCR y/o acorde a su capacidad de infectar a un conjunto de cultivares diferenciales de tomate y la respuesta de susceptibilidad y resistencia frente al aislamiento de Fol (Hirano y Arie, 2006; Arie, 2010; Kashiwa et al., 2016). En el Perú la identificación de las especies de Fusarium se basa en características morfológicas y se asumen formas especiales de acuerdo al síntoma y al huésped de donde se aisló. A nivel mundial existen técnicas moleculares para el diagnóstico de Fol, pero aún no han sido implementadas en el país y por ende no existe una identificación de Fusarium oxysporum f. sp. lycopersici y sus razas. Por lo expuesto, el objetivo de este trabajo es identificar mediante técnicas moleculares y empleo de cultivares diferenciales razas de Fusarium oxysporum asociados a la marchitez de tomate provenientes de la costa del Perú.
Materiales y métodos
Procedencia de las muestras
Los aislamientos de Fusarium fueron proporcionados por la Clínica de Diagnosis de Fitopatología para su identificación molecular. Los aislamientos fueron enviados de las principales zonas productoras: Ica (6), Lima (1) y Arequipa (1) en los años 2012 al 2016. Las muestras fueron colectadas de plantas con síntomas de marchitez y para el envío se tomaron plantas completas acompañadas de sustrato colocadas en bolsas oscuras y enviadas en cajas de Tecnopor; para luego ser procesadas en el laboratorio de la Clínica de Diagnosis de Fitopatología. Los controles positivos, se emplearon las tres razas de F. oxysporum f. sp. lycopersici MAFF 305121 (Raza 1), JCM 12575 (raza 2) y Ch12-1A (raza 3), provistos por la Universidad de Agricultura y Tecnología de Tokio (Tabla 1).
Tabla 1. Aislamientos de Fusarium oxysporum obtenidos del cultivo de tomate que fueron empleados para el estudio
| Nombre del aislamiento | Localización | Fecha de procedencia | |
| HEI 1 | Ica | 11-2013 | |
| HEI 2 | Cachiche-Ica | 12-2013 | |
| HEI 3 | Majes-Arequipa | 06-2013 | |
| HEI 5 | Subtanjalla-Ica | 12-2014 | |
| HEI 6 | Mala-Lima | 03-2016 | |
| HEI 9 | Ica | 12-2014 | |
| HEI 10 | Ica | 03-2012 | |
| HEI 11 | Ica | 04-2016 | |
| MAFF 305121 | Raza 1 | TUAT | Kawabe et al. (2005) |
| JCM 12575 | Raza 2 | TUAT | Inami et al. (2012) |
| Ch12-1A | Raza 3 | TUAT | Inami et al. (2012) |
Material biológico
Los aislamientos fueron obtenidos a partir de plantas con síntomas de marchitez y decoloración vascular del cultivo, los tejidos se lavaron con agua potable para eliminar la materia orgánica presente, seguido con agua destilada estéril, dentro de la cámara de siembra se realizaron cortes de pequeñas porciones de tallo y raíz que se colocaron en hipoclorito de sodio al 0.5 % por 3 minutos, se secó sobre un papel toalla por 20 minutos, transcurrido el tiempo se procedió a sembrar cuatro puntos en medio KOMADA fueron incubadas a 25 °C por 5 días (Carrillo-Fasio et al., 2003; Komada, 1975). De cada aislamiento se realizó un cultivo monospórico, para lo cual una porción de 0.5 cm2 de PDA con tejido de micelio de 7 días se colocó en un tubo de vidrio que contenía 10 mL de agua destilada estéril, se mezcló en vortex y contabilizó por campo visual de 10 X, un número de 10 a 20 conidios, se tomó 50 µL y se extendió sobre un medio agar agua, luego de 18 horas se evaluó y seleccionó las conidios germinadas y se sembró en medios PDA (Duarte et al.,2016).
Identificación morfológica
Para la caracterización morfológica de F. oxysporum se tomaron en cuenta características del desarrollo en medio PDA basados en las claves descritas por Booth (1970), Piontelli y Toro (1992) y Leslie y Summerell (2006). La producción de clamidosporas se realizó a partir de macroconidios producidos en el medio PDA, estos fueron cosechadas y concentrados por centrifugación, luego suspendidos en agua destilada estéril por 20 a 30 días a 25 °C (Dhingra y Sinclair, 1995). Las medidas de estas estructuras fueron comparadas con descriptores empleados por Booth (1970) y Hoog (2000).
Extracción de ADN
El método empleado para la extracción de ADN fue el desarrollado por la Universidad de TUAT, método de mini preparación propuesto por Saitoh et al. (2006) y Liu et al. (2000). De cada aislamiento, se obtuvo una porción de 5-10 mm de agar con micelio en crecimiento activo, los cuales fueron depositados en tubos de 1.5 mL con 500 µL de Buffer de Lisis (100 mM Tris HCl, 10 mM EDTA, 1M KCl; pH 8.0). Se trituró el micelio y se incubó por 10 minutos, luego se agregó fenol-cloroformo-alcohol isoamílico (25:24:1) y se centrifugó 10 minutos a 12000 rpm. Se recuperó la fase acuosa superior, a la cual se añadieron 2 µL de ARNasa y se incubó a 37 °C por 30 minutos, se añadió un volumen similar al colectado de Isopropanol y se colocó a -20 °C por 15 minutos. Se centrifugó a 12000 rpm por 10 minutos a temperatura de ambiente y se descartó el sobrenadante; luego se realizó un lavado con 1 mL de etanol al 70 % y se centrifugó durante 5 minutos. Posteriormente se eliminó el etanol y se secó al vacío. Finalmente, el ADN se resuspendió en 30 µL de agua ultra pura y fue almacenado a -30 °C.
Reacción de PCR
Se preparó un volumen de reacción de 25 µL, que contenía 1 µL de ADN genómico (50 ng-100ng), 4 µL de Buffer de reacción para la PCR (10 X) (ACTaqTM); 2 µL de cada dNTPs (2.5 mM); 0.5 µL de cada oligonucleótido (20 µM) y 0.125 µL de Taq ADN polimerasa (AC-TaqTM) (5 U. µL-1) y se completó con agua ultra pura.
Para la amplificación de la región IGS se emplearon los oligonucleótidos FIGS11/ FIGS y para los genes SIX se emplearon los oligonucleótidos: SIX4F1/R1, SIX3- F1/ R2 Y P12-F1/R1, 12. Las condiciones de la PCR fueron iguales: desnaturalización inicial a 94 °C por 2 min; seguido de 35 ciclos de 30s a 94 °C, 30 s de anillamiento a 61 °C y 30 s de elongación a 72 °C; y un ciclo final de 72 °C por 7 min (Rep et al., 2004; Inami et al., 2012, 2014). Para la amplificación de los genes endo (pg1) y exo (pgx4) poligalacturonasa se utilizaron los oligonucleótidos: uni, spr13 y spr23 y se llevó a cabo con una desnaturalización inicial de 94 °C por 5 minutos; 45 ciclos de segunda desnaturalización a 94 °C por 1 minuto, hibridación a 61 °C por 1 minuto, elongación a 72 °C por 2 minutos; y una extensión final a 72 °C por 10 minutos (Çolak y Bicici, 2013). La reacción de la PCR fue visualizada bajo electroforesis en un gel de agarosa al 2 %.
La purificación del producto de PCR se realizó mediante el Kit Wizard SV Gel and PCR Clean y se sometió a una reacción de calidad de ADN. La secuenciación del ADN se realizó usando el protocolo de temperatura de alargamiento escalonado (STeP) descrito por Platt et al. (2007); Finalmente, la reacción de secuenciación STeP realizó en un volumen de 10 µL con 100 ng de ADN de amplicon y 0.5µL BigDye Terminator v3.1.
Secuenciación
Para obtener la secuencia completa se ensamblaron manualmente los fragmentos continuos generados por los cebadores “Forward” y “Reverse”, usando el programa GENETIX-MAC and ENZYME X. La identificación se determinó en la base de datos del National Center for Biotechnology Information (NCBI), utilizando el programa BLAST (Basic Local Alignment Search Tool; https://blast.ncbi.nlm.nih.gov/Blast.cgi). Las secuencias de la región IGS fueron alineadas para generar árboles filogenéticos (Inami et al., 2012). El árbol filogenético fue generado por el método de máxima Parsimonia con el programa MEGA 7 (Kumar et al., 2016).
Preparación del inóculo
El método empleado fue descrito por Balogun et al. (2008). Se preparó un caldo de papa dextrosa, en volúmenes de 40 mL en matraces Erlenmeyer (100 mL) para ser sometido a 121 °C por 30 min en autoclave. Luego se procedió a colocar en el caldo una pequeña porción (≤ 0.5 cm2) de PDA con micelio del hongo de cuatro días de edad de cada uno de los aislamientos. Este caldo con el inóculo fue incubado a temperatura ambiente por cinco días en un agitador orbital a 120 rpm, para producción de brotes celulares (conidios unicelulares), terminando el período de incubación, se filtró la solución, se centrifugó a 3000 rpm por 15 minutos, se eliminó el sobrenadante y se quedó con el precipitado, donde se encontraron los conidios. La suspensión de conidios que se obtuvo de la preparación, se ajustó a la concentración a 1 x 10 7 conidios mL -1.
Evaluación de la patogenicidad en invernadero
Se sembraron semillas de tomate de cada cultivar diferencial (Ponderosa, Momotaro, Momotaro 8, Block) en contenedores de plástico en un sustrato estéril; se emplearon tres contenedores y cada una contenían dos semillas del cultivar diferencial, haciendo un total de seis plantas de cada cultivar por aislamiento, después de 21 días se procedió a realizar heridas en las raíces introduciendo una espátula de plástico a nivel radicular, seguido se inoculó en la base de cada planta con 1 mL de la suspensión conidial en una concentración de 1 x 10 7 conidios mL-1. A la cuarta semana después de la inoculación se evaluaron los síntomas externos de cada planta, empleando la escala descrita por Inami et al. (2012); así mismo, se evaluó el pardeamiento del cilindro vascular con la escala descrita por Kashiwa et al. (2016) que es empleada en el laboratorio de TUAT. Una vez concluido el experimento se procedió al reaislamiento del patógeno de las plantas inoculadas, para lo cual se empleó el medio KOMADA y medio PDA. Los postulados de Koch se verificaron mediante el reaislamiento del hongo a partir de raíces y tallo con síntomas, para lo cual se emplearon los medios ya descritos para el aislamiento y la corroboración de la identificación del organismo se realizó mediante características morfológicas y moleculares (análisis basados en los genes de la poligalacturonasa) bajo la metodología previamente descrita.
Resultados y discusión
Identificación morfológica
Los ocho aislados en estudio, se identificaron como F. oxysporum, de acuerdo a las características morfológicas y mediciones microscópicas en el medio PDA y se identificaron como HEI 1, HEI 2, HEI 3, HEI 5, HEI 6, HEI 9 y HEI 10. En cuanto a las características morfológicas, el menor y mayor crecimiento de la colonia después de siete días de evaluación fue para los aislamientos HEI 3 y HEI 10 con 5 cm y a 7 cm respectivamente. Para dos aislamientos, HEI 1 y HEI 11, produjeron colonias con micelio de coloración violácea evaluadas sobre y bajo la superficie de la placa; para los aislados: HEI 2, HEI3, HEI 5, HEI 6, HEI 9, HEI 10, la pigmentación fue de una tonalidad blanca sobre la superficie y en el reverso de la placa la coloración fue de violácea clara a oscura. El desarrollo micelial de las colonias se caracterizó por tener un crecimiento radial y concéntrico, con micelio afelpado y algodonoso. En cuanto a las características de medición de las estructuras, los ocho aislamientos presentaron microconidios de forma oval a elíptica, cuyas mediciones oscilaron entre: 6.72 (+0.82) - 11.46 (+1.6) × 2.64 (+0.51) - 3.48 (+0.96) µm., usualmente de 0-1 septo. Los macroconidios de 25.2 (+1.72) - 29.28 (+1.91) × 3.58 (+0.8) - 4.44 (+0.63) µm., con 3-5 septos; ligeramente curvadas con la presencia de una célula pie, las clamidosporas fueron simples y dobles con diámetros de 6.86 (+0.78) - 12.64 (+1.49) µm (Tabla 2). Estas mediciones coincidieron con los descriptores propuestos por Booth (1970), Hoog (2000), Leslie y Summerell (2006) e investigaciones realizadas por Ciampi et al. (2009) y Hafizi et al. (2013) para la especie de F. oxysporum.
Tabla 2. Mediciones de las estructuras de los ocho aislamientos de Fusarium oxysporum aisladas del cultivo de tomate
| Aislamiento | Microconidias | Macroconidias | Clamidospora | |||
| Tamaño A | Septas | TamañoA | Septas | DiámetroB | Descripción | |
| HEI 1 | 11.34 (+1.46) × 3.48 (+0.96) | 0 a 1 | 26.46 (+1.85) × 3.84(+0.84) | 3 a 4 | 10.99 (+1.06) | Simple/Pares |
| HEI 2 | 6.72(+0.82) × 2.64 (+0.51) | 0 a 1 | 27.36 (+1.86) × 4.44 (+0.63) | 3 a 5 | 8.905 (+0.98) | Simple/pares |
| HEI 3 | 10.02 (+1.88) × 3.06 (+0.62) | 0 | 33.18 (+3.1) × 4.26 (+0.65) | 3 a 5 | 7.64 (+1.13) | Simple/Pares |
| HEI 5 | 8.94 (+1.08) × 3.36 (+0.78) | 0 a 1 | 25.2 (+1.72) × 4.38 (+0.62) | 3 a 4 | 8.35 (+0.99) | Simple/Pares |
| HEI 6 | 7.14 (+0.97) × 2.64 (+0.48) | 0 a 1 | 29.28 (+1.91) × 4.38 (+0.58) | 3 a 4 | 9.36 (+1.07) | Simple |
| HEI 9 | 11.46 (+1.6) × 3.06 (+0.6) | 0 a 1 | 26.28 (+1.78) × 4.20 (+0.73) | 3 a 4 | 8.43 (+0.91) | Simple/Pares |
| HEI 10 | 10.9 (+1.4) × 2.8 (+0.57) | 0 a 1 | 25.66 (+1.67) × 3.58 (+0.8) | 3 a 5 | 6.86 (+0.78) | Simple |
| HEI 11 | 8.94 (+1.22) × 3.48 (+0.36) | 0 | 27 (+1.97) × 4.44 (+0.55) | 3 a 4 | 12.64 (+1.49) | Simple |
A = Promedio de longitud y ancho de 100 Microconidias /Macroconidias ± desviación estándar.
B = Promedio de diámetro de 100 clamidosporas ± desviación estándar.
PCR y análisis filogenético de la región IGS
En todos los aislamientos se logró amplificar la región IGS con el uso de los oligonucleótidos FIGS11 y FIGS12, los productos fueron visualizados por electroforesis en gel de agarosa al 1.2 % observándose una única banda de alrededor de 600 bp. Investigaciones previas realizadas por Kawabe et al. (2005), Balogun et al.(2008) e Inami et al. (2014); han demostrado el uso de dichos oligonucleótidos para la región IGS en F. oxysporum, debido a que las especies estrechamente relacionadas pueden mostrar considerable diversidad en la región IGS, manifestándose en variaciones frecuentes en cuanto a longitud y secuencia; lo que permite la diferenciación entre especies cercanas genéticamente (Appel y Gordon, 1995; Gómez, 2008). Al realizar la construcción filogenética, a partir de los alineamientos obtenidos de la región IGS del ADNr de las muestras y de las secuencias obtenidas de formas especiales de F. oxysporum que fueron tomados de la base de datos NCBI, con una prueba de Bootstrap (1000 réplicas) para evaluar el soporte de agrupaciones, se obtuvo que las ocho muestras de estudio fueron agrupadas dentro de F. oxysporum f. sp. lycopersici obtenidos del NCBI y separadas de Gibberella fujikuroi (FGSC 7610) (Figura 1). No obstante Silva et al. (2014) y Alanis-Martínez et al. (2015) mencionaron que las formas especiales de F. oxysporum pueden tener una información genética muy distinta atribuida a su origen polifilético, para el caso de Fol, se presentan tres clados (A1, A2 y A3), lo que sugiere una relación polifilética de nuestros aislamientos con al menos 3 orígenes. Kawabe et al., (2005), manifiesta que no se ha encontrado una relación entre las razas y grupos polifiléticos reportados. Por lo que, el agrupamiento de los aislamientos a nivel de clado no indicarían la patogenicidad del aislamiento, solo refiere el grado de cercanía en relación con los Fol y/o Fo de tomates no patogénicos (Appel y Gordon, 1995; Kawabe et al., 2005). De acuerdo con el trabajo de Arie (2010), Fusarium oxysporum no patogénico dio origen a las formas patogénicas (Fol) durante la asociación saprofítica con el tomate, después del inicio de la domesticación asociado con la actividad de los elementos móviles presentes en el genoma de F. oxysporum que contribuyeron a la emergencia de Fol y a su diversidad patogénica; en ese sentido, sin bien nuestros aislamientos se encuentran cercanamente relacionados con los F. oxysporum f. sp. lycopersici, no se podría atribuir a una forma especial a nivel de esta región; ya que podrían estar cercanamente asociados a Fo no patogénicos; por lo que, solo con la información reportada de la región IGS no brinda precisión al detectar formas especiales, ya que los determinantes de la especificad del hospedero pueden transferirse horizontalmente entre cepas de F. oxysporum de diferentes linajes, este intercambio genético hace que sea difícil desarrollar oligonucleótidos para la detección especifica de una forma especial dada. Por ello, los marcadores moleculares basados en los determinantes genéticos de la patogenicidad, son promisorios para distinguir la forma especial (Van Der Does et al., 2008; Silva et al., 2014).
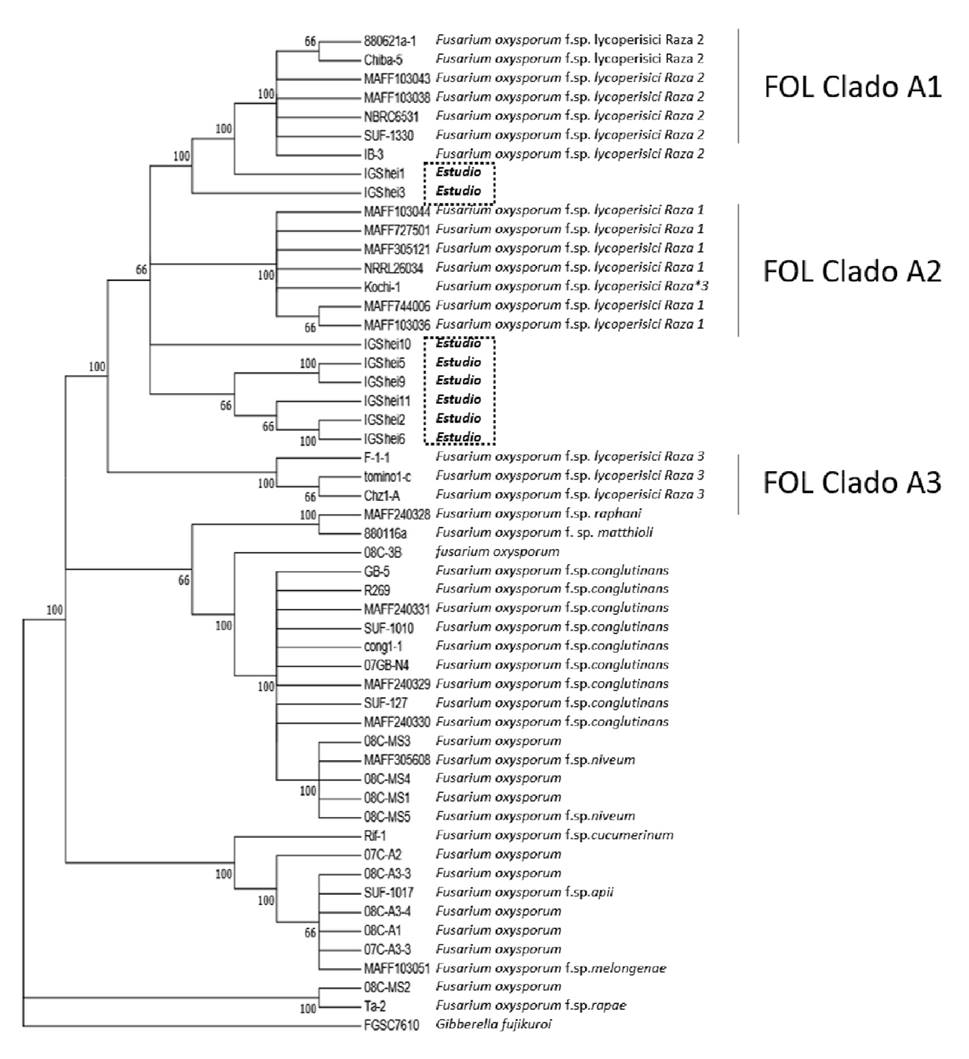
Figura 1. Análisis filogenético utilizando Mega 7. Método de Máxima parsimonia con 1000 replicaciones para el Bootstrap. Se compararon las secuencias espaciadoras internas (IGS) de los aislamientos de Fusarium oxysporum obtenidos en el estudio con las diferentes secuencias de Fusarium oxysporum obtenidas del NCBI y un aislamiento como grupo externo, Gibberella fujikuroi.
PCR para detección de genes de avirulencia (gen SIX) en Fusarium oxysporum
De acuerdo a los resultados el aislamiento HEI 11 es el único en el que se evidenció la presencia de los tres genes de avirulencia (SIX4, SIX3 y SIX1) con un tamaño de banda igual a los controles positivos SIX4 (800bp), SIX3 (570 bp) y SIX1(400bp) y se confirmó su alto grado de identidad con sus respectivos genes SIX reportados para F. oxysporum f. sp. lycopersici con un porcentaje de identidad de 100 % para SIX4, 99 % para SIX3 y 98 % para SIX1, respectivamente. Por otro lado, para los aislamientos HEI 2, HEI 3, HEI 5, HEI 6, HEI 9, HEI 10, se observaron productos de amplificación solo con el juego de los oligonucleótidos SIX3-F1/SIX3-R2 para el gen SIX3 (AVR2); sin embargo, los tamaños amplificados fueron diferentes a los controles positivos (Figura 2). Las proteínas efectoras en hongos son secretadas dentro del espacio extracelular, las cuales pueden actuar a este nivel o pueden ser absorbidos por las células de la planta e interactuar con objetivos intracelulares (Houterman et al., 2007). El grupo de proteína efectoras, determina la virulencia de un patógeno hacia un hospedante en particular (González et al., 2012). Para el caso de las proteínas relacionadas a la patogenicidad de F. oxysporum f. sp. lycopersici, Rep et al. (2003) mencionaron que éstas se encuentran acumuladas en la savia del xilema tras la infección y promueven la virulencia de este patógeno hacia el tomate. Entre ellos se reportaron 3 genes de avirulencia AVR1 (=SIX4), AVR2 (=SIX3) y AVR3 (=SIX1). Por lo que, las razas de F. oxysporum f. sp. lycopersici pueden ser determinada por la combinación de estos genes (Inami et al., 2012). Por lo tanto, la amplificación de los genes de avirulencia en el aislamiento HEI 11, indicarían la presencia de F. oxysporum f. sp. lycopersici. Ya que, numerosas investigaciones, reportan estos genes SIX1, SIX3, SIX4 como parte exclusiva de aislamientos de F. oxysporum f. sp. lycopersici y no en otras formas especiales o aislamientos no patogénicos (Lievens et al., 2009). Los resultados manifiestan que el aislamiento HEI 11 pertenece a la raza 1; ya que, fue el único aislamiento que amplificó el gen de avirulencia AVR1, que solo es detectada en la raza 1, pero no en la raza 2 y 3 (Houterman et al., 2009; Inami et al., 2014). Además, amplificaron para los genes de avirulencia AVR2 y AVR3. Inami et al. (2012), describen que basados en los genes AVR, la presencia de los tres genes de avirulencia: AVR1 (SIX4), AVR2 (SIX 3) y AVR3 (SIX1) corresponderían a la Raza 1 de F. oxysporum f. sp. lycopersici.
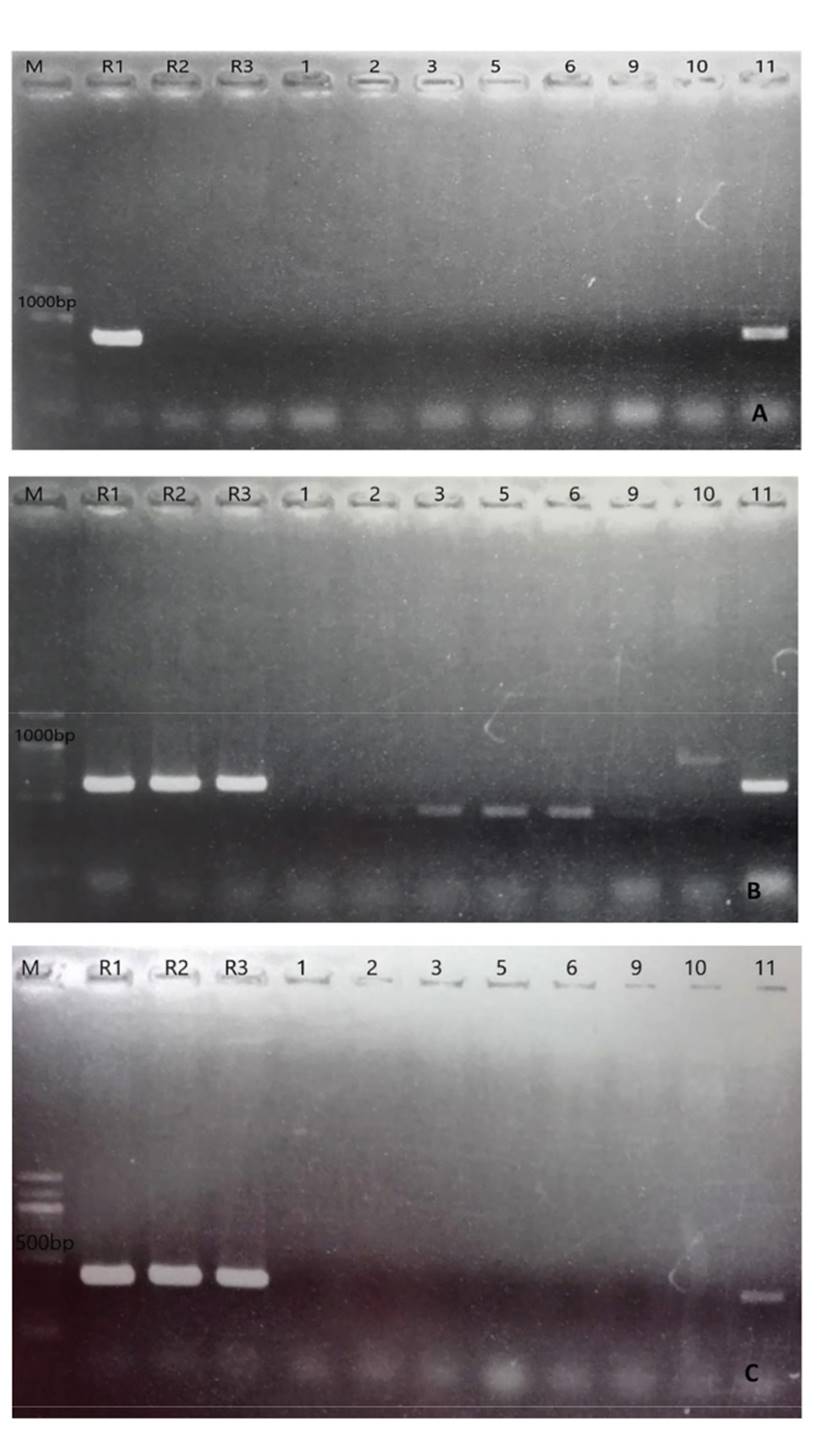
Figura 2. Productos de PCR de los tres genes SIX (SIX4, SIX3, SIX1) del aislado HEI 11 (11) en gel de agarosa con bandas de tamaño esperado a los controles positivos: R1, Fol raza 1; R2, Fol raza 2; R3, Fol raza 3. A: banda correspondiente a SIX4 usando los oligonucleótidos SIX4 F1/SIX4 R1. B: banda correspondiente a SIX3 usando los oligonucleótidos SIX3 F1/SIX3 R2. C: banda correspondiente a SIX 1usando los oligonucleótidos P12 F1/P 12 R1. Las muestras 1, 2, 3,5, 6, 9, 10 y 11 corresponden a los aislados HEI 1, HEI 2, HEI3, HEI 5, HEI 6, HEI 9, HEI 10, HEI 11, respectivamente.
PCR para detección de genes de la poligalacturonasa
Con oligonucleótidos específicos diseñados a partir de genes de la poligalacturonasa para discriminación de razas de Fol empleados para la corroboración de la forma especial del aislamiento HEI 11. Los resultados mostraron la amplificación para el juego de oligonucleótido “uni”, con tamaño de banda de 672 bp. Así también, amplificó para el set de oligonucleótidos “sp13” con un tamaño de banda de 445 bp y no se evidenció ningún producto amplificado para el set de oligonucleótidos “sp23 “(Figura 3). Los tamaños de amplicones mencionados se ajustan a los reportados por Hirano y Arie (2006) para Fusarium oxysporum f. sp. lycopersici raza 1. Las poligalacturonasas son las primeras enzimas involucradas en la degradación de la pared celular durante la patogénesis y juegan un papel importante en el desarrollo de la infección al menos en dos fases: penetración de la corteza de la raíz, con el fin de alcanzar el sistema vascular; y colonización y difusión del hongo hacia la parte superior a través de los vasos del xilema (Bravo, 2013; Srinivas et al., 2019). Por lo que, el diseño de los oligonucleótidos específicos (uni, sp13, sp23, sprl) diseñados por Hirano y Arie (2006) a partir de secuencias nucleotídicas parciales de genes como la endopoligalacturonas (pg 1) y exopoligalacturonasas (pgx 4) de aislamientos de Fol y F. oxysporum f. sp. radicislycopersici, permitieron la confirmación del aislamiento HEI 11 como Fol raza 1. El juego de oligonucleótidos “uni” amplificó un producto con un mismo tamaño de banda con respecto a los controles positivos R1, R2, y R3, confirmando la presencia de F. oxysporum. Además, amplificó para el set de oligonucleótidos sp13 (445 bp) el cual indica que el aislamiento HEI 11, podría pertenecer a la raza 1 o raza 3; sin embargo, al no amplificar con el juego del oligonucleótido sp23 (518 bp), el cual solo amplifica para raza 2 o raza 3, se descartó que HEI 11 pertenezca a la raza 3, con lo que se valida que, a nivel molecular el aislamiento HEI 11 pertenece a F. oxysporum f. sp. lycopersici raza 1. Pruebas similares fueron realizadas por Kawabe et al. (2005) y Baysal et al.(2009).
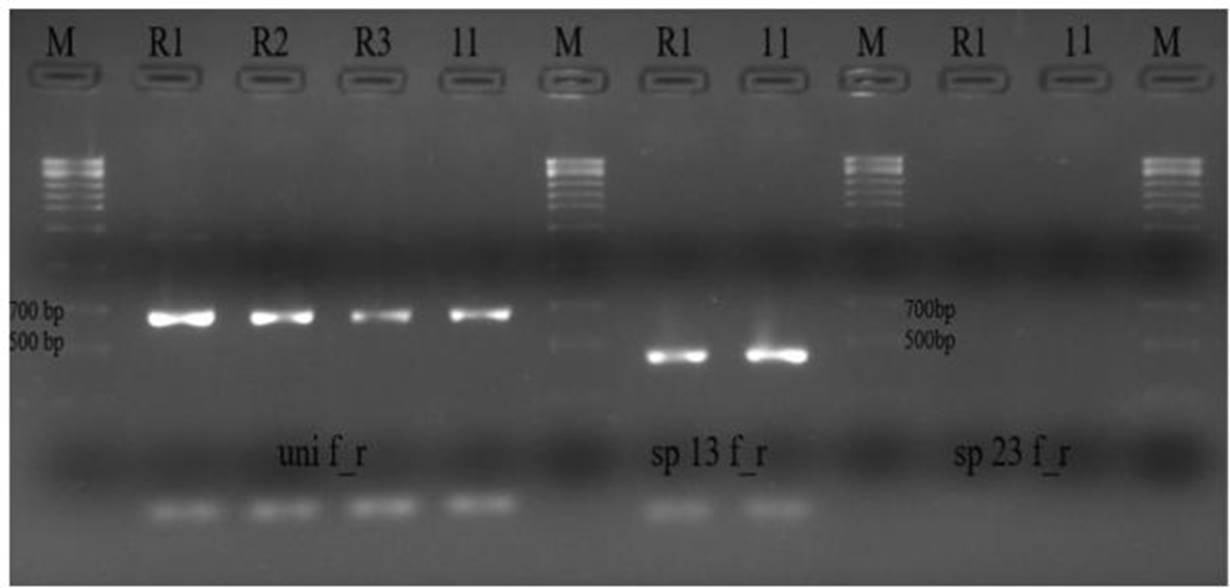
Figura 3. Productos de PCR de electroforesis en gel de agarosa usando los juegos de oligonucleótidos uni f/r, sp13, sp23 específicos para detectar Fol. M: marcador molecular. R1: Fol raza 1. R2: Fol raza 2. R3: Fol raza 3.11: aislamiento HEI 11. HEI 11 con banda de tamaño esperado al control positivo R1 con los oligonucleótidos uni f/r y sp13.
Prueba de patogenicidad
De los ocho aislamientos evaluados, solo HEI 11 causó los síntomas característicos de Fol (Figura 4), los cuales se observaron a partir de la tercera semana (síntomas secundarios) después de la inoculación, solo en el cultivar susceptible a las tres razas (ponderosa) y no en los otros cultivares con resistencia a una de las razas (Momotaro, Momotaro 8 y Block), los síntomas coincidieron con los descritos por Walker (1971) para F. oxysporum f. sp. lycopersici dentro de un invernadero, los cuales comenzaron con una ligera decoloración a nivel de las nervaduras de las hojas, seguido de un decaimiento del peciolo (epinastia) y amarillamiento que inició en las hojas basales y avanzó a las hojas jóvenes; finalmente se observó marchitez y muerte de la planta; al realizar el corte del tallo se evidenció pardeamiento de las células vasculares.

Figura 4. Síntomas de la prueba de patogenicidad en el cultivo de tomate, a la cuarta semana de inoculación con F. oxysporum f. sp. lycopersici. A-B: amarillamiento de las hojas basales, epinastia. C: muerte total de la planta. D-E: decoloración vascular.
Para los aislamientos HEI 1, HEI 2, HEI 3, HEI 5, HEI 6, HEI 9, HEI 10, si bien estos fueron obtenidos de tomate. Sin embargo, no mostró síntomas en el cultivar susceptible a las tres razas; ni en ningún otro cultivar, por lo que no fueron identificadas como razas de F. oxysporum f. sp. lycopersici. Aunque, estos aislamientos son capaces de parasitar plantas de tomate, ya que fueron aislados de este cultivo; no obstante, el análisis molecular demostró que carecen de genes de patogenicidad que les permita causar síntomas en el cultivar susceptible (Negrete, 2013). Algunas investigaciones manifiestan que estos F. oxysporum no patogénicos aislados de tomate pudieron originar a las formas de F. oxysporum f. sp. lycopersici patogénicos durante su asociación saprofítica en el inicio de la domesticación del tomate (Arie, 2010). En la prueba de patogenicidad la determinación del aislamiento HEI 11 como F. oxysporum f. sp. lycopersici raza 1, fue establecida cuando éste, mostró síntomas en el cultivar que carecía de genes de resistencia (Ponderosa) y no se evidenció síntomas en los cultivares: Momotaro (I, i2,i3), Momotaro 8 (I,I2,i3) y Block (I, I2,I3); los cuales poseían genes de resistencia I (Balogun et al., 2008; Inami et al., 2012; Negrete, 2013). Así mismo, en base al análisis molecular se demostró que el aislamiento HEI 11 posee los genes de avirulencia (AVR1, AVR2, AVR3), los cuales pudieron ser reconocidos por al menos uno de los genes de resistencia de los cultivares diferenciales (Momotaro, Momotaro 8 y Block), estableciéndose una relación incompatible entre el patógeno y el hospedante, evidenciándose en la ausencia de la enfermedad en los cultivares resistentes. Por el contrario, en el cultivar susceptible (Ponderosa) al no poseer genes de resistencia se estableció una relación compatible, es decir presencia de la enfermedad. Finalmente, el re aislamiento del aislamiento HEI 11 en el medio selectivo KOMADA y medio PDA de raíces y tallos con síntomas de decoloración vascular y la corroboración de la identidad morfológica y molecular confirmó que se trata de la misma cepa inicial empleada para la inoculación, lo que permitió establecer una relación entre el aislamiento y la enfermedad, completando así el postulado de Koch.
Conclusión
El presente estudio permitió identificar al aislamiento HEI 11 como Fusarium oxysporum f. sp. lycopersici raza 1 y corroborar que las pruebas moleculares basadas en el empleo de oligonucleótidos específicos, son herramientas útiles para la determinación de la F. oxysporum f. sp. lycopersici y de su patogenicidad. Esta investigación servirá de base para futuras investigaciones con fines de estudios del comportamiento poblacional de este patógeno en el país.











 nueva página del texto (beta)
nueva página del texto (beta)



