Introducción
Los snoRNAs son RNAs pequeños no codificantes de aproximadamente 60-300 nt (en los mamíferos) que son predominantemente requeridos para la maduración de los snRNAs (RNAs nucleares)1 y rRNAs (RNAs ribosomales)2; sin embargo, hay evidencia que indica que los snoRNAs tienen otras funciones celulares tales como la regulación postranscripcional «similar a la de los microRNAs (miRNAs)”, pseudouridilación de los mRNAs blanco, modificación de los pre-mRNAs, para dirigir el splicing, y regulación de la edición de los RNAs3,4.
Con base en diferentes criterios, los snoRNAs se clasifican en dos clases: los de cajas C/D y los de cajas H/ACA, los cuales guían (por apareamiento de bases) la metilación de la 2´-O ribosa y la pseudouridilación de nucleótidos específicos de sus RNAs blanco, al formar complejos ribonucleoproteicos (snoRPs) con proteínas particulares5. De forma muy interesante, existen unos snoRNAs que tienen las secuencias consenso de los de tipo C/D y/o de los H/ACA, en adición a una caja CAB (H/ACA scaRNAs) o una G•U/U•G (C/D scaRNAs), las cuales son necesarias para dirigirlos hacia los cuerpos de Cajal (CBs)6. En general, los snoRNAs están altamente conservados en las diferentes especies7, pero tanto su localización genómica como la forma en la que se transcriben es muy variable y compleja8 (Fig. 1). En el cáncer, el estudio de los RNAs no codificantes (ncRNAs, non-coding RNAs) se ha centrado principalmente en los miRNAs y RNAs largos no codificantes (lncRNAs); sin embargo, diversas líneas de evidencia indican la participación de diferentes snoRNAs en estas enfermedades9,10. Estos estudios han demostrado que los snoRNAs pueden funcionar como oncogenes o supresores tumorales y han revelado su posible uso como biomarcadores de diagnóstico y/o pronóstico. Asimismo, algunos estudios han profundizado en la dilucidación de los mecanismos moleculares que subyacen a la función de los snoRNAs en el cáncer y han revelado funciones que no están necesariamente asociadas a la modificación química de otras especies de RNA4,11.
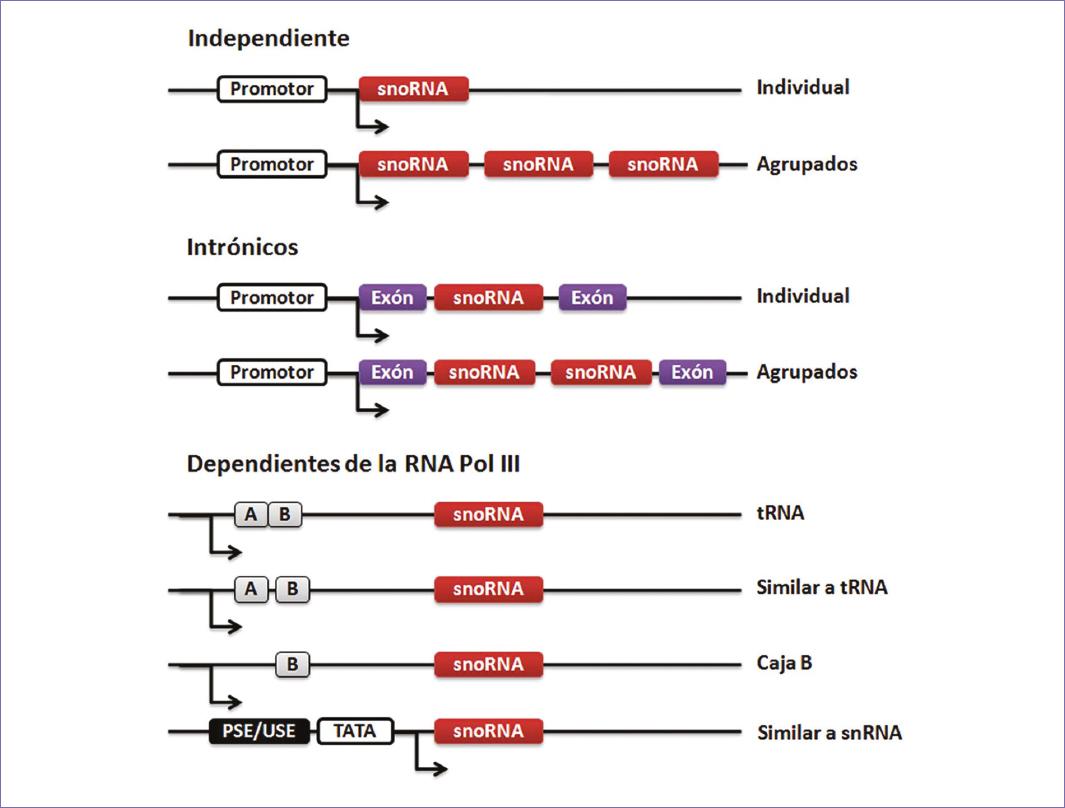
Figura 1 Modos de transcripción de los snoRNAs. La transcripción de los snoRNAs puede ser de forma monocistrónica (transcripción de un producto) o policistrónica (transcripción de varios productos), y puede (dependiente) o no depender (autónoma) del promotor del gen en el que están hospedados. La mayoría son transcritos por la RNA polimerasa II y algunos por la RNA polimerasa III. Interesantemente, algunos snoRNAs se transcriben como dicistrones de tipo: snoRNA-snoRNA, tRNA-snoRNA o snoRNA-miRNA.
snoRNAs de tipo caja C/D y caja H/ACA
Los snoRNAs se localizan principalmente en el nucléolo y subtipos muy particulares de estos, llamados scaRNAs (small Cajal bodies-specific RNAs), en los CBs. No obstante, hay evidencia de la presencia de estos RNAs en el citoplasma celular, tanto en un entorno homeostático como durante condiciones lipotóxicas, por estrés oxidante y ausencia de suero4,12. Las funciones principales de los snoRNAs son la formación del espliceosoma y la maduración del ribosoma al modificar químicamente a los snRNAs y rRNAs, respectivamente13,14. Además de estas tareas, los snoRNAs ejercen otras funciones tales como la regulación postranscripcional «similar a la de los miRNAs», pseudouridilación de sus mRNAs blanco, modificación de los pre-mRNAs (para dirigir el splicing) y regulación de la edición de los RNAs4,12. La función de los snoRNAs está determinada en parte por la presencia de secuencias consenso específicas, por su estructura secundaria, así como por los complejos proteicos a los que se asocian15. De acuerdo a esto, los snoRNAs se clasifican en dos clases, los de caja C/D y caja H/ACA; los primeros poseen una caja C (ugauga), que se localiza en el extremo 5´-terminal, y una D (cuga) que está en el extremo 3´-terminal (Fig. 2). Por su parte, los de tipo H/ACA tienen cajas H (ANANNA) y ACA (aca), ubicadas respectivamente en la «bisagra» y en la región 3´-terminal (Fig. 2). Los snoRNAs de tipo C/D median la 2’-O-metilación al formar snoRPs (complejos ribonucleoproteicos) con las proteínas FBRL (fibrilarina, RNA metilasa), NOP56 (ribonucleoproteína NOP56), NOP58 (ribonucleoproteína NOP58) y NH2L1 (ribonucleoproteína pequeña nuclear 13), mientras que los de tipo H/ACA inducen la pseudouridilación de otras especies de RNA, al formar snoRPs con DKC1 (diskerina pseudouridina sintasa 1), GAR 1 (ribonucleoproteína GAR1), NHP2 (ribonucleoproteína NHP2) y NOP10 (ribonucleoproteína NHP2) (Fig. 2).

Figura 2 snoRNAs y scaRNAs. Los snoRNAs se clasifican en los que tienen cajas de tipo C/D o H/ACA. Cada uno de estos snoRNAs interaccionan con complejos proteicos específicos que determinan su función. Los snoRNAs de tipo C/D median la metilación, mientras que los de tipo H/ACA la pseudouridilación de otras especies de RNA. Por su parte, los scaRNAs pueden ser de tipo C/D o H/ACA, o ambos y se localizan en los cuerpos de Cajal. La secuencia de los snoRNAs que se ejemplifican en la imagen se buscó en el National Center for Biotechnology Information y las estructuras secundarias se visualizaron por medio del servidor RNAfold.
scaRNAs
Los RNAs «guía» que son responsables de la modificación de los snRNAs, son snoRNAs especializados que se localizan en los CBs y se denominan scaRNAs16. Este tipo de snoRNAs poseen todas las características de los de tipo C/D y H/ACA, y motivos adicionales que determinan su localización en los CBs: los scaRNAs H/ACA poseen una caja CAB (ugAC) y los C/D una secuencia G•U/U•G (Fig. 2). La caja CAB es reconocida por la proteína WAP53 (WD repeat containing antisense to TP53) y es necesaria para la localización de los scaRNPs en los CBs6,17; sin embargo, a pesar de que los scaRNAs C/D no tienen caja CAB, también son direccionados por la proteína WAP53 hacia los CBs6. A la fecha se han descrito 29 scaRNAs, de los cuales los de tipo H/ACA son los más abundantes (aproximadamente 20 veces más que los de tipo C/D) y esto concuerda con la abundancia de los scaRNPs de los scaRNAs H/ACA en los CBs, comparado con las proteínas que conforman a los snRNPs de los de tipo C/D (Tabla 1)18,19.
Tabla 1 scaRNAs identificados a la fecha
| Nombre | Nombre alternativo | ID | Tipo | Blanco 1 | Blanco 2 | Blanco 3 | Blanco 4 |
|---|---|---|---|---|---|---|---|
| SCARNA7 | U90 | snoID_0598 | C/DSCARNA | 5.8S-76 | U1.1-70 | ||
| SCARNA28 | snoID_0620 | C/DSCARNA | U2.2-47 | NA | |||
| SCARNA1 | ACA35 | snoID_0603 | H/ACA-SCARNA | NA | 18S-1441 | ||
| SCARNA3 | HBI-100 | snoID_0596 | H/ACA-SCARNA | NA | U6.6-40 | ||
| SCARNA4 | ACA26 | snoID_0595 | H/ACA-SCARNA | U2.3-41 | U2.3-39 | ||
| SCARNA8 | U92 | snoID_0601 | H/ACA-SCARNA | U2.3-34 | U2.3-44 | ||
| SCARNA11 | ACA57 | snoID_0610 | H/ACA-SCARNA | NA | U5.3-41 | ||
| SCARNA14 | U100 | snoID_0612 | H/ACA-SCARNA | NA | U1.1-72 | ||
| SCARNA15 | ACA45 | snoID_0604 | H/ACA-SCARNA | NA | U2.3-39 | ||
| SCARNA16 | ACA47 | snoID_0602 | H/ACA-SCARNA | NA | U1.4-5 | ||
| SCARNA18 | U109 | snoID_0609 | H/ACA-SCARNA | NA | U1.4-6 | ||
| SCARNA18B | snoID_0707 | H/ACA-SCARNA | NA | U1.4-6 | |||
| SCARNA19 | hTR/TERC | snoID_1118 | H/ACA-SCARNA | Telómeros | |||
| SCARNA20 | ACA66 | snoID_0592 | H/ACA-SCARNA | NA | U12.1-27 | ||
| SCARNA21B | snoID_0577 | H/ACA-SCARNA | U12.1-18 | 28S-4426 | |||
| SCARNA22 | ACA11 | snoID_0611 | H/ACA-SCARNA | NA | NA | ||
| SCARNA23 | ACA12 | snoID_0594 | H/ACA-SCARNA | NA | U6.6-40 | ||
| SCARNA26A | snoID_0618 | H/ACA-SCARNA | U4.2-79 | NA | |||
| SCARNA26B | snoID_0625 | H/ACA-SCARNA | U4.1-79 | NA | |||
| SCARNA27 | snoID_0614 | H/ACA-SCARNA | NA | NA | |||
| SCARNA5 | U87 | snoID_0597 | Híbrido | U5.1-39 | 18S-595 | 18S-1530 | U4.1-65 |
| SCARNA6 | U88 | snoID_0613 | Híbrido | U5.1-39 | 18S-1628 | 28S-2861 | 28S-1530 |
| SCARNA10 | U85 | snoID_0608 | Híbrido | 18S-283 | U5.1-44 | 1818S-101 | U5.1-43 |
| SCARNA12 | U89 | snoID_0607 | Híbrido | NA | 18S-917 | 18S-556 | 18S-464 |
| SCARNA21 | ACA68 | snoID_0599 | Híbrido | U2.1-17 | U12.1-18 | U6atac-83 | 28S-4426 |
| SCARNA2 | HBII-382 | snoID_0593 | CD en tándem | U2.1-25 | NA | 18S-1363 | 28S-1963 |
| SCARNA9 | MgU2-19/30 | snoID_0605 | CD en tándem | U2.1-19 | NA | NA | U2.1-30 |
| SCARNA9L | snoID_0600 | CD en tándem | U2.1-19 | NA | NA | U2.1-30 | |
| SCARNA17 | U91 | snoID_0591 | CD en tándem | U12-21 | NA | U4.1-8 | U2.1-43 |
| SCARNA13 | U93 | snoID_0606 | HACA en tándem | NA | U7-7 | U5.1-39 | U2.3-54 |
scaRNAs: small Cajal bodies-specific RNAs (pequeños RNAs específicos de cuerpos de Cajal). Adaptada de Meier, 2017xx.
Organización genómica de los snoRNAs
En los eucariontes se han descrito diversos tipos de organización genómica para las unidades que codifican para los snoRNAs y cada uno corresponde a un modo particular de transcripción. Los snoRNAs se codifican a partir de regiones intrónicas o intergénicas, y se transcriben de forma individual «monocistrónicos» o en grupo «policistrónicos», y dependen (dependientes) o no (autónomos) del promotor del gen en el que están inmersos19-25. En diferentes organismos se ha detectado la presencia de dicistrones de RNA de tipo tRNA-snoRNA26, snoRNA-snoRNA27 y snoRNA-miRNA28. En el caso de los scaRNAs, su organización genómica puede ser en tándem, con cuatro secuencias «guía» potenciales (en lugar de dos) y pueden formar scaRNAs híbridos: un scaRNA H/ACA se inserta en el asa de uno de tipo C/D y esto resulta en la formación de un scaRNA con dos sitios potenciales de pseudouridilación y dos de metilación.
Modos de transcripción
Hasta la fecha se conoce muy poco acerca de la organización de los promotores de los genes que son transcritos de manera autónoma y que codifican para snoRNAs. En las levaduras, estos promotores están formados por octámeros y motivos CCAAT, cajas TATA y Homol D, y elementos ricos en A/T, en donde se unen factores de transcripción tales como Rap1 (RAP1A, miembro de la familia de oncogenes RAS), Abf1p (musculina) y Tbf1; estas regiones son transcritas por la RNA polimerasa II29-31. Se sabe que la caja Homol D es crítica para la transcripción del snoRNA U3, mientras que la caja TATA tiene una gran influencia sobre la eficiencia de la transcripción, pero no es esencial30. Además de estos motivos, hay otros localizados río arriba del U3 que también son capaces de dirigir la transcripción de su promotor29, lo cual denota la complejidad que tiene la transcripción de los snoRNAs.
Por otro lado, también hay snoRNAs, como el SNR52, que son transcritos por la RNA polimerasa III, quien reconoce cajas de control de tipo A y B, que son típicas de regiones que codifican para tRNAs (Fig. 2)32,33. En la Arabidopsis Thaliana y la Oryza sativa, algunos snoRNAs son transcritos de forma dicistrónica para formar especies híbridas de RNA: tRNA-snoRNA26. Específicamente, se identificaron 12 nuevas familias de genes que codifican snoRNAs de tipo C/D (snoR43) y que se localizan río abajo de genes que codifican para el tRNA(Gly). Estas familias perecen transcribirse a partir del promotor del tRNA y producen precursores dicistrónicos tRNA(Gly)-snoR43, los cuales son procesados por la RNAsa Z26,34. Es importante mencionar que el procesamiento de esta especie de RNA dicistrónica depende de las proteínas que forman el snoRNP34.
En las levaduras, se observó que los snoRNAs 190 y U14 son transcritos simultáneamente para dar como resultado un dicistrón de tipo snoRNA-snoRNA, el cual es procesado por la RNAse III. La disrupción de esta enzima resultó en una disminución dramática en los niveles de las formas maduras de estos snoRNAs y en la acumulación del dicistrón snR190-U14. En contraste, la adición de la enzima recombinante de la Rnt1 indujo la hidrólisis de los RNAs dicistrónicos y llevó a la producción de las formas maduras; esto indica que los RNAs dicistrónicos corresponden a precursores funcionales que son parte del procesamiento de ciertos snoRNAs27. De acuerdo a lo anterior, es necesario el estudio de los mecanismos que controlan la transcripción y el procesamiento de los snoRNAs en el humano, ya que esto nos permitirá entender qué mecanismos pudieran estar alterados en el cáncer y esto llevar a la creación de tratamientos efectivos.
Participación de los snoRNAs en el cáncer
Actualmente existen diferentes estudios que demuestran la participación de los snoRNAs en diferentes tipos de cáncer, los cuales se centran en los tipos que tienen una mayor incidencia e índice de mortalidad. Si bien la mayoría de los estudios simplemente demuestran cambios en la expresión de diferentes snoRNAs (comparando tejido tumoral y/o líneas celulares vs. tejido no neoplásico o células no transformadas), los menos han dilucidado los mecanismos por los que estas especies de RNA funcionan como supresores tumorales u oncogenes. Importantemente, los estudios en donde se evaluaron los cambios en la expresión de los snoRNAs han arrojado ciertos biomarcadores diagnósticos y pronósticos (Tabla 2), y se ha propuesto su uso para posibles terapias contra estas enfermedades; no obstante, todavía se está lejos para poder usarlos en la clínica.
Tabla 2 SnoRNAs como potenciales biomarcadores en diferentes tipos de cáncer
| Tipo de cáncer | snoRNAs útiles para la clasificación molecular | snoRNAs asociados con el pronóstico de la enfermedad | snoRNAs asociados con la detección temprana |
|---|---|---|---|
| Leucemias | snoRD 112 snoRD 113 snoRD 114 ACA11} Mannoor, et al., 201440 |
snoRD 6 snoRD 31 snoRD 62 snoRD 71C 201348 snoRD 116-18 SNORA70F SNORA74A}Ronchetti, et al., 201247 |
|
| Cáncer de pulmón | snoRD28 snoRD76 SNORA3 SNORA21 SNORA42 SNORA47 SNORA68 SNORA78}Gao, et al., 201538 |
snoRD33 snoRD66 snoRD76 Su, et al., 201637 snoRD78 |
|
| Cáncer de mama | GAS5 RNAU44 snoRD46 snoRD89}Mei, et al., 201239 |
||
| Cáncer de hígado | SNHG6  Mourtada, et
al., 200941 Mourtada, et
al., 200941ACA11  Gee, et al.,
201142 Gee, et al.,
201142
|
snoRD47  Krishnan, et
al., 201643 Krishnan, et
al., 201643SNORD113-1  Koduru, et
al., 201744 Koduru, et
al., 201744
|
|
| Cáncer de colón | SNORA42  Su, et al.,
201445 Su, et al.,
201445SNORA21  Stepanov, et
al., 201646 Stepanov, et
al., 201646
|
||
| Cáncer de próstata | |||
| Tumores cerebrales | GBM: SNORD76  Ronchetti, et
al., 201247 Ronchetti, et
al., 201247
|
SnoRNAs: Small nucleolar RNAs (RNAs pequeños nucleolares).
CÁNCER DE PULMóN
Un número importante de los estudios que arrojan información acerca de la participación de los snoRNAs en el cáncer, se han realizado en tejidos y/o en líneas celulares de cáncer de pulmón, debido a que es el tipo de cáncer más común en el mundo y la principal causa de muerte asociada a cáncer48. Estos estudios indican que ciertos snoRNAs tienen una expresión aberrante en tejidos tumorales y en líneas celulares que ejemplifican los diferentes subtipos de este cáncer; un dato importante es que la expresión de algunos de estos RNAs puede ser detectada en el suero sanguíneo de los pacientes con cáncer de pulmón, lo cual tiene un significado para la detección no invasiva de la enfermedad49. Por ejemplo, los SNORDs 33, 66, 76 y 78 son útiles para el diagnóstico temprano de este cáncer37, mientras que la expresión diferencial de los snoRDs: snoRAs 3, 21, 42, 47, 68 y 78, y snoRDs 28 y 66, se asocia con la sobrevivencia promedio de los pacientes con cáncer de pulmón; por ende, estos últimos podrían ser snoRNAs biomarcadores para identificar el pronóstico de los pacientes38.
En cuanto a los mecanismos moleculares que se asocian con la expresión aberrante y función de los snoRNAs en el cáncer de pulmón, se sabe que el incremento en la expresión del SNORA42 está relacionado con la amplificación genómica de su gen y con la tumorigénesis al inducir la evasión de la apoptosis que es dependiente de p5350. De igual forma, su sobreexpresión se detectó en células CD133+ y esto se asocia con la inducción de la proliferación celular, la autorrenovación de las células iniciadoras del tumor y la tumorigénesis51. En el mismo sentido, Langhendries, et al.49 revelaron que los snoRNAs U3 y U8 inducen la tumorigénesis al controlar la síntesis de las subunidades pequeña y grande del ribosoma. En resumen, a la fecha existen algunos snoRDs que pudieran funcionar como biomarcadores del diagnóstico temprano y del pronóstico de los pacientes con cáncer de pulmón; sin embargo, aún se conoce muy poco acerca de los mecanismos moleculares involucrados.
CÁNCER DE MAMA
Mourtada-Maarabouni, et al.52, demostraron que el GAS5 (transcrito 5, específico del arresto del crecimiento celular) está expresado a la baja en tejido tumoral de cáncer de mama, lo cual resulta en un crecimiento celular descontrolado y en la evasión de la apoptosis. Un estudio posterior corroboró que la disminución en la expresión de GAS5 y del snoRNA RNU44, se asoció con el mal pronóstico de los pacientes con este tipo de cáncer39. Estudios de secuenciación revelaron que 13 snoRNAs, entre ellos los snoRDs 46 y 89, están relacionados con la supervivencia promedio y/o la libre de recurrencia de los pacientes53. Similarmente, en tumores triple negativos se observó la expresión diferencial de ncRNAs, incluyendo diferentes snoRNAs; sin embargo, los autores no asociaron los cambios en la expresión de lo snoRDs con las características clínicas de los pacientes54. De acuerdo a lo anterior, a la fecha existen snoRDs específicos que pueden funcionar como biomarcadores para predecir la progresión del cáncer de mama y para distinguir el subtipo triple negativo de otros.
Ahondando acerca de los mecanismos moleculares involucrados, Su, et al.55 demostraron que la supresión de la biogénesis de los snoRDs y de la expresión de la fibrilarina inhiben el crecimiento celular y que esto es dependiente de la activación de p53. Asimismo, en células MCF-7 se observó que la expresión ectópica de varios snoRNAs indujo la transcripción de los miRNAs y la activación de vías relacionadas con la respuesta al sistema inmunitario y la apoptosis56. Estos resultados sugieren que los snoRDs están involucrados en el control transcripcional de los genes que codifican para proteínas y/o miRNAs, y que la inducción transcripcional de los miRNAs podría regular la función de diversas vías de señalización celular.
LEUCEMIAS
En los diferentes tipos de leucemias estudiados, la expresión global de los snoRNAs está regulada a la baja y esto es útil para distinguir subtipos particulares de leucemias40,57-60. Por ejemplo, la expresión de los snoRNAs 112-114 es una firma molecular de la leucemia promielocítica aguda40 y la del snoRNA ACA11, de pacientes con mieloma múltiple que son positivos para la translocación cromosómica t(4;14)59. Por su parte, la expresión del scaRNA22 permite diferenciar a los pacientes con mieloma múltiple que tienen la translocación D4 (TC4 mieloma múltiple); la expresión a la alta de los snoRNAs 115 y 116 caracteriza al grupo positivo para células TC258. Algo similar ocurre en las líneas celulares, en donde Teittinen, et al.60 demostraron que cada una de ellas tiene una firma particular de snoRNAs. En adición a esto, la reducción de los niveles de expresión de los snoRNAs se asoció con el pronóstico de pacientes con leucemia linfocítica o leucemia crónica aguda58, por lo que estos snoRNAs podrían emplearse para la detección temprana de la enfermedad.
Además de identificar los niveles de expresión de los snoRNAs en los diferentes tipos y subtipos de leucemias, existen estudios que han intentado identificar la función de estos RNAs en este tipo de cáncer. En este sentido, se sabe que el SNORD114-1 promueve el crecimiento celular al mediar el paso de la fase G0/G1 a la S, vía Rb/p16 y que el snoRD ACA11 induce el crecimiento de las células cancerosas, la resistencia a la quimioterapia y fomenta el estrés oxidativo en pacientes con leucemia promielocítica aguda40 y mieloma múltiple59, respectivamente. Por su parte, Zhou, et al.10 demostraron que la 2’-O-metilación del rRNA, mediada por la activación de los snoRNAs de tipo C/D es crítica para la inducción de la autorrenovación, del potencial clonogénico y de la oncogénesis en la leucemia mieloide aguda. En conjunto, estos resultados indican que estos snoRDs tienen un efecto oncogénico al controlar diferentes procesos celulares en el cáncer de mama.
CÁNCER DE HÍGADO
La inserción genómica de una región viral de 6 kb en el genoma del ratón es capaz de inducir cáncer hepático, lo cual se asocia con un incremento en la expresión de diferentes snoRNAs y miRNAs59. Igualmente, el incremento en la expresión del snoRNA SNHG6 se asocia con el grado histológico y con la oncogénesis, esto al controlar la evasión de la apoptosis, la activación del ciclo celular e inducción de la metástasis por la activación de la transición epitelio mesénquima. De manera muy interesante, el SNHG6 es un regulador negativo de la función del miRNA 101-3p, al competir por el sitio de unión de este miRNA a su mRNA blanco. Asimismo, se sabe que este snoRNA interacciona con la proteína RENT1 (Up-Frameshift Mutation 1 Homolog) y regula la expresión de SMAD7 (miembro 7 de la familia SMAD)41. Estos datos resultan muy interesantes, debido a que revelan nuevos mecanismos por los que los snoRDs están actuando en el cáncer, particularmente en el cáncer hepático.
El incremento en la expresión de otros snoRDs, tales como ACA11 y 47, también está relacionado con la oncogénesis del cáncer hepático. Al igual que para el SNHG6, los niveles de expresión de ACA11 se relacionan con el grado histológico y la activación del ciclo celular, y de la proliferación, migración e invasión celular42. De igual forma, el snoRD47 induce la metástasis e invasión celular, al regular la expresión de marcadores de la EMT, y su expresión se relaciona con la clasificación TNM (tumour, node and metastasis) y con el mal pronóstico de los pacientes43. En contraste, la expresión del SNORD113-1 está a la baja en el tejido neoplásico (debido a la metilación de las islas CpG de su región promotora) y esto se relaciona con el mal pronóstico de los pacientes. La inducción de la expresión de este snoRNA, en células HepG2y Huh7, suprimió el crecimiento celular al controlar la fosforilación de las proteínas ERK1/2 (mitogen-activated protein kinases 3 and 1) y SMAD2/3 (miembros 2 y 3 de la familia SMAD), las cuales son proteínas efectoras de las vías de las proteínas activadas por mitógenos (MAPK) y factor de crecimiento tumoral beta (TGF-b), respectivamente44. En conjunto, estos resultados resaltan el uso de los snoRNAs SNHG6 y ACA11 para la clasificación molecular del cáncer hepático, y de los snoRDs 47 y 113-1 como biomarcadores pronósticos.
CÁNCER DE COLÓN
En este tipo de cáncer se sabe que la hipermetilación de las regiones promotoras de los snoRNAs controla los niveles de expresión de estos RNAs62. Los promotores de los snoRNAs U70C y ACA59B se encuentran hipermetilados en la línea celular de cáncer colorrectal HCT-116 y esto derivó en su silenciamiento transcripcional. Debido a que esto se observó también en otro tipo de tumores, la alteración de la regulación epigenética parece ser un evento común en el cáncer. Por el contrario, también hay snoRNAs que está regulados a la alta en este tipo de cáncer.
Un ejemplo de esto es GAS5, cuya expresión se induce en respuesta al daño del DNA, de manera dependiente de p5363. Similarmente, el incremento en la sobreexpresión del SNORA42 es un factor de riesgo relacionado con la metástasis y el mal pronóstico de los pacientes. Esto se relaciona con un incremento en la proliferación, migración e invasión celular y con la resistencia a la anoikis e inducción de la tumorigénesis45. Otros snoRDs que están expresados a la alta en el cáncer colorrectal son los snoRAs 15, 21 y 41, de los cuales el snoRA21 es un predictor de mal pronóstico46,64. La inhibición de la expresión de SNORA21 disminuyó la proliferación e invasión celular al modular vías relacionadas con el cáncer46.
CÁNCER DE PRÓSTATA
Al igual que en los otros tipos de cáncer, los niveles de expresión de los snoRNAs está alterada en el cáncer de próstata. En tumores metastásicos, del SNORD78 está expresado a la alta y el SNORA55 circulante (en suero sanguíneo) fue un predictor de supervivencia libre de recaída de los pacientes. La disminución forzada de la expresión del SNORD78 inhibió la proliferación y migración celular, al regular vías de señalización asociadas con factores de crecimiento y la expresión de citocinas proinflamatorias65. Entonces, el snoRD78 y el snoRA55 parecen ser respectivamente indicadores de un peor y mejor pronóstico de los pacientes con cáncer de próstata.
TUMORES CEREBRALES
En el neuroblastoma, la amplificación del gen MYCN (MYCN Proto-Oncogene, BHLH Transcription Factor) es una marca de mal pronóstico para los pacientes66. Como era de esperarse, los tumores con MYCN amplificado tienen un transcriptoma distinto al de los tumores sin la amplificación, lo cual se relaciona con la activación de la vía mTOR (Blanco de la Rapamicina). Con base en lo anterior, Schramm, et al.67 sugieren el uso de inhibidores de esta vía para el tratamiento de los pacientes con este subtipo tumoral. Por otro lado, Cheng, et al.47 demostraron que la expresión a la baja del SNORD76 se asocia con la clasificación y un fenotipo tumoral más agresivo del glioblastoma multiforme. La inducción de la expresión de este snoRNA inhibió la proliferación y el crecimiento de las células tumorales, lo cual se vio reflejado en una disminución tanto del tamaño como del volumen del tumor.
En gliomas pediátricos de alto grado, la expresión del clúster del snoRNA HBII-52 está disminuida y esto fue específico para los tumores que no tienen la mutación H3FA68. Un estudio reciente, realizado en astrocitomas pediátricos, mostró que estos tumores tienen una expresión aberrante de diferentes snoRNAs; no obstante, el significado biológico no se conoce69. Con base en lo anterior, podemos decir que el SNORD76 es un biomarcador pronóstico del glioblastoma multiforme y que los niveles del clúster HBII-52 son una marca molecular adicional para los gliomas pediátricos de alto grado que son negativos para la mutación H3FA. No obstante, el estudio de los snoRNAs en los tumores cerebrales es escaso y son necesarios más estudios para comprender la importancia que tienen estas especies de RNA en el cáncer del sistema nervioso.
LINFOMA
Inicialmente, Tanaka, et al.70 sugirieron que el snoRNA U50HG podría tener una participación en el linfoma de células B, ya que este se localiza en el punto de rotura de la translocación cromosómica t(3;6)(q27;q15), la cual es una marca molecular de este tipo de cáncer. Posteriormente, en el linfoma de células periféricas (que tiene un mal pronóstico) se demostró una disminución en la expresión global de los snoRNAs. En contraste, el incremento en expresión del snoRNA HBII-239 se asoció con un pronóstico favorable de los pacientes con linfoma angioinmunoblástico de células T y en linfoma que no es de células periféricas61. Entonces, la expresión de los snoRNAs podría ser usada para distinguir los subtipos de linfoma y para pronosticar a los pacientes con esta enfermedad.
CáNCER DE PáNCREAS
En el cáncer de páncreas, Cui, et al.71 demostraron que la expresión del SNORA23 se detectó únicamente en los tejidos neoplásicos. Los niveles de este snoRA se correlacionaron con la invasión y de manera inversa con el tiempo de supervivencia, libre de la enfermedad, de los pacientes con adenocarcinoma pancreático ductal. Dentro de los mecanismos que podría estar regulando el snoRA23 en el PDAC, es el control de la expresión de SYNE2 (spectrin repeat-containing nuclear envelope 2), que participa en el control de la invasividad dependiente del anoikis. El bloqueo de la actividad de este snoRA (por un RNA antisentido) disminuyó el crecimiento de los tumores, la expresión tumoral de SYNE2 y la diseminación tumoral y la metástasis hacia el hígado71. Con base en esto, este snoRA parece tener una participación central en la oncogénesis del adenocarcinoma pancreático ductal y podría ser utilizado para determinar el pronóstico de los pacientes.
OSTEOSARCOMA Y CARCINOMA ADRENOCORTICAL
En ratones mutantes para p53, que desarrollan osteosarcoma, se demostró el incremento en la expresión de un clúster de snoRNAs que son regulados transcripcionalmente por el factor ETS2 (Proto-Oncogene 2, Transcription Factor). Como era de esperarse, la eliminación de este factor de transcripción redujo los niveles de estos snoRNAs y revirtió el fenotipo prometastásico; sin embargo, no afectó el desarrollo del osteosarcoma72. Estos datos muestran nuevamente la importancia de los SNORDs en el cáncer y revela la participación del Ets2 en la regulación de su expresión.
Por su parte, el carcinoma adrenocortical mostró la expresión diferencial de 19 snoRNAs, los cuales podrían ser usados como molécula biomarcadoras65.
Conclusiones
Los snoRNAs tienen funciones relativamente bien identificadas como son la 2’-O-metilación y pseudouridilación de otras especies de RNA, y otras que no son tan conocidas, pero que indican que la función celular de los snoRNAs es más compleja de lo que se creía. Importantemente, ahora se sabe que su expresión es aberrante en diferentes tipos de cáncer y que esta podría usarse pronto en la parte clínica para el diagnóstico y/o pronóstico de este grupo de enfermedades. No obstante, aún falta mucho por conocer acerca de los mecanismos que rigen su función celular y se están alterados en el cáncer.











 nueva página del texto (beta)
nueva página del texto (beta)


