Introducción
La sensibilidad y resistencia antimicrobiana es un tema de salud pública mundial que cobra cada vez mayor relevancia con el aumento en la esperanza de vida, la pluripatología en pacientes ancianos, la automedicación y el uso indiscriminado de esquemas antimicrobianos en pacientes hospitalizados, entre otros factores.1
La identificación de organismos específicos que causan gran morbimortalidad a nivel mundial (por ejemplo, organismos productores de β-lactamasas de espectro extendido (BLEE) o aquéllos con el acrónimo ESKAPE (acrónimo para las siguientes bacterias: Enterococcus faecium, Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa e Enterobacter spp.) ha logrado enfocar los esfuerzos multidisciplinarios en su monitoreo epidemiológico y la búsqueda de estrategias terapéuticas que reduzcan su prevalencia. La creciente resistencia a carbapenémicos, sobre todo en Latinoamérica y otras regiones del mundo de países en vías de desarrollo, crea una problemática adicional por la limitación económica en las opciones terapéuticas disponibles.2,3
Las enterobacterias BLEE se han considerado como la principal causa de infecciones nosocomiales por décadas; sin embargo, también ha aumentado su prevalencia en infecciones comunitarias. La atención de pacientes en unidades de cuidados intensivos (UCI) por procesos infecciosos severos, ya sea de origen comunitario o nosocomial, impone un reto adicional. En las UCI es común que la mayoría de los pacientes tengan algún antibiótico como parte de su terapéutica por cursar con una infección severa, confirmada o bajo sospecha, que ocasionó su ingreso a dicha unidad, o por tratarse de pacientes graves, pluripatológicos con alto riesgo de adquirir una infección nosocomial.4
El perfil de resistencias bacterianas puede diferir de acuerdo al lugar, población y situaciones ambientales. Por lo tanto, todas las guías de recomendaciones terapéuticas de enfermedades infecciosas hacen hincapié en la necesidad de conocer la microbiología y perfil de sensibilidad y resistencia antimicrobiana locales. Existe una relación causal entre el uso de cierto antibiótico y la emergencia de resistencia al mismo, por lo que se espera que el índice de resistencias locales sea más alto con respecto a los antibióticos que más se utilizan. Anteriormente el uso de antibióticos de espectro reducido controlaba la presión selectiva de resistencia, pero se perdió con el uso cada vez mayor de antibióticos de amplio espectro. Todo esto contribuye a que los agentes causales de infecciones en pacientes en las UCI sean multidrogorresistentes.5,6
El objetivo del presente estudio fue documentar el perfil de sensibilidad en la unidad de cuidados intensivos adultos (UCIA) de nuestro hospital, comparando infecciones comunitarias con nosocomiales, infección con colonización, antibióticos con mayor uso y factores asociados a resolución del proceso infeccioso.
Material y métodos
Se realizó un estudio de cohorte, observacional y analítico en pacientes ingresados a la UCIA de 2018 a 2020 a quienes se les solicitó cultivos. Se incluyeron sólo pacientes con aislamientos bacterianos, y se excluyeron los cultivos negativos y repetidos. Se identificó desde su solicitud el tipo de cultivo solicitado, el sitio de cultivo, el diagnóstico del paciente y su estado clínico, documentando variables clínicas (fiebre o hipotermia, hipotensión, taquicardia, alteración del estado de alerta), bioquímicas (leucocitos en biometría hemática, proteína C reactiva y procalcitonina) e imagenológicas (radiografías, ultrasonido, tomografías). A cada paciente a quien se le realizó algún cultivo, se le siguió de manera prospectiva, vigilando su comportamiento y evolución de las variables clínicas, bioquímicas e imagenológicas durante su estancia. Una vez que se obtenía el resultado del cultivo, se analizó a la luz de la evolución del paciente y se determinó si correspondía a infección o colonización/contaminación. De los casos que correspondían a infección, se documentó la evolución de acuerdo al esquema antimicrobiano utilizado, si se realizaron ajustes con base en el antibiograma, y al desenlace del proceso infeccioso.
Los aislamientos bacterianos y su sensibilidad antimicrobiana fueron identificados con el equipo automatizado VITEK 2 Compac (bioMérieux Marcy l’ Étoile, France), siguiendo los criterios del Clinical and Laboratory Standards Institute (CLSI) para reportar los aislamientos como susceptibles o resistentes, y realizando controles de calidad de manera estandarizada.
Se creó una base de datos en Excel para la captura de datos para su posterior análisis.
Los resultados se expresan como cantidades (n), porcentajes (%), valores de referencia de acuerdo al nivel de medición, y se hizo una comparación de medias y proporciones con χ2, t de Student y ANOVA. Se obtuvieron razones de desventajas (OR) para identificar variables asociadas a resolución. Se consideró un valor de p < 0.05 para significancia estadística. Se utilizó el paquete IBM©SPSS©Statistics V24 para en análisis estadístico.
Resultados
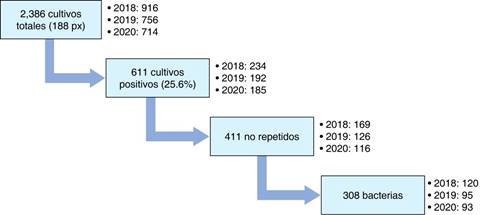
La purga y selección de cultivos a lo largo de 3 años se describe en la Figura 1. Se enfatiza que el análisis final se realizó con 308 aislamientos bacterianos.
Las bacterias gram-negativas predominaron en todo el periodo de estudio, con relación de 65 a 35%. Al considerar los principales aislamientos, las bacterias más frecuentes fueron Escherichia coli (n = 59, 19.16%), Pseudomonas aeruginosa (n = 40, 12.99%) y Klebsiella pneumoniae (n = 28, 9.09%) (Tabla 1).
Tabla 1: Principales aislamientos por año.
| 2018 | 2019 | 2020 | Total | |
|---|---|---|---|---|
| n (%) | ||||
| Escherichia coli | 33 | 11 | 15 | 59 (19.16) |
| Pseudomonas aeruginosa | 18 | 9 | 13 | 40 (12.99) |
| Klebsiella pneumoniae | 14 | 7 | 7 | 28 (9.09) |
| Staphylococcus epidermidis | 8 | 11 | 6 | 25 (8.12) |
| Enterococcus faecalis | 7 | 12 | 5 | 24 (7.79) |
| Enterococcus faecium | 3 | 5 | 8 | 16 (5.19) |
| Stenotrophomonas maltophilia | 6 | 2 | 5 | 13 (4.22) |
| Staphylococcus aureus | 3 | 7 | 3 | 13 (4.22) |
| Acinetobacter baumannii | 3 | 2 | 5 | 10 (3.25) |
| Staphylococcus hominis | 2 | 5 | 1 | 8 (2.60) |
| Proteus mirabilis | 2 | 3 | 2 | 7 (2.27) |
| Enterobacter cloacae | 2 | 2 | 3 | 7 (2.27) |
| Serratia marcescens | 1 | 1 | 4 | 6 (1.95) |
| Staphylococcus haemolyticus | 2 | 1 | 3 | 6 (1.95) |
Los cultivos realizados fueron principalmente de origen respiratorio (expectoración, aspirado traqueal y lavado bronquioalveolar), seguido de urocultivo y hemocultivos. En conjunto representaron 76.7% del total. Las Tablas 2 y 3 resumen el promedio de sensibilidad a las principales bacterias, tanto gram-positivas como gram-negativas, a los antibióticos con mayor uso en nuestra unidad.
Tabla 2: Mapa de sensibilidad a aislamientos principales (gram-positivos).
| Staphylococcus epidermidis, % | Enterococcus faecalis, % | Enterococcus faecium, % | Staphylococcus aureus, % | Staphylococcus hominis, % | Staphylococcus haemolyticus, % | |
|---|---|---|---|---|---|---|
| N = 25 | N = 24 | N = 16 | N = 13 | N = 8 | N = 6 | |
| Gentamicina | 52 | 53 | 39 | 83 | 88 | 0 |
| Tigeciclina | 100 | 100 | 100 | 100 | 100 | 80 |
| Ceftarolina | 100 | 0 | 0 | 100 | 0 | 100 |
| Clindamicina | 13 | 0 | 0 | 54 | 0 | 0 |
| Levofloxacino | 21 | 62 | 38 | 54 | 29 | 0 |
| Moxifloxacino | 26 | 62 | 71 | 54 | 43 | 0 |
| Linezolid | 86 | 100 | 87 | 100 | 100 | 83 |
| Oxacilina | 17 | 50 | 0 | 62 | 0 | 0 |
| Rifampicina | 92 | 0 | 0 | 92 | 71 | 40 |
| Tetraciclina | 79 | 30 | 19 | 92 | 86 | 67 |
| Vancomicina | 96 | 100 | 94 | 100 | 100 | 100 |
| Daptomicina | 100 | 100 | 0 | 100 | 0 | 100 |
| Doxiciclina | 67 | 50 | 67 | 100 | 100 | 100 |
Tabla 3: Mapa de sensibilidad a aislamientos principales (gram-negativos).
| Escherichia coli, % | Pseudomonas aeruginosa, % | Klebsiella pneumoniae, % | Stenotrophomonas maltophilia, % | Acinetobacter baumannii, % | Proteus mirabilis, % | Enterobacter cloacae, % | Serratia marcescens, % | |
|---|---|---|---|---|---|---|---|---|
| N = 59 | N = 40 | N = 28 | N = 13 | N = 10 | N = 7 | N = 7 | N = 6 | |
| Amikacina | 93 | 77 | 92 | 46 | 0 | 100 | 100 | 80 |
| Aztreonam | 32 | 0 | 47 | 0 | 0 | 50 | 67 | – |
| Cefepima | 43 | 60 | 56 | 39 | 30 | 57 | 86 | 60 |
| Ceftazidima | 61 | 47 | 63 | 40 | 20 | 33 | 75 | 50 |
| Ceftriaxona | 43 | 0 | 54 | 0 | 13 | 57 | 71 | 60 |
| Ertapenem | 95 | 0 | 96 | 100 | – | 100 | 86 | 100 |
| Meropenem | 96 | 45 | 96 | 15 | – | 86 | 100 | 80 |
| Piperacilina/tazobactam | 82 | 56 | 59 | 14 | 0 | 100 | 50 | – |
| Tobramicina | 43 | 71 | 63 | 57 | 33 | 100 | 100 | 100 |
| Colistina | 100 | 100 | 100 | 83 | 100 | 0 | 100 | 0 |
| Ciprofloxacino | 16 | 72 | 50 | 46 | 20 | 67 | 86 | 80 |
| Fosfomicina | 86 | 75 | 64 | 50 | 22 | 50 | 50 | 100 |
| Tigeciclina | 100 | 0 | 75 | 75 | 63 | 0 | 100 | 100 |
| Ceftolozano/tazobactam | 89 | 89 | 80 | 75 | 25 | – | – | 100 |
| Ceftazidima/avibactam | 94 | 90 | 86 | 71 | 22 | 100 | 100 | 100 |
El porcentaje general de infección comparado con colonización/contaminación fue de 69.8 versus 30.2%, y comparando gram-positivos con gram-negativos, el porcentaje de infección fue mayor en bacterias gram-negativas (71.8 versus 66%, OR 1.10, IC 95% de 0.913-1.32, p = 0.297). Las bacterias que se asociaron a infección con mayor frecuencia (> 75%) fueron Enterococcus faecalis, Staphylococcus haemolyticus, Acinetobacter baumannii, Escherichia coli, Staphylococcus aureus y Enterococcus faecium. En cambio, aquéllas con mayor asociación a colonización (≥ 50%) fueron Serratia marcescens y Staphylococcus epidermidis (Tabla 4). Por sitio de origen del cultivo, los que se asociaron con mayor frecuencia a infección (> 80%) fueron bilis (100%), líquido pleural (91%), sitio quirúrgico (89%), hemocultivo por punción (83%) y líquido peritoneal (82%). En cambio, el cultivo de expectoración y de punta de catéter venoso central tuvo < 50% de asociación a infección (Tabla 5).
Tabla 4: Proporción de infección y colonización por aislamiento.
| Infección, % | Colonización, % | N | |
|---|---|---|---|
| Enterococcus faecalis | 83.3 | 16.7 | 24 |
| Staphylococcus haemolyticus | 83.3 | 16.7 | 6 |
| Acinetobacter baumannii | 80.0 | 20.0 | 10 |
| Escherichia coli | 79.7 | 20.3 | 59 |
| Staphylococcus aureus | 76.9 | 23.1 | 13 |
| Enterococcus faecium | 75.0 | 25.0 | 16 |
| Klebsiella pneumoniae | 71.4 | 28.6 | 28 |
| Proteus mirabilis | 71.4 | 28.6 | 7 |
| Enterobacter cloacae | 71.4 | 28.6 | 7 |
| Pseudomonas aeruginosa | 65.0 | 35.0 | 40 |
| Staphylococcus hominis | 62.5 | 37.5 | 8 |
| Stenotrophomonas maltophilia | 61.5 | 38.5 | 13 |
| Serratia marcescens | 50.0 | 50.0 | 6 |
| Staphylococcus epidermidis | 28.0 | 72.0 | 25 |
| Total | 68.5 | 31.5 | 262 |
Tabla 5: Infección/colonización por sitio de origen del cultivo.
| Tipo de cultivo | Infección, % | Colonización, % | N |
|---|---|---|---|
| Bilis | 100.0 | 0.0 | 13 |
| Líquido pleural | 90.9 | 9.1 | 11 |
| Herida quirúrgica | 89.3 | 10.7 | 28 |
| Hemocultivo por punción | 83.3 | 16.7 | 24 |
| Líquido peritoneal | 81.8 | 18.2 | 22 |
| Hemocultivo por CVC | 69.0 | 31.0 | 29 |
| LBA | 64.3 | 35.7 | 28 |
| Urocultivo | 57.8 | 42.2 | 83 |
| Aspirado traqueal | 52.9 | 47.1 | 102 |
| Expectoración | 44.9 | 55.1 | 49 |
| Punta de CVC | 9.1 | 90.9 | 11 |
CVC = catéter venoso central.
Al discernir entre infección y colonización, la procedencia comunitaria se asoció más a infección que la nosocomial (85 versus 61.7%, OR 3.5, IC 95% 1.93-6.45, p < 0.001).
La comparativa entre el patrón de sensibilidad a los principales antibióticos utilizados de infecciones comunitarias y nosocomiales se presenta en la Tabla 6.
Tabla 6: Sensibilidad a infecciones comunitarias y nosocomiales.
| Origen | p | ||
|---|---|---|---|
| Comunitario | Nosocomial | ||
| n (%) | n (%) | ||
| Amikacina | 69 (89.9) | 113 (73.5) | 0.022 |
| Aztreonam | 35(34.3) | 32 (34.4) | 0.994 |
| Cefepima | 69 (52.2) | 120 (56.7) | 0.430 |
| Ceftazidima | 22 (59.1) | 56 (58.9) | 0.787 |
| Ceftriaxona | 61(41.0) | 86 (40.7) | 0.334 |
| Ertapenem | 54 (96.3) | 54 (92.6) | 0.401 |
| Meropenem | 68 (91.2) | 112 (65.2) | < 0.001 |
| Piperacilina/tazobactam | 46 (71.7) | 60 (51.7) | 0.073 |
| Colistina | 23 (100.0) | 64 (87.5) | 0.075 |
| Ciprofloxacino | 95 (46.3) | 181 (44.8) | 0.742 |
| Gentamicina | 91 (67.0) | 176 (54.0) | 0.036 |
| Fosfomicina | 28 (64.3) | 73 (68.5) | 0.906 |
| Trimetroprim/sulfametoxazol | 71 (46.5) | 108 (54.6) | 0.556 |
| Tigeciclina | 79 (81.0) | 133 (74.4) | 0.543 |
| Ceftolozano/tazobactam | 13 (84.6) | 32 (81.3) | 0.286 |
| Ceftazidima/avibactam | 20 (90.0) | 63 (81.0) | 0.539 |
| Ceftarolina | 9 (22.2) | 35 (40.0) | 0.406 |
| Clindamicina | 17 (47.1) | 42 (11.9) | 0.012 |
| Levofloxacino | 35 (57.1) | 67 (37.3) | 0.154 |
| Moxifloxacino | 24 (62.5) | 54 (38.9) | 0.142 |
| Linezolid | 32 (100.0) | 60 (91.7) | 0.244 |
| Oxacilina | 11 (72.7) | 41 (14.6) | < 0.001 |
| Rifampicina | 13 (100.0) | 39 (82.1) | 0.260 |
| Tetraciclina | 32 (50.0) | 64 (59.4) | 0.383 |
| Vancomicina | 32 (96.9) | 62 (100.0) | 0.162 |
| Daptomicina | 5 (100.0) | 8 (100.0) | 0.000 |
| Doxiciclina | 3 (66.7) | 12 (83.3) | 0.519 |
En cuanto al uso de antibióticos en la unidad, el antimicrobiano que más se utilizó fue meropenem, con 56% en los pacientes con cultivo positivo, con una media de 8.47 días de duración del tratamiento, seguido de linezolid (30%), vancomicina (20%), piperacilina/tazobactam (19%) y metronidazol (19%), ceftriaxona (16%), tedizolid (15%), ertapenem (13%) tigeciclina (13%), y ciprofloxacino (12%), para completar la lista de los 10 antimicrobianos más utilizados en la UCIA. Se identificaron seis antibióticos (meropenem, colistina, daptomicina, vancomicina, ceftazidima/avibactam y tobramicina nebulizada) que tuvieron un máximo de administración ≥ 28 días.
De los 187 aislamientos en los que se realizó antibiograma para meropenem, 48 fueron resistentes (25.7%), pero 26 (54.2%) de ellos recibió meropenem como tratamiento, y sólo en 13 (50%) se realizó ajuste de antibiótico al tener disponible el resultado del antibiograma.
En cuanto a resolución, la Tabla 7 muestra las variables asociadas. Las infecciones por bacterias gram-positivas tuvieron mayor porcentaje de resolución.
Tabla 7: Variables relacionadas con resolución.
| Resolución | OR | IC 95% | p | |||
|---|---|---|---|---|---|---|
| % | % | Inferior | Superior | |||
| Gram positivo vs gram negativo | 82.1 | 68.8 | 2.07 | 1.16 | 3.70 | 0.019 |
| Infección comunitaria vs nosocomial | 82.2 | 68.7 | 2.11 | 1.18 | 3.77 | 0.016 |
| Infección vs colonización | 70.7 | 80.0 | 0.6 | 0.33 | 1.09 | 0.09 |
| Procalcitonina ≥ 0.5 ng/mL vs < 0.5 | 72.5 | 79.1 | 0.69 | 0.038 | 1.25 | 0.23 |
| PCR ≥ 6 mg/L vs < 6 | 76.3 | 87.5 | 0.45 | 0.05 | 3.89 | 0.46 |
| Leucocitos ≥ 12,000 o ≤ 4,000/mcL vs 4,000-12,000 | 74.7 | 75.5 | 0.96 | 0.55 | 1.67 | 0.88 |
PCR = proteína C reactiva.
Discusión
El objetivo del presente estudio fue documentar el perfil de sensibilidad en la UCIA de nuestro hospital, comparando infecciones comunitarias con nosocomiales, infección con colonización, los antibióticos con mayor uso y los factores asociados a resolución del proceso infeccioso.
El porcentaje de cultivos positivos se mantuvo constante durante los tres años, en promedio de 25%, así como el predominio en la proporción de gram-negativos contra gram-positivos de 65.35%, lo cual es una referencia de estabilidad y homogeneidad de las condiciones generales durante el periodo de estudio. Comparando los resultados con otro reporte de resistencias bacterianas en México (a pesar de tomar en cuenta sólo infecciones nosocomiales y de haberse realizado tanto en pacientes de la terapia intensiva como de piso general de todas las edades), la proporción de gram-negativos y gram-positivos es muy similar (67:33%).7
Casi la mitad de los cultivos eran provenientes del tracto respiratorio (43%), y la combinación de respiratorio, urinario y hemocultivo representaron 76.7% del total, por lo que los resultados del presente estudio aplican sobre todo a infecciones relacionadas a esos aparatos o sistemas. Dicho hallazgo se relaciona también con el hecho de que los principales aislamientos encontrados (41% del total) fueron bacterias gram-negativas: E. coli (19.2%), P. aeruginosa (13%) y K. pneumoniae (9.1%), que suelen detectarse en cultivos respiratorios y urinarios.
El sitio de origen de los cultivos es muy parecido al reportado en el estudio mencionado previamente de Gutiérrez-Muñozy colaboradores (respiratorio 40.7%, hemocultivo 18.1% y urocultivo 10.1%, y su sumatoria de 68.9%).7
En cuanto a los principales agentes aislados, tanto en el estudio de Gutiérrez-Muñoz como en el Informe Anual 2015 de la Red Hospitalaria de Vigilancia Epidemiológica de la Dirección General de Epidemiologia (este último reportó sólo los aislamientos de las UCIA), el principal aislamiento fue Pseudomonas aeruginosa (24 y 17%, respectivamente), seguido de Acinetobacter baumannii (12 y 14%, respectivamente). La E. coli aparece en tercer lugar en el informe de la RHOVE (11%) y en sexto lugar en el estudio de Gutiérrez-Muñoz (7.5%).7,8
Al realizar un comparativo con el reporte de otro estudio nacional contemporáneo, se observa que en E. coli y Klebsiella pneumoniae en nuestra UCI hay mayor sensibilidad a cefalosporinas (cefepime y ceftriaxona 43 versus 31% y ceftazidima 61 versus 31%, respectivamente) y piperacilina/tazobactam (82 versus 77%), pero menor sensibilidad a ciprofloxacino (16 versus 30%), amikacina (93 versus 98%) y carbapenémicos (95 versus 100%). Sin embargo, el porcentaje de sensibilidad a cefalosporinas y quinolonas, incluso a piperacilina/tazobactam es muy bajo.9
En Pseudomonas aeruginosa es muy evidente la baja sensibilidad en nuestro medio a todos los fármacos analizados (amikacina 77%, cefepime 60%, ceftazidima 47%, ciprofloxacino 72%, meropenem 45% y piperacilina/tazobactam 56%), en comparación con lo reportado en el estudio mencionado (amikacina 85%, cefepime 78%, ceftazidima 76%, ciprofloxacino 82%, meropenem 70% y piperacilina/tazobactam 78%). Para Acinetobacter baumannii es aún peor el escenario en ambos estudios, sobre todo en el nuestro, con sensibilidad de 0% a amikacina y piperacilina/tazobactam (comparado con 53 y 40%, respectivamente) y 30% a cefepime y 20% a ciprofloxacino (comparado con 32 y 29%, respectivamente). Otros estudios revisados, tanto nacionales como internacionales, también son consistentes con estos resultados.10-13
El porcentaje general de infección comparado con colonización/contaminación fue de 69.8 versus 30.2%. A pesar de que aislar una bacteria gram-negativa se relacionó con mayor frecuencia a infección (71.8 versus 66%), las bacterias que de manera individual tuvieron mayor asociación con infección fueron gram-positivos (E. faecalis y S. haemolyticus, 83.3%). Esto puede explicarse en parte por la ausencia de significancia estadística en la diferencia porcentual observada (OR 1.10, IC 95% de 0.913-1.32, p = 0.297) así como por el sitio de origen de los mismos, ya que dichas bacterias se aislaron con mayor frecuencia en los cultivos con mayor tasa de infección (Tabla 7).
En el comparativo de infecciones comunitarias y nosocomiales fue evidente mayor asociación entre el aislamiento de un cultivo de origen comunitario con infección comparado con uno nosocomial (85 versus 61.7%, OR 3.5, IC 95% 1.93-6.45, p < 0.001). Esto podría relacionarse con el hecho de que los pacientes hospitalizados, sobre todo aquéllos con larga estancia, suelen colonizarse con flora nosocomial y resultan con cultivos positivos a pesar de no tener algún síndrome infeccioso. El mayor número de cultivos realizados en pacientes hospitalizados obedece a la conducta de realizar cultivos ante la ausencia de criterios de infección, o como comúnmente suele denominarse: «por rutina». A pesar de que desde hace décadas se ha insistido por parte de infectología y microbiología en la optimización de la toma de cultivos y la calidad de los mismos, ha sido difícil su implementación en el ámbito clínico cotidiano.14 El enfoque de medicina defensiva en la práctica actual de la medicina occidental también influye en la toma deliberada de cultivos, sobre todo en el contexto de pacientes críticos. La identificación de criterios predictores de elevada probabilidad pre-prueba para procesos infecciosos se ha identificado sobre todo para hemocultivos, por lo que su implementación sería de gran ayuda en este contexto.15,16
El concepto comúnmente aceptado de que infecciones comunitarias tienen un perfil de sensibilidad mayor que el de infecciones nosocomiales, no se pudo sostener en nuestros resultados. Aunado a la baja sensibilidad a los antimicrobianos, las bacterias comunitarias muestran comportamiento muy parecido a las nosocomiales en la mayoría de los casos. En el análisis realizado sólo se obtuvo mayor sensibilidad con significancia estadística en infecciones comunitarias versus nosocomiales en amikacina (89.9 versus 73.5%, p = 0.022), meropenem (91.2 versus 65.2%, p < 0.001), clindamicina (47.1 versus 11.9%, p = 0.012) y oxacilina (72.7 versus 14.6%, p < 0.001). No es algo aislado que pasa sólo en nuestro medio. Martin-Loeches y colaboradores también lo mencionan en su revisión, destacando el hecho de la migración de la atención hospitalaria al domicilio del paciente o a asilos, así como la mayor interacción de manera frecuente con centros de atención médica (unidades de hemodiálisis o quimioterapia) y el reingreso hospitalario cada vez más frecuente en población añosa y con pluripatología crónica.6
Sin embargo, a pesar de la similitud en la sensibilidad entre infecciones comunitarias y nosocomiales, las primeras tuvieron mayor probabilidad de resolución (82.2 versus 68.7%, OR 2.11, IC 95% de 1.18-3.77, p = 0.016), al igual que las infecciones causadas por bacterias gram-positivas (82.1 versus 68.8%, OR 2.07, IC 95% de 1.16-3.70, p = 0.019). Esto deja en evidencia la influencia de múltiples factores en el desenlace clínico del proceso infeccioso más allá de la sensibilidad antimicrobiana.
Derivado de los mismos resultados, las opciones para tratamiento empírico con sensibilidad mayor de 80% en infecciones comunitarias fueron amikacina (89.9%), ertapenem (96.3%), meropenem (91.2%), colistina (100%), tigeciclina (81%), ceftolozano/tazobactam (84.6%), ceftazidima/avibactam (90%), linezolid (100%), rifampicina (100%), vancomicina (96.9%) y daptomicina (100%). Para infecciones nosocomiales, la lista se reduce a ertapenem (92.6%), colistina (87.5%), ceftolozano/tazobactam (81.3%), ceftazidima/avibactam (81%), linezolid (91.7%), rifampicina (82.1%), vancomicina (100%), daptomicina (100%) y doxiciclina (83.3%).
Dichas opciones son congruentes con las recomendaciones de las guías internacionales de tratamiento empírico en la UCI en el contexto de la alta prevalencia de Pseudomonas aeruginosa y enterobacterias bramnegativas multirresistentes en nuestro medio. Y debido a nuestra baja prevalencia de cocos gram-positivos, y de manera específica de Staphylococcus aureus resistente a meticilina, las opciones empíricas para su tratamiento también son congruentes y suficientes.17,18
Conclusiones
Las bacterias predominantes en nuestra unidad de cuidados críticos son bacilos gram-negativos multidrogorresistentes, provenientes principalmente de infecciones respiratorias, urinarias y de torrente sanguíneo. Es más probable encontrar cultivos positivos relacionados a infección en pacientes con infecciones comunitarias que en nosocomiales. La tasa de sensibilidad es muy similar en infecciones comunitarias y nosocomiales, siendo muy baja en cefalosporinas y quinolonas, y cada vez más en piperacilina/tazobactam, amikacina y carbapenémicos. Dichos antibióticos también fueron los más utilizados. Las infecciones por gram-positivos y adquiridas en la comunidad tuvieron mayor posibilidad de resolución.











 nueva página del texto (beta)
nueva página del texto (beta)