Introducción
El adn antiguo (adna) es el material genético que sobrevive en restos orgánicos recuperados de contextos arqueológicos, históricos o paleontológicos. A pesar de que el estudio del adna comenzó hace más de 30 años [Hagelberg et al. 2015], en la última década se ha visto un crecimiento explosivo del campo. Esto se debe en gran medida al desarrollo de las tecnologías de secuenciación masiva y avances en métodos para muestrear, aislar y capturar el adn preservado en especímenes antiguos. Estos avances han dado pie al desarrollo de la paleogenómica, campo que se enfoca en reconstruir y analizar genomas completos y parciales de restos arqueológicos humanos y no humanos [Sarkissian et al. 2015].
Durante las tres últimas décadas se han llevado a cabo investigaciones antropológicas alrededor del mundo que han muestreado restos esqueléticos humanos utilizando, aproximaciones basadas en el análisis del adna y recientemente de la paleogenómica, lo cual ha fomentado que dichas aproximaciones se conviertan en parte integral de las investigaciones interdisciplinarias que abordan preguntas de interés antropológico, histórico y bioarqueológico. Algunas aplicaciones de la paleogenómica en la bioarqueología incluyen el uso de los datos genómicos para trazar procesos migratorios antiguos [Barquera et al. 2020; Raghavan et al., 2015], confirmar diagnósticos paleopatológicos [Schuenemann et al. 2018], estudiar la evolución de microbios patogénicos o comensales [Bravo et al. 2020], reconstruir la dieta [Jensen et al. 2019], estudiar procesos de domesticación y coevolución [Vallebueno et al. 2016], y determinar el sexo o las relaciones de parentesco entre individuos en contextos mortuorios [Sánchez et al. 2019].
La aplicación de la paleogenómica al estudio del abundante patrimonio cultural de México representa una gran oportunidad para enriquecer los estudios arqueológicos y antropológicos en América Latina. El establecimiento de laboratorios nacionales especializados en paleogenómica, con la infraestructura requerida para la recuperación de adna y la capacitación de recursos humanos locales en metodologías de laboratorio y análisis de datos, ha sentado los cimientos para el desarrollo de una paleogenómica local capaz de liderar estudios nuevos y colaborar de forma equitativa con centros de investigación en el extranjero. No obstante, la obtención de adna es un proceso laborioso, costoso y destructivo, por lo tanto, tiene consecuencias irreversibles para la preservación de las colecciones esqueléticas y el manejo ético y sustentable del patrimonio cultural. Por esta razón es fundamental desarrollar colaboraciones interdisciplinarias entre los laboratorios de paleogenómica y el Instituto Nacional de Antropología e Historia (inah) que fomenten un balance entre la importancia de la investigación y la necesidad de proteger el patrimonio cultural de México. Debido a esto, las investigaciones interdisciplinarias que utilicen aproximaciones de adna deben estar guiadas por tres principios básicos: 1) planteamiento de preguntas de investigación bien definidas; 2) estrategias de muestreo que respondan directamente a los objetivos del estudio, y 3) minimizar la destrucción de los restos óseos [Austin et al. 2019; Fox et al. 2019; Prendergast et al. 2018].
En las siguientes páginas presentamos un breve resumen de la historia del campo del adn antiguo, desde sus comienzos hasta la era de la Paleogenómica. Hacemos énfasis en el uso del adna en la antropología y destacamos ejemplos de estudios de adna realizados en México, que han generado nueva información sobre las dinámicas poblacionales antiguas y la presencia de enfermedades infecciosas en el pasado. Finalmente, discutimos la necesidad de una cultura ética en la investigación paleogenómica, especialmente con relación a las prácticas de muestreo sustentable, la destrucción de los restos óseos, y las políticas de repatriación y devolución de muestras. Concluimos con una reflexión sobre el futuro de la paleogenómica en México.
Historia breve del adn antiguo: desde amplicones hasta genomas completos
Los primeros intentos de obtener adn de restos orgánicos antiguos se llevaron a cabo hace más de 30 años. En 1981, Wang y compañía recuperaron adna del cuerpo preservado de una mujer china de 2 000 años de antigüedad [Wang et al., 1981]. Posteriormente, Higuchi y colaboradores [1984] obtuvieron adna de los restos preservados de un cuagga, una cebra extinta [Higuchi et al.1984]. Posteriormente se publicaron estudios de adna humano aislado de una momia egipcia [Pääbo 1985] y de tejido cerebral humano de 8 000 años de antigüedad [Doran et al. 1986]. En 1989, Hagelberg y compañía demostraron que el adna también se preserva en restos esqueléticos, aunque en muy pocas cantidades y en condiciones de extrema fragmentación [Hagelberg et al. 1989]. A pesar de las limitadas capacidades tecnológicas de la época, estos primeros estudios establecieron el gran potencial del adna para las futuras investigaciones evolutivas y arqueológicas [Hagelberg et al. 2015; Pääbo 1989].
La invención de la reacción en cadena de la polimerasa (pcr), la cual permite amplificar millones de copias de fragmentos de adn a partir de una sola molécula [Saiki et al. 1988], impulsó el rápido desarrollo del campo del adna en la década de los noventa. Combinando la pcr con la secuenciación Sanger [Sanger et al. 1977] fue posible recuperar fragmentos cortos de adna de entre 50 y 200 pares de bases (pb). Estos fragmentos se solapan para ensamblar secuencias de hasta miles de pb. Debido a la mala preservación del adna, las investigaciones de esta época se enfocaron en recuperar fragmentos del genoma mitocondrial, el cual se hereda por la línea materna y se encuentra en mayores cantidades en los tejidos antiguos que el adn nuclear [Giles et al. 1980]. Así se llevaron a cabo los primeros estudios de poblaciones humanas antiguas en contextos arqueológicos del continente americano [Stone et al. 1993] y el Pacífico [Hagelberg et al. 1993].
El estudio del adna del Neandertal a finales de los noventa [Krings et al. 1997] permitió establecer estándares para prevenir la contaminación y asegurar la autenticidad de los resultados de adna [Cooper et al. 2000]. Los criterios de autenticidad de adna actualmente establecidos son: 1) la separación física de áreas de trabajo para procedimientos preamplificación y postamplificación por pcr; 2) la inclusión de controles negativos en cada paso del procesamiento de muestras antiguas en el laboratorio; 3) la examinación de la conducta molecular apropiada del adn recuperado: los tamaños de los fragmentos de adna deben ser pequeños, entre 50-150 pb, debido a los procesos de degradación y deben tener marcas características de daño (figura 1), 4) la cuantificación de la cantidad de adna recuperado; 5) la estimación de posible contaminación, y 6) el requerimiento de reproducibilidad o replicación independiente de los resultados obtenidos [Cooper et al. 2000; Llamas et al. 2017; Orlando et al. 2021].
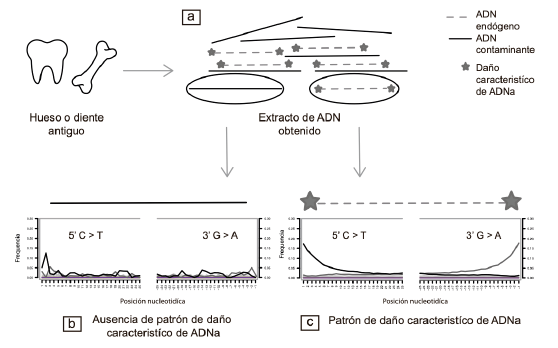
Fuente. Miriam Bravo López.
Figura 1. Perfil de daño característico de adna. a) La estimación del patrón de daño del adna parte de la obtención de secuencias de dnaa de un resto arqueológico por método de ngs. Estas secuencias pueden estar constituidas por adn endógeno (en menor proporción) o adn contaminante (mayor proporción). La estimación del patrón de daño se obtiene a través del alineamiento de estas secuencias con un genoma de referencia del organismo de interés. b) y c) El eje de x” indica la posición nucleotídica de la secuencia, que va en dirección 5’ a 3’ (del lado derecho) y dirección 3’ a 5’ (lado izquierdo). El eje de “y” indica la frecuencia de las modificaciones en las bases de las secuencias de adna: el cambio de la base nucleotídica citosina (c) por timina (t) (línea color negro) y los cambios de la base nucleotídica guanina (g) por adenina (a) (línea color gris). La estimación del patrón de daño de adna se puede obtener a través de diversos programas bioinformáticos.
La llegada de las tecnologías de secuenciación masiva en el 2005, también conocidas como secuenciación de segunda generación o ngs, por sus siglas en inglés, revolucionó el campo del adna e inauguró la era Paleogenómica [Sarkissian et al. 2015]. Mientras que los métodos basados en pcr sólo podían producir copias de un fragmento de adna proveniente de una sola molécula a la vez, los secuenciadores de ngs son capaces de sintetizar las secuencias de miles de moléculas simultáneamente. Esto incrementa la cantidad de secuencias que se pueden generar de una sola corrida, reduciendo el tiempo y costo total del proceso de secuenciación [Goodwin et al. 2016].
Utilizando los métodos de ngs, en el 2010 se publicó el primer genoma completo de un humano antiguo, recuperado del cabello de un hombre que vivió hace 4 000 años en Groenlandia [Rasmussen et al. 2010]. Este gran logro fue seguido de otros, como la secuenciación del genoma completo del Neandertal [Green et al., 2010] y el descubrimiento de una nueva especie de homínidos —los denisovanos— identificados exclusivamente por medio del adna [Krause et al. 2010: Reich et al. 2010]. Los métodos de ngs también permitieron una mejor caracterización de los patrones de daño inherentes al adna y facilitaron la secuenciación de muestras demasiado fragmentadas para la amplificación exitosa por medio de pcr [Briggs et al. 2007].
Durante este periodo también hubo grandes avances en las metodologías de laboratorio. Se descubrió que el adna se preserva mejor en elementos óseos densos como la porción petrosa del hueso temporal [Gamba et al. 2014; Pinhasi et al. 2015] y se mejoraron las técnicas de muestreo para dientes, cálculo dental y huesos largos [Damgaard et al. 2015; Llamas et al. 2017]. También se desarrollaron protocolos de extracción de adna capaces de aislar fragmentos muy pequeños de adna (hasta 25 pb) y de remover sustancias inhibidoras o contaminantes [Dabney et al. 2013; Glocke et al. 2017]. Por último, el uso de métodos para transformar los extractos de adna en genotecas (también conocidas como librerías), combinado con el desarrollo de técnicas para enriquecimiento y captura, han permitido la recuperación de fragmentos de adna endógeno o de agentes patogénicos en muestras mal preservadas o con altas proporciones de adn moderno contaminante (figura 2) [Horn 2012; Orlando et al. 2021]. Todos estos avances han sido acompañados por mejoras en los procesos bioinformáticos para manejar la gran cantidad de datos generados por las plataformas de ngs y el desarrollo de métodos estadísticos para lidiar con la degradación y daño inherentes al adna [Peltzer et al. 2016; Slatkin 2016].
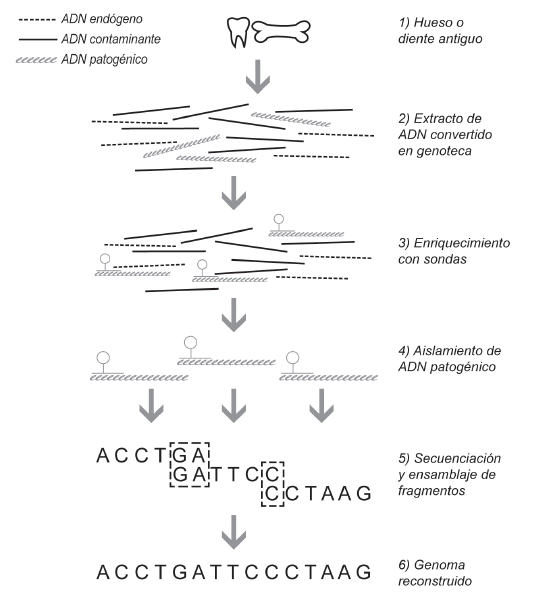
Fuente: Gilberto Martínez Vicentini y María Nieves-Colón.
Figura 2. Proceso de enriquecimiento de adn antiguo de agentes patogénicos mediante el uso de sondas. Las sondas se adhieren únicamente al adn del patógeno de interés permitiendo la remoción de adn contaminante o endógeno.
Mediante la combinación de estas poderosas herramientas, se han secuenciado miles de genomas completos recuperados en contextos arqueológicos y paleontológicos de hasta un millón de años de antigüedad [Valk et al., 2021] y de casi todas partes del mundo; aunque con una mayor preferencia por restos arqueológicos de Europa [Hofreiter et al. 2015]. Se han estudiado temas que se pensaban imposibles hace sólo una década, como, por ejemplo, la diversidad genética de homínidos del Pleistoceno [Meyer et al. 2016], la evolución del microbioma humano [Warinner et al. 2015] o la caracterización de agentes patogénicos relacionados con epidemias antiguas como la peste negra y la tuberculosis prehispánica [Bos et al., 2019]. A pesar de que se continúan utilizando los métodos de pcr y Sanger, la secuenciación por ngs y las técnicas de enriquecimiento se han convertido en estándar en el campo del adna [Orlando et al. 2021]. La era de la paleogenómica llegó para quedarse.
Aplicaciones del adna y la paleogenómica a la bioarqueología en México
Estudios de historia poblacional humana
En México el adna se ha utilizado como herramienta para reconstruir diversos aspectos de las sociedades prehispánicas, incluyendo la estructura poblacional, la composición étnica y las costumbres funerarias. Entre los primeros estudios de adna llevados a cabo en México se encuentran los trabajos pioneros de Vargas Sanders y colaboradores [1989, 1995, 1996], quienes demostraron la preservación de ácidos nucleicos en restos esqueletales humanos de 650 a 750 años de antigüedad excavados en un sitio arqueológico de Iztapalapa, Ciudad de México. Estos primeros estudios (cuadro 1) fueron el precedente para otros que buscaban investigar la diversidad mitocondrial de poblaciones antiguas del norte de México [Morales et al. 2017], la cuenca de México y áreas limítrofes [Aguirre et al. 2017; Kemp et al. 2005; Mizuno et al. 2017; Morales et al., 2019; Solórzano et al. 2011], la costa del Pacifico [Román et al. 2015] y la región Maya hacia el sur del país [González Oliver et al. 2001; Merriwether et al. 1997; Ochoa et al. 2016]. En su gran mayoría estas investigaciones buscaron reconstruir la historia poblacional de los antiguos centros urbanos de México (para una discusión detallada sobre los estudios de adna en Mesoamérica véase [Roca-Rada et al. 2020]).
Cuadro 1. Estudios de adn antiguo en sitios prehispánicos mesoamericanos
| Sitio o atribución cultural | N | Frecuencia de haplogrupos mitocondriales | Fecha | ¿Estimación de sexo? | Referencia | |||
| A | B | C | D | |||||
| Tula | 130 | No determinado | 700-1250 d. C. | Sí | Vargas Sanders et al., 1996 | |||
| Copán | 9 | 0 | 0 | 88 | 12 | 700-1300 d. C. | No | Merriwether et al., 1997 |
| Xcaret | 24 | 88 | 4 | 8 | 0 | 600-1521 d. C. | No | González Oliver et al., 2001 |
| Tlalelcolco | 23 | 65 | 13 | 4 | 18 | 1325 d. C. | No | Kemp et al., 2005 |
| Tlatelolco | 14 | 57 | 21 | 7 | 14 | 1454-1457 d. C. | Sí | De La Cruz et al., 2008 |
| Pre-Azteca | 10 | 30 | 30 | 0 | 40 | 1240-1541 d. C. | No | Mata Míguez et al., 2012 |
| Post-Azteca | 15 | 60 | 20 | 6 | 13 | |||
| Hoyo Negro | 1 | 0 | 0 | 0 | 1 | ~10,500 a. C. | No | Chatters et al., 2004 |
| Teopancazco | 29 | 55 | 21 | 17 | 7 | 200-500 d. C. | Sí | Álvarez Sandoval et al., 2015 |
| Pericúes, Piedra Gorda | 6 | 0 | 3 | 3 | 0 | 800-300 d. C. | Sí | Raghavan et al., 2015 |
| Sierra Tarahumara | 2 | 0 | 0 | 2 | 0 | ~1,500 d. C. | ||
| Xcambó | 2 | 2 | 0 | 0 | 0 | 250-550 d. C. | ||
| Bonampak | 5 | 5 | 0 | 0 | 0 | 580-800 d. C. | ||
| Palenque | 9 | 5 | 0 | 3 | 1 | 750-800 d. C. | ||
| El Rey Quintana Roo | 5 | 3 | 0 | 2 | 0 | 1200-1500 d. C. | ||
| Comalcalco | 8 | 4 | 0 | 3 | 1 | 700-900 d. C. | No | Ochoa Lugo et al., 2016 |
| Tenosique | 5 | 3 | 0 | 2 | 0 | 700-900 d. C. | ||
| Sueños de Oro (El Ciebo), Tenosique | 2 | 0 | 0 | 1 | 1 | 700-900 d. C. | ||
| Calicanto Jalapa | 1 | 1 | 0 | 0 | 0 | 700-900 d. C. | ||
| Peje Lagarto Huimanguillo | 1 | 1 | 0 | 0 | 0 | 700-900 d. C. | ||
| Teotihuacan | 36 | 58 | 25 | 14 | 3 | 300-700 d. C. | No | Aguirre Samudio et al., 2017 |
| Jícaro y La Cascabel | 4 | 0 | 3 | 1 | 0 | 800-1250 d. C. | No | Morales Arce et al., 2017 |
| Paquimé y Convento | 5 | 0 | 2 | 3 | 0 | 1450-1200 d. C. | ||
| Saki Tzul | 2 | 0 | 0 | 0 | 2 | ~5300 a. C. | Sí | Posth et al., 2018 |
| Mayahak Cab Pek | 1 | 0 | 0 | 0 | 1 | ~7300 a. C. | ||
| Tlatelolco | 11 | 55 | 18 | 9 | 18 | 1350-1519 d. C. | Sí | Morales Arce et al., 2019 |
| Cholula | 12 | 42 | 42 | 16 | 0 | 240-1400 d. C. | ||
| Cueva del Terror de Medianoche | 41 | 14 | 2 | 1 | 0 | 1000BP-550 d.C. | Sí | Verdugo, et al., 2020 |
Fuente: Miguel Ángel Contreras-Sieck
Por ejemplo, en una serie de estudios asociados al proyecto “Teotihuacan: élite y gobierno”, Álvarez-Sandoval y colaboradores [2015] caracterizaron un fragmento de 149 pb del genoma mitocondrial de 29 individuos enterrados en Teopancazco, un conjunto residencial en la periferia de la ciudad de Teotihuacán [Álvarez et al. 2015]. Esta investigación encontró una alta diversidad de linajes mitocondriales en la población de Teopancazco que se mantiene constante a través del tiempo, desde la etapa más temprana de ocupación (Tlamilolpa: 150-350 d. C.) hasta la más tardía (Xalolpan: 350-550 d. C.). En este estudio también analizaron el adn mitocondrial de individuos enterrados durante el periodo transicional, el cual se caracteriza por la aparición de nuevas costumbres funerarias como el enterramiento de individuos decapitados, en su mayoría adultos masculinos provenientes de zonas circundantes a Teotihuacán y la costa del golfo. Los linajes mitocondriales encontrados en este periodo se diferencian de aquellos observados en las otras dos etapas (Tlamilolpa y Xalolpan). Estos hallazgos son consistentes con la evidencia arqueológica, la cual sugiere que los conjuntos residenciales periféricos al centro urbano de Teotihuacan eran de carácter multiétnico y multicultural [Manzanilla 2015].
En el estudio de Álvarez-Sandoval y colaboradores también se utilizó el adna para identificar el sexo de 16 infantes encontrados en el sitio de Teopancazco. Gracias a esta identificación se descubrió que, en una de las fosas con individuos decapitados, los infantes masculinos y femeninos estaban posicionados en lados opuestos de la tumba. Este hallazgo sugiere una posible relación entre el sexo del infante y el tipo de enterramiento [Álvarez et al. 2014, 2015].
El adna también se ha utilizado para caracterizar las dinámicas poblacionales e investigar las costumbres funerarias de comunidades prehispánicas en otros centros urbanos como Xaltocan [Mata et al. 2012], Cholula y Tlatelolco [De la Cruz et al. 2008, Morales et al. 2019]. En el caso de Xaltocan, Mata-Míguez y colaboradores documentaron un cambio drástico en los linajes mitocondriales de individuos enterrados en conjuntos residenciales habitados antes y después de la conquista Azteca. Esto sugiere que el imperialismo Azteca propició grandes cambios demográficos y genéticos, tanto en la ciudad-estado de Xaltocan como en otras partes de la cuenca de México. En un estudio más reciente, Morales Arce y colaboradores [2019] caracterizaron la diversidad genética de las poblaciones prehispánicas de Cholula y Tlatelolco. Esta investigación encontró que, aunque la evidencia arqueológica indica similitudes entre la arquitectura y cultura material de Teotihuacan y Cholula durante el periodo clásico (250-600 d. C.) existe una gran diferenciación en la diversidad mitocondrial de las poblaciones prehispánicas de Cholula y Teopancazco.
Otra contribución notable de este estudio fue la determinación del sexo de 15 infantes encontrados en contextos de sacrificio ritual en Tlatelolco. Los análisis demostraron que el 83% de las víctimas eran niñas. Este hallazgo contrasta con un estudio previo de adna el cual encontró que la gran mayoría de los individuos sacrificados en el templo R de Tlatelolco eran infantes masculinos [De la Cruz et al. 2008]. Morales Arce y colaboradores sugieren que esta discrepancia podría reflejar la variedad de costumbres funerarias de Tlatelolco, donde diferentes rituales podrían requerir distintos tipos de víctimas. De forma similar, la aplicación de la paleogenómica ha provisto nueva evidencia sobre el sexo de víctimas seleccionadas para el sacrificio ritual en otros contextos arqueológicos de Mesoamérica, como, por ejemplo, en la Cueva del Terror de Medianoche en Belice [Verdugo et al. 2020].
Por otra parte, el poblamiento de América también ha sido uno de los temas de interés antropológico donde las aproximaciones paleogenómicas han contribuido con nueva información. En México, varios estudios se han enfocado en los debates con respecto a la temporalidad, el número de migraciones, y la población ancestral según propuesta por el modelo paleoamericano. A partir del análisis de la morfología craneal, el modelo paleoamericano sugiere que el poblamiento del continente americano fue llevado a cabo por dos grupos genéticamente distintos: los primeros paleoamericanos, seguido por los ancestros de los grupos nativo americanos actuales [González José et al. 2003].
El primer aporte de la paleogenómica a este debate corresponde al análisis de los restos humanos de “Naia”, una mujer adolescente que vivió hace aproximadamente 12 000 años y cuyos restos fueron recuperados del cenote de Hoyo Negro en la Península de Yucatán. Los análisis de adna permitieron identificar que “Naia” llevaba el haplogrupo mitocondrial d1 [Chatters et al. 2014]. Actualmente, este haplogrupo se encuentra con alta frecuencia en poblaciones indígenas de América del Sur, lo cual contrasta con la asignación de los restos de “Naia” al grupo paleoamericano a partir de su morfología craneofacial, y evidencia la complejidad de la dispersión y evolución de los primeros pobladores de América. Más aún, Raghavan y colaboradores [2015] exploraron la hipótesis del modelo paleoamericano mediante la obtención de datos genómicos antiguos a partir de restos humanos pertenecientes a dos poblaciones que se afirma son “relictos” de los paleoamericanos: seis individuos pericúes del período histórico de Piedra Gorda en Baja California con aproximadamente 800 a 300 años de antigüedad y 11 individuos fueguinos de la Patagonia datando del siglo xix. Las comparaciones con poblaciones nativas americanas modernas y con adna obtenido de dos momias prehispánicas de la Sierra Tarahumara revelaron que los individuos paleoamericanos se agrupan dentro de la diversidad nativa americana. Específicamente los pericúes y las dos momias mostraron mayor afinidad genética con los tarahumaras (rarámuri) y huicholes del norte de México, y con los nahuas centrales contemporáneos. Estos resultados son consistentes con una única migración inicial de todos los nativos americanos y la posterior diversificación dentro del continente americano de una rama norte y sur hace aproximadamente 13 000 años [Raghavan et al. 2015].
Estudios de paleopatología
La historia de movimientos poblacionales prehispánicos en México, sus grandes y densamente poblados centros urbanos antiguos, así como las ricas colecciones arqueológicas que han sobrevivido hasta hoy, hacen de este país un lugar idóneo para estudiar las dinámicas evolutivas de los patógenos humanos. La detección de adna de patógenos y la secuenciación de sus genomas permiten identificar agentes infecciosos, confirmar diagnósticos paleopatológicos, datar la antigüedad del patógeno, reconstruir cambios temporales en su diversidad y estudiar los orígenes zoonóticos de las enfermedades humanas [Bos et al. 2019]. Varios estudios recientes ilustran la utilidad de la paleogenómica para investigar la filogeografía y las dinámicas evolutivas de los patógenos antiguos en contextos arqueológicos de México [Barquera et al. 2020; Bravo et al. 2020; Guzmán et al. 2020; Vågene et al. 2018].
Los primeros acercamientos en la identificación del adna de patógenos en México se basaron en el uso de la técnica de pcr para recuperar segmentos cortos del genoma exclusivos de ciertos patógenos. Varios estudios utilizaron esta técnica para analizar los restos esqueletales de individuos con lesiones indicativas de tuberculosis y treponematosis excavados en contextos arqueológicos de Pajones, Zacatecas, y Tamtoc, San Luis Potosí. En el primer estudio, Martínez Mora y colaboradores [2014] amplificaron un marcador característico del complejo de Mycobacterium tuberculosis, el elemento de inserción is6110, en un individuo de Pajones que presentaba lesiones indicativas de tuberculosis extrapulmonar. Por otra parte, Steffani Vallejo [2014] logró amplificar otro marcador de M. tuberculosis (is1081) en un individuo de Tamtoc. Sin embargo, la autenticidad de los resultados obtenidos por estos estudios es cuestionable dado que estos dos elementos de inserción comparten identidad de secuencia con micobacterias ambientales [Garfias 2016; Müller et al. 2015].
La investigación paleogenómica en México también ha contribuido al estudio de las enfermedades treponémicas o treponematosis, que incluyen cuatro síndromes o presentaciones clínicas causadas por subespecies de Treponema pallidum: la sífilis venérea (T.p. pallidum), la frambesia (T.p. pertenue), el bejel o sífilis endémica (T.p. endemicum) y la pinta (T.p. careteum). Tres de las cuatro subespecies de treponemas (T.p. pallidum, T.p. pertenue y T.p. endemicum) causan lesiones óseas y son detectables en restos esqueléticos humanos. A pesar de que estas enfermedades se han estudiado por décadas, los orígenes y la distribución global antigua de estas tres subespecies continúan siendo debatidos. La similitud genética entre subespecies de treponemas y el solapamiento de sus presentaciones clínicas dificulta su estudio en contextos modernos y antiguos [Mulligan et al. 2008; Noordhoek et al. 1990].
La evidencia bioarqueológica de treponematosis hallada en contextos prehispánicos y coloniales de México ha sido extensamente documentada e investigada [Mansilla et al. 1995, 2005; Márquez et al. 2015; Molto 2005; Salas 1982]. Mansilla y Pijoan [2005] analizaron las colecciones esqueletales de nueve sitios arqueológicos de varios estados de México datando del 900 a. C. hasta el 1520 d. C.. Este estudio demostró la presencia de treponematosis en el México prehispánico y encontró que las presentes enfermedades incrementaron en frecuencia a partir del 1000 d. C. Algunos de los ejemplos más notables de enfermedades treponémicas fueron encontrados en individuos excavados en la cueva de la Candelaria en Coahuila (1100-1300 d. C.) [Pineda et al. 2009]. En este sitio se recuperaron 116 cráneos, de los cuáles 60 presentaban lesiones líticas con formación de hueso reactivo en la bóveda craneal. Además, dos de los 60 cráneos también presentaban caries sicca, una lesión patognomónica de la infección treponémica [Pineda et al. 2009] (figura 3).
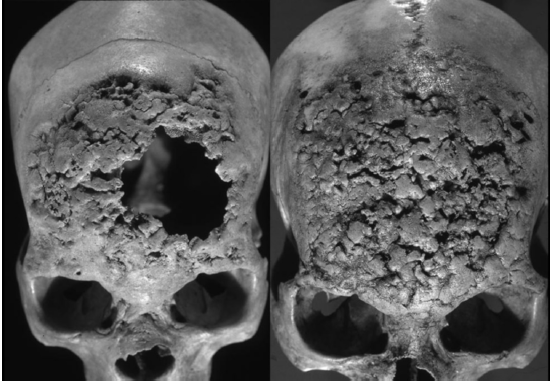
Fuente: Pineda y colaboradores 2009 e inah-daf.
Figura 3. Caries sicca en dos cráneos humanos recuperados de la Cueva de la Candelaria.
En el 2018, Schuenemann y colaboradores reconstruyeron los primeros genomas antiguos completos de T. p. pallidum y T. p. pertenue a partir de los restos óseos de tres infantes enterrados en el convento de Santa Isabel en la Ciudad de México entre los siglos xvii y xix. Hasta este momento, se pensaba que era improbable recuperar genomas completos de T. pallidum antiguo en restos esqueléticos humanos, ya que estudios anteriores identificaron pocas espiroquetas en las lesiones óseas [Hunnius et al. 2007]. Schuenemann y colaboradores [2018] también reportaron evidencia de un posible caso de frambesia congénita, por tanto, una posible coocurrencia de la sífilis venérea y la frambesia en contextos históricos de la Ciudad de México. El diagnóstico diferencial de la sífilis venérea, la frambesia y el bejel, no es posible considerando sólo la evidencia esquelética [Hackett 1976]. Por lo tanto, este trabajo representa una gran contribución al estudio de las treponematosis antiguas, pues mediante la colaboración cercana entre bioarqueólogos y genetistas, la implementación del muestreo paleopatológico informado y los métodos de paleogenómica, se logró la identificación del patógeno y de la subespecie específica de T. pallidum presente en estos casos.
Recientemente, el estudio de Barquera y colaboradores [2020] reconstruyó otro genoma antiguo de T. p. pertenue a partir de restos óseos de un individuo de ancestría africana enterrado en el hospital colonial San José de los Naturales de la Ciudad de México (1500-1700 d. C.). Este genoma está estrechamente relacionado con la cepa de T. p. pertenue identificada previamente por Schuenemann y colaboradores [2018]. Ambos genomas se agrupan filogenéticamente con cepas de T. p. pertenue que se encuentran actualmente en África occidental, lo que plantea más preguntas sobre el origen de este patógeno y la direccionalidad de la transmisión durante la era de la exploración.
Otro ejemplo de investigación interdisciplinaria, donde se combina la paleogenómica y la paleopatología es el estudio de Teposcolula Yucundaa, un sitio posclásico y colonial de la Mixteca Alta de Oaxaca [Spores et al. 2007]. En Teposcolula, la investigación bioarqueológica de fosas con enterramientos múltiples excavados en la gran plaza reveló una curva de mortalidad catastrófica con una tasa irregularmente alta de fallecimientos entre adolescentes y adultos jóvenes [Warinner et al., 2012]. Dado que en el análisis paleopatológico no se encontró evidencia de trauma o infección crónica, Warinner y compañía concluyeron que estos individuos murieron súbitamente debido a una infección viral, posiblemente durante la epidemia de cocoliztli de 1544-1550 d. C.
Para investigar más a fondo la posible causa de la epidemia de cocoliztli, Vågene y colaboradores[2018] extrajeron adna de la pulpa dental de 24 individuos excavados en la gran plaza de Teposcolula. Utilizando técnicas paleogenómicas de vanguardia, incluyendo la secuenciación masiva y el enriquecimiento, en este estudio encontraron que 10 de los 24 individuos resultaron positivos para Salmonella enterica. Con secuenciación a mayor profundidad, se logró reconstruir el genoma completo de S. enterica serovar Paratyphi C, una subespecie de salmonella que infecta sólo a los humanos y causa fiebre paratifoidea. A partir de este resultado, Vågene y colaboradores sugirieron que este patógeno pudo haber causado una o varias de las epidemias documentadas en el registro etnohistórico. Sin embargo, los síntomas de la fiebre paratifoidea no son consistentes con las descripciones históricas de cocoliztli, por lo que este estudio generó diversas controversias [Zhang 2018]. Este patógeno, S. enterica serovar Paratyphi C, también fue detectado en restos humanos antiguos que datan del periodo Neolítico hasta la época Medieval en Europa y Asia [Haller et al., 2021; Key et al. 2020; Zhou et al. 2018]. Esto sugiere que la fiebre paratofoidea surgió en el hemisferio oriental y fue introducida al continente americano durante la época del contacto europeo [Zhou et al. 2018]. De manera interesante, Bravo López [2021] identificó el mismo serovar en una osamenta colonial excavada en el centro de México, lo cual sugiere una amplia distribución geográfica para este patógeno durante la época de la Nueva España.
En su mayoría, los estudios paleogenómicos de patógenos se han enfocado al análisis de bacterias debido a que las infecciones bacterianas tienen mayor probabilidad de dejar lesiones en el esqueleto e indicar su presencia en el registro bioarqueológico. No obstante, como queda demostrado con el caso de S. enterica, los recientes avances en métodos paleogenómicos de laboratorio y análisis también permiten la identificación de patógenos que no dejan marcas esqueletales [Bos et al. 2019]. Esto se logra primordialmente con la extracción del adna preservado en la pulpa dentaria o en el cálculo dental; dichos adelantos han sentado las bases para la detección y el análisis de los virus antiguos. En México, Guzmán Solís y colaboradores [2020] utilizaron esta técnica para identificar dos casos de parvovirus en individuos de ancestría africana enterrados en el Hospital San José de los Naturales (el mismo hospital muestreado por Barquera y colaboradores [2020]). Los análisis filogenéticos de esta cepa de parvovirus indican una relación cercana con un linaje viral africano lo cual sugiere que esta infección fue introducida a México por medio del comercio transatlántico de esclavos.
Hasta el momento son muy escasos los estudios paleogenómicos de patógenos humanos realizados por grupos de investigación mexicanos [Bravo et al. 2020; Guzmán et al. 2020]. Sin embargo, estos estudios representan un precedente para que la investigación paleogenómica en México produzca más hallazgos novedosos e invaluables sobre la historia evolutiva de las enfermedades infecciosas que circulaban en poblaciones antiguas de México y que continúan contribuyendo a la carga global de enfermedades actualmente.
Hacia una paleogenómica ética y sustentable: el manejo de restos esqueletales humanos
Indudablemente, la aplicación de la paleogenómica a la bioarqueología está revolucionando nuestro entendimiento sobre el pasado de formas novedosas e inesperadas, sin embargo, estos avances en conocimiento requieren la destrucción parcial o completa de restos esqueléticos antiguos, un recurso no renovable y patrimonio cultural de enorme significado que además está legalmente bajo resguardo y preservación por parte del inah. Más aún, cuando un resto arqueológico orgánico (hueso, diente, cálculo dental, coprolito) es consumido por el proceso de muestreo, se pierden oportunidades de obtener más información sobre el mismo y la posibilidad de que futuras generaciones puedan llevar a cabo investigaciones aplicando futuros avances tecnológicos [Fox et al. 2019]. Por tanto, al igual que los arqueólogos consideran las consecuencias de la excavación destructiva sobre la preservación de los sitios antiguos [Harris 2006], los antropólogos y los paleogenetistas deben construir espacios de diálogo donde se considere de manera interdisciplinaria el impacto del muestreo paleogenómico de restos humanos para la preservación de las colecciones esqueléticas [Austin et al., 2019; Fox et al. 2019]. Consideramos que la investigación paleogenómica con restos arqueológicos debe estar guiada por preguntas de investigación bien definidas y adherirse a tres principios básicos: 1) ética y respeto; 2) colaboración y consulta, y 3) sustentabilidad.
Ética y respeto:
Los restos humanos no son artefactos, son los vestigios de individuos o ancestros que podrían tener descendientes o hasta familiares que aún viven. En países como México, los restos forman parte del patrimonio cultural y nacional de todos los ciudadanos [González Sobrino et al. 2011]. Por tanto, la investigación con restos humanos debe llevarse a cabo con respeto y considerar las perspectivas de diversas partes interesadas [Bardill et al. 2018]. En América Latina estas partes pueden incluir el estado, las instituciones gubernamentales y museos que manejan el patrimonio cultural, las comunidades indígenas o descendientes, el gremio científico y el público en general, entre otros.
Antes de comenzar un estudio paleogenómico, es necesario entender las leyes y regulaciones que rodean los restos humanos en el país o comunidad en el que se está trabajando. Estas regulaciones pueden tener vigencia a distintos niveles de organización y jerarquía. En el caso de México, el inah a través de la Ley Federal sobre Monumentos y Zonas Arqueológicas, Artísticos e Históricos [1975] es el principal responsable de la custodia, preservación e investigación que involucre el análisis de restos humanos provenientes de sitios arqueológicos o contextos históricos. Por otra parte, en 2019 el inah publicó los “Lineamientos generales para el manejo y resguardo de restos humanos” donde se estipulan las consideraciones para regular la protección, el traslado, destino, depósito, seguridad y gestión de los restos arqueológicos humanos en el territorio nacional. Estos lineamientos se encuentran en la normateca interna del sitio web del inah y representan un primer paso para desarrollar protocolos éticos de investigaciones paleogenómicas con restos humanos antiguos, por ejemplo, uno de los requisitos indispensables para realizar el muestreo de algún resto arqueológico es su inscripción ante el Sistema Único de la Dirección del Registro Público de Monumentos y Zonas Arqueológicas e Históricas. Las instituciones que se encargan del cumplimiento de estos lineamientos son las Coordinaciones Nacionales de Arqueología y de Antropología, la Dirección de Antropología Física y los Centros inah, así como el Consejo de Arqueología.
El Consejo de Arqueología es un consejo asesor y regulador compuesto por 13 funcionarios: un representante de cada dirección del inah (Salvamento Arqueológico, Registro Arqueológico, Dirección de Estudios Arqueológicos, Coordinación Nacional de Conservación del Patrimonio Cultural y la Coordinación Nacional de Arqueología), cuatro representantes de los centros regionales del inah, tres académicos o investigadores y un presidente designado. El Consejo de Arqueología es el responsable de hacer cumplir las regulaciones del inah, incluidas las directrices generales antes mencionadas, a través de la aprobación, supervisión y regulación de todos los proyectos arqueológicos mexicanos [Ortega et al. 2011]. Si bien, el permiso para realizar investigaciones no destructivas en colecciones arqueológicas se deja a discreción de la institución que cura la colección, sólo el Consejo de Arqueología puede otorgar o denegar el permiso para los análisis destructivos de materiales arqueológicos, incluidos restos humanos, así como su exportación internacional. El Consejo facilita la evaluación de todas las propuestas de investigación que solicitan análisis destructivos a través de una junta de revisión de investigadores externos. Aunque las propuestas se revisan externamente, el Consejo toma la decisión final sobre si las muestras se pueden exportar o si se pueden realizar análisis destructivos.
En otros países como, por ejemplo, en Estados Unidos, la investigación bioarqueológica con restos esqueléticos de poblaciones indígenas debe cumplir con las estipulaciones de leyes federales como la Native American Graves Protection and Repatration Act (nagpra). Además, si los restos están afiliados a comunidades indígenas descendientes, éstos también son regulados por las leyes y códigos de ética de los gobiernos tribales soberanos [Claw et al. 2018]. Por último, los museos o universidades que custodian los restos tienen sus propias regulaciones y procesos de revisión institucional para la investigación destructiva [Austin et al. 2019]. Es la responsabilidad del investigador o investigadora entender quiénes son las partes interesadas en los restos esqueléticos humanos en cuestión y obtener los permisos y consentimientos necesarios antes de comenzar un proyecto.
El manejo y estudio de restos humanos también debe tomar en cuenta los posibles impactos de la investigación para las comunidades descendientes. Los datos genéticos y paleogenómicos pueden tener fuertes implicaciones para el entendimiento del pasado, la identidad y la autovisión de las comunidades [González Sobrino et al. 2011]. Por ejemplo, los hallazgos de la investigación con adna pueden afectar nociones de identidad y pertenencia, revelar características que pueden ser estigmatizantes (propensidad a enfermedades) o entrar en conflicto con creencias ancestrales o ideologías espirituales [Bardill et al. 2018].
Más aún, la investigación científica, especialmente los estudios genómicos, están profundamente contextualizados por los diferenciales históricos de poder entre científicos y sujetos. Muchas comunidades históricamente marginadas, como las poblaciones indígenas en Australia y Estados Unidos, han sido víctimas de prácticas poco éticas o negligentes de parte de la comunidad científica, que ha creado un clima de desconfianza, primordialmente en lo que concierne al manejo de restos humanos o materiales biológicos [Claw et al. 2018; Guglielmi 2019; Phillips 2019]. Para evitar reproducir estos esquemas de desigualdad, antes de comenzar un proyecto de investigación en paleogenómica se deben balancear y considerar sus posibles impactos y consecuencias. Teniendo en cuenta que el adna puede influir en la percepción que las personas tienen del pasado y por consiguiente de su relación con el presente, la comunidad científica debe preguntarse: ¿Quiénes se reconocen como descendientes de los restos humanos analizados? ¿Cuáles son los potenciales beneficios del estudio versus sus posibles consecuencias para las comunidades descendientes involucradas? ¿Cuál es el beneficio del proyecto para la comunidad científica y el público en general? ¿Cómo se pueden reconciliar estos dos aspectos de manera equitativa?.
En México hay actualmente pocas discusiones en torno a reclamos de partes interesadas sobre restos humanos arqueológicos, que puede atribuirse al sentimiento de que la herencia mexicana pertenece a todos los ciudadanos mexicanos y es una fuente fundamental de orgullo nacional. Cucina [2013] explica que este patrimonio cultural compartido en el ámbito nacional se debe en gran parte a la asimilación durante la colonización y la historia reciente de México, en comparación con el borrado sistemático y el genocidio que ocurrió durante la colonización europea de Estados Unidos y Canadá. A pesar del sentimiento de patrimonio nacional que comparten muchos mestizos e indígenas [Whittaker 2020], no se puede ignorar que las regiones más empobrecidas de México tienen poblaciones predominantemente indígenas. El énfasis nacional en una identidad multicultural compartida después de la Revolución mexicana, combinado con las crecientes disparidades de riqueza entre las áreas rurales y urbanas, pudo haber provocado la disolución de las identidades de las minorías étnicas [López 2009]. Los grupos que pudieron haber reconocido la continuidad con sus antepasados prehispánicos carecieron del poder colectivo para organizar y expresar sus preocupaciones. Una encuesta a los habitantes de Quintana Roo reveló que los mayas que viven hoy en día no sienten una conexión significativa con los antiguos mayas y, por lo tanto, con los restos esqueléticos encontrados en sitios arqueológicos de la región [Ortega Muñoz 2010]. Sin embargo, Ortega Muñoz [2011] argumenta que podría deberse a décadas de mercantilización de los recursos culturales por parte del gobierno nacional que ha condensado diversas herencias culturales en un “bien de mercado”, eliminando vínculos significativos que pudieran haber tenido los habitantes locales. Por lo tanto, aunque muchos mexicanos no identifican los restos esqueléticos arqueológicos como sus antepasados, los investigadores deben considerar las formas en cómo el discurso nacional y la desigualdad de recursos pueden haber cortado las conexiones culturales y espirituales que alguna vez se sostuvieron entre las poblaciones modernas y antiguas.
Consultar y colaborar
La colaboración y consulta aseguran que las investigaciones con aproximaciones paleogenómicas consideren preguntas de relevancia y beneficio mutuo para las diversas partes interesadas [Claw et al. 2018; Matisoo Smith 2019; Tackney et al. 2019]. Por consulta nos referimos a la acción de considerar como consultores a representantes de las diversas partes involucradas en el estudio de los restos humanos, que puede incluir arqueólogos y antropólogos, curadores de museo, paleogenetistas, y miembros de las comunidades descendientes o indígenas, entre otros. La consulta con estos últimos puede promover la creación de lazos colaborativos a largo plazo y potenciar proyectos futuros con un aporte comunitario. La comunidad también puede contribuir al diseño del proyecto científico con perspectivas locales, ayudando a formular preguntas e hipótesis de interés mutuo, proporcionando información crucial sobre el contexto cultural, etnohistórico y arqueológico de los restos o ayudando a interpretar los datos y llegar a conclusiones informadas por su cosmovisión o experiencias locales [Claw et al. 2018; Matisoo et al. 2019; Tackney et al. 2019]. La colaboración equitativa entre comunidades e investigadores lleva a la construcción de competencia cultural entre los científicos, asegura el respeto mutuo y fomenta el intercambio bidireccional de conocimiento e información; también puede llevar al desarrollo de oportunidades educativas y de difusión, por consiguiente, promover la creación de capacidad local para la investigación [Claw et al. 2018; Wade 2018].
Esto último es particularmente importante en el contexto de los estudios paleogenómicos, los cuales usualmente se llevan a cabo mediante colaboraciones internacionales debido a que requieren acceso a grandes recursos tecnológicos y financieros. En su peor forma, las colaboraciones reproducen los esquemas colonialistas e imperialistas de desigualdad e inequidad que han marcado la ciencia occidental [Ávila 2018; Prendergast et al. 2018]. Para contrarrestar, es importante fomentar y apoyar la creación de capacidad mediante colaboración equitativa y bidireccional con científicos y partes interesadas locales
En el caso de México, es necesario entrenar estudiantes e involucrar a las instituciones antropológicas y laboratorios locales en el proceso de investigación, fomentar la transferencia de tecnología y garantizar el acceso a la información genómica generada, en lugar de utilizar estos recursos solamente para exportar muestras antiguas [Claw et al. 2018]. La colaboración equitativa también tiene el beneficio de reunir equipos científicos interdisciplinarios los cuales, al contar con diversas experiencias y perspectivas, y fomentar la consulta y colaboración con las partes interesadas, están mejor equipados para reconstruir el pasado de las poblaciones humanas de forma holística e integral [Downes 2019; Tackney et al. 2019].
Sustentabilidad
El proceso de obtener adna de restos esqueléticos humanos requiere la destrucción de dientes, huesos y otros tejidos antiguos. Aunque esto representa una pérdida irreversible para el patrimonio cultural, la paleogenómica sustentable busca el balance entre minimizar la destrucción, maximizar el potencial beneficio del descubrimiento científico, y salvaguardar los restos para investigaciones futuras, así que es necesario tener un estrecho vínculo entre la estrategia de muestreo y la pregunta de investigación [Fox et al. 2019; Prendergast et al. 2018]. Antes de destruir una muestra histórica o arqueológica, el investigador debe preguntarse: ¿Qué información proveen los datos genéticos que no puede obtenerse con otros métodos no destructivos? ¿Cómo contribuye esta información a responder la pregunta central o probar la hipótesis principal del estudio? Estas preguntas son fundamentales ya que en la última década se han publicado un número de protocolos de muestreo de adna [Xavier et al. 2021] en diferentes elementos óseos al igual que de sedimentos proximales a los restos humanos antiguos [Epp et al. 2019] evidenciando el rápido avance del campo y la necesidad de que la estrategia de muestreo esté enfocada a responder directamente los objetivos de la investigación.
El muestreo debe tomar en cuenta el contexto arqueológico y las condiciones de preservación de los restos, por ejemplo, se deben considerar cuáles sitios, individuos y/o contextos fúnebres representan la mejor oportunidad para contestar las preguntas de investigación. Dado el contexto tafonómico y la preservación macroscópica, es posible que distintas partes del esqueleto devenguen distintos resultados [Hansen et al. 2017]. En general, se debe evitar muestrear elementos anatómicos únicos, morfológica o culturalmente informativos como aquellos usados para la estimación de sexo o edad, elementos con marcadores de trauma, actividad, modificación cultural intencional o cambios patológicos. Si el objetivo del estudio es paleopatológico, el investigador debe considerar muestrear sólo aquellos elementos que probablemente posean la mayor concentración de agentes patogénicos [Margaryan et al. 2018], como ejemplo, no se debe muestrear un fémur si el fin del estudio es obtener un patógeno que sólo afecta los dientes. Finalmente, durante el muestreo se debe prevenir la contaminación de los restos, mediante la utilización de guantes y mascarillas, y la esterilización de superficies de trabajo, entre otras precauciones [Llamas et al. 2017; Prendergast et al. 2018].
Antes de la destrucción del elemento óseo, éste debe ser documentado rigurosamente, especialmente si contiene marcadores informativos de algún proceso biológico, patológico o cultural (figura 4). Esta documentación debe incluir un registro fotográfico, descripciones macroscópicas, mediciones de longitud y peso, también se pueden tomar escaneos 3d o producir moldes de los elementos óseos para preservar sus características morfológicas. Toda esta información debe ser almacenada en una base de datos asociada al estudio, la cual debe ser accesible a los investigadores y a las instituciones custodias de los restos, como museos, universidades, osteotecas o gobiernos tribales (figura 5).

Fuente: Kelly E. Blevins.
Figura 4. Ejemplo de documentación fotográfica durante el muestreo de elementos óseos patológicos para el análisis de adn antiguo. Antes (a) y después (b) de la toma de muestra.
Fuente: Kelly E. Blevins.
Figura 5. Ejemplo de base de datos documentando el muestreo de restos esqueléticos para un proyecto de investigación en paleopatología molecular.
Para garantizar el desarrollo de la paleogenómica sustentable en el marco de colaboraciones internacionales, los proyectos también deben considerar la transferencia tecnológica como un punto fundamental de la colaboración con países en vías de desarrollo con el fin de garantizar que se tengan los equipos y elementos necesarios para garantizar el cumplimiento de estos estándares de documentación. De esta forma, las instituciones locales que desempeñan el importante papel de seguimiento de los restos podrán garantizar la integración de cualquier elemento devuelto nuevamente a la colección. Mas aún, la información documental provista por los investigadores o investigadoras ayudará a monitorear cuáles individuos en la colección han sido muestreados, de parte de qué institución, para qué proyecto y qué resultados se obtuvieron (positivos o negativos), para poder prevenir la destrucción repetida de elementos óseos de los mismos individuos [Austin et al. 2019; Fox et al. 2019; Prendergast et al. 2018].
Finalmente, el investigador debe tener claro cuáles son las políticas de devolución y/o repatriación para las muestras y el material genético restante al final de la investigación. Los primeros pedidos para el establecimiento de políticas de retorno estandarizadas y acuerdos para garantizar la accesibilidad de la investigación internacional a los investigadores mexicanos datan de la década de los noventa [Márquez 1999], pero no se establecieron reglas formales hasta la publicación de los lineamientos generales para el manejo de restos humanos del inah en el 2019 [inah 2019]. Aunque la decimoquinta disposición de este reglamento establece que el material residual debe ser devuelto y cualquier publicación resultante debe compartirse con el inah, el proceso no especificado puede hacer que esta estipulación sea difícil de hacer cumplir y conduce a una interpretación subjetiva de “material residual”.
La falta de devolución de restos óseos daña la integridad a largo plazo de las colecciones esqueléticas. La devolución y almacenaje de elementos óseos no utilizados, polvo o extracto existentes puede potenciar estudios futuros con la misma colección sin necesidad de repetir el muestreo destructivo [Austin et al. 2019]. Las políticas de devolución también pueden promover relaciones mutuamente beneficiosas entre los investigadores y comunidades indígenas o descendientes. En algunos casos las comunidades pueden considerar que el material genético o los restos humanos han sido prestados a los investigadores, pero siguen siendo de su propiedad y deben ser devueltos al finalizar el estudio (“dna on loan”) [Arbour et al. 2006; Matisoo 2019]. La estipulación de acuerdos claros de devolución es esencial para la sustentabilidad a largo plazo de las relaciones de mutuo beneficio entre la investigación paleogenómica, las comunidades descendientes y las instituciones custodias del patrimonio cultural.
Conclusiones: El futuro de la paleogenómica en México
En los últimos 30 años, el estudio del adn antiguo se ha convertido en parte integral de la bioarqueología. Los adelantos tecnológicos y metodológicos de la era Paleogenómica han permitido la generación de grandes cantidades de datos genéticos y genómicos, mientras que el desarrollo de métodos de muestreo sustentables ha comenzado a minimizar la destrucción de los restos óseos y asegurar la preservación futura de los recursos culturales; los presentes avances facilitan la integración de la paleogenómica a proyectos bioarqueológicos multidisciplinarios que combinan diversas líneas de evidencia para reconstruir la experiencia humana en la antigüedad.
En México, el campo de la paleogenómica está creciendo gracias al establecimiento de laboratorios y grupos de investigación especializados en adna en instituciones como el Laboratorio Internacional de Investigación sobre el Genoma Humano (liigh-unam), el Laboratorio Nacional de Genómica para la Biodiversidad (langebio-cinvestav) y el Laboratorio de Genética Molecular de la Escuela Nacional de Antropología e Historia (enah) [dgcs 2018]; el desarrollo de programas de entrenamiento en adna en centros educativos como la enah [inah 2019] y el apoyo del inah hacia la investigación de adna [inah 2017]. Este crecimiento de capacidad local guarda gran potencial para el desarrollo de proyectos futuros liderados por científicos locales y de colaboraciones equitativas con científicos del exterior.
Gracias a su rico patrimonio cultural y el crecimiento de capacidad local para la investigación esperamos que en las próximas décadas México se posicione como un líder de la paleogenómica. Esperamos que el liderazgo se distinga por el desarrollo de proyectos colaborativos entre antropólogos y paleogenetistas que cuenten con preguntas y objetivos bien definidos, consideren diversos intereses y puntos de vista, y fomenten prácticas éticas y sustentables para la investigación con adn antiguo.











 nueva página del texto (beta)
nueva página del texto (beta)


