Antecedentes
En diciembre de 2019, una escalada en los casos de infecciones respiratorias se registró en la ciudad de Wuhan, provincia de Hubei, China (Huang, Chaolin et al., 2020). Esta neumonía atípica rememora las infecciones respiratorias emergentes de 2002 y 2012, conocidas como SARS (severe acute respiratory syndrome) y MERS (Middle East respiratory syndrome), respectivamente (Cascaella et al., 2020). Para el 31 de diciembre de 2019, el gobierno chino reportó el brote a la Organización Mundial de la Salud (OMS), señalando como agente etiológico un virus, el cual fue nombrado por el Comité Internacional de Taxonomía de Virus como síndrome respiratorio agudo severo por coronavirus 2 (SARS-CoV-2) (WHO, 2020a). En febrero de 2020, la OMS denomina al síndrome respiratorio como enfermedad por coronavirus de 2019 (Covid-19, por su abreviatura en inglés) (WHO, 2020a). El inicio del brote fue rastreado hasta un mercado de productos frescos y animales vivos en Wuhan, lo que aportó información para clasificar la infección como zoonótica y de alta transmisibilidad entre humanos; ello con base en sólida evidencia genética y epidemiológica (Andersen et al., 2020). El número de reproducción básica (R0), un indicador de transmisibilidad del virus, ha sido estimado tanto por la OMS como por diversos estudios académicos independientes, encontrando un valor promedio de 3.28, con una mediana de 2.79 (Liu et al., 2020; He et al., 2020). Este valor es mayor a aquellos observados para la influenza H1NI (1.14-1.36) (Glass et al., 2012) y el SARS (2.2-3.6) (Lipsitch et al., 2003), y es indicativo de que, en un entorno sin medidas de contención, un individuo infectado es capaz de contagiar a más de dos personas en un periodo de tres a cuatro días (Liu et al., 2020; He et al., 2020). No obstante, el valor de R0 es dinámico y puede aumentar o descender en función de las medidas gubernamentales de contención social, seguimiento epidemiológico y tratamiento de los enfermos (Fang et al., 2020). El 11 de marzo de 2020, la OMS declara a Covid-19 como una pandemia (WHO, 2020a), lo que lleva el reto a cotas globales, obligando a los gobiernos a replantearse sus relaciones con el mundo y a encarar uno de los aspectos más negativos de la sociedad globalizada, la rápida diseminación de las enfermedades emergentes. Al respecto, se prevé que Covid-19 llegue a convertirse en una enfermedad endémica en todo el mundo, para lo cual el Centro de Control de Enfermedades de los Estados Unidos ha usado el neologismo «híper endémico». Para mediados de mayo de 2020, la OMS reportó más de 4.5 millones de casos de Covid-19 confirmados a nivel mundial, incluyendo 307,337 defunciones (WHO, 2020b). En comparación, los coronavirus causantes de SARS y MERS produjeron cerca de 10,000 infectados, con tasas de mortalidad del 10 y 37%, respectivamente (Peeri et al., 2020). Hasta el momento de la publicación de este trabajo, los casos de Covid-19 han superado en 450 veces a las infecciones emergentes previas, mientras que la mortalidad se mantiene en alrededor del 7% (WHO, 2020b).
Los objetivos de este artículo de revisión son los siguientes:
Proporcionar al lector una visión general de la enfermedad Covid-19 y su agente etiológico, el virus SARS-CoV-2.
Dar a conocer las aportaciones de la nanociencia en la construcción del actual cuerpo de información que se tiene sobre la naturaleza del virus, su interacción con el cuerpo humano y los métodos de detección y cuantificación actualmente disponibles para corroborar el diagnóstico de los pacientes y dar seguimiento a la epidemia con información fidedigna.
Presentar algunos casos en los que la nanociencia y la nanotecnología han provisto de mejoras a los actuales métodos de medición del SARS-CoV-2 en ensayos moleculares varios que pueden garantizar la detección temprana y certera de la infección.
Divulgar las potencialidades de la nanotecnología para encarar y/o dar soluciones a los grandes retos que trae consigo esta pandemia, siendo uno de ellos la formulación de una vacuna eficaz para inmunizar a la población global.
Sobre el virus y la enfermedad
El SARS-CoV-2 es un virus perteneciente al género β-coronavirus, cuyo material genético es una cadena de ácido ribonucleico (ARN). El material genético del virus (aproximadamente 30 kb) se halla protegido del medio por cuatro proteínas estructurales principales (ver Figura 1), las cuales se describen a continuación (Bar-On et al., 2020).
La proteína E (envoltorio) es un componente transmembrana del virus, el cual está involucrado en el intercambio de iones entre el interior de la partícula viral y el entorno. También interviene en el proceso de ingreso del virus a la célula hospedera mediante interacciones con las proteínas de la membrana celular.
La proteína M (membrana) es una glicoproteína que funge como membrana de la partícula viral y da soporte a la proteína E. La proteína M es la más abundante en las partículas virales, y consiste de tres dominios transmembrana. Esta proteína es crucial en el ensamblaje de las partículas virales, pues provee el andamiaje que da la forma y estructura al virus.
La proteína S (espiga) es una glicoproteína transmembrana que sobresale de la partícula viral, poseyendo un ectodominio mayor a los 10 nm. Esta proteína está formada hasta por 300 monómeros, cuyas subunidades S1 y S2 son las que llevan al anclaje del virus en los receptores de las células hospederas.
La proteína N (nucleocápsula) está unida al material genético del virus, formando la «nucloecápside», la cual protege al ARN del medio, y es fundamental para la liberación del ARN en el citoplasma de la célula infectada.
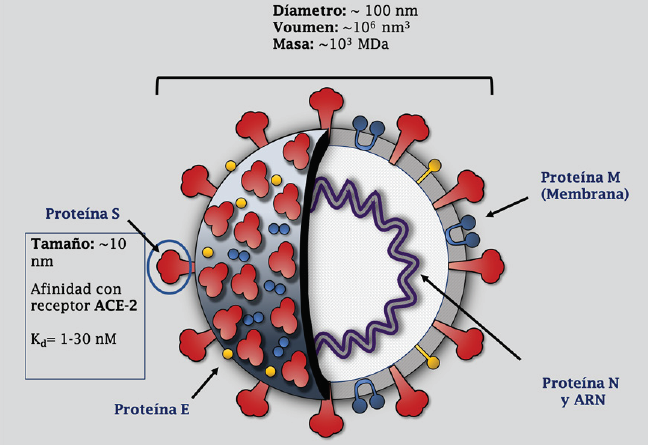
Nota: Se muestra el material genético ARN y las cuatro proteínas estructurales más importantes: membrana (M), envoltorio (E), nucloepéptido (N) y espiga (S). También se provee información estructural del virus y su principal dominio de enlace con las células hospederas.
Fuente: Elaboración de los autores.
Figura 1 Representación esquemática del virus SARS-CoV-2.
El virus cuenta también con 10 a 14 proteínas no estructurales o funcionales, conocidas como ORF (open reading frame), cuyas funciones permanecen aún poco conocidas (Bar-On et al., 2020). La identidad genética del virus mantiene un 97% de similitud con uno de los β-coronavirus que infecta a los murciélagos, mientras que se guarda una menor compatibilidad genética con los virus SARS-CoV (75-80%) y MERS-CoV (50%) (Zheng, 2020; Bar-On et al., 2020).
Normalmente, los β-coronavirus infectan a animales silvestres como los murciélagos, los camellos o los pangolines. No obstante, desde la década de los 60 del siglo XX, se tiene conocimiento de mutaciones que hacen a los virus infecciosos para los seres humanos, siendo conocidos como «coronavirus humanos» (HCoV) (Hamre y Procknow, 1966). Los primeros cuatro HCoV reportados en humanos (HKU1, OC43, NL63 y 229E) son causantes de síntomas respiratorios y gastrointestinales leves a moderados (Corman et al., 2018). No obstante, a partir del siglo XXI, los nuevos HCoVs son capaces de desencadenar complejos cuadros de síndromes respiratorios severos, iniciando en 2002 con el SARS-CoV, después en 2012 con el MERS-CoV y en 2019 con el SARS-CoV-2 (Bar-On et al., 2020).
Los estudios genéticos hasta ahora realizados relacionan la patogenicidad de los coronavirus hacia los humanos con una mutación en los dominios de unión con el receptor (DUR) que se hallan en la glicoproteína S (Tang et al., 2020; Wrapp et al., 2020). De este modo, la fisiopatología del virus inicia con el enlace entre el dominio de unión de la proteína S con los receptores de la enzima convertidora de angiotensina 2 (ECA2) (Lan et al., 2020), los cuales son abundantes en las células neumocito tipo II de los alveolos (Kuba et al., 2005). Esta unión se ve facilitada por la proteína TMPRSS2, misma que actúa como co-factor al separar a dos de los monómeros de la proteína S para incrementar la eficiencia de fusión entre la proteína S y el receptor ECA2, como se muestra en la Figura 2 (Hoffmann et al., 2020). Cabe mencionar que los receptores ECA2 están presentes en células del corazón, riñón, intestino delgado, bazo, colon e hígado, resultando en padecimientos concomitantes relacionados con estos órganos durante la infección (Hamming et al., 2004). Por esta razón, algunos casos de Covid-19 cursan con diarrea; además, el daño a nivel celular en otros órganos puede generar disrupción a nivel tisular, acompañado de una respuesta exacerbada del sistema inmune innato, resultando en una tormenta de citosinas, como se ha observado en algunos pacientes en etapas avanzadas de la infección. Aunque la principal entrada del virus al cuerpo es la vía aérea, este puede alcanzar otros órganos migrando al torrente sanguíneo tras cruzar la barrera hemato alveolar (Ziegler et al., 2020).

Nota: El dominio de unión al receptor de la proteína S en la superficie del virus queda expuesto por la interacción de la proteína TMPRSS2, la cual expone a la proteína S1 y facilita el contacto con el receptor ECA2. Posteriormente, ocurre la endocitosis del virus, fracturando la membrana celular.
Fuente: Elaboración de los autores.
Figura 2 Entrada del SARS-CoV-2 a la célula hospedera.
El virus penetra en la célula hospedera por endocitosis, rodeándose por la membrana celular para crear un endosoma ácido, el cual se transporta por el citosol para posteriormente desensamblarse y liberar al material genético viral (ARN) en el citoplasma. Con ayuda de los ribosomas de la célula hospedera, el ARN viral se traduce para generar poliproteínas no estructurales, mismas que son transformadas por proteasas en la enzima ARN polimerasa dependiente del ARN, la cual replica al material genético y a las proteínas estructurales del virus, como partes separadas en el citosol (Yuki et al., 2020). Las partes del virus son llevadas a los organelos celulares, como el aparato de Golgi o el retículo endoplasmático, para ensamblar a una partícula viral activa. Los virus ensamblados salen de la célula, llevando consigo parte de la membrana celular, lo que causa estrés celular (Fu et al., 2020). Esto genera una respuesta por parte de las células dañadas, marcada por la generación de interleucinas, llevando en algunos casos a una respuesta exacerbada del sistema inmune innato, la cual puede terminar en una tormenta de citoquinas con potencial de llevar a la muerte (Fu et al., 2020; Yuki et al., 2020).
Aportes de la nanociencia y la nanotecnología para el seguimiento y control de Covid-19
El inicio de este milenio ha dejado en claro a la humanidad que las enfermedades emergentes son un agente disruptor del statu quo. Este asunto prioritario ha sido sistemáticamente dejado de lado por las instituciones gubernamentales solo para retornar con mayor fuerza. En 2002, inician los brotes de SARS en el sureste asiático; tras contener el brote epidémico, la inversión para la investigación del agente infeccioso se redujo sustancialmente sin que se llegara a desarrollar una vacuna. Posteriormente, llegaron las olas de influenza aviar (H5N1) y porcina (H1N1), para en 2012 dar paso al MERS en Medio Oriente, epidemia que fue vista como un problema desvinculado de occidente. En los inicios de la tercera década del siglo aparece el Covid-19, con una sorprendentemente alta tasa de transmisión, generando un síndrome respiratorio agudo y en ocasiones una exacerbada respuesta del sistema inmune innato. De cara a este panorama, pareciera que los virus están afinando sus mecanismos de propagación para lograr una propagación global.
Por su parte, la especie humana también ha alcanzado un alto grado de sofisticación, manipulando, tanto en el laboratorio como en ensayos in vitro e in vivo, nanopartículas más pequeñas que los propios virus. Estos nanomateriales proveen a la medicina de un enorme arsenal para: a) caracterizar y cuantificar virus; b) alcanzar estándares adecuados de sanitización; c) mejorar los tratamientos contra algunas enfermedades, y, d) lograr inmunizar de manera eficaz contra algunas enfermedades infecciosas.
Aun cuando la pandemia lleva menos de seis meses, una importante cantidad de trabajos se han realizado para abordar estos rubros desde la nanociencia y la nanotecnología. Estas investigaciones parten de estudios previos realizados para otros HCoV. Por ejemplo, Warnes et al. (2015) encontraron un 70% de inactivación del virus 229E en superficies recubiertas con nanopartículas de cobre y de aleaciones de cobre, lo cual se atribuyó a la generación de especies reactivas de oxígeno (ROS, por sus siglas en inglés) por parte de los iones Cu2+ en la superficie de las nanopartículas. En abril de 2020, la empresa Nanotech Surface llevó a cabo la sanitización en edificios y calles de Milán empleando una suspensión de TiO2 y nanopartículas de plata. Los promotores de esta solución aseguran que las superficies tratadas pueden inactivar al virus SARS-CoV-2 hasta por 2 años (Nanotech Surface, 2020). Las propiedades desinfectantes del TiO2 como fotocatalizador (Zan et al., 2007) y agente sanitizante en espacios no iluminados es ampliamente conocida (Mazurkova et al., 2010), y se basa tanto en la producción de ROS como en las interacciones iónicas del semiconductor con las proteínas estructurales de los virus. En el caso del desarrollo empleado por la compañía Nanotech Surface, no se ofrece mayor información sobre el funcionamiento del producto a largo plazo. Los autores especulan que se puede tratar de una pintura que contiene TiO2 y nanopartículas de plata de liberación prolongada, lo cual mantendría a la superficie en un continuo proceso de esterilización. Esto no necesariamente es garantía de la inactivación del SARS-CoV-2 en superficies por un periodo tan prolongado, por lo que estudios a largo plazo son necesarios.
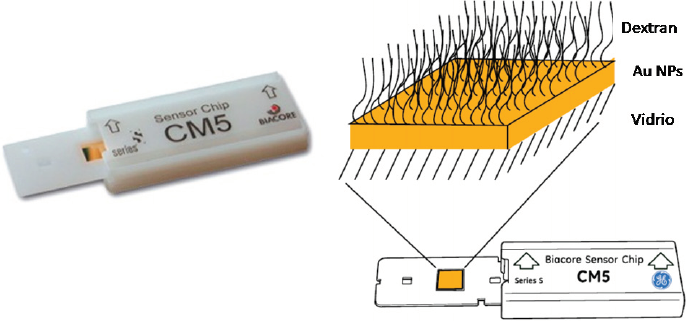
Las propiedades únicas que presentan las nanopartículas metálicas, como la resonancia del plasmón superficial (RPS), han sido aprovechadas para la identificación de biomoléculas y sus interacciones con otras macromoléculas. Existen en el mercado dispositivos espectrométricos para el seguimiento de las fluctuaciones de la señal plasmónica emitida por nanopartículas metálicas tras ser funcionalizadas con moléculas orgánicas e interactuar con biomoléculas en muestras acuosas. Ejemplos de estos dispositivos son el sensor «chip CM5» y el sistema «P4SPR», los cuales cuentan con recubrimientos de nanopartículas de oro. Estos dispositivos han sido empleados para la determinación de interacciones de la proteína S del SARS-CoV-2 con los receptores moleculares en las membranas de las células hospederas.
En las primeras semanas de la epidemia, se determinó la afinidad de la proteína S del SARS-CoV-2 al receptor ECA2, mostrando una cinética de unión de 10 a 20 veces más alta que lo observado en su momento para el virus SARS-CoV (Wrapp et al., 2020), explicando la alta virulencia del segundo. En un estudio más reciente, enfocado en el tratamiento de la infección, Mycroft-West et al., (2020) observaron una interacción química entre el DUR de la proteína S del SARS-CoV-2 con el anticoagulante heparina, usando un equipo P4SPR, lo cual puede dar sustento bioquímico a una alternativa de tratamiento de la infección empleando heparina de bajo peso molecular (Yin et al., 2020). Este protocolo de tratamiento ya se ha comenzado a implementar en hospitales de China, Europa y América, con el fin de disminuir el riesgo de coagulemia, al mismo tiempo que se saca provecho del efecto antiinflamatorio de la heparina. Es posible que el uso de este anticoagulante en etapas tempranas del virus disminuya la magnitud de la infección.
Mejora en las técnicas de diagnóstico para Covid-19.
Estado del conocimiento
La eficiencia en el diagnóstico ha sido sustancial para promover la prevención, el seguimiento y la contención de brotes epidémicos. Este paso es crucial para obtener información que permita tomar las medidas sanitarias necesarias para que el sistema de salud provea atención adecuada a los infectados sin llegar a colapsar. Así, es posible asegurar atención a los pacientes en diferentes etapas de la enfermedad, sobre todo cuando no existe tratamiento o vacuna, como es el caso de Covid-19.
El protocolo aprobado por la OMS para la detección del SARS-CoV-2 en muestras de mucosas de vías respiratorias humanas es conocido como «Protocolo Berlín», el cual se basa en la detección y cuantificación del virus mediante el método de la reacción en cadena de la polimerasa con transcriptasa inversa (RT-PCR, por sus siglas en inglés) (Corman et al., 2020). El procedimiento inicia con la extracción del material genético del virus, removiendo las proteínas estructurales y recuperando secuencias específicas de ARN. Posteriormente se promueve la transcripción reversa del ARN, para formar una molécula de ADN, seguido por la reacción en cadena de la proteína polimerasa que amplifica esta secuencia a lo largo de ciclos alternados de calentamiento: a 55 ºC para la transcripción inversa del ARN, y a 95 y 58 ºC en la replicación del ADN. Para lograr resultados cuantitativos se adiciona el agente primer FAM (6-carboxifluoresceina) unido a un oligonucleótido para producir fluorescencia durante la apertura de la doble cadena en la replicación del ADN. Para SARS-CoV-2, las secuencias objetivo usadas en esta prueba suelen ser la de la RNA polimerasa dependiente del RNA (RdRp), la proteína estructural E, y en ocasiones la proteína N o la secuencia ORF1ab. En un análisis cuantitativo de PT-PCR en tiempo real, la sensibilidad suele ser alta, con valores tan bajos como 3.7 copias para detectar la secuencia RdRp (Chan et al ., 2020). No obstante, estudios recientes de los mismos desarrolladores del Protocolo Berlín advierten de posibles errores en la detección y cuantificación del método al emplear la secuencia RdRp, abogando más por el uso de la secuencia de proteínas estructurales E y N (Corman y Drosten, 2020). Una alternativa al RT-PCR es la técnica conocida como RT-LAMP (reverse transcription loop-mediated isothermal amplification), en la cual las cadenas de ADN son amplificadas a una temperatura constante entre 60 a 65 ºC. Un notable incremento en la replicación de las cadenas de ADN se obtiene por la adición de loop-primers durante la replicación. Al igual que para RT-PCR, la cuantificación se realiza por espectrofotometría y otras técnicas ópticas.
Los métodos moleculares de detección del virus tienen desventajas, como la necesidad de personal calificado e instalaciones especiales para la realización de las pruebas, por lo que en la mayoría de los casos no se cuenta con unidades móviles en los centros de salud o en comunidades remotas, lo cual lleva a un importante retraso en la realización en serie de las pruebas y en el manejo de los resultados. Por esta razón, las pruebas de detección del SARS-CoV-2 se mantienen como confirmatorias, y se aplican solamente después de un diagnóstico de Covid-19 por una valoración clínica. Asimismo, cabe la posibilidad de obtener falsos negativos. En un estudio de 205 pacientes clínicamente diagnosticados para Covid-19, la prueba confirmatoria por RT-PCR fue mayor en muestras de lavado broncoalveolar (93%), seguido de muestras de esputo (72%), muestras tomadas con hisopo nasal (63%) y el más bajo fue para las muestras tomadas solamente con hisopo faríngeo (32%). Los resultados falsos negativos aparecen cuando las muestras son recolectadas al inicio de la enfermedad y la deficiencia en la técnica de muestreo, especialmente de hisopados nasofaríngeos (Wang et al., 2020).
Junto con los ensayos genéticos para la detección del SARS-CoV-2, existen pruebas serológicas que evalúan la aparición de los anticuerpos generados por las células linfocíticas en virtud de la exposición a los antígenos generados por las subunidades virales. En el caso del SARS-CoV-2 los anticuerpos son generados para los antígenos de las proteínas N y S (Long et al., 2020). Estos ensayos miden principalmente las inmunoglobulinas M y G, las cuales aparecen en diferentes estadios de la enfermedad y tras la recuperación del paciente, a través de ensayos inmunológicos (e.g. ELISA). Las pruebas serológicas son mucho menos precisas que las genéticas, especialmente porque las inmunoglobulinas comienzan a aparecer 7 días después del contagio; aunque este lapso puede variar en función de la severidad de la infección. En contraste, los métodos RT-PCR y RT-LAMP pueden medir el agente patógeno desde los primeros días de la infección y en muestras variadas, como mucosas, esputo o heces (Figura 4).

Fuente: Elaboración de los autores con información de: Nalla et al. (2020), Zheng et al. (2020), Wölfel et al. (2020), Wang et al. (2020), Guo et al. (2020), To et al. (2020) y Pan et al. (2020).
Figura 4 Cinética de la respuesta a SARS-CoV-2 mediante ensayos moleculares de tipo RT-PCR y serológicos para la determinación de inmunoglobulinas.
El uso de materiales nanométricos puede incrementar la eficiencia de las pruebas convencionales para la detección de SARS-CoV-2
Previo a los ensayos moleculares para la detección del virus, se requiere la extracción cuantitativa del ARN viral a partir de muestras de mucosas. El método convencional de extracción y purificación de ARN emplea una columna giratoria de sílice, iniciando con la lisis de las partículas virales, seguido por varios ciclos de centrifugación, lavado y elución de la columna. Una baja eficiencia de extracción conlleva a obtener señales pobres durante la amplificación y con ello a falsos negativos. Además, la presencia de impurezas de la muestra puede incrementar la concentración de inhibidores de la polimerasa, ocasionando lecturas poco confiables. Como alternativa, se presentan los métodos de extracción del material genético empleando nanopartículas magnéticas, lo que elimina los pasos de centrifugación en la extracción del material genético. Después del lisado, las moléculas de ARN se adsorben específicamente sobre las nanopartículas funcionalizadas con grupos polares. Entonces, en presencia de un campo magnético, los ácidos nucleicos se separan rápidamente de la mayoría de las impurezas, y se lleva a cabo un paso rápido de lavado utilizando una solución iónica para liberar los ácidos nucleicos purificados. Al respecto, Zhao et al. (2020) proponen un método de extracción de ARN de SARS-CoV-2 utilizando nanopartículas magnéticas de hierro (MNP) funcionalizadas con grupos carboxilo (PC), sobre las cuales realiza la lisis y extracción en un solo paso. Además, eliminando el paso de elución, al permitir que el complejo PC-MNP-ARN se introduzca directamente en el proceso RT-PCR. Esto reduce drásticamente el tiempo de análisis, de 3 horas a aproximadamente 30 minutos. Los complejos obtenidos son compatibles con otros métodos de amplificación isotérmica, como RPA y LAMP, por lo que podrían utilizarse en el desarrollo de dispositivos portátiles.
Ensayos colorimétricos
Los materiales nanométricos ayudan a incrementar la sensibilidad de las técnicas de diagnóstico existentes, al incrementar los efectos ópticos y electrónicos que proveen la señal usada para la cuantificación de material genético o de anticuerpos (inmunoglobulinas). Por ejemplo, a través del fenómeno de resonancia del plasmón de superficie (RPS), la señal de fluorescencia puede incrementar en los procesos cuantitativos de RT-PCR y RT-LAMP. Asimismo, las propiedades conductoras y semiconductoras de los nanomateriales proveen un campo para la fabricación de sensores eléctricos o colorimétricos. Esto es de gran importancia, pues estos sensores tienden a miniaturizar el proceso de análisis, requiriendo menos espacio y una menor cantidad de muestra para realizar el diagnóstico. Del mismo modo, no se precisa de personal altamente calificado para realizar las pruebas, por lo que estas se pueden llevar a cabo en domicilios, en consultorios médicos y en espacios designados por las autoridades sanitarias. Existe un nutrido cuerpo de literatura acerca de los principios y la incursión de la nanotecnología en las técnicas moleculares de identificación de diferentes virus (Draz y Shafiee, 2018; Lee et al., 2017; Mokhtarzadeh et al., 2017). Concretamente, en la detección y cuantificación de coronavirus, la historia inicia en 2004 con el desarrollo de métodos colorimétricos de diagnóstico. En estos métodos se empleaban suspensiones coloidales de nanopartículas de oro, en cuya superficie se depositaban secuencias de ARN de SARS-CoV. Más adelante, se adicionaba la cadena complementaria para obtener ADN, mismo que se desorbía, provocando la desestabilización de cargas y con ello la aglomeración de las nanopartículas de oro. El cambio de color, de rojo a morado equivalía a un diagnóstico positivo (Li y Rothberg, 2004).
Después de casi dos décadas, estas pruebas diagnóstico han ido ganando sofisticación hasta llegar a los dispositivos de flujo lateral (Figura 5), los cuales son una manera rápida de obtener un diagnóstico, ya sea midiendo anticuerpos o material genético viral. En estos dispositivos, de dimensiones milimétricas, se coloca una pequeña cantidad de muestra clínica (del orden de los microlitros), junto con una solución buffer de fosfatos enriquecida con albúmina de suero bovino. La mezcla comienza entonces a avanzar lateralmente hacia la zona de conjugación, en donde se encuentra un conjugado de antígeno de SARS-CoV-2 (generalmente de proteína S), funcionalizado con nanopartículas de oro. Este conjugado se elabora antes de cada ensayo, empleando tanto antígeno humano como antígeno de otro mamífero, como ratón o conejo. En caso de que la muestra sea positiva para Covid-19, los anticuerpos IgM y/o IgG interactuarán químicamente con los antígenos funcionalizados. La muestra llega entonces a la zona reactiva, la cual consiste de una membrana de nitrocelulosa que presenta tres ranuras paralelas, en las que se encuentran las contrapartes de los anticuerpos IgM e IgG, así como para la inmunoglobulina del mamífero. La unión anticuerpo-Au-antígeno-anticuerpo lleva a la expresión de la RPS, haciendo que las líneas en donde se presenten estas uniones químicas adquieran un color rojo a rosado.

Nota: En este ejemplo se ha colocado una muestra positiva para Covid-19, por lo que tiene a los anticuerpos IgM e IgG. En la parte de abajo se muestran las representaciones de los posibles resultados de la prueba.
Fuente: Elaboración de los autores, adaptado de Li et al. (2020).
Figura 5 Sistema de diagnóstico de flujo lateral.
Como se muestra en la Figura 5, la ranura control siempre debe mostrar color, caso contrario la prueba se considera defectuosa y por lo tanto inválida. De acuerdo con lo reportado por Li et al. (2020), esta prueba puede ser empleada para analizar muestras de sangre extraída por punción en el dedo pulgar. Con un tiempo de ensayo de aproximadamente 15 minutos, este ensayo ha mostrado un 88.67% de sensibilidad entre pacientes con Covid-19 confirmado e individuos sanos, así como un 91% de especificidad (Li et al., 2020). Por su parte, Huang, Chao et al. (2020) encontraron un valor de sensibilidad de 100% y una especificidad ligeramente inferior al 95%. El ensayo mostró una notable selectividad en la detección de IgM de SARS-CoV-2 sin interferencias por otros virus. Algunos autores están probando nanopartículas diferentes a las de oro para evaluar la presencia de las inmunoglobulinas que se presentan durante la enfermedad (IgG). En el estudio más reciente se emplean nanopartículas de poliestireno dopadas con lantano (Chen et al., 2020).
Zhu et al. (2020) llevaron la técnica colorimétrica más allá, al acoplarla a RT-LAMP. Los autores iniciaron con la extracción y amplificación de los plásmidos F1ab y de la proteína N del SARS-CoV-2 mediante la técnica LAMP. Posteriormente, los productos de la reacción en cadena de la polimerasa pasaron al módulo de flujo lateral para unirse a antígenos conjugados con nanopartículas poliméricas funcionalizadas con estretavidina. Con este ensayo clínico se logró un 100% de selectividad y especificidad para SARS-CoV-2, aunque el tiempo de la prueba incrementó hasta 1 hora, lo cual sigue siendo menor que el tiempo tomado por RT-PCR, de aproximadamente 3 horas (Zhu et al., 2020).
Biosensores
Los biosensores son también un método rápido que aprovecha las propiedades conductoras y semiconductoras de los nanomateriales para determinar la presencia y la concentración de material genético, proteínas o partículas virales en muestras complejas. Para el diagnóstico de SARS-CoV-2 se ha propuesto el uso de biosensores de transistor de efecto de campo (Bio-FET, por sus siglas en inglés), los cuales funcionan por el acoplamiento de un transistor con una biomolécula que sirve como receptor selectivo del analito, en una dinámica similar a la unión antígeno-anticuerpo. En estos arreglos, el transistor de efecto de campo es un material semiconductor, el cual se halla separado del receptor del analito por una capa de material aislante. Toda vez que el analito se une con el agente receptor, el potencial electrostático del semiconductor cambia, lo cual puede ser medido por dos electrodos que se hallan a cada lado de la celda de medición. Estos cambios en la conductancia se relacionan con la presencia y concentración del analito medido, ya sea el anticuerpo de la proteína S del SARS-CoV-2, una parte de la secuencia genética del virus, una partícula viral o una proteína del virus (Dutta et al., 2017). Un ejemplo de esta clase de dispositivos lo reportan Seo et al. (2020), quienes soportaron una película delgada de grafeno (10,000 µm2 y 1-1.5 nm de grosor) sobre un soporte de SiO2. Después, la superficie del semiconductor fue funcionalizada con 1-pirenobutanoato de succinimidilo (PBASE, por sus siglas en inglés), el cual sirvió para, luego, inmovilizar al antígeno de la proteína S del SARS-CoV-2 (Figura 6).
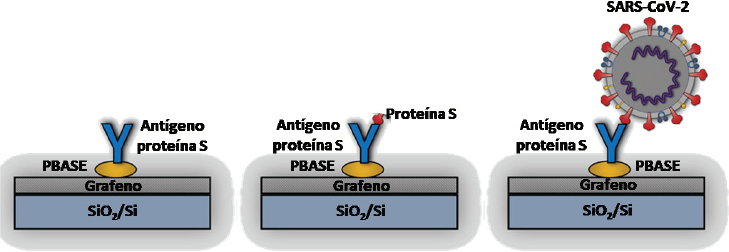
Fuente: Elaboración de los autores, adaptado de Seo et al. (2020).
Figura 6 Ejemplo de biosensor de transistor de efecto de campo (Bio-FET), usando grafeno para la detección de SARS-CoV-2. Izquierda: semiconductor funcionalizado. Centro: celda midiendo proteína S de SARS-CoV-2 en solución salina. Derecha: electrodo midiendo partículas virales de SARS-CoV-2 en muestras de mucosa.
Después, se midieron los cambios en la conductancia debido a la funcionalización, la adición del antígeno de la proteína S y, finalmente, la respuesta del sensor al ligarse el antígeno con la proteína S. El sensor mostró un límite de detección de 1 fg/ml para la proteína S, usando una solución buffer como matriz, mientras que, en muestras de exudado nasofaríngeo, este valor aumentó hasta 100 fg/ml. El sensor mostró alta selectividad al mezclar muestras de proteína S de SARS-CoV-2 y MERS-CoV. Por último, al medir partículas virales completas en muestras de pacientes con diagnóstico positivo a Covid-19 se encontró un límite de detección de 242 copias/ml (Seo et al., 2020).
Debido a sus propiedades conductoras, las nanopartículas metálicas de oro y el grafeno son ideales para la construcción de electrodos selectivos para componentes virales de SARS-CoV-2. Mahari et al. (2020) construyeron un electrodo, usando FTO para soportar nanopartículas esféricas de oro, las cuales fueron funcionalizadas con el antígeno de la proteína S1 de SARS-CoV-2. El electrodo mostró una respuesta de 10 a 30 segundos, con un límite de detección de 90 a 120 fM en muestras de buffer de fosfato y saliva, respectivamente. Hasta ahora, las pruebas de detección de antígenos y anticuerpos en saliva son escasos para SARS-CoV-2, la mayoría de estos estudios son limitados debido al bajo número de participantes, los cuales transitan por diferentes etapas de la enfermedad, y en virtud de ello la carga viral es variable (Azzi et al., 2020; To et al., 2020). Debido a ello, es aún imposible hacer una comparación de estos resultados con lo que se puede encontrar en una muestra de saliva en un paciente infectado con Covid-19.
Biosensores plasmónicos fototérmicos
Qiu et al. (2020) han desarrollado un biosensor con nanopartículas de oro, el cual aprovecha las propiedades de RPS y lo complementa con el efecto fototérmico que ocurre en las nanopartículas de metales nobles en virtud de la expresión de la resonancia localizada del plasmón de superficie (RLPS). El arreglo experimental propuesto se muestra en la Figura 7a, el cual cuenta con una fuente de luz blanca para inducir el efecto de la resonancia localizada del plasmón de superficie en las nanopartículas de oro soportadas en un chip. Algo parecido a lo mostrado previamente en la Figura 3 para el chip CM5. Posteriormente, las nanopartículas de oro fueron funcionalizadas con un tiol y una secuencia específica del material genético del SARS-CoV-2 (proteína E y proteína funcional ORF1ab, Figura 7b), produciendo un incremento en la intensidad de la banda plasmónica. Una solución buffer con la secuencia complementaria del ARN funcionalizado fue adicionada en la superficie del chip. Al complementarse las cadenas de oligonucleótidos, la intensidad de la RLPS incrementó ligeramente. Cuando la secuencia de ARN adicionada no es complementaria a la cadena funcionalizada se produce una rápida separación, y por ello la señal plasmónica se mantiene sin cambios.

Fuente: Elaboración de los autores.
Figura 7 a) Dispositivo del biosensor empleado para medir RLPS y el efecto termoplasmónico en un chip recubierto con nanopartículas de oro funcionalizadas con un tiol y una secuencia de ARN; b) nanopartícula esférica de oro funcionalizada con un tiol y una secuencia de ARN que codifica para una proteína de SARS-CoV-2.
El equipo de sensado cuenta con un láser de diodo (532 nm y 40 mW), colocado en la parte superior del chip, con una inclinación de 72º (Figura 7a). Este láser fue empleado para generar la respuesta termoplasmónica en las nanopartículas de oro. El incremento de temperatura llegó hasta los 41 ºC, aumentando la velocidad de ensamble de las cadenas de oligonucleótidos; del mismo modo, aceleró su repulsión en caso de no ser secuencias complementarias. Mediante el fenómeno de incremento de la señal plasmónica, tras la complementación de la cadena de ARN soportada en la nanopartícula de oro, fue posible identificar la presencia del SARS-CoV-2 en 82 pacientes previamente diagnosticados con Covid-19. El límite de detección del método fue establecido en 2.26 × 104 copias de secuencias de RNA, mientras que un paciente en las primeras etapas de la enfermedad llega a presentar hasta 1 × 106 copias/ml. Por último, el método de diagnóstico mostró ser poco proclive a reaccionar con secuencias espurias (falsos positivos). Este método resultó ser tan sensible, selectivo y eficiente como los ensayos RT-PCR y ELISA (Qiu et al., 2020).
Desarrollo de una vacuna para Covid-19.
Estado del conocimiento
Hasta mediados del mes de mayo de 2020 han sido presentados ante la OMS 118 candidatos para lograr una vacuna eficaz que logre inmunizar a humanos contra el SARS-CoV-2. Estos esfuerzos de investigación son realizados, tanto por universidades como por compañías farmacéuticas en los cinco continentes, sobre las bases de los conocimientos adquiridos a partir de los brotes de SARS y MERS. De estas propuestas, 110 se encuentran en ensayos preclínicos, en tanto que ocho se hallan en una etapa de mayor avance, como en fase de evaluación clínica a nivel ½, 1 o 2. Las plataformas propuestas para la vacuna varían, poniendo sobre la mesa los siguientes enfoques: a) exposición directa a partículas virales inactivas o atenuadas; b) administración de un vector viral no replicante; c) exposición de células linfocíticas a subunidades virales (principalmente a la proteína S), y, d) administración de secuencias de material genético (ADN o ARN) que codifica para antígenos que serán «presentados» a las células del sistema inmune para la generación de anticuerpos contra el virus (WHO, 2020c).
Las vacunas basadas en la administración de partículas virales inactivas o atenuadas son las más eficientes para generar inmunidad tanto extracelular (humoral) como intracelular, siendo las más comúnmente empleadas para inmunizar contra diferentes tipos de influenza. Dado que el genoma del SARS-CoV-2 ha sido secuenciado desde enero de 2020 (No. MN908947.3 en GenBank) (Khailany et al., 2020), es posible llevar a cabo cultivos en masa del virus, para posteriormente inactivarlo mediante irradiación con luz UV o reacción con formaldehido (Jiang et al., 2005). No obstante, en algunos casos el patógeno atenuado puede reactivarse en el cuerpo humano, trayendo consigo algunos de los síntomas de la enfermedad, por lo que los individuos con algún tipo de inmunosupresión podrían ser vacunados usando solamente cargas de virus inactivo. En el caso de Covid-19, cuya fisiopatología sigue en proceso de ser descifrada, padecer la enfermedad por una inmunización inadecuada es un riesgo que no se debería correr. Por otra parte, la preparación de esta clase de vacunas precisa de laboratorios con condiciones de bioseguridad tipo 3, por lo que su producción en masa, aunque posible, llega a ser costosa y centralizada (Saif, 2020).
Otras aproximaciones optan por el desarrollo de inmunidad a través de la exposición de las células del sistema inmune a las proteínas estructurales del virus, logrando la producción de anticuerpos por los linfocitos T CD4+/CD8+ (Shang et al., 2020). Estimular la generación de anticuerpos por exposición a la proteína S del virus ha mostrado ser la estrategia de inmunidad adecuada contra otros HCoV en ensayos clínicos. Por ejemplo, Bukreyev y colaboradores reportaron inmunización eficaz en monos de la especie Chlorocebus sabaeus tras el suministro de la proteína S del virus SARS-CoV (Bukreyev et al., 2004). No obstante, las vacunas basadas en subunidades de virus generan solamente inmunidad humoral, la cual puede no ser suficiente para el agente SARS-CoV-2.
Por último, las vacunas basadas en material genético, como el ARN mensajero (ARNm) conjuntan las ventajas de las vacunas mencionadas, sin los riesgos asociados de inmunización inadecuada o el riesgo inherente de trabajar con el agente infeccioso, aun en laboratorios de alto nivel de bioseguridad.
En las últimas décadas, las vacunas de ARNm se han erigido como una opción para complementar (y en algunos casos reemplazar) a las vacunas tradicionales. La síntesis de ARNm es sencilla, rápida, de bajo costo, por lo que es posible alcanzar niveles de producción que satisfagan las demandas globales (Saif, 2020). Para lograr una inmunización eficaz, el ARNm debe mantenerse intacto mientras viaja a través del espacio intersticial y alcanza a las células objetivo para lograr la transfección. Una vez en el citosol, el material genético codifica para generar antígenos, moduladores y señaladores celulares, los cuales se transfieren hacia la membrana celular o permanecen en el citosol, desarrollando tanto inmunidad intracelular como humoral (Reichmuth et al., 2016).
La vacuna para SARS-CoV-2 puede estar formulada con nanopartículas
El principal reto de las vacunas de ARNm es la facilidad con la que el material genético puede hidrolizarse dentro y fuera de la célula, por lo que es necesario encapsularlo en una bio-carcasa resistente a las condiciones fisiológicas, sea capaz de penetrar la doble capa lipídica de las células y desensamblarse en el citoplasma; algo similar al proceso de infección viral. Una estrategia ampliamente aceptada es el desarrollo de nanopartículas de lípidos, las cuales son capaces de unirse con el ARNm, condensando al material genético en un volumen de unos cuantos nm3. Estas nanopartículas transportan al ARNm a través de las mucosas, la sangre y el espacio intersticial, dirigiéndose hacia las células objetivo con ayuda de una biomolécula (normalmente una proteína) que funciona como antena en la superficie de la nanopartícula. Esta molécula ayuda a la localización del dominio de unión con el receptor en la membrana celular para posteriormente ingresar por endocitosis; algo similar al proceso de entrada de un virus a la célula. Una vez que el endosoma se encuentra en el espacio citoplasmático, la nanopartícula lipídica sufre un cambio estructural que favorece su desensamble para liberar al material genético (Figura 8). Las principales células objetivo de estas nanopartículas son las células presentadoras de antígenos, como las células endoteliales, los linfocitos B, los macrófagos o las células dendríticas, siendo estas últimas las más relevantes (Reichmuth et al., 2016). Dentro del citoplasma, el ARNm es expresado, y el antígeno generado es procesado por el proteosoma, generando determinantes antigénicos (epítopos), los cuales son cargados en los complejos de histocompatibilidad y llevados a la cara exterior de la doble membrana lipídica de la célula. Este proceso convierte a la célula transductada en una célula presentadora de antígeno, la cual interacciona con un linfocito T para generar anticuerpos específicos para el antígeno (Saif, 2020).

Fuente: Elaboración de los autores, adaptado de Reichmuth et al. (2016).
Figura 8 Las nanopartículas lipídicas protegen al ARNm y coadyuvan a su inserción en las células objetivo en forma de endosomas, los cuales son desensamblados en el citosol, dejando libre al ARNm para codificar antígenos endógenos. A) Los antígenos exógenos pueden también endocitar, formando endosomas de los cuales no necesita emerger. B) Nótese cómo el ARNm que no está asociado con nanopartículas lipídicas no es capaz de penetrar la membrana celular.
Las nanopartículas de lípidos son micelas que cuentan con un centro acuoso rodeado por una bicapa lipídica que funciona como coraza (Li y Szoka, 2007). La mayor parte de las formulaciones hasta ahora reportadas emplean lípidos catiónicos, pues presentan afinidad con el RNAm de carga negativa; no obstante, en algunos casos se emplean lípidos neutros o negativamente cargados. En algunos casos se emplean lipidoides con grupos amino para mantener una carga eléctrica variable. Dentro del citoplasma, los endosomas son estructuralmente modificados debido al cambio de pH entre el líquido intersticial y el citosol, lo cual lleva a la desestabilización de las bicapas lipídicas, llevando a la liberación del ARNm (Reichmuth et al., 2016). Además de los lípidos ionizables, estas nanopartículas se componen de fosfolípidos, colesterol y polietilenglicol. Los primeros tienen una función estructural, ayudando con la formación de la bicapa lipídica y la eventual disociación del endosoma en el citoplasma. Asimismo, el fosfato contenido en los fosfolípidos, junto con las aminas, ayudan a mantener la carga de la nanopartícula lipídica. El colesterol tiene una función de estabilización estructural y juega un papel crucial en la transfección de las células. El colesterol ayuda a la transición de la fase laminar de la nanopartícula hacia la fase hexagonal, lo cual es clave para la liberación del ARNm en el citoplasma. El polietilenglicol modificado con lípidos se deposita en la superficie de la nanopartícula como una barrera que aporta estabilidad a la nanopartícula lipídica y evita que se una a proteínas, tanto en el espacio intersticial como el citoplasma (Li y Szoka, 2007). Las cargas de colesterol y polietilenglicol deben ser optimizadas, pues ello impacta en la estabilidad de la nanopartícula lipídica, en su capacidad de transfección y en el potencial de liberación del ARNm. Por ejemplo, al incrementar la carga de colesterol se facilita la transición de fase laminar a hexagonal en la nanopartícula, con el respectivo impacto en la liberación del ARN mensajero. Por otra parte, incrementar la carga de polietilenglicol lleva a un mayor tiempo de circulación de la nanopartícula en sangre, lo cual reduce la capacidad de la misma para ser asimilada por las células objetivo (Reichmuth et al., 2016).
Las nanopartículas lipídicas son sintetizadas mediante el proceso de formación de micelas, al mezclar lentamente una solución acuosa con etanol. El ARNm está presente en la fase acuosa, mientras que los lípidos se hallan disueltos en la fase alcohólica. Conforme se agrega el agua a la solución de etanol se forman vesículas circulares, las cuales van creciendo por el proceso de fusión micelar, hasta formar partículas estables en donde se encuentra encapsulado el ARNm. Los factores que más impactan en el tamaño y estructura de la nanopartícula del lípido son: i) la velocidad de mezclado de las soluciones acuosa y alcohólica; ii) el volumen de cada una de las soluciones, y, iii) la composición de lípidos en la fase alcohólica (Reichmuth et al., 2016). Las técnicas más avanzadas para generar estas nanopartículas echan mano de microfluidica para desarrollar reactores de rejilla que favorecen las condiciones hidrodinámicas para obtener un mezclado rápido que genera nanopartículas uniformes con un tamaño promedio de 50 a 70 nm, y una baja distribución de tamaño (Stroocky McGraw, 2004).
Desde la década pasada, el grupo de investigación de Dong, en la Universidad de Ohio, ha desarrollado y optimizado un método de síntesis de nanopartículas de lípidos, las cuales han llamado TT3. Estas nanopartículas han funcionado éxitosamente como acarreadores de ARNm en ensayos in vivo (Li et al., 2015; Luo et al., 2017). Las nanopartículas esféricas de N1, N3, N5-tris(2-aminoetil)benceno-1,3,5-tricarboxiamida (TT3) están compuestas por un anillo bencénico central sustituido por tres cadenas aminolipídicas (ver Figura 9).
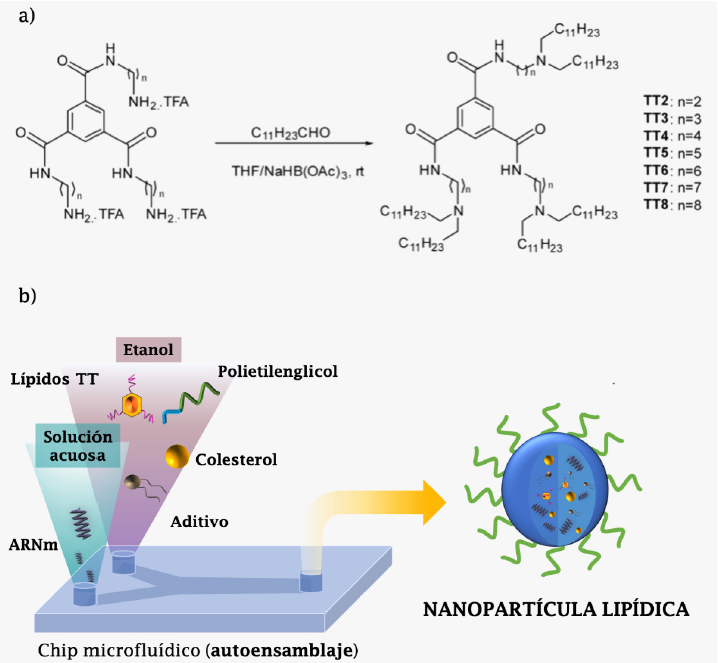
Fuente: Elaboración de los autores, modificado de Li et al. (2015).
Figura 9 Proceso de síntesis de las nanopartículas lipídicas TT3.
La formulación de las nanopartículas TT3, compuestas por 2-dioleoil-sn-glicero-3-fosfoetanolamina (DOPE), colesterol y 1,2-dimiristoil-sn-glycerol, metoxipolietilenglicol (DMG-PEG2000), en una proporción molar TT3/DOPE/ colesterol/DMG-PEG2000 = 20/30/40/0.75, se encuentran en fase clínica para la aplicación y entrega de ARNm que codifica para la generación de anticuerpos para proteínas S y ORF1ab del SARS-CoV-2. En estas pruebas se emplean células linfocíticas de ratón, mostrando resultados prometedores (Zeng et al., 2020), con una entrega del ARNm 70 veces mayor a lo obtenido para otras nanopartículas lipídicas. Más aún, el ARNm encapsulado en las nanopartículas TT3 puede ser preservado hasta por tres meses en congelación con nitrógeno líquido, adicionando 5% (p/v) de sucrosa o trehalosa (Zhao et al., 2020).
A la fecha de publicación de este artículo, existen dos prospectos de vacuna basados en ARNm encapsulado en nanopartículas lipídicas, los cuales se hallan en fase de ensayos clínicos. La aproximación de la compañía Moderna ha concluido la fase 1 (NCT04283461) y se mueve éxitosamente hacia la fase 2. Por su parte, el desarrollo a cargo del consorcio BioNTech/Fosun/Pharma/Pfizer se encuentra en fase clínica ½ (2020-001038-36 NCT04368728) (WHO, 2020c). En enero de 2020, Moderna recibió el código genético del virus SARS-CoV-2, a partir del cual desarrolló el ARNm-1273, una pieza sintética de material genético que codifica para la proteína S. La síntesis del ARNm y la formulación de las nanopartículas lipídicas se realizó en tan solo 130 días (Cohen, 2020). La vacuna se suministra mediante una inyección intramuscular en el brazo, y las nanopartículas lipídicas son químicamente dirigidas hacia los ganglios linfáticos más próximos, mediante la funcionalización de la nanopartícula con señaladores químicos. Las nanopartículas, con un tamaño menor a los 100 nm pasan a través del espacio intersticial hasta alcanzar las células dendríticas, en donde son asimiladas por endocitosis (Tu et al., 2020). En abril de 2020, se llevó a cabo el primer ensayo de la vacuna en una población humana reducida (52 individuos). Si el ensayo es exitoso, para el otoño de 2020 se estarían haciendo pruebas con 5,000 voluntarios. La vacuna puede tomar de 12 a 18 meses para estar lista, pues se tienen que optimizar la dosis, la vía de administración y ponderar los efectos secundarios. En los primeros ensayos se ha visto que, con dosis de 1 microgramo se alcanza la tan ansiada inmunidad, en ensayos in vivo, por lo que 1 gramo puede inmunizar hasta a 1 millón de personas (Cohen, 2020). Otras propuestas de vacunas basadas en el uso de nanopartículas son realizados por instituciones públicas y privadas en todo el mundo, como Pharma, BIOCAD, Novavax, Curevac las Universidades de Fundan y Shangai en China y de Tokio; no obstante, estas se encuentran en una etapa menos avanzada de desarrollo (Amanat y Krammer, 2020). Es probable que tras la pandemia de Covid-19, la humanidad inicie una etapa de vacunas basadas en nanopartículas, dando un paso adelante en el desarrollo de la nanomedicina. Lo único seguro es que nadie sabe cuál o cuáles vacunas funcionarán eficazmente.
Perspectivas
Ante el reto que plantea la pandemia de Covid-19, consideramos que los desarrollos en nanociencia y nanotecnología deben enfocarse en cuatro ejes principales.
Mejora en los métodos de diagnóstico. La nanotecnología puede ayudar a mejorar las técnicas moleculares empleadas en la detección y cuantificación del virus, o de los anticuerpos en suero. Las propiedades conductoras y ópticas de las nanopartículas metálicas proveerán menores límites de detección, alta selectividad, y en general una mayor confiabilidad en el diagnóstico. Las nanopartículas de oro y otros conductores pueden ser empleados para la fabricación de biosensores. Por otro lado, el efecto de RSP mostrado por las nanopartículas metálicas puede ser aprovechado para incrementar la señal de fluorescencia en los ensayos RT-PCR o utilizarse para evaluar las interacciones de los componentes del virus con diversas biomoléculas, lo cual puede ser útil incluso para probar la efectividad de un medicamento para neutralizar al virus. La nanotecnología puede también miniaturizar los dispositivos de prueba para llevarlos a las zonas urbanas marginales y las áreas rurales. Sitios a donde el virus inevitablemente llegará en el futuro próximo.
Desarrollo de equipo de protección personal. Los actuales equipos de protección personal, como caretas, mascarillas N95, trajes de aislamiento y guantes, son una barrera adecuada para proteger al personal de salud que trata con los enfermos más graves, así como para las familias que comparten espacio con infectados. Las nanopartículas inorgánicas de metales de transición y óxidos metálicos han mostrado una notable capacidad para la inactivación de agentes patógenos, incluyendo virus (Galdiero et al., 2011; Joe et al., 2016). La disponibilidad de mascarillas con capacidad autolimpiadora es atractiva desde la perspectiva económica y práctica. En una exploración rápida en el motor de búsqueda Google Patents se encontraron alrededor de 7,780 patentes relacionadas con materiales nanoparticulados para la limpieza de equipo de protección personal, 25% de las cuales abordan la funcionalización de mascarillas con nanopartículas de plata para la inactivación de patógenos que atacan las vías respiratorias.
Tratamiento. Hasta el momento de redacción de este trabajo, diferentes medicamentos han sido probados para el tratamiento de Covid-19. Sustancias como mesilato de camostat, azitromicina, hidroxicloroquina, lopinavir ivermectina, remdesivir, tocilizumav y sarilumab han sido propuestas para bloquear la fusión del virus con las células hospederas, la replicación viral y la infección en general. En este sentido, las nanopartículas inorgánicas funcionalizadas pueden entregar a los medicamentos de manera eficiente, al ser dirigidos a las células afectadas por el SARS-CoV-2, principalmente los neumocitos tipo II.
Desarrollo de vacunas. Hasta el 15 de mayo de 2020, más de un centenar de alternativas de vacuna se están desarrollando en todo el mundo, ocho de ellas en la etapa de ensayos clínicos (WHO, 2020c). Las nanopartículas de biomoléculas, como proteínas y lípidos, pueden sustituir a las vacunas que usan al virus inactivado o atenuado, lo cual sería una garantía de bioseguridad.
Conclusiones
Indudablemente, la nanociencia y la nanotecnología tienen un papel importante en la caracterización del SARS-CoV-2, las interacciones que tiene con las células del cuerpo y la fisiopatología de la enfermedad que produce. Aunque ha pasado poco tiempo desde el inicio del brote, mucho se ha avanzado en el desarrollo de técnicas analíticas robustas, confiables y portátiles. En ese sentido, la nanociencia y su persistente enfoque en miniaturizar los sensores es una ventaja para el desarrollo de sistemas de diagnóstico portátil, que puedan ser comercializados y que cada individuo pueda hacer el test en casa. Asimismo, uno de los campos en los que se lleva un mayor avance es en el desarrollo de vacunas. Esta podría ser la primera vez que una vacuna esté formulada con nanopartículas, lo cual representaría uno de los mayores hitos en la historia de la nanociencia y la nanotecnología. No obstante, aún existen áreas del conocimiento inexploradas para SARS-CoV-2, por ejemplo, el desarrollo de equipo de protección personal eficiente para salvaguardar la salud de los profesionales de la salud que están en la primera línea de cuidado de los enfermos más graves. Paralelamente, se debe explorar la nanocatálisis como una alternativa para remover al virus del aire en espacios cerrados, como aviones, oficinas, subterráneos o asilos de ancianos.
La pandemia por Covid-19 ha puesto a la humanidad en una encrucijada, en la que se tienen que ponderar los pros y contras de una sociedad hiperglobalizada. Al mismo tiempo, los dirigentes, los tomadores de decisiones e incluso los medios de comunicación tienen que voltear la mirada hacia la comunidad científica, pues son los únicos que pueden dar una solución al problema. En menos de cinco meses de emergencia sanitaria, la comunidad científica ha descifrado el genoma del virus, rastreado su origen, propuesto una serie de medidas para el tratamiento de Covid-19 y avanzado a pasos agigantados para la elaboración no de una sino de 118 propuestas de vacuna. Como científicos, debemos apropiarnos de estos logros y no permitir que se nos invisibilice una vez controlado el problema y hasta que llegue la siguiente pandemia o los embates más crudos del cambio climático. Este es el momento de mostrar a la sociedad, a los altos funcionarios y a los medios de comunicación el papel tan importante que tienen la ciencia y la tecnología en la vida de prácticamente todos los seres humanos que habitan el planeta, e incluso de los que están en órbita.











 nueva página del texto (beta)
nueva página del texto (beta)