Introducción
Una de las áreas que más retos impone a la nanotecnología es la de las ciencias biomédicas. En un organismo, cada fenómeno ocurre en la escala del nanómetro, es decir, en el orden de 10-9 m, que equivale a la millonésima parte de un milímetro. Aunque estas dimensiones invisibles a nuestros ojos pudieran parecernos fuera de nuestro contexto habitual, es un hecho que cada una de nuestras células está compuesta de estructuras nanométricas, y que es precisamente en esta escala que ocurren las interacciones en cada una de ellas (Jain, 2008). Resulta así que no solamente el control de nanoestructuras es imprescindible para incidir en un fenómeno celular, sino que las células constituyen una fuente de inspiración para las nanociencias, y han dado lugar a materiales bioinspirados, es decir, materiales inspirados en estructuras biológicas, así como a materiales biomiméticos, los cuales imitan estructuras y funciones biológicas. Las nanopartículas para fines farmacéuticos pueden ser administradas a través de una variedad de rutas, incluyendo la vía oral, la intravenosa, la intramuscular y la intradérmica. Uno de los principales retos para la entrega de fármacos es el direccionamiento en el blanco adecuado, esto es, en el órgano, tejido y sitio de acción específicos. En un organismo, el direccionamiento de células y sustancias es un hecho cotidiano. Hormonas, vesículas extracelulares y otras sustancias, tales como mediadores de inflamación, viajan por el torrente sanguíneo a velocidades que, en el cuerpo humano, van de 0.1 - 30 cm/seg (McCall, 2010) donde, a pesar de su largo trayecto, son capaces de dirigirse a sitios precisos. La manera en que tal especificidad ocurre es a través de interacciones moleculares de muy alta afinidad, por ejemplo, de una enzima con su ligando, o de una hormona con su receptor. No sin sorpresa, la explotación de interacciones biológicas ha constituido una de las principales estrategias de direccionamiento para un nanomedicamento. Pero durante su trayecto al sitio diana, las nanopartículas deben enfrentarse con uno de los principales sistemas de un organismo: su sistema de defensa o sistema inmune. Dentro de un animal, cualquier agente extraño, tal como una bacteria o una nanopartícula serán reconocidas como una sustancia extraña, y bastarán unos cuantos segundos para que tales agentes sean destruidos a través del sistema inmune (Sompayrac, 2012; Puntes y Saldaña, 2015). Más aún, las células encargadas de reconocer lo extraño podrían salirse de control, activarse de manera exacerbada y conducir a una respuesta inflamatoria capaz de acarrear efectos letales para el individuo (Peri et al., 2012). Debido a ello, con el diseño de nanopartículas se busca con frecuencia el camuflaje, es decir, el recubrimiento de partículas de tal manera que su paso por el organismo pueda permanecer inadvertido por parte del sistema inmune hasta llegar al blanco. A la fecha, se han desarrollado distintos recubrimientos para el camuflaje de nanopartículas (López Marín et al., 2013). La manera más comúnmente utilizada para camuflajear nanopartículas es el uso de polímeros altamente hidrofílicos, como el polietilenglicol, los cuales generan un impedimento estérico entre nanopartículas y células del sistema inmune. En contraste, otra de las acciones a buscar en un nanomedicamento corresponde a la acción de activar la respuesta inmune de manera deliberada. Por ejemplo, la activación de la respuesta inmune resulta sumamente útil para fines de vacunación (Karch, Burkhard, 2016), o bien en el marco de una inmunoterapia contra el cáncer (Kim et al., 2017). Por último, en cualquiera de ambos casos, los nanomedicamentos deben estar exentos de toxicidad, tanto a nivel celular como organísmico. Sin embargo, algunas veces una misma sustancia es capaz de tener tanto efectos positivos y negativos en un organismo. En este artículo hablaremos de una clase de ligandos con carácter dual en nanomedicina, los patrones moleculares asociados a patógenos (PAMPs, por sus siglas en inglés). Los describiremos a fin de dar cuenta de su carácter tóxico, pero también por su potencial como potentes activadores de la respuesta inmune, y del uso emergente que esto último conlleva para la producción de nanovacunas.
¿Cómo es la respuesta de defensa en contra de organismos patógenos?
Sistemas de defensa en contra de agentes dañinos han sido necesarios en todos los organismos vivientes. Las bacterias, por ejemplo, están dotadas de enzimas que son capaces de destruir el DNA de virus invasores, inactivándolos ya sea de manera generalizada (es decir, con rupturas sobre casi cualquier DNA foráneo) (Kelly y Smith, 1970), o bien mediante un sistema de “memoria molecular”, el cual reconoce específicamente el DNA de virus que han tenido contacto previo con la bacteria para luego fragmentarlo (Barrangou et al., 2007).
En organismos vertebrados, incluyendo el humano, ocurren también mecanismos de defensa de dos tipos: uno de ellos actúa de manera inmediata, específicamente a través de actores moleculares que se encuentran dispuestos de manera innata, y un segundo tipo de respuesta corresponde a eventos en donde el individuo es capaz de prepararse para infecciones contra un agente en particular. A este último tipo de respuesta se le llama respuesta inmune adquirida y, a pesar de ser sumamente compleja, fue la primera de estas dos respuestas en ser puesta en evidencia. Corría la última década del siglo XIX cuando Emil von Behring y Shibasaburo Kitasato descubrieron que los animales previamente expuestos a una bacteria patógena producían en su sangre sustancias capaces de neutralizar las toxinas producidas por ese mismo microbio (Kaufmann, 2017). A dichas sustancias neutralizantes se les denominó anticuerpos, y pronto quedó demostrado que los anticuerpos eran sustancias capaces de neutralizar el mismo agente patógeno si se transferían de un individuo a otro, lo que fue llamado seroterapia (Kaufmann, 2017). Actualmente se sabe que los anticuerpos (así como otras respuestas específicas hacia patógenos) se producen únicamente al contacto del individuo ante las sustancias capaces de desencadenar esa respuesta inmune (o antígenos). La respuesta inmune de esta categoría, esto es, la respuesta inmune adquirida, ha dado pie a una gran variedad de desarrollos tecnológicos. Un ejemplo es el uso de anticuerpos que, inmovilizados sobre nanopartículas, son utilizados para direccionar éstas hacia un sitio u órgano en particular (Pietersz et al., 2017). Hasta fines del siglo pasado, la inmunidad adquirida había dominado prácticamente todos los estudios y aplicaciones en inmunología. Sin embargo, ahora sabemos que la respuesta inmune adquirida no apareció evolutivamente sino cientos de millones de años después de que organismos multicelulares habían comenzado a poblar el planeta, defendiéndose ya de agentes infecciosos (Flajnik y Du Pasquier, 2004; Cooper y Herrin, 2010; Buchmann, 2014). La estrategia general de esa respuesta, en donde no se requiere de una preparación previa, consiste en el despliegue de receptores (de manera innata) dedicados a detectar moléculas distribuidas en muchos microrganismos a la vez. Curiosamente, aunque el hecho de conferir protección ante múltiples infecciones representa una gran ventaja, hasta ahora poco se ha explotado este tipo de respuesta en nanomedicina. Y es que el sistema inmune innato es un actor que no fue puesto en evidencia sino hasta fines del siglo XX (Medzhitov et al., 1997).
El sistema inmune innato: un combate inmediato ante el peligro
Como muchos otros descubrimientos, las bases de lo que hoy conocemos como sistema inmune innato salieron a la luz por serendipia. A finales del siglo pasado se investigaba el rol de un gen llamado toll sobre el desarrollo embrionario de uno de los modelos animales más utilizados en biología, la mosca de la fruta o Drosophila melanogaster. Se observó entonces que al producirse mutaciones del gen toll, la ausencia de la proteína correspondiente provocaba en el insecto una susceptibilidad muy exacerbada ante infecciones por hongos. Es decir que, en Drosophila, la proteína Toll correspondía a una molécula involucrada en el sistema de defensa de la mosca. En efecto, pronto se sabría que Toll es una sustancia capaz de inducir inflamación, y que promueve una respuesta eficaz en contra de infecciones (Belvin y Anderson, 1996). Además, poco después se encontró que Toll cuenta con proteínas homólogas en el humano que funcionan de manera similar: localizadas en la superficie de células del sistema inmune, las proteínas tipo Toll (o TLRs, del inglés Tol-like receptors) monitorean señales de peligro y, al quedar ambas partes acopladas, se desencadena una cascada de eventos intracelulares que terminan por activar genes e inducir la producción de proteínas pro-inflamatorias, es decir, activan procesos destinados a destruir una sustancia extraña (Medzhitov et al., 1997). Los receptores de la respuesta inmune innata no se restringen a los TLR. Se ha identificado toda una variedad de receptores que, en conjunto, son denominados receptores de reconocimiento de patrones (PRRs, del inglés pattern recognition receptors). Los hay anclados a biomembranas y secretados a circulación. Entre los principales PRRs se conocen los TLRs, los receptores tipo Nod (NLRs, por sus siglas en inglés), receptores que unen manosas (MBL, DC-SIGN, MR) y los receptores lectina tipo-C (CLRs, del inglés C-type lectin receptors) (Akira et al., 2006, Demento et al., 2011, Silva et al., 2017). Como muchas otras interacciones biológicas, los receptores de inmunidad innata se unen a sus ligandos mediante interacciones no covalentes. En la Figura 2 se muestra la estructura esquematizada de una proteína tipo Toll acoplada a su ligando, un lipoglicano de bacterias.
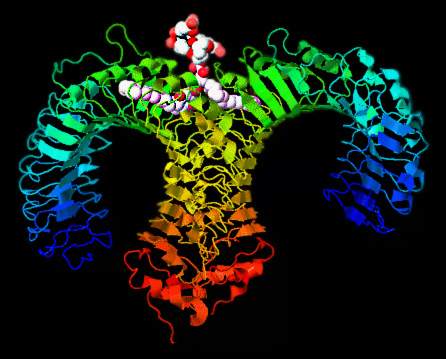
En esta imagen se esquematiza la estructura 3D de un PRR, la proteína TLR-4 (representada en colores) acoplada a un lipopolisacárido de bacterias Gram negativas (un PAMP), con átomos simbolizados por esferas blancas y rojas. Los TLRs son proteínas que atraviesan la membrana y, luego de la interacción con su ligando en la superficie celular, transmiten una señal al interior de la célula a través de cambios estructurales en su dominio intracelular (aquí no mostrado).
Fuente: Imagen obtenida del portal educativo de uso libre PDB-101 (Berman et al., 2000).
Figura 1 La interacción entre un receptor de reconocimiento de patrones (PRR) en una célula inmune y un patrón molecular asociado a patógenos (PAMP) ocurre por autoensamblaje molecular.
Estructura, distribución y toxicidad de los PAMPs
¿Y si esta mezcla no funciona en absoluto?…
¿Qué pasaría si se tratara de un veneno?
Romeo y Julieta, W. Sheakespeare
Los patrones moleculares asociados a patógenos (PAMPs) no pertenecen a un solo tipo de estructuras moleculares. Los hay que corresponden a lípidos glicosilados, a lipofosfoglicanos, a oligonucleótidos (CpG DNAs, dsRNAs), lipoglicanos, glicanos, proteínas y péptidos (Akira et al., 2006, Demento et al., 2011, Silva et al., 2017). Por otro lado, la dilucidación de PAMPs constituye un área en desarrollo continuo, en la que frecuentemente se reportan nuevas moléculas capaces de ser reconocidas por receptores de la respuesta inmune innata. Ejemplos de ello son los hallazgos de parches de membranas microbianas que actúan como PAMPs, pero de los que aún se desconoce su identidad (Basto et al., 2012), o la descripción de que varias proteínas del virus de inmunodeficiencia humana son reconocidas como PAMPs (Lee, 2013).
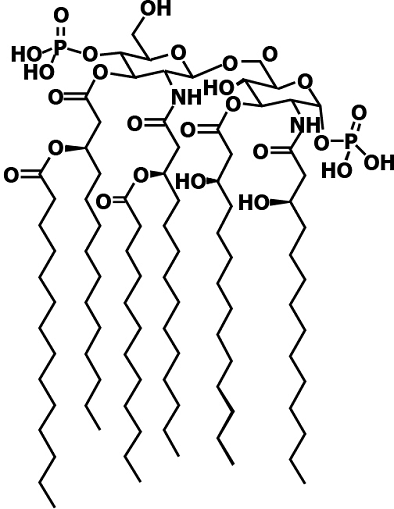
Los primeros PAMPs en ser identificados fueron polisacáridos con motivos lipídicos de bacterias gram negativas. En la Figura 2 se muestra la estructura de uno de estos PAMPs, también conocidos como lipopolisacáridos (LPS). Químicamente, dicha estructura es mucho más estable que una proteína o un nucleótido. Resiste al calor moderado y a la desecación, básicamente debido a que su conformación no depende de condiciones específicas en un medio acuoso, como es el caso de moléculas con grupos protonables. La distribución de LPS en el mundo microbiano es muy abundante. Además, los microrganismos Gram negativos se encuentran en prácticamente cualquier lado. Por estas razones, durante la preparación de una formulación farmacéutica es fácil que los LPS aparezcan como productos contaminantes (Bennett y Beeson, 1950), lo cual ha sido también reportado durante la preparación de nanomedicinas (Vallhov et al., 2006). El problema resulta crítico si el producto farmacéutico se destina a una formulación administrada por vía intravenosa, pues la activación del sistema inmune innato en el endotelio vascular podría salirse de control, y llevar al individuo a un choque anafiláctico. Bajo este panorama, los PAMPs, y, en particular, los LPS han constituido durante décadas uno de los elementos más temidos en la industria farmacéutica. Por otro lado, sin embargo, la capacidad de estas sustancias para activar el sistema inmune innato constituye un punto muy deseable para el desarrollo de productos tales como sistemas de vacunación o inmunoterapias contra el cáncer.
Una de las razones fundamentales para que el sistema inmune innato haya cobrado la gran relevancia que actualmente tiene fue el hallazgo de su función como el único puente que conduce al correcto funcionamiento de la inmunidad de tipo adquirido (Medzhitov y Janeway, 1997). Este concepto ha hecho cambiar por completo las estrategias para el diseño de vacunas, y ha implicado el traslado de los PAMPs desde el banquillo de los acusados, a un importante sitio donde compiten por lugares estelares para desarrollos inmunoterapéuticos. En el siguiente apartado resumiremos cómo el uso de PAMPs en el área de vacunas constituye un área emergente en nanomedicina.
PAMPs y la activación de la respuesta inmune para nanovacunas
La activación del sistema inmune innato representa un elemento esencial para lograr una respuesta inmune integral, por lo cual buscar condiciones para cumplir dicha condición resulta el paso lógico para el desarrollo de vacunas. Gracias a la nanotecnología, es posible diseñar partículas multifuncionales que permitan modular la respuesta inmune tal como se desea. Se dispone de un sinnúmero de materiales capaces de encapsular y proteger antígenos, los cuales pueden además ser dirigidos a las células de particular interés, tales como macrófagos, células dendríticas o linfocitos B. Por otro lado, las nanovacunas pueden ser diseñadas de tal forma que la presentación de antígenos ocurra en un contexto biomimético. En este sentido, una de las estrategias más prometedoras actualmente es el despliegue de PAMPs en la superficie de la nanovacuna. Cada vez más desarrolladores de vacunas están utilizando PAMPs para asegurar la activación de la respuesta inmune innata a través de sus formulaciones. En la Figura 3 se esquematiza el principio de la activación del sistema inmune innato a través del uso de PAMPs en el diseño de una nanovacuna.
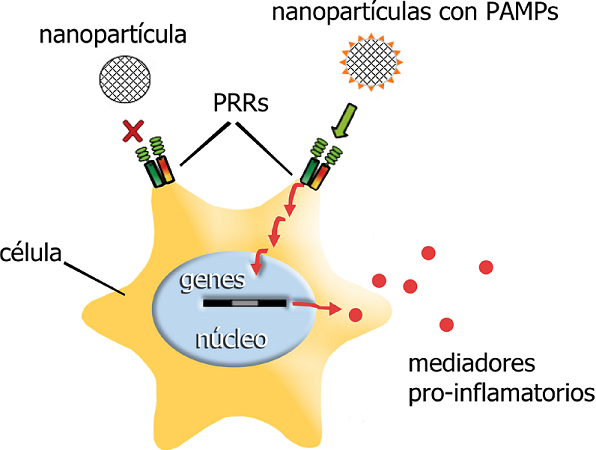
El despliegue de patrones moleculares asociados a patógenos (PAMPs) en una nanopartícula promueve la interacción de ésta con los llamados receptores de reconocimiento de patrones (PRRs) en una célula. Este reconocimiento produce una cascada de señales intracelulares (flechas rojas) que terminan por translocarse al núcleo, en donde en cuestión de minutos se induce la expresión de genes que codifican mediadores que activan el sistema inmune.
Fuente: Elaboración de los autores.
Figura 3 Interacciones entre una célula presentadora de antígenos y nanopartículas que despliegan PAMPs.
Se sabe que en varias de las vacunas desarrolladas hasta ahora, los PAMPs forman parte de la formulación, y a ellos se atribuye, al menos en parte, una buena eficacia de vacunación. Entre estas preparaciones se encuentran vacunas en contra de bacterias Gram negativas y de amibas (Massari et al., 2006, Lee et al., 2010, Cervantes-Barragan et al., 2009, Wong-Baeza et al., 2010). En lo que se refiere a productos obtenidos mediante nanotecnología, el uso de PAMPs fue inicialmente explorado por Demento y colaboradores, quienes propusieron utilizar LPS como adyuvante para una vacuna de subunidad en contra del virus del Nilo Occidental (Demento et al., 2009). A partir de este trabajo, toda una serie de propuestas de nanovacunas decoradas con PAMPs han visto la luz en la última década. Algunos autores han optado por la utilización de vesículas extracelulares de bacterias (las cuales contienen PAMPs ) (Unal et al., 2011, Basto et al., 2012, Tirado et al., 2015) o por LPS desplegados en la superficie de liposomas (Wiley et al., 2012). Otros, en cambio, han desplegado PAMPs en nanopartículas poliméricas, de sílice, de silicio poroso o de oro coloidal (Demento et al., 2011). Con respecto a los PAMPs utilizados para decorar nanoestructuras, los reportes incluyen polinucleótidos, como el poli(I:C) que es un agonista del TLR3 (Ultimo et al., 2015), DNA plasmídico y oligonucleótidos que unen el TLR9, LPS con variaciones estructurales finas, ß-glucano, los lípidos mpla, factor cuerda, y el lipopéptido sintético Pam3cag (Demento et al., 2011). De manera interesante, se sabe que algunos receptores de PAMPs tienen un patrón de reconocimiento bastante promiscuo, dando lugar a una respuesta inmunológica de amplio espectro, que resultaría útil para combatir distintos patógenos. En nuestro laboratorio exploramos el uso de PAMPs obtenidos de micobacterias, organismos que han sido empleados como inmunoestimulantes, por ejemplo para inmunoterapia durante tratamientos de cáncer de vejiga (Talat Iqbal y Hussain, 2014). En conclusión, son múltiples las posibilidades que ofrece el desarrollo de nanoestructuras híbridas, capaces de explotar mecanismos de la respuesta inmune adquirida, pero también de la respuesta innata. Más investigaciones en el área serán necesarias para que nanovacunas capaces de inducir una respuesta inmune robusta, pero controlada, sean una realidad.











 nova página do texto(beta)
nova página do texto(beta)