Las bacterias expresan sistemas de secreción indispensables para transportar proteínas, como, por ejemplo, toxinas o enzimas, y otras macromoléculas hacia el espacio extracelular (González-Pedrajo y Dreyfus, 2003). Existen siete sistemas de secreción descritos que se pueden encontrar en bacterias Gram-positivas y bacterias Gram-negativas (Figura 1). La diferencia que puede ser más notable entre estos complejos multiproteicos es que las bacterias Gram-positivas necesitan realizar un simple proceso de secreción a través de una sola membrana mientras que las bacterias Gram-negativas transportan la molécula biológica a través de la doble membrana que las rodea hacia el medio extracelular, mediante un mecanismo más complejo. Sin embargo, los genes que codifican para proteínas que participan en el proceso de secreción de bacterias Gram-positivas muestran un alto grado de similitud con los genes identificados originalmente en Escherichia coli (Ibidem). Debido a esto, el criterio que se utiliza para diferenciar los sistemas de secreción radica en el tipo de transporte realizado por la bacteria, independientemente de si se trata de una bacteria Gram-positiva o Gram-negativa.
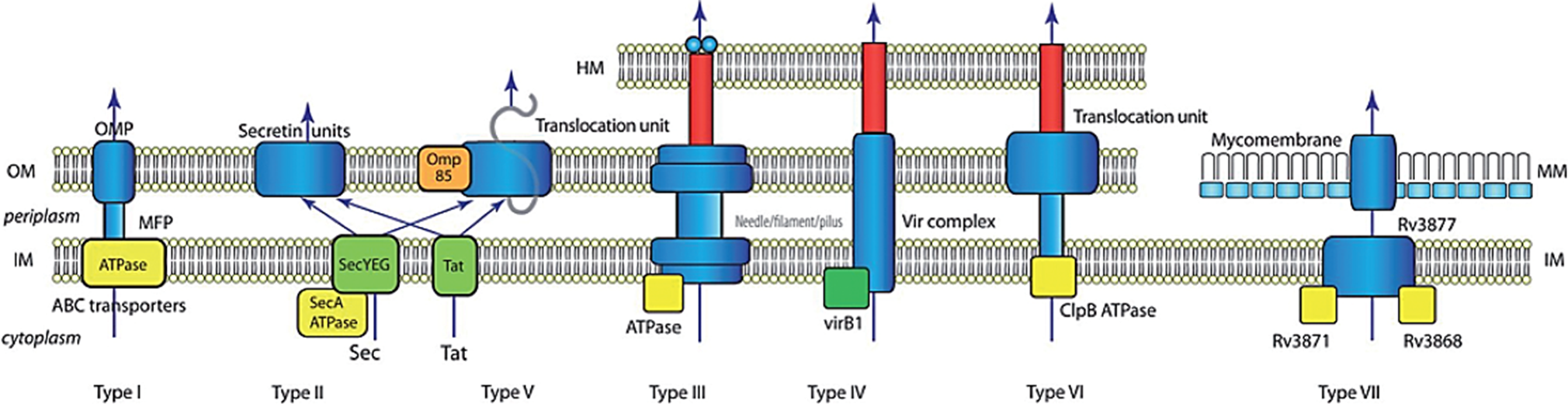
Fuente: Tomada de: Tsai-Tien, et al.; BMC Microbiology, 2009.
Figura 1 Sistemas de secreción bacterianos. Esquema simplificado de los 7 sistemas de secreción descritos. HM: membrana de la célula hospedadora; OM: membrana externa; IM: membrana interna; MM: micomembrana; OMP: Proteína de membrana externa; MPF: Proteína de fusión de membrana. Las ATPasas y chaperonas se muestran en amarillo.
El tipo de transporte se puede realizar mediante dos vías, la vía Sec-dependiente y la vía Sec-independiente. En la vía Sec-dependiente la secreción de sustratos depende de la secuencia señal ubicada en el extremo amino terminal que posee la proteína a secretar, pues por medio de esta secuencia, la proteína es reconocida y transportada por el sistema de secreción Sec. La vía Sec-independiente se caracteriza por la translocación de la proteína sin la presencia de la secuencia señal en el amino terminal o de un intermediario periplasmático (Koster et al., 2000). La vía Sec-independiente engloba los sistemas de secreción tipo I, III y IV, los cuales forman complejos multiproteicos que evitan la presencia del efector en el periplasma por lo que los sustratos son secretados desde el citoplasma hasta el medio extracelular (Kanonenberg et al., 2013; González-Pedrajo y Dreyfus, 2003). El sistema de secreción tipo I (SST1) es capaz de transportar polipéptidos de más de 800 kDa que ostentan una señal de secreción incluida en los últimos 53 residuos del extremo carboxilo terminal, la cual les permite ser reconocidas por el translocador (compuesto de un transportador ABC, una proteína de fusión a membrana MFP y una proteína trimérica de membrana externa), y ser translocadas mediante el aporte de energía de la hidrólisis de ATP mediada por el ATP-binding cassette (conocido como transportador ABC) (Holland et al., 2005) (Figura 1). El SST1 exporta péptidos y factores de virulencia, tales como metaloproteasas, glicanasas (Delepelaire, 2004) y proteínas relacionadas a la formación de biofilms (Russo et al., 2006). El sistema de secreción tipo II (SST2) sólo se encuentra en proteobacterias, en especies simbiontes obligadas, bacterias patógenas y especies de vida libre, sin necesariamente pertenecer a un grupo particular (Cianciotto, 2005). El SST2 es necesario como agente virulento de Vibrio cholerae, Legionella pneumonphila, E. coli enterotoxigénica, Ralstonia solanacearum, Pectobacterium atrosepticum, y Xanthomonas campestris (Filloux, 2004). El sistema de secreción tipo III (SST3) se encuentra en bacterias Gram-negativas que interactúan con plantas y animales como agentes patógenos o mutualistas (Grant et al., 2006). La función principal de este sistema de secreción es transportar proteínas efectoras del citosol bacteriano a la célula hospedera que, una vez translocadas, intervienen en la alteración del metabolismo celular (Mota y Cornelis, 2005). El sistema de secreción tipo IV (SST4) es el único sistema de secreción que, además de transportar proteínas, es capaz de secretar ácidos nucléicos en plantas, animales, levaduras y otras bacterias (Christie y Cascales, 2005). El SST4 está presente en varios organismos patógenos como Agrobacterium tumefaciens, Helicobacter pylori, Pseudomonas aeruginosa, Bordetella pertussis, Legionella pneumophila y Mesorhizobium loti (Ibidem). El sistema de secreción tipo V (SST5) secreta proteínas de 40 a 400 kDa de tamaño que poseen un péptido señal en el dominio amino terminal conservado. Las proteínas son autotransportadas al periplasma bacteriano. Posteriormente, la translocación hacia el espacio extracelular de la proteína es mediada por la inserción del dominio barril beta de su extremo C-terminal conservado en la membrana externa, con lo cual se forma un poro a través del cual se permite su translocación (Henderson et al., 2004; Jacob-Dubuisson et al., 2004). El SST5 permite la secreción de adhesinas en E. coli, Haemophilus influenzae, Yersinia enterocolitica, Bordetella pertussis; toxinas en Helicobacter pylori; proteasas en Neisseria meningitidis, Shigella flexneri y Serratia marcescens (Tseng et al., 2009). El sistema de secreción tipo VI (SST6) al igual que el SST3, se asemeja a un inyectisoma y es capaz de translocar proteínas efectoras directamente al citosol de la célula hospedera (Bingle et al., 2008). El modelo propuesto del inyectisoma del SST6 incluye una proteína chaperona con actividad ATPasa, un canal que comunica la membrana interna con la membrana externa y una aguja que tiene una proteína que forma un poro (Shrivastava y Mande, 2008). Requerido para la virulencia, el SST6 es utilizado por Vibrio cholerae, Edwardsiella tarda, Pseudomonas aeruginosa, Francisella tularensis, y Burkholderia mallei para infectar humanos y plantas por igual, así como sólo en plantas por Agrobacterium tumefaciens, Pectobacterium atrosepticum y Xanthomonas oryzae (Bingle et al., 2008). El sistema de secreción tipo VII (SST7) se encuentra codificado en cinco grupos de genes de las bacterias Gram-positivas del genero Mycobacterium, y, además, se encuentra estrechamente relacionado con los patógenos Corynebacterium diphtheriae y Nocardia (Abdallah et al., 2007) (Figura 1).
Los sistemas de secreción tipo III
Las bacterias Gram-negativas son ampliamente estudiadas para conocer con mayor especificidad los procesos celulares que modifican en organismos eucariotas, principalmente las bacterias que provocan enfermedades en animales y plantas de interés económico, y, sobre todo, de bacterias que alteran procesos celulares en el humano mediante los mecanismos de secreción. Los sistemas de secreción tipo tres (SST3) son “nanojeringas” o “inyectisomas” de origen proteico y escala nanométrica que se ensamblan en las membranas celulares de las bacterias que las producen, y que permiten la entrega de proteínas de virulencia bacteriana a las células eucariotas que infectan (Figura 2). El SST3 está compuesto por más de 20 proteínas diferentes. El inyectisoma se ha identificado en especies de Bordetella, Chlamydia, Erwinia, E. coli, Pseudomonas, Ralstonia, Rhizobia, Salmonella, Shigella, Xanthomonas y Yersinia, (González-Pedrajo y Dreyfus 2003), y originalmente identificado en Salmonella typhimurium (Kubori et al., 1998), es un complejo que se encuentra parcialmente conservado en Salmonella enterica, Shigella flexneri y E. coli enteropatógena, por lo que el SST3 no difiere en su estructura en los géneros antes mencionados. Los componentes estructurales del SST3 y el cuerpo basal del flagelo bacteriano muestran bajo microscopía electrónica gran similitud, esto podría significar que existe una relación evolutiva entre ambos sistemas y un mecanismo común de reconocimiento y de exportación de sustratos (González-Pedrajo y Dreyfus 2003). Sin embargo, las proteínas efectoras que alteran el metabolismo celular hospedero son únicas para cada sistema (Galán y Wolf-Watz, 2006).
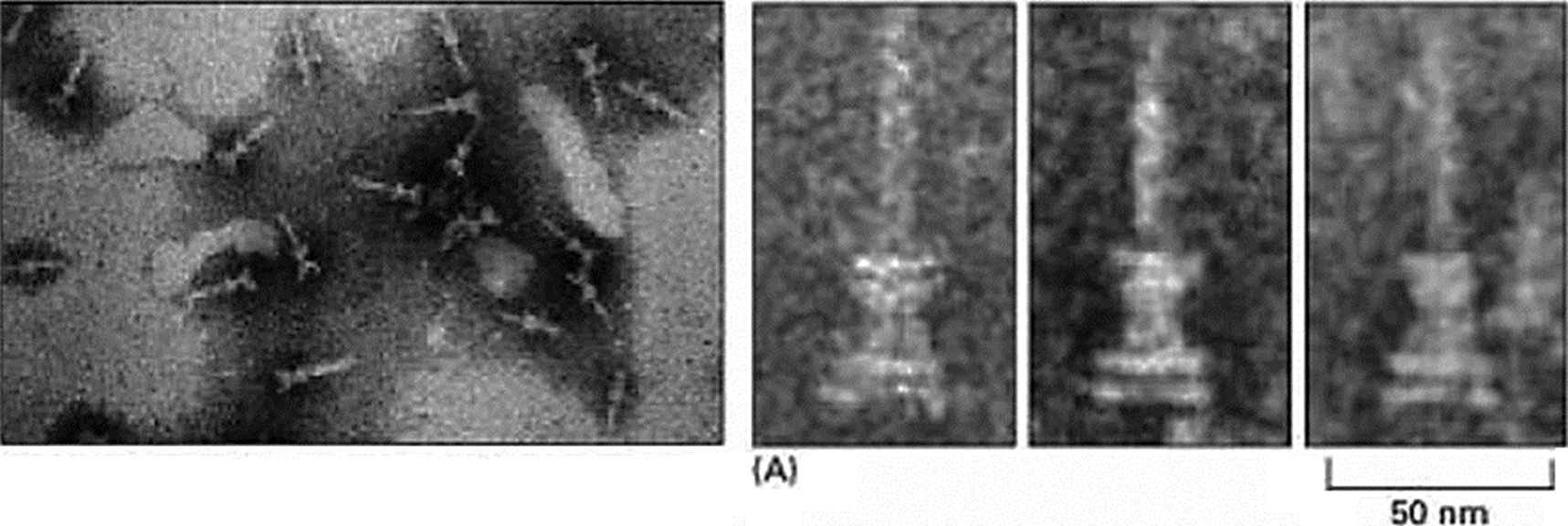
Fuente: Tomada de Tamano et al., 2000.
Figura 2 Micrografía electrónica de transmisión (TEM) de inyectisomas bacterianos. Nano-jeringas moleculares cuya base está formada de anillos proteicos que se ensamblan sobre las membranas interna y externa de la bacteria. La aguja se prolonga de dicha base hacia el exterior para hacer contacto con la célula hospedera.
La estructura o cuerpo basal de este sistema de secreción se extiende desde la membrana interna (MI) a la membrana externa (ME) y tiene un diámetro aproximado de 50 nm (Tamano et al., 2000) (Figura 2). La constitución básica de la estructura del sistema se compone por dos anillos transmembranales anclados a las membranas interna (MI) y externa (ME), respectivamente, comunicados a través de proteínas que forman un tubo que se extiende desde el primer anillo multiproteico (MI) a lo largo del periplasma microbiano hasta el segundo anillo ubicado en la ME de la bacteria. El cuerpo basal de los inyectisomas se conecta con una aguja hueca en Yersinia; con un filamento, como en Salmonella; o bien, mediante un pilus, en P. syringe. Cada una de estas estructuras mide en promedio 60 nm de longitud y se conecta con proteínas que forman un poro de translocación en la membrana plasmática de las células del hospedero (Cornelis, 2006). El complejo multiproteico del SST3 alcanza en su conjunto una longitud aproximada de 60-80 nm (Blocker et al., 1999).
La familia de patógenos A/E
Escherichia coli enteropatógena (EPEC), enterohemorrágica (EHEC) y Citrobacter rodentium (un patógeno de ratón) provocan en el epitelio intestinal una lesión histopatológica A/E (attaching and effacing) o de adherencia y destrucción que se caracteriza por la pérdida de microvellosidades intestinales y la formación de una estructura en forma de copa o pedestal, donde la bacteria se une íntimamente a la célula hospedera (Figura 3). La isla de patogenicidad LEE (Locus of Enterocyte Effacement) (Rocha-Gracia et al., 2005), además de contener los genes que codifican para el SST3, cuenta con genes que codifican proteínas que son secretadas y translocadas mediante este sistema y proteínas que regulan su propia expresión (Garmendia et al., 2005; Lara-Ochoa et al., 2010). El SST3 transloca proteínas efectoras que modifican diferentes estructuras y procesos celulares como la interrupción de la función de la barrera intestinal, el incremento en la permeabilidad de uniones estrechas acompañadas de la disminución de la resistencia transepitelial, pérdida del potencial de membrana mitocondrial, la inhibición de la transición del ciclo celular G2/M y la inducción de apoptosis (Medrano-López, et al., 2012).
Modificación del SST3 de EPEC para su uso con fines terapéuticos
La eficiente translocación de proteínas a través de los SST3 ha motivado su estudio y utilización con fines médicos y biotecnológicos. Los SST3 de diferentes bacterias (Salmonella typhimurium, Pseudomonas aeruginosa, Pseudomonas syringe) han sido utilizados con éxito como nanoinyectores para la translocación de antígenos hacia células epiteliales, tales como núcleoproteínas de la cápside del virus de la influenza (Russmann et al., 1998), de ovoalbúmina (Epaulard et al., 2006 y 2008), con la finalidad de estimular la respuesta inmune y generar protección. También se ha logrado la translocación de TNFα, anticuerpos y nanoanticuerpos para la inactivación de proteínas blanco en el interior de la célula eucariote o para inmunoterapia antitumoral con resultados alentadores (Nishikawa et al., 2006; Yoon et al., 2011; Blanco-Toribio et al., 2010). Éstos y otros ejemplos de la utilización de estas nanomáquinas biológicas, si bien han tenido éxito, presentan ciertas limitaciones y retos: a) Se han utilizado bacterias atenuadas, es decir, bacterias patógenas con ciertas modificaciones genéticas que reducen su patogenicidad, lo cual tiene un riesgo intrínseco de reversión (que dichas bacterias patógenas recuperen su patogenicidad y causen daño al huésped); b) las proteínas efectoras, sustrato de estos SST3, son entregadas a la célula blanco, provocando las modificaciones en las vías de señalización y las modificaciones estructurales del citoesqueleto, ampliamente documentadas; c) la generación de respuesta inmune contra estas bacterias, lo que limita su utilidad a largo plazo, y, d) algunas bacterias utilizadas, como S. typhimurium, tienen la capacidad de internalizarse en el organismo, lo que complica su control.
Tomando ventaja de la posibilidad de expresar de manera funcional al SST3 de EPEC en una bacteria Escherichia coli comensal, podemos evitar el riesgo de la reversión presente en los casos en que se utilizan bacterias patógenas atenuadas; además, es posible evitar la translocación de proteínas efectoras de dichas bacterias patógenas. Por otro lado, mediante la eliminación de los genes que codifican para proteínas efectoras dentro de la isla de patogenicidad LEE, se puede prevenir la secreción y translocación de dichas proteínas. Aunado a lo anterior, mediante la construcción de proteínas quiméricas, a las que se les adiciona un péptido señal para su reconocimiento y secreción a través del SST3, es posible translocar proteínas específicas con fines terapéuticos. Por ejemplo, para terapias anti-cancerígenas, algunas variedades de las citocromo oxidasas (CYP´s), que se ha demostrado que se expresan en E. coli y que participan en la biotransformación de fármacos anticancerígenos (Rodríguez-Antona e Ingelman-Sundberg, 2006). Si logramos enviar directamente a células de cáncer de colon citocromo oxidasas producidas en bacterias comensales, podremos eliminar dichas células. Otra posibilidad, es la expresión y el envío de asparaginasas, cuyo efecto anticancerígeno ha sido demostrado sobre células de leucemia linfocítica aguda infantil (Moreno-Enríquez et al., 2012; Huerta-Saquero et al., 2013; Labrou et al., 2010). El uso controlado de los SST3 bacterianos es un campo muy prometedor para la entrega dirigida de medicamentos o biomoléculas con fines terapéuticos.











 nueva página del texto (beta)
nueva página del texto (beta)



