Introducción
La resistencia a antimicrobianos se refiere a los procesos que presentan los microorganismos (bacterias, virus, hongos y parásitos) que inciden y hacen ineficaces a los medicamentos utilizados en su tratamiento1.
La escasez, y en algunos casos ausencia, de medicamentos eficaces para el tratamiento de enfermedades infecciosas tiene consecuencias graves. Los datos de la Organización Mundial de la Salud (OMS) indican que la resistencia bacteriana ocasionó 700,000 muertes en 2015; esta cifra, según el estudio realizado por O'Neill2, podría ascender a 10 millones en 2050 si no se llevan a cabo intervenciones para revertir la tendencia. Según proyecciones recientes de la OMS, en los siguientes 30 años las muertes por bacterias resistentes podrían ser más frecuentes que las atribuidas al cáncer3,4. En terminología económica, la resistencia es una externalidad negativa, es decir, tiene un efecto indeseable en otras personas además de la inmediata en el consumidor del antibiótico5.
Un creciente número de infecciones, como neumonía, tuberculosis, septicemia y gonorrea, son cada vez más difíciles y a veces imposibles de tratar, a medida que los antimicrobianos van perdiendo eficacia1,6. Nunca la amenaza de resistencia a los antimicrobianos ha sido más inmediata y la necesidad de soluciones más urgente, dice el Dr. Tedros Adhanom Ghebreyesus, Director General de la OMS.
Si bien los informes de agentes antibacterianos en desarrollo son innovadores, pasarán años antes de que lleguen a los pacientes y los nuevos tratamientos por sí solos no serán suficientes para combatir la amenaza de la resistencia a los antimicrobianos; por tal motivo, la OMS trabaja con diferentes países para mejorar la prevención y el control de las infecciones, y para fomentar el uso apropiado de los antibióticos existentes y futuros7,8.
La resistencia a los antimicrobianos puede ser natural (intrínseca) o adquirida. La resistencia natural es resultado de propiedades específicas del microorganismo y su aparición es previa al uso de los antibióticos. La resistencia adquirida se define como la pérdida de la sensibilidad a las concentraciones terapéuticas utilizadas de un medicamento particular. En sentido estricto, se considera multirresistente (MDR, multidrug-resistant) al microorganismo que presenta resistencia adquirida in vitro a más de un fármaco antibacteriano, pero esta MDR puede estratificarse en niveles. Las definiciones internacionales de MDR, resistencia extendida (XDR, extensively drug-resistant) y panresistencia (PDR, pandrug-resistant) fueron propuestas en 20129. Se considera MDR per se al microorganismo no sensible al menos a un antimicrobiano de tres o más familias farmacológicas. La XDR la presenta el microorganismo no sensible a un fármaco de todas las categorías, menos dos o una categorías. La PDR se refiere al microorganismo resistente a todos los fármacos de todas las categorías. El uso adecuado de estas definiciones permite una mejor comprensión de la extensión del problema de la resistencia y hace posible la comparación de los datos de vigilancia epidemiológica entre instituciones, regiones y países9. Para lograr diseñar y llevar a cabo intervenciones dirigidas cuyos resultados puedan ser medidos, es imprescindible que la información acerca de la resistencia a los antimicrobianos se organice en un conocimiento específico de las características y la magnitud del problema. El presente estudio tiene como objetivo conocer el perfil de resistencia bacteriana en un hospital privado de tercer nivel.
Método
Se realizó un estudio observacional, transversal y retrospectivo durante el periodo comprendido de febrero de 2016 a abril de 2019 en un hospital privado de tercer nivel de atención, ubicado en la ciudad de Chihuahua, Chihuahua, México. Se calculó el tamaño de la muestra de acuerdo con el número de muestras clínicas con cultivo existentes durante el mencionado periodo. Se aislaron 166 bacterias (Tabla 1). La identificación de los microorganismos se efectúo a partir de diferentes cultivos y su sensibilidad a los antibióticos se determinó con el sistema automatizado BD Phoenix cumpliendo con las políticas internas del laboratorio de la institución y los estándares del Clinical and Laboratory Standards Institute (CLSI)10. Los microrganismos aislados se clasificaron en MDR, XDR o PDR de acuerdo con las definiciones del Centro Europeo para la Prevención y Control de Enfermedades (ECDC) y los Centers for Disease Control and Prevention (CDC) de los Estados Unidos de Norteamérica9-11. Se extrajo información adicional de las historias clínicas, como el servicio de ingreso. Las variables de interés se registraron en una base de datos.
Tabla 1 Tipo de muestras clínicas, número de aislados bacterianos y nivel de resistencia
| Muestra clínica | N.º de muestras | N.º de aislados | Nivel de resistencia | |||
|---|---|---|---|---|---|---|
| MDR | XDR | PDR | No MDR | |||
| Orina | 23 | 25 | 16 | 0 | 0 | 9 |
| Exudado de herida/escara | 21 | 24 | 17 | 1 | 0 | 6 |
| Hemocultivo/catéter | 48 | 48 | 31 | 4 | 1 | 12 |
| Expectoración/secreción bronquial | 48 | 53 | 40 | 2 | 1 | 10 |
| Otras | 16 | 16 | 10 | 0 | 0 | 6 |
| Total | 156 | 166 | 114 | 7 | 2 | 43 |
MDR: multirresistencia; PDR: panresistencia; XDR: resistencia extendida.
Se contó con la aprobación del Comité de Investigación de la Facultad de Medicina y Ciencias Biomédicas (CI-057-19), y no se utilizaron datos personales de pacientes. Las pruebas estadísticas empleadas fueron de tipo descriptivo. Se obtuvieron valores porcentuales por especie de bacterias y área hospitalaria.
Resultados
Se cultivaron 156 muestras clínicas de diversos fluidos biológicos: orina (n = 23), exudado de heridas o escaras (n = 21), sangre y punta de catéter (n = 48), expectoración y secreción bronquial (n = 48), y otros (p. ej., líquido cefalorraquídeo, peritoneal y pulmonar; n = 16). A partir de estas muestras se caracterizaron 166 cepas bacterianas debido a que en 10 de las muestras clínicas analizadas se aislaron dos bacterias (Tabla 1).
El 58% (96/166) de las bacterias fueron gramnegativas, encontrándose entre las más frecuentes Escherichia coli (29/166), Pseudomonas aeruginosa (18/166) y Klebsiella pneumoniae (14/166); entre las bacterias grampositivas (70/166) más comunes se encontraron: Staphylococcus epidermidis (22/166), Enterococcus faecalis (14/166) y Staphylococcus aureus (10/166) (Tabla 2).
Tabla 2 Clasificación de las bacterias aisladas según la tinción de Gram y especies más frecuentes
| Clasificación | n (%) | Principales representantes (en orden descendente) |
|---|---|---|
| Gramnegativas | 96 (58) | E. coli, P. aeruginosa, K. pneumoniae |
| Grampositivas | 70 (42) | S. epidermidis, E. faecalis, S. aureus |
| Total | 166 (100) |
Se empleó el perfil de sensibilidad a antibióticos para clasificar las bacterias como no MDR (cuando no presentaron multirresistencia), MDR, XDR o PDR. La figura 1 muestra que 75 bacterias gramnegativas (78%) presentaron algún nivel de multirresistencia: se encontraron 66 con MDR, siete con XDR y, notoriamente, dos con PDR. Por su parte, 48 (69%) aislados de bacterias grampositivas resultaron ser MDR. La caracterización por especie y su nivel de MDR se muestran en la tabla 3. Las 166 bacterias pertenecieron a una de 33 especies distintas; solo tres de las 33 especies aisladas no presentaron ningún nivel de MDR (Pseudomonas oryzihabitans, Citrobacter freundii y Staphylococcus schleiferi). P. aeruginosa mostró los tres niveles de resistencia múltiple (MDR: ocho aislados; XDR: tres aislados; PDR: dos aislados). Al menos un aislado de K. pneumoniae, Klebsiella aerogenes, Acinetobacter baumannii y complejo Burkholderia cepacia presentó MDR y XDR. Por último, 25 especies mostraron al menos un aislado con MDR. Esto nos demuestra que la multirresistencia en la muestra abarca todos los niveles y casi todas las especies incluidas.
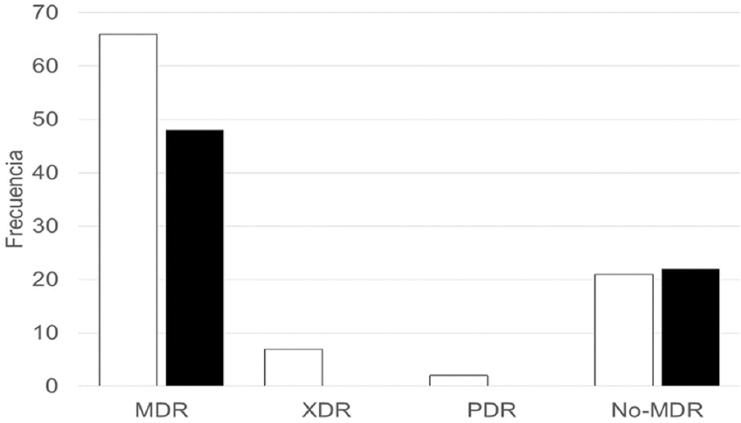
Figura 1 Distribución de aislados por nivel de multirresistencia y tinción. En blanco, aislados gramnegativos; en negro, aislados grampositivos. MDR: multirresistentes; PDR: panresistentes; XDR: con resistencia extendida.
Tabla 3 Distribución de los aislados por especie y nivel de resistencia
| Especie | Tinción de Gram | Frecuencia | Nivel de resistencia | |||
|---|---|---|---|---|---|---|
| MDR | XDR | PDR | No MDR | |||
| Escherichia coli | − | 29 | 23 | 0 | 0 | 6 |
| Staphylococcus epidermidis | + | 22 | 17 | 0 | 0 | 5 |
| Pseudomonas aeruginosa | − | 18 | 8 | 3 | 2 | 5 |
| Enterococcus faecalis | + | 14 | 7 | 0 | 0 | 7 |
| Klebsiella pneumoniae | − | 14 | 11 | 1 | 0 | 2 |
| Staphylococcus aureus | + | 10 | 7 | 0 | 0 | 3 |
| Staphylococcus hominis | + | 9 | 5 | 0 | 0 | 4 |
| Staphylococcus haemolyticus | + | 7 | 6 | 0 | 0 | 1 |
| Klebsiella aerogenes | − | 5 | 4 | 1 | 0 | 0 |
| Enterobacter cloacae | − | 4 | 2 | 0 | 0 | 2 |
| Acinetobacter baumannii | − | 4 | 2 | 1 | 0 | 1 |
| Klebsiella oxytoca | − | 3 | 2 | 0 | 0 | 1 |
| Stenotrophomonas maltophilia | − | 3 | 3 | 0 | 0 | 0 |
| Proteus mirabilis | − | 2 | 1 | 0 | 0 | 1 |
| Bacillus cereus | + | 2 | 2 | 0 | 0 | 0 |
| Pseudomonas putida | − | 2 | 1 | 0 | 0 | 1 |
| Streptococcus agalactiae | + | 2 | 1 | 0 | 0 | 1 |
| Serratia liquefaciens | − | 1 | 1 | 0 | 0 | 0 |
| Alcaligenes faecalis | − | 1 | 1 | 0 | 0 | 0 |
| Enterobacter aerogenes | − | 1 | 1 | 0 | 0 | 0 |
| Serratia marcescens | − | 1 | 1 | 0 | 0 | 0 |
| Staphylococcus lentus | + | 1 | 1 | 0 | 0 | 0 |
| Staphylococcus cohnii | + | 1 | 1 | 0 | 0 | 0 |
| Pseudomonas oryzihabitans | − | 1 | 0 | 0 | 0 | 1 |
| Citrobacter freundii | − | 1 | 0 | 0 | 0 | 1 |
| Staphylococcus schleiferi | + | 1 | 0 | 0 | 0 | 1 |
| Providencia rettgeri | − | 1 | 1 | 0 | 0 | 0 |
| Achromobacter spp. | − | 1 | 1 | 0 | 0 | 0 |
| Enterococcus faecium | + | 1 | 1 | 0 | 0 | 0 |
| Salmonella spp. | − | 1 | 1 | 0 | 0 | 0 |
| Complejo Burkholderia cepacia | − | 1 | 0 | 1 | 0 | 0 |
| Pantoea agglomerans | − | 1 | 1 | 0 | 0 | 0 |
| Sphingobacterium multivorum | − | 1 | 1 | 0 | 0 | 0 |
MDR: multirresistencia; PDR: panresistencia; XDR: resistencia extendida.
En cuanto al tipo de muestra biológica y el nivel de resistencia (Tabla 1), todos los aislados procedentes de orina que presentaron multirresistencia pertenecieron al nivel MDR, es decir, no hubo aislados de orina con XDR ni PDR. En las muestras de exudado de herida o escara, por cada 17 aislados MDR se aisló uno con XDR. En las muestras de hemocultivo o catéter, por cada siete aislados MDR se aisló alrededor de uno con XDR. En las muestras de expectoración o secreción bronquial, por cada 20 aislados MDR se encontró uno con XDR. Los dos aislados de P. aeruginosa con PDR se obtuvieron de muestras de hemocultivo o catéter y de expectoración o secreción bronquial.
En la figura 2 se muestran las cinco especies más frecuentes en cada nivel de multirresistencia. En orina, como ya se ha mencionado, todos los aislados multirresistentes presentaron MDR y todos correspondieron a E. coli. En las muestras tomadas de exudado de herida o escara, las cepas multirresistentes más frecuentemente aisladas con nivel MDR fueron E. coli (cinco aislados) y S. epidermidis (cuatro aislados); hubo un solo aislado con XDR, que correspondió a P. aeruginosa, y no hubo aislados PDR. Los aislados obtenidos de hemocultivo o catéter fueron los que mostraron más variedad; E. coli siguió estando entre las bacterias más frecuentes con MDR (dos aislados), pero S. epidermidis se aisló con mayor frecuencia (6 aislados), y entre los aislados con MDR que les siguieron en frecuencia se encontraron P. aeruginosa (un aislado), K. pneumoniae (un aislado), K. aerogenes (un aislado) y Acinetobacter baumannii (un aislado). Por último, como se mencionó anteriormente, uno de los dos aislados PDR de P. aeruginosa provino de una muestra de hemocultivo o catéter y el otro de expectoración o secreción bronquial. En este último fluido, el perfil de bacterias semejó en composición al de hemocultivo o catéter; se encontraron siete aislados de S. epidermidis, cuatro aislados de K. pneumoniae y tres aislados cada uno de E. coli, P. aeruginosa y K. aerogenes. Entre los aislados con XDR de expectoración o secreción bronquial más frecuentes se identificaron P. aeruginosa y miembros del complejo B. cepacia. Como material suplementario, en el Anexo 1 se detallan todos microorganismos aislados por especie, la muestra de donde proceden y el nivel de resistencia (MDR, XDR o PDR).
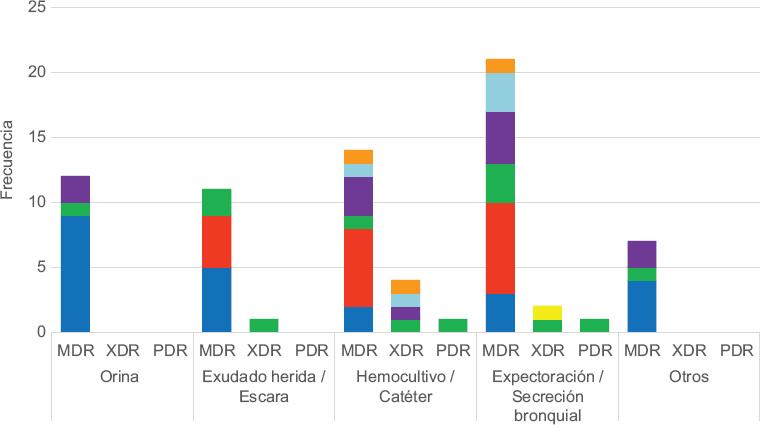
Figura 2 Microorganismos resistentes más frecuentes y distribución según el origen de la muestra: Escherichia coli (azul oscuro), Klebsiella pneumoniae (violeta), Pseudomonas aeruginosa (verde), Staphylococcus epidermidis (rojo), Acinetobacter baumannii (naranja), Klebsiella aerogenes (azul claro) y complejo Burkholderia cepacia (amarillo). MDR: multirresistentes; PDR: panresistentes; XDR: con resistencia extendida.
Con el propósito de explorar la magnitud del problema de la multirresistencia en las distintas secciones del hospital, los aislados se clasificaron de acuerdo con el servicio y el nivel de multirresistencia (Tabla 4). Se encontró un 53% a 83% de aislados con algún nivel de multirresistencia en los diferentes servicios, siendo la unidad de terapia intensiva (83.3%), el servicio de ginecología y obstetricia (80%), y el servicio de cirugía (73.9%) los más afectados en cuanto a proporción de aislados con multirresistencia. La unidad de cuidados intensivos neonatales presentó aislados bacterianos con los tres niveles de multirresistencia; de este servicio se aislaron las dos cepas de P. aeruginosa con PDR (Tablas 3-4). No hubo aislados bacterianos procedentes del servicio de urgencias.
Tabla 4 Distribución de aislados por servicio de ingreso y nivel de resistencia
| Servicio | N.º de aislados | Aislados MDR n (%) | Aislados XDR n (%) | Aislados PDR n (%) | Total MDR+XDR+PDR n (%) | Total no MDR n (%) |
|---|---|---|---|---|---|---|
| Cirugía | 23 | 17 (74) | 0 | 0 | 17 (74) | 6 (26) |
| Medicina interna | 30 | 16 (53) | 0 | 0 | 16 (53) | 14 (47) |
| Ginecología y obstetricia | 5 | 4 (80) | 0 | 0 | 4 (80) | 1 (20) |
| Pediatría | 5 | 2 (40) | 0 | 0 | 2 (40) | 3 (60) |
| Unidad de cuidados intensivos neonatales | 31 | 19 (61) | 3 (10) | 2 (6) | 24 (77) | 7 (23) |
| Unidad de terapia intensiva | 72 | 56 (78) | 4 (5) | 0 | 60 (83) | 12 (17) |
MDR: multirresistentes; PDR: panresistentes; XDR: con resistencia extendida.
Discusión
La resistencia bacteriana es un problema altamente significativo de trascendencia mundial que amerita su vigilancia y control continuo para limitarla. La OMS ha implementado estrategias12,13 que son primordiales para direccionar las acciones. Los resultados de este estudio muestran que, así como ocurre en todo el mundo14-16, la multirresistencia en los aislados de nuestro hospital afecta tanto a grampositivos como a gramnegativos (Fig. 1). Como en otros estudios17, E. coli y Klebsiella sp. se encontraron entre los gramnegativos multirresistentes más frecuentes, y Pseudomonas ocupó el segundo lugar en cuanto a frecuencia entre los multirresistentes gramnegativos, y en extensión de multirresistencia ocupó el primer lugar, ya que dos aislados clínicos presentaron PDR. En cuanto a los microorganismos grampositivos, S. epidermidis fue la bacteria con multirresistencia más frecuentemente aislada, a diferencia de otros estudios17 en los que S. aureus suele ocupar el primer lugar; 17 de los 22 aislados clínicos de S. epidermidis en esta muestra presentaron MDR, lo cual es de considerar dado su potencial para participar en infecciones nosocomiales y formación de biopelículas. E. faecalis también se aisló de manera frecuente y el 50% de estos aislados presentaron MDR.
Es alarmante observar que únicamente uno de cada cuatro aislados clínicos de esta muestra no presenta multirresistencia (44 de 166 aislados) (Fig. 1), y que de las 33 especies identificadas solo tres no presentaron ningún nivel de multirresistencia (Tabla 3). Este hecho señala una resistencia generalizada y confirma la gravedad del problema en la región norte del país17.
El análisis de la multirresistencia según el fluido señala que el problema es de mayor complejidad y gravedad en los pacientes con infecciones en la sangre (o al menos en un catéter) o en las vías respiratorias, ya que en estos fluidos fue donde se encontró el mayor porcentaje de aislados multirresistentes de cada nivel (MDR, XDR y PDR), la mayor variedad de bacterias y los dos aislados PDR.
En cuanto a los servicios hospitalarios, los de terapia intensiva, ginecología y obstetricia, y cirugía requieren atención por la alta proporción de aislados bacterianos con multirresistencia en muestras clínicas provenientes de pacientes de dichos servicios; además, el servicio de cuidados intensivos neonatales requiere atención especial por ser fuente de aislados PDR.
La información obtenida permitirá implementar medidas dirigidas a los servicios con mayores problemas de multirresistencia y enfocar el uso adecuado de los antibióticos en este hospital para el tratamiento de las enfermedades infecciosas ocasionadas por los microorganismos que se encontraron con mayor multirresistencia, tanto en frecuencia como en gravedad. El estudio de otros factores posiblemente involucrados se encuentra en proceso.
Conclusiones
En nuestro estudio, la multirresistencia a los antibacterianos abarca hasta el 75% de los aislados clínicos y alcanza una extensión de PDR en el 1.2% de los casos. Los aislados multirresistentes más frecuentes fueron E. coli, P. aeruginosa, S. epidermidis y K. pneumoniae; estas especies y los servicios de cuidados intensivos, cirugía, ginecología y obstetricia, y la unidad de cuidados intensivos de neonatos son áreas de prioridad para el estudio de factores de riesgo y la implementación de intervenciones.











 nueva página del texto (beta)
nueva página del texto (beta)


