INTRODUCCIÓN
La anaplasmosis bovina, es una enfermedad hemobacteriana infecciosa que afecta a los bovinos y algunos rumiantes silvestres, causada por rickettsias del género Anaplasma spp. La bacteria es transmitida biológicamente por garrapatas del género Rhipicephalus spp. y Dermacentor spp. (Potgieter y van Rensburg 1987). La anaplasmosis se caracteriza por causar anemia severa, pérdida de peso, disminución en la producción de leche, ictericia y fiebre; además de abortos en el último tercio de gestación y la muerte en ausencia de tratamiento oportuno. En algunos casos los animales que presentaron una infección aguda pueden recuperarse y fungen como reservorios de la enfermedad lo que permite la trasmisión a animales susceptibles (Aubry y Geale 2011). En México se considera a la anaplasmosis como responsable de hasta el 26% de la mortalidad de animales manejados en programas de mejoramiento genético (Rodríguez et al. 2009), y una pérdida económica anual estimada en más de 300 millones de pesos (Rodríguez-Vivas et al. 2017).
En diferentes países se han realizado investigaciones referentes al control de la anaplasmosis por vacunación, sin embargo, a la fecha no existe ninguna vacuna debido a la amplia diversidad antigénica y genética de A. marginale (Ocampo Espinoza et al. 2006). En los últimos años se han utilizado estrategias para el diseño y generación de vacunas con base en las proteínas que se encuentran en la superficie de la bacteria, identificándose proteínas homologas al Sistema de Secreción Tipo IV (SSTIV) descrito por primera vez en Agrobacterium tumefaciens; constituido por 11 proteínas codificadas por el operón virB que incluye a VirB1 - VirB11 y VirD4 los cuales se clasifican en categorías de acuerdo con sus funciones como: activación, accesorios, proteínas asociadas al phylum y de secreción (Zhao et al. 2016).
La proteína VirD4, facilita el contacto del sustrato con el canal de translocación, por lo que está clasificada dentro de los miembros de proteínas de acoplamiento o receptoras de sustrato (González-Pedrajo y Dreyfus 2003). La proteína cuenta con los motivos funcionales Walker A y B, conservados a través de 13 genomas; estos motivos son importantes para la unión con nucleótidos y la hidrólisis del ATP, por lo que las mutaciones en estos motivos provocan pérdida en la función de translocación (Willadsen et al. 1989). También se ha reportado actividad ATPasa en A. tumefaciens, la cual es estimulada por DNA de cadena simple y doble (Atmakuri et al. 2003). También se identificó un homólogo a VirD4 presente en Helicobacter pylori necesario para la secreción de CagA por el sistema tipo IV (Stein et al. 2000), por lo que las funciones de estas proteínas no se limitan a las reacciones de transferencia de ADN, sino que tendrían un papel más general en el tráfico del sustrato a través de las maquinarias tipo IV. Con base en esta información, el objetivo del presente trabajo fue analizar la presencia y grado de conservación del gen virD4 en cepas mexicanas de A. marginale, mediante análisis in silico y su identificación en membranas de eritrocitos infectados con A. marginale.
MATERIALES Y MÉTODOS
Selección de muestras y extracción de ADN genómico de A. marginale
Se utilizaron las siguientes cepas mexicanas previamente caracterizados mediante los marcadores moleculares msp1 α y msp4: Playa Vicente, Veracruz (Mex-30-130-01); Puente de Ixtla, Mor., (Mex-17-017-01); Soto la Marina, Tamps, (Mex-28-037-01); Tlapacoyan-2, Ver. (Mex-30-184-03); Aguascalientes, Ags, (Mex-01-001-01) y Guayaboso, Tamps, (Mex-28-037-02) (Nomenclatura del INEGI de Banco de Germoplasma de la unidad de Anaplasmosis). El DNA genómico se extrajo a partir de sangre infectada de bovino (3.9 x 1010 eritrocitos infectados) mediante el Kit UltracleanTM Blood SpinTM ADN Isolation (MO BIO) siguiendo las instrucciones del fabricante.
Amplificación por PCR del gen virD4
Se diseñaron dos juegos de iniciadores basados en las secuencias reportadas en GenBanck (AMF_RS09935 antes NC_004842 REGION: 1154957.1157377 mediante el programa Integrated ADN Technologies, Inc. (IDT). Los iniciadores fueron: sentido 5’ATGCACAGTAGCTCTAATCACATAC3’ y, antisentido 5’TTACGGGTTGTCATCATCACG3’, usados bajo las siguientes condiciones 1 µL de iniciadores, 13 µL de Taq Green (Promega), 11 µL de agua libre de nucleasas y 1 pg de DN, utilizando el siguiente programa 95 oC 1 min; 57 oC, 75 oC 45 s. 30 ciclos; 72 oC 10 min. Los amplicones se visualizaron por electroforesis en geles de agarosa al 1%, los cuales se purificaron mediante el kit Wizard R Sv Gel and PCR Clean Up System, (Promega) y clonados mediante el kit cloneJET PCR Cloning Kit, (Thermo Fisher Scientific). El producto de la clonación también se utilizó para la transformación de células competentes E. coli Top10 ® 8878 Thermo-Scientific.
Secuenciación de los fragmentos amplificados
Las células transformadas E. coli Top10 ® positivas por PCR a virD4, se incubaron en 2 mL de medio LB con ampicilina (50 mg mL -1) durante toda la noche a 37○ C en agitación constante a 200 rpm. Posteriormente, el cultivo se centrifugó a 13 000 x g durante 2 min a temperatura ambiente para realizar la extracción de DNA plasmídico mediante el kit comercial GeneJET Plasmid Miniprep, Thermo scientific, siguiendo las instrucciones del fabricante. Los plásmidos obtenidos se secuenciaron en ambos sentidos, utilizando oligonucleótidos del promotor T7 (sentido y antisentido) en la Unidad de Secuenciación del Instituto de Biotecnología IBT, UNAM, campus Morelos. Debido al tamaño de la secuencia, ésta se dividió en tres fragmentos para lograr la secuenciación completa del gen virD4.
Análisis in silico de la secuencia nucleotídica y aminoacídica de VirD4
El porcentaje de conservación entre las secuencias obtenidas de los aislados se determinó mediante el programa ClustalW (Thompson et al. 1994), se utilizó el gen putativo virD4 de A. marginale cepa St. Maries reportado en el NCBI-GENE como referencia. La secuencia deducida de aminoácidos se obtuvo a través de Swissprot Expassy (Gasteiger et al. 2005), utilizando la secuencia de nucleótidos de la cepa Tlapacoyan-2 Mex-30-184-03. Posteriormente se predijeron los dominios extracelulares expuestos con el programa TMHMM (Moller et al. 2001) la presencia de péptido señal y la presencia de epítopos lineales tipo B mediante los programas BCEPred, ABCPred (Saha y Raghava 2004, 2006) y NETNGlyc (Gupta et al. 2004).
Síntesis de Péptidos a partir de la proteína VirD4
El péptido diseñado a partir de las ponderaciones más alta en el grado de inmunogenicidad y antigenicidad que arrojaron las herramientas computacionales ABCpred y ABCpred se sintetizó comercialmente en forma de tetrámero ramificado, mediante el sistema de Péptidos Antigénicos Múltiples (MAP’S). La síntesis se realizó por American Peptide Company, Inc. Identificado como péptido MAPVirD4.
Producción de anticuerpos específicos contra MAPVirD4
Se utilizaron para cada tratamiento 3 conejos de la raza Nueva Zelanda de dos meses de edad albergados en el bioterio del Instituto Nacional de Salud Pública, en Cuernavaca, Morelos. Se mantuvieron en jaulas con agua y alimento ad libitum. La inmunización consistió en la mezcla de 50 µg de tetrámero MAPVirD4 en 1 mL de PBS 1x y emulsificado con 1ml de adyuvante Montanide ISA 71VG (Seppic ® )por vía subcutánea. La dosis se administró en sitios cercanos a los nódulos linfáticos inguinales y axilares, utilizando una jeringa de 3 ml con aguja de calibre 22 Como control negativo se utilizó el suero de cada animal previo a la inmunización. Los animales fueron inmunizados los días 0 (T0), 8 (T1), 15 (T2), 22 (T3), 29 (T4) y 36 días (T5). Para seguimiento, se colectó suero posterior a cada inmunización a partir de T1. Los sueros se evaluaron mediante técnica inmunoensayos de ELISA, para determinar el momento idóneo en la producción de anticuerpos.
Reconocimiento del péptido MAPVirD4 mediante la técnica inmunoenzimática de ELISA
Para determinar la cinética de la respuesta inmune, placas de 96 pozos (Corning ® ), se recubrieron con 10 µg de MAPVirD4 a una dilución 1:200 y se dejó incubar 18 h a 4 oC. Posteriormente, se lavó tres veces con SSAF Tween 20 al 0.05%, y se colocaron los sueros T0, T3, T4 y T5 a dilución 1:100, durante una hora. Una vez transcurrido el tiempo se realizaron lavados, y se agregó anti-IgG conejo-fosfatasa alcalina (Phosphatase-Conjugated AffiniPure Goat Anti-Bovine, Jackson Immuno Research, Laboratories, INC.) diluido 1:5000 y se dejó incubar por 1 h a 37 oC, se retiró el conjugado y se lavó la placa. Por último, como sustrato, se agregó p-nitrofenilfosfato al 0.075% en solución Tris 100mM pH 9.5, la reacción se dejó incubar por 30 min a 37 oC. para luego leer a una absorbancia de 405 nm en un lector de ELISA (Microplate reader Model 680 BIO-RAD).
Reconocimiento de la proteína VirD4 en eritrocitos infectados de bovino por inmunofluorescencia
En un frotis de sangre infectada con A. marginale Tlapacoyan-2 Mex-30-184-03 se colocaron 10 µL de los sueros de conejo anti-MAPVirD4 del T0 y T5 a una dilución de 1:50 en PBS (pH 7.2). La laminilla se incubó en cámara húmeda y obscura en posición horizontal a 37 oC por 30 min, se lavó tres veces por 5 min con PBS y un minuto con agua destilada. Una vez seca, se colocaron 10 µL de anticuerpo conjugado FITC Anti-Conejo (Jackson ImmunoResearch Laboratories) en cada una de las muestras. El exceso de conjugado se eliminó lavando la laminilla tres veces con PBS 1X pH 7.4 y secó a temperatura ambiente. Para su observación al microscopio de fluorescencia, se colocaron unas gotas de glicerina 1:10 en PBS pH 7.2. La laminilla se observó al microscopio con objetivo de inmersión (100x). Paralelamente, una laminilla de sangre no infectada (control) se trató como se describió anteriormente para verificar que la fluorescencia fuera específica.
RESULTADOS
A la amplificación del gen virB4 por PCR se observaron amplicones con una longitud calculada de 2421pb (Figura 1), lo que coinciden con los reportados en NCBI para el gen virD4 de la cepa St. Maries de A. marginale. Las secuencias de nucleótidos obtenidas para el gen virD4 de A. marginale de los aislados mexicanos mostraron una identidad y similitud del 100% mediante el programa ClustalW.

Figura 1: Amplificación del gen virD4 en seis aislados mexicanos con los iniciadores diseñados: PM) Marcador de Pesos molecular, C-) Control negativo, 1) Playa Vicente; 2) Puente de Ixtla, Mor.; 3) Soto la Marina Tamps; 4) Tamaulipas Guayaboso, 5) Aguascalientes 6) Tlapacoyan-2, Ver. Se observó un amplificado en todos los aislados de aproximadamente 2421 pb, el cual coincide con lo reportado para la secuencia de St Maries GenBank: AMF_RS09935 antes NC_004842 REGION: 1154957..1157377.
En cuanto a las características estructurales de la secuencia proteica, se predijo una composición de 806 aminoácidos, con un peso molecular de 91 kDa y un pI de 4.36 (Figura 2, EXPASY); mostro seis posibles regiones transmembranales entre el aminoácido 1 al 300 (Figura 3, TMHMM). Por otro lado, se observó la ausencia de péptido señal (Figura 4, SignalP). Pero mediante el programa PSIPRED se identificó un número mayor de alfa hélices que están representadas por cilindros de color rosa en la estructura de la proteína (Figura 5).
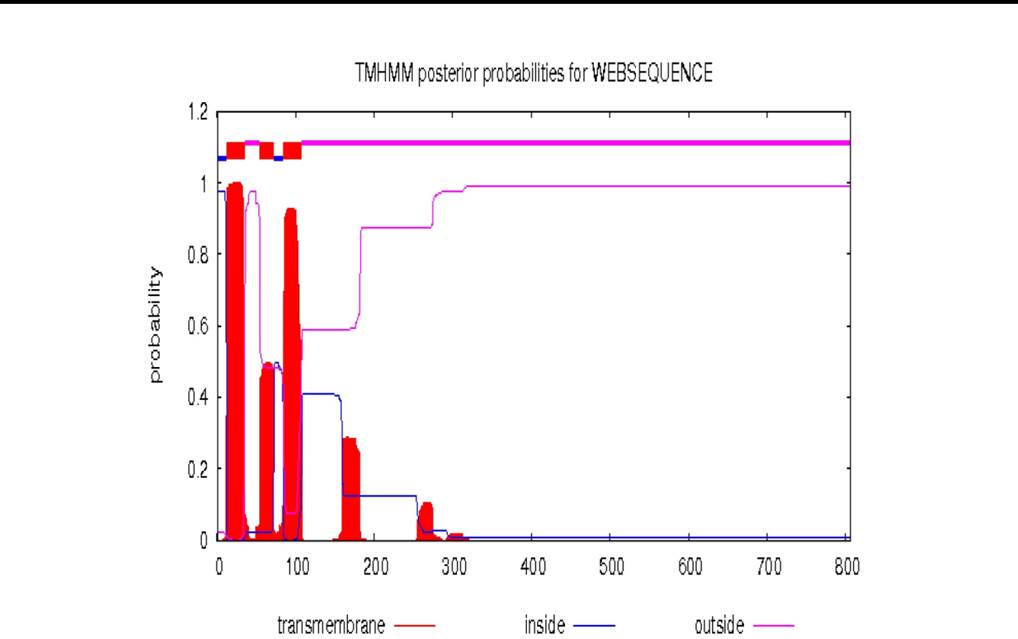
Figura 3 Predicción de regiones transmembranales. Se observan tres posibles regiones transmembranales del aminoácido 1 al 275. Cada color representa lo siguiente: rojo regiones transmembranales; azul regiones intracitoplásmicas; rosa regiones extramembranales El eje de las x corresponde a la posición de los aminoácidos y el de las y a la puntuación que otorga el programa.
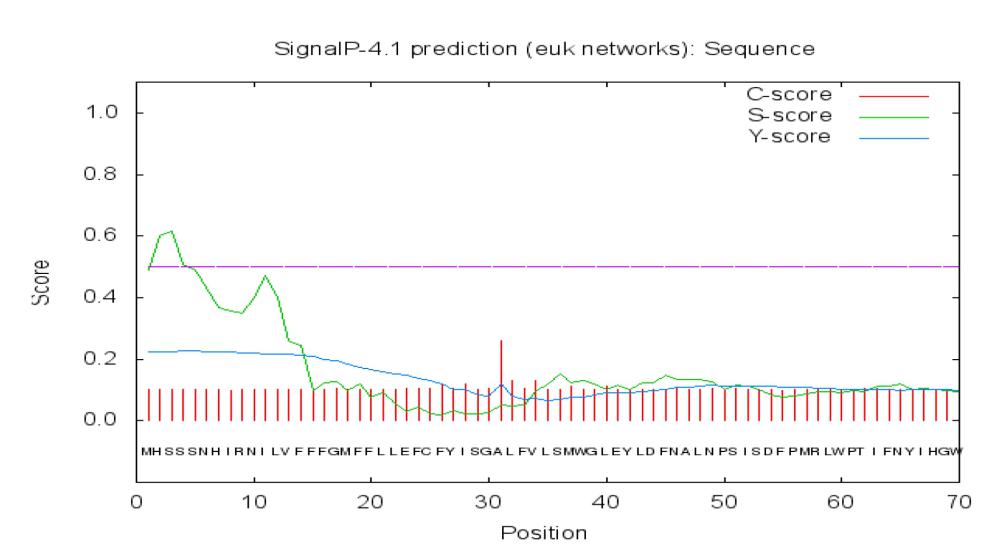
Figura 4 Predicción del péptido señal. No hay Identificación de péptido señal por el programa SignalP. El eje de las x corresponde a la cadena de aminoácidos y el de las y a la puntuación que otorga el programa. Score C: Nos indica el primer aminoácido presente en la proteína madura. Score S: Distingue la posición del péptido señal, diferenciándolo de la proteína madura. Score Y: Es una combinación de la C -score y la pendiente de la S-score, lo que resulta en una mejor predicción del sitio de corte que el C -score por sí solo.
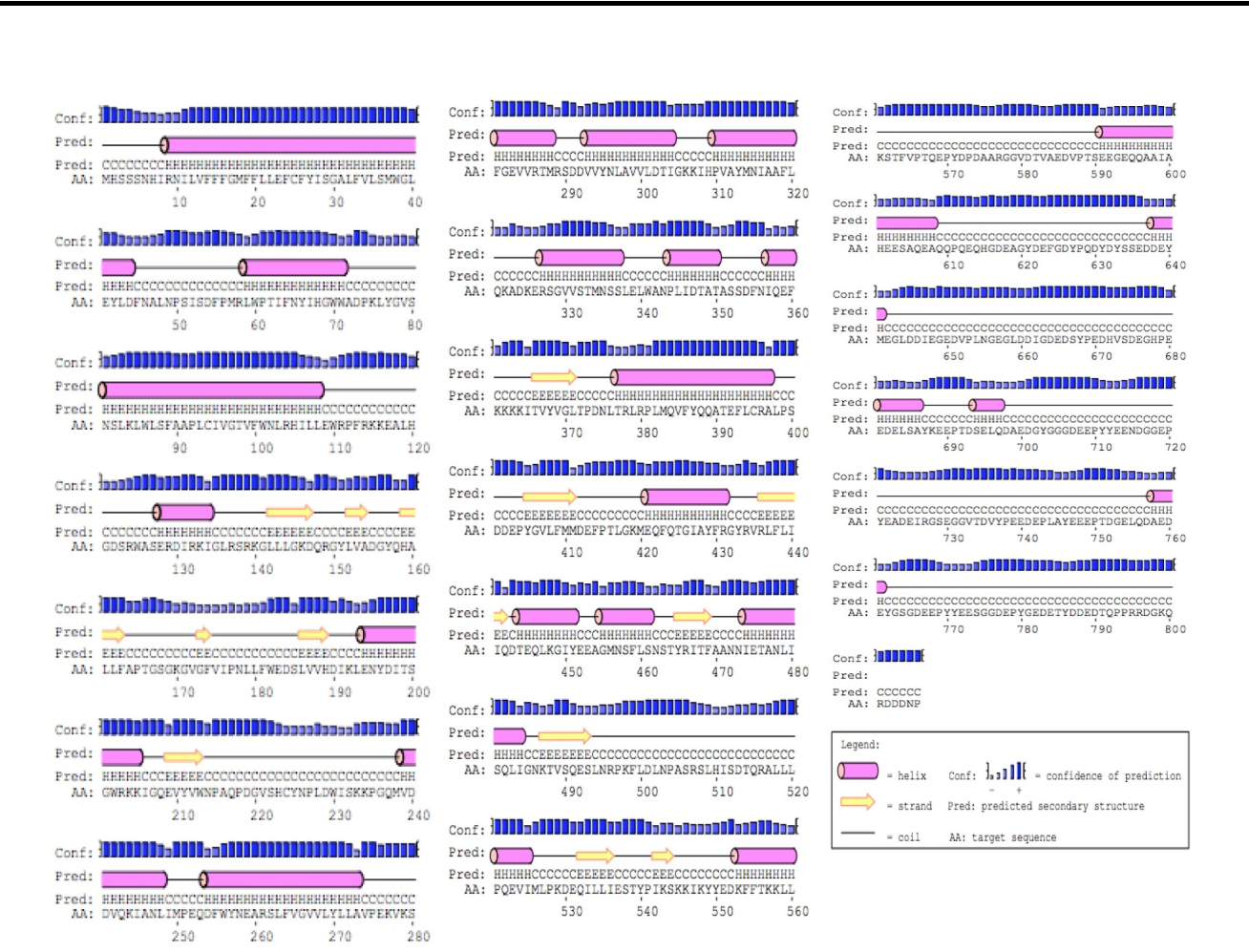
Figura 5 Predicción de estructura secundaria. Las regiones α-hélices se muestran en tonos rosados, mientras que las estructuras β-plegada se muestran representadas con una flecha amarilla. Las líneas negras representan bobinas o vueltas y la figura en azul, representa la confiabilidad de la predicción.
Con respecto a las regiones inmunogénicas y antigénicas, los programas ABCPred y BCEPred identificaron seis secuencias correspondientes a epítopos lineales tipo B (Tabla 1), tres de las secuencias predichas por los programas se encontrarón en las regiones trasnmembranales observadas en el programa TMHMM (posición 68-83 aa., 73-83 aa., 272-286 aa.), por lo que se descartaron como candidatos. Los restantes tres péptidos se encontraron en el dominio extracelular de proteína VirD4, característica importante en la búsqueda de un candidato vacunal. Se analizaron las características de los aminoácidos presentes en cada una de las secuencias restantes obtenidas con los programas y se seleccionó el péptido PTDGELQDAEDEYG (749-763 aa.) por presentar hidrofilicidad, solubilidad aminoacídica y facilidad de síntesis; así como un tamaño y ubicación adecuada para considerarse como candidato a vacuna.
Tabla 1 Comparación de epítopos para VirD4 con los programas ABCpred y BCE-Pred. Se observan los epítopos predichos por ambos programas computacionales. Los epítopos subrayados coinciden en ambos programas y en negritas se observa el epítopo seleccionado para la síntesis.
| Péptido | Posición | Tamaño (aa) |
| LAVPEKVKSFGEVVRT | 272-286 | 16 |
| PKDEQILLIESTYPIKS | 527-543 | 17 |
| EPTDGELQDAEDEYG | 749-763 | 15 |
| HGWWADPKLYGVSNSL | 68-83 | 16 |
| PKLYGVSNSL | 73-83 | 10 |
| DEIRGSEGGVTDVYPEEDEPLAYEEEPTD | 724-762 | 29 |
Los anticuerpos generados contra MAPVirD4 se evaluaron por la técnica de ELISA usando el mismo MAPVirD4 como antígeno. La respuesta se observó con el suero T3, y fue aumentando hasta el día 36 (T5) post-inoculación (Figura 6). Estos resultados demuestran la capacidad del péptido MAPVirD4 de ser reconocido por el sistema inmune del conejo en los diferentes tiempos y el aumento de estos anticuerpos a la par del reto antigénico. Así mismo se observó que el formato de Péptidos antigénicos Múltiples (MAP) es útil para evaluar la producción de anticuerpos, asi como el uso de los programas bioinformáticos para predecir capacidades antigénicas e inmunogénicas.

Figura 6: Evaluación de la generación de anticuerpos contra MAPVirD4 en conejos mediante la técnica de ELISA. Los sueros de conejo se diluyeron 1/100. Se observa el aumento de anticuerpos en el T5. Control (-) Suero de conejo Pre-inmune, T3) Suero de conejo Tiempo 3 de inmunización, T4) Suero de conejo Tiempo 4 de inmunización, T5) Suero de conejo Tiempo 5 de inmunización.qwertyuiop.
Los anticuerpos generados contra MAPVirD4, reaccionaron con formas intraeritrocíticas de la cepa Tlapacoyan-2 de A. marginale, como se observa en el panel D de la Figura 7 y no se observó reacción contra eritrocitos no infectados, esto indica que no existe respuesta cruzada con alguna otra proteína presente en la estructura del eritrocito (Figura 7 C). Así mismo, no se observó respuesta con el suero preinmune contra eritrocitos infectados, esto significa que los anticuerpos se produjeron posterior al contacto con el péptido (Figura 7 B). Estos resultados permiten asumir que VirD4 al ser reconocida por anticuerpos específicos por inmunofluorescencia tendría que ser estudiada más a detalle para determinar su potencial imunogénico en el bovino.

Figura 7: Microscopía de fluorescencia. Reconocimiento de A. margianle mediante suero de conejo inmunizado con MAPVirD4. A) Suero de conejo preinmune contra eritrocitos sanos; B) Suero de conejo preinmune contra eritrocitos infectados; C). Suero de conejo T5 contra eritrocitos sanos y D) Suero de conejo T5 contra eritrocitos infectados (Las fechas indican formas infectantes de A. marginale en eritrocitos de bovinos, identificadas).
DISCUSIÓN
Los patógenos bacterianos intracelulares cuentan con mecanismos para su replicación dentro de las células eucariontes, de los cuales muchos de ellos son desconocidos (Sinai et al. 1997). Algunos de estos mecanismos están relacionados con la transferencia de moléculas entre el microrganismo y el hospedero, como en los sistemas de secreción, que juegan un papel importante en la invasión y patogenia de organismos (Gillespie et al. 2009, Juhas et al. 2012, Redzej et al. 2017). El conocimiento detallado sobre estos sistemas de secreción podría ser usado para plantear nuevas estrategias terapéuticas en contra de estos microorganismos.
Los alineamientos de las secuencias del gen virD4 de seis aislados mediante herramientas bioinformáticas tuvieron un 100% de identidad entre ellas y con las secuencias de reportados en NCBI, este grado de conservación permite especular que este gen podría estar asociado a una actividad importante en Anaplasma, considerando que estas seis cepas presentan variabilidad respecto de otros marcadores moleculares (Jiménez-Ocampo et al. 2012). Previamente se ha demostrado que mutaciones en los genes del sistema de secreción tipo IV en Haemophilus influenzae (Gram negativo) disminuyen la capacidad de conjugación entre cepas, en comparación con las cepas no modificadas (Juhas et al. 2012). De manera particular al mutar el antígeno CagA, dominio presente en la proteína VirD4 de H. pylori, esta pierde su función durante la interacción con su huésped (Stein et al. 2000). Por otro lado, se demostró recientemente un papel estabilizador de VirD4 hacia el complejo VirB1-11, lo cual sugiere una función importante de VirD4 sobre la virulencia en las bacterias que la expresan (Redzej et al. 2017).
Para seleccionar una proteína que pueda ser blanco del sistema inmune, ya sea para pruebas diagnósticas o para el control de la anaplasmosis es primordial conocer su estructura y su capacidad inmunogénica (Araújo et al. 2008). Los resultados de los análisis in silico de VirD4 mostraron regiones transmembranales que podrían funcionar como anclaje de la proteína a la membrana, como en el caso de Agrobaterium tumefaciens (Redzej et al. 2017). Asimismo, se observó la ausencia de péptido señal, resultado que afirma lo ya publicado con respecto a la clasificación de esta proteína en el sistema de secreción tipo IV, las cuales no dependen de secuencias señal para la translocación de sustratos desde el citoplasma hasta el medio extracelular (Wilson et al. 2011). Además, se observó un número mayor de alfa hélices, característico de proteínas que cuentan con motivos estructurales de unión a DNA (Acosta et al.1996), dominios que se mencionan en los estudios previos de esta proteína en otros organismos y tres secuencias representativas de epítopos tipo B conservados y expuestos hacia las regiones extracelulares (Llosa et al. 2002).
La inoculación de conejos con estos tetrámeros antigénicos múltiples (MAPVirD4) inducen la producción de anticuerpos específicos. Estos sueros reaccionan con eritrocitos infectados con A. marginale en un ensayo de inmunofluorescencia. Lo que corrobora no solo que MAPVirD4 es expresada por la rickettsia durante su estancia en eritrocitos, sino que se muestra de manera indirecta su localización.
CONCLUSIONES
El análisis bioinformático demostró que las secuencias obtenidas presentan un 100% de conservación e identidad entre los seis aislados mexicanos secuenciados; así como entre las secuencias reportadas en NCBI. Se identificó la presencia de epítopos B conservados entre las seis cepas mexicanas de A. marginale y las reportadas en NCBI. Los anticuerpos específicos reaccionaron por ELISA con el MAPVirD4 y con cuerpos iniciales de A. marginale en formas intraeritrocíticas de A. marginale por fluorescencia directa. De acuerdo con sus características, las proteínas son ideales para ser candidatos a vacunas contra la Anaplasmosis bovina.











 nueva página del texto (beta)
nueva página del texto (beta)




