Introducción
El programa de conservación de una raza en peligro de extinción debe considerar como punto de partida y elemento fundamental para la formación y mantenimiento de poblaciones puras con el fin de conservar la variabilidad genética existente en la raza/especie (Santana 2017). Tradicionalmente la diversidad genética se define como la variedad de alelos o diferentes genotipos presentes en una población, los cuales reflejan las diferencias morfológicas, fisiológicas y de comportamiento que se pueden apreciar entre los individuos y poblaciones (Frankham et al. 2002).
El mantenimiento de la diversidad genética es de gran importancia en la crianza de animales debido a que la heterocigosidad y la diversidad alélica se pueden perder a una tasa acelerada cuando se cuenta con poblaciones pequeñas, cerradas y seleccionadas (Sheikhlou y Abbasi 2016). En Colombia, la gran variedad de regiones agroclimáticas presentes ha favorecido el desarrollo de especies animales y vegetales autóctonas, entre las cuales se encuentran incluidas tres razas porcinas, tres ovinas y siete bovinas (Ocampo et al. 2017). Entre las razas porcinas criollas, se cuenta con las razas Zungo en la costa Atlántica, Casco de Mula en los llanos orientales y San Pedreño en el departamento de Antioquia y el viejo Caldas, los cuales tuvieron su origen a partir del cerdo ibérico español que fue introducido por los conquistadores españoles en 1493 (Oslinger 2006).
Los cerdos criollos colombianos se caracterizan por presentar altas tasas reproductivas, tolerancia a enfermedades parasitarias y gran adaptación a las condiciones del trópico que les permiten producir y reproducirse, contrarrestando factores adversos como la alimentación deficiente, escasez de agua, enfermedades y manejo precario; por todas estas características, los cerdos criollos poseen un papel muy importante en la economía y alimentación campesina (Oslinger 2006). Pero a pesar de todas las bondades y extraordinaria capacidad de adaptación que poseen, las razas criollas porcinas se encuentran en riesgo de desaparecer, debido al uso preferente de razas mejoradas foráneas en los sistemas de producción porcícolas, estimándose el número de ejemplares de la raza San Pedreño en el país en menos de 200 individuos, además se presume que las poblaciones presentan altas tasas de consanguinidad y alto grado de parentesco, lo que situaría a la raza en estado de riesgo de acuerdo a las categorías definidas por la FAO (FAO 2013).
La Corporación Colombiana de Investigación Agropecuaria AGROSAVIA es la institución encargada de conservar y fomentar el uso de los recursos genéticos autóctonos del país, y para ello dispone del Sistema de Bancos de Germoplasma de la Nación para la Alimentación y la Agricultura (SBG- NAA), proyecto financiado por el Ministerio de Agricultura y Desarrollo Rural en convenio con el Instituto Colombiano Agropecuario ICA (Martínez et al. 2012). Actualmente, la población de cerdos San Pedreño del banco de germoplasma está compuesto por 125 individuos, distribuidos en 6 grupos familiares que se manejan bajo el sistema de apareamiento circular cíclico (Nomura y Yonezawa 1994). Debido a que se trata de un núcleo cerrado, se puede tener un aumento considerable de la consanguinidad, lo cual puede conllevar a efectos negativos sobre las características o aptitudes productivas y reproductivas del animal (Martínez et al. 2012).
El impacto de esta estrategia de manejo reproductivo sobre la evolución de la diversidad genética puede evaluarse en el nivel genealógico a través del pedigrí de los animales utilizando criterios basados en la probabilidad de identidad por descendencia de genes y criterios basados en probabilidades de origen genético (Sheikhlou y Abbasi 2016). Por lo anterior el objetivo de este estudio fue analizar la información de pedigrí de los cerdos San Pedreño del banco de germoplasma en el centro de investigación el Nus de AGROSAVIA para cuantificar la diversidad y estructura genética poblacional en términos de niveles de consanguinidad, tamaño efectivo de la población, intervalo generacional, coeficientes de parentesco y contribución de los fundadores a la población actual, para definir recomendaciones sobre la estrategia de conservación y políticas de reproducción.
Materiales y métodos
Información utilizada
Se utilizó información colectada de los libros de campo de los cerdos San Pedreño (Figura 1) del SBGNAA en el centro de investigación el Nus de AGROSAVIA (San Roque, Antioquia, Colombia) entre los años 2008 y 2017. Cada registro completo en la base de datos estuvo conformado por el código de identificación del individuo, sexo, fecha de nacimiento e identificación del padre y madre. La base de datos incluyó 1 794 animales (901 hembras y 893 machos). Todos los parámetros poblacionales se estimaron considerando la población de referencia definida como los animales que nacieron entre 2015 y 2017 (n = 664), lo que equivale a los animales nacidos durante el último intervalo generacional.
Intervalo generacional
El intervalo generacional se define como la edad promedio de los padres cuando nace su descendencia. Este parámetro se calculó para las cuatro vías genéticas posibles: padre-hijo, padre-hija, madre-hijo y madre-hija. Mientras que el intervalo de generación promedio la población se define como el promedio de las cuatro vías.
Nivel de integridad del pedigrí
La integridad de pedigrí expresa cuanta información generacional en promedio se dispone de cualquier individuo del pedigrí. El número de generaciones para determinado individuo se puede calcular de tres maneras: número de generaciones completas dado como el número de generaciones en que se conocen ambos padres; número de generaciones máximas (número total de generaciones posibles si los padres son conocidos o desconocidos) y número de generaciones equivalentes, que se calculó de la siguiente manera:
Donde: n es el número de generaciones en las que el animal está separado de cada antepasado con un registro conocido (Maignel et al. 1996).
La comparación entre los números de generaciones completas y máximas permite verificar la integridad de la información de pedigrí, por lo que los números cercanos para ambos indican la existencia de pocos animales desconocidos en todo el pedigrí.
Probabilidad de origen genético
La contribución genética esperada de cada fundador de la población de referencia se definió como la probabilidad de que un gen tomado al azar dentro de la población de referencia proceda de un fundador determinado. El número total de fundadores (f) contiene información limitada con respecto a la diversidad genética de la población porque algunos fundadores se han utilizado más intensamente y, por lo tanto, contribuyen más a la población de referencia que otros fundadores. Para tener en cuenta esto, se estimó el número efectivo de fundadores (fe). Este número se definió como el número de fundadores que contribuyen por igual y que se espera que produzcan la misma diversidad genética en las poblaciones bajo estudio (Lacy 1989). La estimación de este parámetro permite medir el equilibrio de las contribuciones genéticas entre los fundadores. El número efectivo de fundadores se calculó como:
Donde: f representa el número de fundadores y q k es la contribución genética del fundador kth a la población de referencia (Gutiérrez y Goyache 2005). Para un número total dado de fundadores, cuanto más equilibradas sean sus contribuciones genéticas esperadas, mayor será el número efectivo de fundadores.
Los ancestros principales (fundadores o no) fueron estimados con el método propuesto por Boichard et al. (1997). La contribución marginal esperada (qj) de cada ancestro principal (j) se calculó como su contribución genética esperada, independientemente de las contribuciones de los otros antepasados. El número efectivo de antepasados (fa), es el número mínimo de antepasados (incluidos los fundadores y los no fundadores) necesarios para explicar la diversidad genética de la población, se estimó de la siguiente manera:
Donde: qj es la contribución marginal de un antepasado j; en otras palabras, la contribución genética realizada por un antepasado que no fue explicada por otros ancestros elegidos previamente (Gutiérrez y Goyache 2005). Calculado de esta manera, f a es una medida de la diversidad genética que explica los cuellos de botella recientes y, por lo tanto, explica en parte la pérdida de la diversidad alélica desde la fundación de la población (Sørensen et al. 2005).
La proporción entre el número efectivo de ancestros y fundadores
Parámetros poblacionales relacionados con el coeficiente de consanguinidad
Coeficiente de parentesco promedio
El coeficiente de parentesco promedio de un individuo es la probabilidad de que un alelo seleccionado al azar de la población pertenezca a un determinado animal. Este coeficiente puede interpretarse como la representación del animal en el pedigrí (Goyache et al. 2003, Gutiérrez y Goyache 2005). De acuerdo con Gutiérrez et al. (2003), este parámetro se calcula como el promedio de los coeficientes en la fila de la matriz de parentesco del numerador correspondiente al individuo.
Coeficiente de consanguinidad
La consanguinidad (F) es la probabilidad de que un individuo tenga dos alelos idénticos por descendencia, para su calculo se utilizó el algoritmo desarrollado por Meuwissen y Luo (1992). El aumento de consanguinidad por generación se calculó de la siguiente manera:
Donde: F t y F t-1 son la consanguinidad promedio en las generaciones t th y t th-1 , respectivamente.
Tamaño efectivo de la población
El tamaño efectivo de la población (N e ) es el número de individuos que contribuyeron de manera efectiva dejando descendientes, transmitiendo genes a la siguiente generación y manteniendo la diversidad genética (Gutiérrez y Goyache 2005). El tamaño efectivo poblacional se calculó de la siguiente manera para la población de referencia:
Donde: ΔF es el aumento relativo de endogamia por generación.
Software utilizado
La integridad del pedigrí, el intervalo generacional, los parámetros derivados de la probabilidad de origen génico, los parámetros poblacionales rela cionados con el coeficiente de consanguinidad y el índice de conservación genética se estimaron con el programa ENDOG 4.8 (Gutiérrez y Goyache 2005).
Resultados
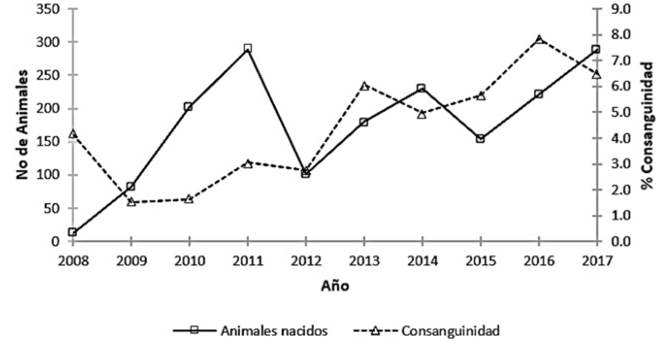
La evolución de los individuos registrados por año de nacimiento es presentado en la Figura 2. A través de los años de estudio se puede observar que el número nacimientos ha variado considerablemente, presentándose la mayor cantidad de nacimientos en el 2011 con 289 nacimientos, en tanto que el menor número de nacimientos se registró en el 2008 con 12 nacimientos pero a partir de dicho año se presentó un incremento progre sivo del número de nacimientos por año. El intervalo generacional promedio fue de 2.13 años. En cuanto al intervalo generacional promedio en las 4 vías posibles fueron: padre-hijo = 2.01 años, padre-hija = 2.05 años, madre-hijo = 2.19 años, madre-hija = 2.27 años. Presentando el menor intervalo generacional la vía padre-hijo(a).
Nivel de integridad del pedigrí
El análisis de integridad del pedigrí reveló que de los 1794 registros, el número de animales con ambos padres conocidos fue de 1 754 individuos, lo que corresponde al 97.77% de la población total, presentando un solo progenitor desconocido 10 animales, lo que corresponde al 0.56% de los registros. Mientras que el análisis de integridad del pedigrí para toda la población reveló 8 generaciones máximas, 4 generaciones completas y 5.18 generaciones equivalentes (Tabla 1).
Tabla 1 Estructura del pedigrí en la población de cerdos San Pedreño del banco de germoplasma entre 2008-2017.
| Parámetro | Valor |
|---|---|
| Número de Animales | 1794 |
| Número de machos | 893 |
| Número de hembras | 901 |
| Número de padres | 45 |
| Número de madres | 85 |
| Animales con progenie | 130 |
| Animales sin progenie | 1664 |
| Animales con padre y madre conocidos | 1754 |
| Animales con solo padre desconocido | 10 |
| Animales con solo madre desconocida | 0 |
| Machos con padre y madre desconocidos | 13 |
| Hembras con padre y madre desconocidos | 17 |
| Generaciones máximas | 8 |
| Generaciones completas | 4 |
| Generaciones equivalentes | 5.18 |
Probabilidad de origen genético
En la población de referencia se identificaron 22 animales que fueron identificados como fundadores, mientras que el número efectivo de fundadores para la misma población fue de 13 individuos. El número de ancestros que explica el 100% de la variabilidad genética de la población de referencia es de 18 individuos (fundadores o no), en tanto que solo 3 individuos explicaron el 50% de la variabilidad genética. Respecto a la proporción del número efectivos de ancestros y fundadores en la población de referencia fue 0.54 y por lo tanto inferior a 1, lo que indica la ocurrencia de posibles cuellos de botella en la población (Tabla 2).
Tabla 2 Resumen de parámetros calculados para la probabilidad del origen genético en la población de referencia.
| Parametro | Valor |
|---|---|
| Número total de animales fundadores | 22 |
| Número efectivo de animales fundadores fe | 13 |
| Número de ancestros | 18 |
| Número de ancestros que explican el 50% del pool genético | 3 |
| Número efectivo de ancestros 𝑓𝑎 | 7 |
| Contribución del principal ancestro (%) | 18.2 |
| Relación 𝑓𝑎 𝑓𝑒 | 0.54 |
Parámetros poblacionales relacionados con el coeficiente de consanguinidad
La consanguinidad promedio para todos los animales del pedigrí y la población de referencia fue de 4.66 y 6.73%, respectivamente. De acuerdo con la Figura 2, la consanguinidad promedio por año para todos los animales en el pedigrí del banco de germoplasma ha venido presentado un incremento progresivo en la consanguinidad desde el 2010, siendo el 2016 el año que se presentó la consanguinidad histórica más elevada en la población con un 7.80%.
En la Tabla 3 se observa que la consanguinidad, el número de animales consanguíneos y el parentesco aumentó a lo largo de las generaciones completas, siendo el incremento de la con sanguinidad por generación completa del 2.25%. De igual manera se observa que en la cuarta generación completa todos los animales de la población bajo estudio son consanguíneos. El tamaño efectivo de la población (Ne) fue de 25.31, valor que está por de bajo del tamaño efectivo poblacional mínimo definido por la FAO de 50 individuos como nivel crítico.
Tabla 3 Número de animales por generación (N), coeficiente de consanguinidad promedio (F), porcentaje a animales consanguíneo, coeficiente de consanguinidad para animales consanguíneos y parentesco promedio (A) de la población de cerdos San Pedreño del banco de germoplasma.
| Generaciones completas | No. de animales | F promedio (%) | % de consanguíneos | F promedio para animales consanguíneos (%) | A promedio (%) |
|---|---|---|---|---|---|
| 0 | 40 | 0 | - | - | 4.2 |
| 1 | 41 | 1.2 | 4.9 | 25 | 7.1 |
| 2 | 556 | 2.2 | 21 | 10.6 | 11.2 |
| 3 | 583 | 5 | 87 | 5.8 | 15.6 |
| 4 | 574 | 7.3 | 100 | 7.3 | 17.3 |
Discusión
En el banco de germoplasma de la nación colombiana los nacimientos de cerdos San Pedreño del 2008 al 2017 ha oscilado, debido a que los recursos que se destinan para el mantenimiento de los animales de los bancos de germoplasma se ha reducido, lo que no permite mantener altas poblaciones de animales. En el 2008 el bajo número de nacimientos observados (12 lechones) obedece a que el banco de germoplasma se creó ese año con animales jóvenes, no aptos para la reproducción en su momento. Por otro lado, entre el 2011 y 2017 se presentó el mayor número de nacimientos (288 y 289 lechones, respectivamente), lo que permitió realizar actividades de fomento entre los productores del departamento de Antioquia encaminados a incrementar el número de cerdos criollos San Pedreño en el país.
Con respecto al intervalo generacional, cuando se estimó por las cuatro vías posibles, el intervalo generacional fue mayor para la vía de la madre-hijo(a) que cuando se calculó para la vía padre-hijo(a). El uso del sistema de apareamiento circular en el banco de germoplasma puede ser la causa de estos patrones, debido a que la selección de las hembras de reemplazo tiene lugar después de la selección de los machos, lo que resulta en intervalos generacionales mayores para las hembras que los machos. El intervalo generacional promedio fue de 2.13 años, valor que es menor que lo reportado por Santana et al. (2017) para el cerdo criollo cubano de 2.16 años y los intervalor de 2.43, 2.47 y 2.49 años reportado por Posta et al. (2016) para las razas criollas húngaras de cerdos Mangalica rubio, rojo y vientre de golon drina. Pero al compararlo con razas mejoradas utilizadas en los sistemas de producción porcícolas, el intervalo generacional promedio encontrado para la raza San Pedreño es considerablemente mayor que los valores de 1.60 y 2.07 años reportados por Melka y Schenkel (2010), Welsh et al. (2010) y Tang et al. (2013) para las razas Yorkshire, Duroc, Hampshire y Landrace. Lo que indica que la diferencia presentada en estas razas podría deberse a la mejora en el manejo reproductivo y la menor longevidad en estas razas. El mayor intervalo generacional en los cerdos San Pedreño del banco de germoplasma refleja los problemas relacionados con la reducción drástica en el censo de animales de esta raza en los últimos años, lo que retrasa la sustitución de los animales en la población.
Para el análisis de integridad del pedigrí, de los 1 794 individuos que componen el pedigrí del banco de germoplasma, 10 animales presentaron padre desconocido y los 30 que poseen padre y madre desconocidos en el pedigrí forman parte de los individuos con los que inicio el programa de conservación o los padres fundadores. Para el número de generaciones obtenidas a partir del pedigrí, no se observó una diferencia considerable entre el número de generaciones máximas (8), equivalentes (5.18) y generaciones completas (4), lo cual que indica que no hay pérdida considerable de información genealógica en los individuos del pedigrí (Danchin-Burgue et al. 2012). La falta de información en los datos genealógicos puede hacer que se subestimen los coeficientes de consanguinidad y parentesco en el promedio de la población y en los animales más antiguos.
En la probabilidad del origen genético de la población de referencia (animales nacidos entre 2015 y 2017) la proporción entre el número efectivo de ancestros y fundadores indica como los animales han contribuido al desarrollo de la población; si el número efectivo de fundadores es mayor que el número efectivo de ancestros, es muy posible que se hayan presentado cuellos de botella en la formación de la población (Sorensen et al. 2005). Para los cerdos San Pedreño del banco de germoplasma proporción se tuvo un valor de 0.54, lo que indica que la población se ha visto afectada por cuellos de botella. Valores inferiores fueron encontrados por Posta et al. (2016) para las razas criollas húngaras Mangalica Rubia y Mangalica Rojo para los cuales se reportaron proporciones de ancestros y fundadores de 0.44 y 0.53, respectivamente. Mientras que un mayor desbalance entre el número de ancestros y fundadores fueron observado en el trabajo realizado por Janssens et al. (2005) en poblaciones de las razas Landrace Belga y Pietrain con proporciones de ancestros y fundadores de 0.35 y 0.32, respectivamente.
En lo referente a la consanguinidad promedio de todos los animales del pedigrí (4.66%) y de la población de referencia (6.73%), se ha presentado un incremento progresivo en los últimos años, lo que ha superado el límite recomendado por la FAO del 5% para poblaciones cerradas (FAO 2013). En el análisis de las generaciones completas, se observó que la consanguinidad y el parentesco promedio se incrementan conforme aumenta el número de generaciones completas. El incremento de la consanguinidad por generación completa (2.25%) es superior al 1% recomendado por la (FAO (2013), lo que indica que se puede evitar la depresión por endogamia y por consecuencia la pérdida de la variabilidad genética en la población (Ocampo y Cardona 2013, Bernardes et al. 2016, Sheikhlou y Abbasi 2016).
El Ne estimado para la población de referencia fue de 25.31 individuos. Teniendo en cuenta que el tamaño efectivo poblacional sugerido por la FAO de 50 individuos y lo reportado por Meuwissen quien concluyo que debido a la mutación y la deriva genética el Ne crítico debería estar entre 50 y 100 animales. Por lo que el tamaño efectivo poblacional encontrado en el presente estudio es bajo, lo que representa una situación desfavorable para la población de cerdos del banco de germoplasma e implica la necesidad de prácticas de conservación.
Conclusiones
El análisis del pedigrí es una herramienta de monitoreo del estado de la diversidad genética de las poblaciones. Con la correcta supervisión de los parámetros poblacionales se puede evitar la pérdida de la diversidad genética. Para la población de cerdos San Pedreño del banco de germoplasma, que se maneja como población cerrada, sin entrada de animales ajenos al banco de germoplasma, el análisis de la información indica que la consanguinidad y el tamaño efectivo poblacional no se encuentran dentro de los valores recomendados para poblaciones de animales domésticos. Mientras que el número efectivo de fundadores es mayor que el número efectivo de ancestros, lo que ha derivado en cuellos de botella. Por lo que mantener la diversidad genética a largo plazo dependerá del esfuerzo para incrementar el tamaño efectivo de la población, equilibrar la contribución de los ancestros y evitar el apareamiento de animales emparentados para reducir la consanguinidad, por lo que es recomendado incluir individuos no emparentados de otras poblaciones.











 nova página do texto(beta)
nova página do texto(beta)