Introducción
La maduración del ARNm primario (pre-ARNm) consta de diferentes etapas: la adición de la CAP extremo 5’, la poliadenilación y el splicing[1]. Particularmente, el splicing constitutivo es un proceso co-transcripcional de corte de intrones y empalme de exones. A diferencia del splicing alternativo, que tiene la capacidad de incluir exones, intrones o parte de ellos [2]. Además, se ha observado que el 95% de los genes humanos llevan a cabo este proceso, contribuyendo así a la diversidad proteica[3]. Sin embargo, las alteraciones durante el splicing alternativo se manifiestan en el desarrollo de enfermedades genéticas y cáncer[4]. El cáncer es una problemática de salud pública, debido a los niveles elevados de incidencia y mortalidad en el mundo. En México, el cáncer de mama es la primera causa de muerte por cáncer en mujeres[5], [6]. El cáncer de mama es un tumor maligno originado por el crecimiento desmedido de células mamarias, capaz de migrar e invadir tejidos lejanos[7]. Molecularmente, el splicing alternativo en cáncer de mama, se ha correlacionado con la progresión tumoral, debido a que favorece la angiogénesis, invasión y metástasis, además, altera el ciclo celular e inhibe la apoptosis[8]. Recientemente, se han reportado terapias que restablecen el splicing alternativo mediante el empleo de oligonucleótidos antisentido para el tratamiento de múltiples enfermedades. En este trabajo, analizamos la expresión de isoformas involucradas en la proliferación, invasión, transición epitelio-mesénquima e inhibición de la apoptosis y de actividades supresoras en cáncer de mama. Además, presentamos el uso de oligonucleótidos antisentido como futuro tratamiento contra cáncer de mama.
Splicing alternativo y su mecanismo
El splicing alternativo (SA) es un proceso nuclear que participa en el desarrollo, diferenciación de la célula, regulando la expresión génica[4]. La diversidad proteómica está determinada por los diferentes eventos de SA que surgen como: la retención de intrones, salto de exón, uso de sitio de splicing alternativo 5’, sitio de splicing alternativo 3’ y exones mutuamente excluyentes (Fig.1) [9]
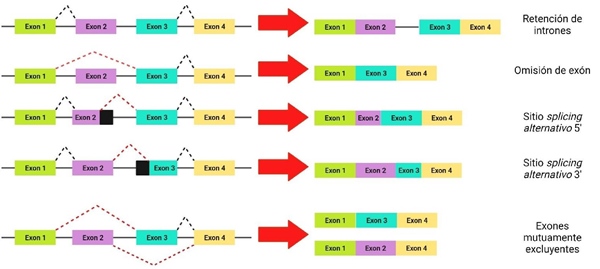
El siguiente esquema muestra los diferentes eventos de splicing alternativo que se presentan en el núcleo celular como: retención de intrones, salto de exón, sitio splicing alternativo 5, sitio splicing alternativo y exones mutuamente excluyentes
Fig 1 Tipos de splicing alternativo en ARNm
Este proceso es catalizado por un complejo multiproteico, llamado spliceosoma. El spliceosoma está conformado por las ribonucleoproteínas nucleares pequeñas (snRNP) U1, U2, U4, U5 y U6. Las snRNP son las encargadas de catalizar la eliminación de intrones y el empalme de exones, reconocen los sitios de splicing 5’ y 3’. El SA es un mecanismo altamente regulado, que involucra a otras proteínas o factores en trans[2]. Estos factores comprenden a las proteínas SR (proteínas ricas repetidos de residuos arginina/ serina) y hnRNP’s (proteínas ribonucleares heterogéneas) ambas son las responsables de activar y/o inhibir el SA, respectivamente[10]. Sin embargo, esto varía según su unión a elementos potenciadores de splicing intrónico (ISE), elementos potenciadores de splicing exónico (ESE), elementos silenciadores de splicing intrónico (ISS) y elementos silenciadores de splicing exónico (ESS), ya que la unión de las proteínas SR a la región ISS y ESS inhiben el SA [2], [3]. No obstante, las afectaciones en estas proteínas reguladoras también pueden alterar la actividad proteica y comprometer la integridad celular, lo que conlleva al desarrollo de diversas enfermedades, especialmente el cáncer de mama.
Cáncer de mama
El cáncer de mama es la neoplasia más frecuente en mujeres, sin embargo, no es exclusiva de este género ya que también se presenta en hombres. Este ha sido un problema de salud mundial ya que presenta una prevalencia del 15.4%[5].
El cáncer de mama es el crecimiento anormal de células epiteliales mamarias que ocurre dentro de los conductos, lóbulos y/o lobulillos mamarios[7], [11]. Curiosamente, la localización anatómica del tumor juega un papel importante, pues se le ha asociado con el grado de invasividad y la supervivencia del paciente; se ha observado que el desarrollo del tumor dentro de los conductos o lóbulos mamarios limita el crecimiento celular, aumenta la supervivencia del paciente y reduce la reincidencia del cáncer; en cambio, las células tumorales que invaden la región ducto-lobulillar hacia el estroma, promueven la invasividad[11]. Existen cinco subtipos de cáncer de mama, los cuales se clasifican conforme a la expresión positiva o negativa del receptor de estrógeno (ER), receptor de progesterona (PR) y receptor del factor de crecimiento epidérmico (HER2)[1], [2], clasificándose en: 1) luminal A (ER+, PR+, HER2-), 2) luminal B (ER+, PR-, HER2- o ER+, PR+ / PR-, HER2+), 3)HER2 positivo (ER-, PR-, HER+), 4) TNBC o triple negativo (ER-, PR-, HER2-) y 5) tumores de tipo normal[1], [2], [11]. Y cada uno tiene distintos pronósticos y respuestas a terapias[1], [2].
Desregulación del splicing alternativo en genes asociados a cáncer de mama
A pesar de la amplia regulación del SA, este puede verse gravemente afectado por mutaciones génicas, desarrollando isoformas oncogénicas e inhibidoras de la supresión de tumores. No obstante, el descubrimiento de estas isoformas ayuda en la detección de nuevos marcadores contra cáncer de mama y el descubrimiento de nuevas dianas terapéuticas.
Receptor de estrógenos (ER)
En cáncer de mama, ERα es un gen que expresa tres isoformas; ERα66 (longitud larga), ERα36 y ERα46 (Fig.2)[12]. Cada una de ellas se distingue por la presencia de los dominios AF1 y AF2, encargados de regular la transcripción de ER[2], [13], [14]. Curiosamente, ERα46 y ERα66 presentan secuencias de aminoácidos similares que van desde la posición 174 a la 595[2]. Además, la expresión esta inversamente correlacionada, es decir, cuando ERα66 se expresa poco, ERα46 se sobreexpresa[12]. Sin embargo, existen particularidades que las diferencian, ya que ERα46 posee dos veces mayor afinidad al ligando, participando en la oncogénesis y resistencia a los fármacos[2], [13].
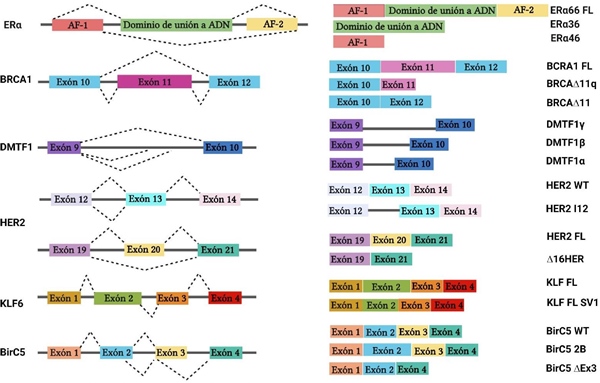
El esquema representa múltiples eventos de splicing alternativo que generan isoformas oncogénicas. De lado izquierdo, se representa el pre-ARNm que será procesado. De lado derecho, se muestra las isoformas resultantes. Las líneas oscuras simbolizan intrones y los rectángulos esquematizan exones. Las líneas punteadas indican la unión de los exones. FL: Isoforma de larga longitud. WT: Wild Type
Fig. 2 Eventos de splicing alternativo involucrados en el desarrollo de cáncer de mama.
Cáncer de mama tipo 1 (BRCA1)
BRCA1 es un gen supresor de tumores, localizado en el núcleo y encargado de reparar el ADN por medio de recombinación homologa[2], [15]. BRCA1 expresa las isoformas BRCA1 FL (longitud larga), BRCA∆11 y BRCA∆11q, que se caracterizan por la inclusión/exclusión del exón 11, lo cual determina la localización de BRCA1(Fig.2 )[15]. En diferentes tumores de cáncer de mama, se ha observado una sobreexpresión de BRCA∆11q y una reducción de BRCA1 FL[16].
DMTF1
DMTF1 es un supresor tumoral activado por la vía RAS/ERBB2[17]. La presencia de la región homologa myb (MHR) distingue las variantes DMTF1α, DMTF1β y DMTF1γ. DMTF1α es la isoforma larga que posee actividad supresora de tumores[18]. DMTF1β y DMTF1γ son las variantes más cortas, ya que presentan una reducción de MHR, que afecta la unión al ADN[15]. En cáncer de mama, el SA de DMTF1 se produce en el 30% de los casos, donde se observa a DMTF1α disminuida y DMTF1β aumentada. Por medio de secuenciación de ARNm se ha reportado que DMTF1β aumenta su expresión entre 43%-55%[15], [17]. Además, la sobreexpresión de DMTF1β y DMTF1γ se ha asociado con la reducción de la tasa de supervivencia a largo y corto plazo[18].
HER2
HER2 es un oncogén que pertenece a la familia de receptores tirosina cinasa[15], [19]. La sobreexpresión del receptor lo clasifica como subtipo HER2+, que representa del 15-20% de los casos diagnosticados en cáncer de mama[2], [19]. Además, la sobreexpresión de HER2 es del ~30% en cáncer de mama primario[13], [15]. Generalmente, HER2 expresa la variante ∆16HER2, que carece del exón 20, predominante en cáncer de mama metastásico y asociada a la resistencia a fármacos (Fig.2)[2], [15]. Se ha observado que la co-expresión de HER2 larga (FL) y ∆16HER2 incrementa la heterogeneidad del cáncer, por lo que altera su biología, curso clínico y respuesta al tratamiento[2]. Estudios recientes, reportan la sobreexpresión de la isoforma HER2-I12 que contribuye en la tumorigénesis al mejorar la proliferación, migración e invasión celular, además que activa las vías de señalización RAS/MAPK y PI3K/Akt.
Factor 6 tipo Kruppel (KLF6)
KLF6 es una proteína supresora de tumores, que posee dominios de unión al ADN, llamados “dedos de zinc”[20], [21]. En tejidos normales, KLF6 presenta cuatro exones que constituyen la isoforma larga (KLF6 FL). En cáncer, se expresan las isoformas: KLF6-SV1, KLF6-SV2 y KLF6-SV3 que participan en la oncogénesis, progresión del tumor y evasión de la apoptosis[2], [15], [21]. La importancia de estas isoformas radica en la presencia y/o ausencia del dominio de localización nuclear (NLS), pues se ha observado que KLF6- SV1 permanece en el citoplasma debido a la pérdida del exón 2 durante el SA[2]. Cabe resaltar que KLF6-SV1 es la isoforma mayor predominante en cáncer de mama, que antagoniza la función de KLF6-FL, promueve la progresión tumoral, contribuye en la transición epitelio-mesénquima y se asocia con un mal pronóstico (Fig.2 )[15], [16], [20].
Survivina (BIRC5)
BIRC5 codifica para la proteína multifuncional survivina, implicada en el control de la mitosis, respuesta al estrés celular y regulación de la apoptosis[15], [22]. Generalmente, la survivina se expresa durante el desarrollo fetal y no en tejidos diferenciados y sanos. En cáncer de mama, la sobreexpresión de la proteína está involucrada al inhibir la apoptosis, promover la angiogénesis y proliferación celular[15], [23]. BIRC5 tiene seis isoformas: survivina WT (Wild Type), 2α, 2B, 3α, 3B y ∆ex3 (Fig.3) [15]. Particularmente, la survivina ∆ex3 muestra una sobreexpresión en tumores metastásicos y/o estadio IV, por lo que se relaciona con una enfermedad agresiva y pronostico desfavorable en cáncer de mama[24], [25]. Además, se ha encontrado que la proteína Sam68 participa en la exclusión del exón 3, al actuar como hnRNP, unirse al pre-ARNm e impedir la unión de los factores de splicing, de esta manera contribuye en la formación de ∆ex3 y podría utilizarse como una excelente diana terapéutica [8]. Por otra parte, la survivina 2B es la isoforma mayor expresada en tumores primarios de mama, que responden con éxito al tratamiento, por lo que lo posiciona como un excelente biomarcador(Fig. 2 y 3) [8], [24], [25].

La siguiente representación gráfica muestra la composición e isoformas de la proteína survivina A) BirC5 codifica para la proteína survivina, que se caracteriza por la presencia de un dominio BIR y una hélice superenrollada B) El splicing alternativo que sufren los pre-ARNm de la survivina, generan seis isoformas. De lado izquierdo, se esquematiza la región pre-ARNm que será procesada. De lado derecho, se observa el tamaño y función de la proteína resultante.
Fig 3 Isoformas de la proteína survivina
Oligonucleótidos antisentido (ASO´S) como tratamiento contra cáncer de mama
Durante mucho tiempo, el tratamiento de elección contra cáncer ha sido la radioterapia, quimioterapia y los tratamientos hormonales, sin embargo, su uso compromete la integridad del paciente. Ante estos acontecimientos, los investigadores han buscado terapias alternas que mejoren la efectividad del tratamiento y disminuyan el sufrimiento del paciente.
Los oligonucleótidos antisentido (ASO’s) son secuencias cortas específicas de 15 a 30 nucleótidos que se unen al ARNm para formar híbridos bicatenarios[1]. La hibridación de los oligonucleótidos inhibe la expresión génica, mediante bloqueo estérico o por degradación del hibrido a causa de la RNasa H[26], [27]. Estos han sido modificados químicamente en la cadena principal y los anillos de azúcar, que incluyen modificaciones fosforotiato (PS), fosforodiamidato morfolino(PMO) 2’-O-metoxietilo (MOE) y 2’-O-metilo (OMe), dando mejor resultado 2’-O-metilo y 2’’-O-metoxietilo al proporcionar una mayor especificidad, afinidad al ARNm diana y resistencia a la degradación por endonucleasas [26], [28], [29]. Por lo tanto, estas modificaciones son una excelente opción para el recubrimiento de ASO’s utilizados contra cáncer. Además, se ha observado la participación de los ASO’s en la modificación de eventos de SA, que podrían reestablecer el procesamiento de isoformas antitumorales. En este caso, los oligonucleótidos interruptores de splicing (SSO) son secuencias cortas, que compiten contra los factores de splicing por la unión a elementos reguladores, por lo que contribuye en la activación o inhibición del SA (Fig4.)[1], [29]. Los oligonucleótidos dirigidos a potenciadores de splicing (TOES) son elementos que dirigidos a proteínas SR y promueven la inclusión de exones. En diferentes estudios, los TOES han sido utilizados para promover la inclusión del exón 7 de SMN2 en pacientes con Atrofia Muscular Espinal (AME)[29]. Los potenciadores de splicing exón-especifico por efectores quiméricos pequeños (ESSENCE) son moléculas que se unen al dominio serina/arginina, de las proteínas SR, en el extremo 3’ para favorecer la inclusión de exones[1]. Además, se ha demostrado la eficacia de estas moléculas al redirigir el SA de SMN2 en AME, el exón 18 y Bcl-x en BRCA1[1], [29]. En cambio, los oligonucleótidos dirigidos a silenciadores de splicing (TOSS) son elementos que reconocen proteínas hnRNP A1/A2, que afectan la unión de U1 al pre-ARNm e inhibe el splicing alternativo. Cabe resaltar que los TOSS ya ha sido probados y dirigidos a hnRNP A1, reprimiendo la exclusión del exón 8 y favoreciendo la inclusión del exón 7 de SMN2 en AME[30].
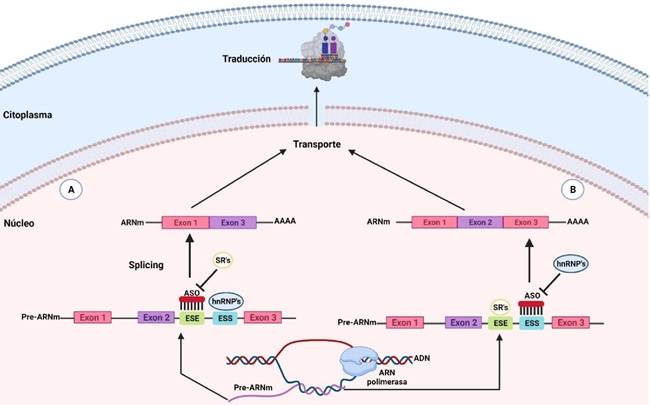
a) El oligonucleótido antisentido se une a elementos silenciadores de splicing que impiden la inclusión del exón 2. b) El oligonucleótico antisentido se une a elementos potenciadores del splicing, que favorecen la inclusión del exón 2. Posteriormente, el ARNm es transportado hacia el citoplasma para ser traducido a proteína en el ribosoma.
Fig 4 Mecanismo de acción de los oligonucleótidos antisentido en splicing alternativo
Los SSO también han sido probados en diversos genes asociados a cáncer. En glioblastoma, los SSO favorece la inclusión del exón 14a en Mnk, lo que conlleva al aumento de la expresión Mnk2a (supresor de tumores) y disminución de la isoforma Mnk2b (pro-oncogénica)[31]. Por otra parte, el diseño de TOSS, dirigido a hnRNP A1, muestra una reducción de la isoforma Bcl-xL (Longitud completa) y aumento de Bcl-xS (longitud corta) en líneas celulares de cáncer de próstata, colon y mama, de manera que favorece la apoptosis[30]. Además, OT-101 es un ASO, diseñado para inhibir el ARNm de TGF-β2, que mostró una mejoría en la supervivencia general de los pacientes con cáncer de páncreas[32]. En este sentido, la unión de ASO’s a elementos reguladores restablecería el SA y disminuiría la expresión de oncogenes en cáncer de mama.
Conclusión
La desregulación del splicing alternativo en cáncer ha sido evidenciado en diferentes estudios. Ejemplo de esto, son las diferentes isoformas de los genes BirC5 y KLF6 que favorecen la oncogénesis en cáncer de mama. Afortunadamente, el desarrollo de ASO’s brinda una nueva y mejor opción como terapia antitumoral. Los estudios de protección de los ASO´s sugiere que la modificación 2-O-ME es una excelente opción, ya que evitan la degradación por nucleasas y favorece la vida media del fármaco. Por último, las evidencias apuntan que los ASO’s serían el tratamiento ideal contra cáncer de mama, ya que tienen la capacidad de restablecer el splicing alternativo y han demostrado ser específicos, efectivos, no deterioran la salud y mejoran la supervivencia del paciente.











 nueva página del texto (beta)
nueva página del texto (beta)



