Introducción
El síndrome de transfusión gemelo a gemelo (the twin-twin transfusion síndrome o TTTS por sus siglas en inglés) es el resultado de un flujo sanguíneo desigual a través de anastomosis (que es la conexión de los vasos sanguíneos) vasculares placentarias que enlazan las dos circulaciones fetales. Estas anastomosis vasculares en la placenta compartida están presentes en prácticamente todos los embarazos gemelares monocoriónicos (monochorionic twins o MC por sus siglas en inglés), pero solo en aproximadamente 10 % conducen al síndrome de transfusión gemelo a gemelo.
La transfusión neta de sangre sucede a expensas del llamado gemelo donante, el cual se vuelve hipovolémico y anémico, mientras que el gemelo receptor se vuelve hipervolémico y policitémico. Esto conduce al rápido desarrollo de una discordancia significativa en el volumen de líquido amniótico entre los gemelos que sin la debida intervención puede ser fatal para ambos gemelos (Spruijt, Lopriore, Steggerda, Slaghekke y van Klink, 2019).
Actualmente, el síndrome de transfusión gemelo a gemelo es una de las circunstancias más mortales en medicina fetal, y sigue siendo un gran reto para obstetras y neonatólogos de todo el mundo. En tal sentido, el mejor tratamiento lo representa la cirugía con láser fetoscópico. La técnica, actualmente vigente, utiliza dicho láser para interrumpir el flujo sanguíneo a través de las comunicaciones vasculares con el fin de coagular solo los vasos anastomóticos en el ecuador vascular que está a cierta distancia de la membrana entrelazada debido a los volúmenes discordantes de líquido amniótico entre los gemelos (Glennon, Shemer, Palma-Dias y Umstad, 2016).
La cirugía láser para síndrome de transfusión gemelo a gemelo se puede dividir en dos pasos fundamentales: 1) identificación endoscópica de las anastomosis vasculares placentarias, y 2) ablación con láser de las anastomosis. Por lo tanto, el objetivo de la cirugía con láser es identificar y extirpar correctamente todas las anastomosis vasculares. Esto plantea dos cuestiones: 1) ¿se pueden identificar todas las anastomosis vasculares placentarias?, y 2) ¿se pueden extirpar todas las anastomosis vasculares placentarias? (Quintero, Kontopoulos y Chmait, 2016).
Ahora bien, centrándose en la detección endoscópica de las anastomosis vasculares placentarias, surge la siguiente pregunta: ¿cómo se pueden identificar las anastomosis? Algunos autores sugirieron que las anastomosis podrían distinguirse según su apariencia, es decir, en función de patrones específicos, ángulos, con dibujos destinados a ayudar en este proceso, esto es, la identificación basada en el reconocimiento de patrones. Desafortunadamente, esto no fue posible para los médicos debido a la gran cantidad de patrones que las anastomosis pueden tener (De Lia, Kuhlmann, Cruikshank y O'Bee, 1993).
De lo anterior surgen las siguientes preguntas de investigación: ¿puede una herramienta computacional ayudar en la identificación de las anastomosis dentro de un video fetoscópico?, y ¿qué tan exacta puede ser dicha identificación? El presente trabajo plantea como hipótesis que una técnica computacional puede identificar las anastomosis con cierto porcentaje de exactitud.
Distinguir las anastomosis para realizar una adecuada intervención en la cirugía láser para síndrome de transfusión gemelo a gemelo constituye una problemática para el médico con menos experiencia, por lo que, en aras de atender esta necesidad, se plantean los siguientes objetivos: demostrar que una herramienta computacional puede ayudar en la identificación de anastomosis en un video fetoscópico con cierto porcentaje de exactitud, y crear una herramienta computacional que ayude en la clasificación automática de anastomosis, de manera que sirva al médico novel como herramienta de apoyo en su etapa de entrenamiento y habilitación.
La ciencia de solucionar problemas clínicos, a través del análisis de imágenes generadas en la práctica clínica, se conoce como análisis de imágenes médicas, y su objetivo es extraer información de manera eficiente para mejorar el diagnóstico clínico. En el presente artículo se pone de manifiesto este avance, el cual consiste en la aplicación de técnicas de aprendizaje automático para el análisis de imágenes médicas. Presenta, en la primera sección, una descripción del desarrollo del trabajo de investigación realizado con varios apartados que dan claridad sobre el proceso seguido en aras de alcanzar los objetivos previstos, destacando la manera en que el aprendizaje profundo se utiliza con éxito como una herramienta para el aprendizaje automático, donde una red neuronal es capaz de aprender características automáticamente. De las técnicas de aprendizaje profundo, las redes convolucionales profundas se usan activamente para el análisis de imágenes médicas. Esto incluye áreas de aplicación como segmentación, detección de anormalidades, clasificación de enfermedades, diagnóstico asistido por computadora y recuperación (Anwar et al., 2018). Pero ¿a qué se hace referencia cuando se habla de redes neuronales convolucionales?
Una red neuronal convolucional es un tipo de modelo de aprendizaje profundo para procesar datos que tiene un modelo de cuadrícula -como las imágenes- inspirado en la organización de la corteza visual animal y diseñado para aprender de forma automática y adaptativa jerarquías espaciales de características, desde niveles de patrones bajos hasta de alto nivel. Una neuronal convolucional es una construcción matemática que generalmente se compone de tres tipos de capas (o bloques de construcción): convolución, agrupación y capas completamente conectadas. Las dos primeras realizan extracción de características, mientras que la tercera -una capa totalmente conectada- asigna las características extraídas a la salida final, como la clasificación.
Una capa de convolución juega un papel clave en la neuronal convolucional, que se compone de una pila de operaciones matemáticas, como la convolución, un tipo especializado de operación lineal. En las imágenes digitales, los valores de píxeles se almacenan en una cuadrícula bidimensional (dos dimensiones), es decir, una matriz de números, y se aplica una pequeña cuadrícula de parámetros llamada kernel, un extractor de características optimizable, en cada posición de imagen, lo que hace que las neuronales convolucionales sean altamente eficientes para el procesamiento de imágenes, ya que puede aparecer una característica en cualquier lugar de la imagen. A medida que una capa alimenta su salida a la siguiente capa, las entidades extraídas pueden volverse jerárquicas y progresivamente más complejas. El proceso de optimización de parámetros -como los núcleos- se denomina capacitación, que se realiza para minimizar la diferencia entre las salidas y las etiquetas de verdad del terreno a través de un algoritmo de optimización llamado propagación hacia atrás y descenso de gradiente, entre otros (Yamashita, Nishio, Do y Togashi, 2018).
Método
En la presente sección se explica la metodología que se siguió para proponer una forma de resolver la problemática presentada, desde la recolección de los datos de entrada, su tratamiento y transformación hasta la propuesta de una herramienta que ayude a su resolución; en la Figura 1 se puede visualizar en forma gráfica la metodología seguida.
Creación de la fuente de datos
En el contexto de este trabajo, el proceso general del origen de datos proporcionados fueron videos en formato MPG, grabados durante la intervención quirúrgica a pacientes reales cuando se les aplicó la cirugía fetal; los videos fueron extraídos directamente del fetoscópio por el médico a cargo de la intervención e incluyen los siguientes datos: fecha, tratamiento, cirujano, ID del paciente, fecha de nacimiento y sexo, en un archivo XML o HTML. Son generalmente N videos con una estructura de nombre Video_001.mpg, …Video_00N.mpg para cada paciente; para esta investigación se tuvo acceso a N videos de 12 pacientes.
Posteriormente, se creó un programa para unir todos los videos separados de cada paciente (de dicho proceso se obtuvieron 12 videos completos), de ese modo se pudo tenerlos en formato MP4 para su posterior uso de clasificación de anastomosis con la herramienta Videolabel por el médico experto.
Aunado a esto, de los 12 videos completos se extrajeron los frames de cada uno y se renombraron con una nomenclatura adecuada para tener un dataset de imágenes sin clasificar. La nomenclatura fue la siguiente:
F = Frame.
P1, P2, P3… PN = Paciente.
0000000001 = Número consecutivo.
Ejemplo:
F-P1-0000000001.png
Para concluir, en esta fase se elaboró un programa en MatLab, donde se revisó todo el directorio que contiene imágenes y se las recortó de manera automática con la finalidad de dejar solo la imagen en sí y eliminar lo sobrante. Esta actividad se realizó porque se trabaja con miles de imágenes, las cuales tendrán que ser procesadas por la red neuronal convolucional. Esta tarea de eliminación de segmentos permite ahorrar tiempo, y arroja como resultado un DataSet de imágenes sin clasificar.
Etiquetado manual
Para el etiquetado del DataSet de imágenes se desarrolló un software que ayuda a clasificar las imágenes (llamado VideoLabel). Este software clasificador fue usado por los expertos médicos con conocimientos del tema; su funcionamiento, en forma general, consiste en leer un video ya procesado. El software permite a un médico experto -en caso de identificar alguna anastomosis- pausar el video, marcar el elemento y guardar el tipo de anastomosis identificada mientras internamente el programa guarda dicha imagen clasificada. Las clases de anastomosis que ya tiene cargado el programa VideoLabel son las siguientes: normal, arterio-venosa, veno-arterial, arterio-arterial, veno-venosa, borde placentario, membrana interfetal, inserción del cordón del gemelo donante, inserción del cordón del gemelo receptor, capilar.
La salida del programa VideoLabel es el almacenamiento de las imágenes clasificadas en una carpeta que conforman el DataSet de imágenes etiquetadas por el experto. En este trabajo de investigación se trabajó con las categorías arterio-venosa, capilar y veno-arterial; en la Figura 2 se pueden visualizar los tres tipos de anastomosis con los que se trabajó:
Cabe resaltar que no se presentan todos los tipos de anastomosis en las intervenciones; de igual manera, es complicado reunir un DataSet de imágenes numeroso, ya que el tiempo dedicado al etiquetado manual por el médico experto es escaso. En la siguiente sección se especifican las cantidades de imágenes reunidas y etiquetadas por el experto.
Aumento de datos
En general, las redes neuronales convolucionales son demasiado profundas como para entrenarlas desde cero con conjuntos de datos pequeños (se necesitan al menos mil imágenes clasificadas por categoría) para entrenamiento desde cero. Para eso, se recomiendan las técnicas de transferencia de aprendizaje y aumento de datos; en ese sentido, la técnica de aumento de datos reside en incrementar artificialmente el grupo de imágenes de entrenamiento empleando diversos reajustes a las imágenes originales, como modificar el brillo, escalar, acercar, girar, reflejar vertical y horizontalmente, etc. Las imágenes obtenidas deben ser lo más aceptablemente reales como para que un espectador externo no pueda distinguir entre una imagen creada por aumento de datos y una original (Gómez-Ros et al., 2019).
Aunque existen diversas estrategias para aumento de datos, las anteriores se han explorado solo con imágenes naturales, y no con imágenes médicas. Entre las estrategias para aumento de datos en imágenes médicas se recomiendan las siguientes, ya que fueron las que dieron mejores resultados, pues incrementaron el porcentaje de efectividad en el entrenamiento de redes neuronales convolucionales: flips (dar la vuelta), rotate (rotación), gaussian blur (difuminado gaussiano) (Hussain, Gimenez, Yi y Rubin, 2018).
En esta investigación se crearon 2320 imágenes artificiales para aumentar el número de imágenes totales por categoría; en la Tabla 1 se muestra el conjunto de las imágenes reales y artificiales que forman el DataSet.
Transferencia de aprendizaje
La transferencia de aprendizaje es una estrategia común y efectiva para entrenar una red con un conjunto pequeño de datos, donde hay una red preentrenada en un conjunto de datos extremadamente grande, como AlexNet, que contiene 1.2 millones de imágenes con 1000 clases, que luego se reutiliza y aplica a la tarea de interés dada. La suposición subyacente en la transferencia de aprendizaje es que las características genéricas (es decir, lo aprendido en un conjunto de datos lo suficientemente grande) se pueden compartir entre conjuntos de datos aparentemente dispares. Esta portabilidad de características genéricas aprendidas es una ventaja única del aprendizaje profundo que es útil en varias tareas de dominio con pequeños conjuntos de datos (Yamashita et al., 2018). De igual manera, se realizó transferencia de aprendizaje de la red AlexNet preentrenada, donde se ajustaron las últimas capas (esto se explica en siguientes secciones de este artículo).
Arquitectura de la red neuronal convolucional
Desde hace algunos años, se han expuesto varios modelos de redes neuronales convolucionales profundas, como AlexNet, ZFNet, VGG, GoogleNet, Residual Net, DenseNet, FCN y U-Net. Dichos modelos no solo han proporcionado un rendimiento de vanguardia para diversas tareas como la clasificación de imágenes, la segmentación, la detección de objetos y las tareas de seguimiento, sino que también han ofrecido un nuevo punto de vista para la fusión de imágenes (Zhou, Ruan y Canu, 2019).
Para este trabajo de investigación se ha usado la red preentrenada AlexNet -como se sugiere en Tajbakhsh et al. (2016)-, ya que es considerada como adecuada para realizar transferencia de aprendizaje en imágenes médicas. Además, como los conjuntos de datos siguen siendo demasiado pequeños, en lugar de ajustar todos los pesos de las capas, se han añadido dos capas completamente conectadas al final de las capas anteriores, para lo cual se agregó una capa a la arquitectura de la red neuronal convolucional. Esta capa se llama fully connected layer (23 en el listado de capas para AlexNet), la cual se encarga de clasificar los datos en varias clases donde las capas anteriores a ella (de la 1 a la 22) realizan la tarea de extraer las características de las imágenes.
En lo que respecta a la classification output layer (25 en el listado de capas de AlexNet), son las encargadas de calcular la pérdida de entropía cruzada para problemas de clasificación de múltiples clases con clases mutuamente excluyentes. La capa infiere el número de clases del tamaño de salida de la capa anterior.
Cabe señalar que la transferencia de aprendizaje ayuda a entrenar a las dos capas que se hayan modificado según las necesidades, y no a toda la red completa; en la Figura 3 se puede visualizar la arquitectura de capas final de la red neuronal convolucional.
Entrenamiento de la red neuronal convolucional
Para la realización tanto del entrenamiento de la red neuronal convolucional como de las pruebas en general, se presentan las características técnicas del equipo de cómputo usado: procesador Intel Core i5 7200U a 2.5Ghz, memoria RAM de 8 Gb, sistema operativo Windows 10 de 64 bits, y tarjeta gráfica Nvidia GeForce 940mx 2 Gb de RAM.
El total de imágenes (entre reales y artificiales) para entrenar a la red neuronal convolucional fue de 2465. Al respecto, es importante resaltar que para probar la red una vez entrenada se emplearon 23 imágenes inéditas (es decir, la red no las conocía).
A continuación, se enumeran las actividades seguidas para proceder al entrenamiento de una red neuronal convolucional:
Inicializar todos los parámetros o pesos con valores aleatorios de las primeras capas de la red.
Utilizar un grupo de imágenes de entrenamiento y usarlas en el modelo.
Computar el total de error de las probabilidades consecuencia del modelo.
Extender hacia atrás para computar el error de gradiente de todos los pesos en la red y emplear el gradiente descendiente para modificar dichos valores y disminuir el error de salida (Quintero, Merchán, Cornejo y Sánchez Galán, 2018).
Un parámetro importante se refiere al número de épocas que tendrá la red neuronal convolucional; una época se refiere a una sola pasada del conjunto de entrenamiento completo (Schilling, 2016). Conocer el número de épocas para entrenar a la red neuronal convolucional en imágenes médicas es un factor importante para tener un parámetro de decisión; algunos autores recomiendan 50 épocas (Kumar, Kim, Lyndon, Fulham y Feng, 2017; Petscharnig y Schöffmann, 2018).
La Figura 4 muestra la red neuronal convolucional realizando el entrenamiento para un máximo de 50 épocas. Como resultado se obtuvo un porcentaje de exactitud de 100 % y el tiempo empleado por el programa fue de 2 horas y 21 minutos aproximadamente.
Entrenamiento de un clasificador
Catalogar un objeto consiste en designarlo a una de las clases disponibles. Los objetos se pueden determinar por una lista de particularidades, como la textura, el tamaño o el color de sus pixeles; para lograr catalogar objetos es necesario precisar los límites entre las diferentes clases. Normalmente estos límites se computan mediante un tratamiento de entrenamiento en el que se usan las particularidades de una serie de modelos de ejemplo de las clases. Se mencionan los límites por claridad; en general, un clasificador deduce unos patrones de decisión durante el entrenamiento.
De eso se desprende que catalogar un objeto desconocido consiste en designarlo a la clase en la cual las particularidades usadas durante el entrenamiento tienen más equivalencia con las particularidades del objeto (Garrido Satué, 2013).
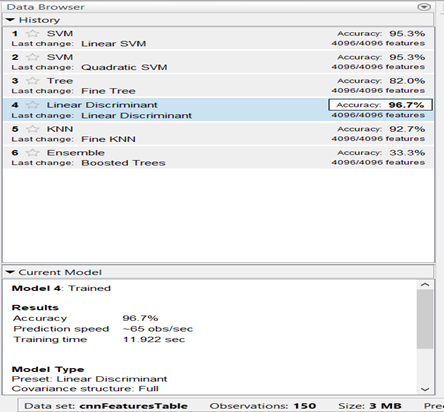
Una vez que se ha terminado el entrenamiento de la red neuronal convolucional, al resultado de dicho entrenamiento se le aplica el entrenamiento de un clasificador, ya que se pueden explorar los datos, seleccionar particularidades, especificar esquemas de validación cruzada, entrenar modelos y evaluar resultados. Es probable realizar entrenamiento automático para averiguar el tipo preferible de modelo clasificador (como árboles de decisión, análisis discriminante, máquinas de vector soporte, regresión logística, vecinos próximos, clasificación ensamblada). La Figura 5 muestra el clasificador seleccionado con los datos de diversos clasificadores entrenados y cuál obtiene el mejor modelo de clasificador para los datos presentados.
Funcionamiento del prototipo
Se desarrolló un prototipo para probar el funcionamiento de la red neuronal convolucional, lo cual se hizo pensando en un usuario final que no tuviera muchos conocimientos informáticos. La pantalla cuenta con tres botones de acción: examinar, pausar y salir. En la Figura 6 se puede ver una pantalla del prototipo funcionando en la clasificación de anastomosis.
La fuente de datos debe ser una carpeta que contenga el video de alguna intervención quirúrgica en formato MP4; posteriormente, el prototipo reproducirá el video de forma automática mientras la red neuronal convolucional selecciona cada frame o fotograma, lo analiza, y al encontrar alguna coincidencia con las categorías entrenadas las marcará con un color (verde para arterio-venosa, amarillo para capilar y azul para veno-arterial); al final guardará dicho frame en el dispositivo. La nomenclatura con la que se guarda la imagen es la siguiente, según las categorías seleccionadas:
AV-fecha-hora.png para la categoría arterio-venosa.
C-fecha-hora.png para la categoría capilar.
VA-fecha-hora.png para la categoría veno-arterial.
La salida del prototipo es un conjunto de imágenes clasificadas y etiquetadas por la red neuronal convolucional, de acuerdo con las tres categorías previamente mencionadas. Con el uso del prototipo, el médico especialista pueda tener resultados para validarlos.
Resultados
La primicia principal de este trabajo de investigación era determinar si una herramienta computacional podría ayudar a identificar las anastomosis dentro de un video fetoscópico y qué tan exacta sería dicha identificación. En tal sentido, durante el desarrollo se obtuvieron algunos resultados del trabajo realizado. Por ejemplo:
Se obtuvieron 12 videos unidos completos de las intervenciones quirúrgicas con más de ocho horas de video fetoscópico.
De los 12 videos se obtuvo un DataSet de 111 273 imágenes sin clasificar.
Producto del etiquetado manual por parte del experto médico, se obtuvieron las imágenes clasificadas por categorías mostradas en la Tabla 1.
Una parte importante de los resultados permite sostener que la solución propuesta funciona adecuadamente para lo que fue diseñada, por lo que se consideró esencial aplicar técnicas como la matriz de confusión, la curva ROC y el AUC para indicar si dicha metodología es aceptable.
En tal sentido, y de manera general, se puede mencionar que una matriz de confusión es una herramienta que se usa comúnmente para delinear el rendimiento de un sistema de clasificación en un cúmulo de datos en el que se conocen como los valores verídicos. La matriz abarca información de las clasificaciones reales y las predichas elaboradas por el sistema, y se emplea para computar el desempeño del algoritmo. Otra forma de examinar los rendimientos de los clasificadores son los gráficos ROC (receiver operating characteristics), método para visualizar, organizar y seleccionar los clasificadores fundamentado en su rendimiento, donde la curva ROC contiene toda la información de la matriz de confusión (Fos Guarinos, 2016). Asimismo, el área bajo la curva AUC (area under curve) es el estadístico por excelencia para medir la capacidad discriminante de la prueba y para comparar pruebas entre sí y determinar cuál es la más eficaz (Del Valle Benavides, 2017).
En la Figura 7 se puede ver la matriz de confusión resultante de la experimentación llevada a cabo con tres clasificaciones distintas en la red neuronal convolucional después de entrenar la red. Véase que para la clasificación arterio-venosa se tiene 94 % de clasificación de verdaderos positivos y 6 % de falsos negativos en el conjunto de valores de prueba proporcionados a la red neuronal convolucional. Para la clasificación capilar se tiene 96 % de clasificación de verdaderos positivos y 4 % de falsos negativos en el conjunto de valores de prueba suministrados a la red neuronal convolucional. Finalmente, para la clasificación veno-arterial se tiene 100 % de clasificación de verdaderos positivos y 0 % de falsos negativos en el conjunto de valores de prueba proporcionados a la red neuronal convolucional.
En cuanto a la curva ROC, se alcanzaron los siguientes resultados para cada una de las tres clasificaciones (la Tabla 2 muestra la AUC que se obtuvo por cada categoría).
Tabla 2 Resultados de AUC por clasificación
| CLASIFICACION | AUC |
| Arterio-venosa | 0.97 |
| Capilar | 0.97 |
| Veno-arterial | 0.98 |
Fuente: Elaboración propia
Un resultado de la ROC y su AUC cuyo valor esté entre 0.5 y 1 indica que la predicción del clasificador es correcta (si tiende a 1 es casi perfecta); por el contrario, un resultado por debajo de 0.5 indica que existen inconsistencias en la clasificación de los datos. En la Figura 8 se muestra cada una de las categorías con su curva ROC y su AUC.
Discusión
Como se mencionó en los resultados, se lograron porcentajes de efectividad superiores a 90 % en general para las tres categorías, con una tasa de imágenes reales de cerca de 150. Una limitación y área de debilidad de la investigación fue el relativo bajo número de imágenes reales que se solventó con la técnica del aumento de datos; pero ¿qué pasaría si el número de imágenes reales aumentara? Aventurando una hipótesis, se puede suponer un incremento en la tasa de efectividad.
No obstante lo anterior, la gran fortaleza de la investigación fueron los porcentajes de efectividad obtenidos teóricamente por las técnicas mencionadas, las cuales se pudieron demostrar con el prototipo de software creado. En consecuencia, se puede afirmar que se logró conseguir el objetivo planteado, es decir, desarrollar una herramienta computacional para contribuir a la clasificación automática de anastomosis dentro de un video fetoscópico mediante redes neuronales convolucionales, las cuales sirven de soporte en la etapa de entrenamiento del médico novel.
Conclusiones
En el presente trabajo se explicó la creación de una herramienta computacional para ayudar al médico novel en la identificación de anastomosis dentro de un video fetoscópico, lo cual forma parte de su preparación para realizar una adecuada intervención con cirugía láser para el síndrome de transfusión gemelo a gemelo.
En este sentido, cabe indicar que la creación de un prototipo que lee y reproduce videos fetoscópicos contiene dentro de sí una red neuronal convolucional debidamente entrenada con tres categorías diferentes de imágenes de anastomosis, lo cual permite clasificarlas con un porcentaje de efectividad superior a 90 % mientras se reproduce el video. Sin embargo, vale apuntar que la capacidad de fluidez del prototipo está ligada directamente al equipo de cómputo usado, por lo que se sugiere uno con altas prestaciones (p. ej., con disco duro de estado sólido de 500 Gb como mínimo, al menos 8 Gb de memoria RAM y un sistema operativo de 64 bits).
Como producto adicional, se lograron obtener dos DataSets de imágenes: uno sin clasificar y otro debidamente clasificado por el médico o experto, que fue la base con la que se entrenó a la red neuronal convolucional empleada en la herramienta generada. Por eso, se puede asegurar que el prototipo creado será de gran ayuda para que el personal médico novel en proceso de entrenamiento realice cirugías fetoscópicas, ya que le permitirá contar con una herramienta de apoyo.
Futuras líneas de Investigación
Finalmente, como líneas de investigación futuras se puede realizar un estudio comparativo y pruebas con otras arquitecturas de red neuronal convolucional (aparte de AlexNet), como VGGNet, GoogleNet, ResNet y ZFNet con la finalidad de comparar sus resultados, incrementar el número de categorías para que sea más completo el prototipo, aumentar el número de imágenes de las categorías para optimizar la exactitud de los resultados, así como aplicar aprendizaje no supervisado para clasificar videos con el objetivo de realizar ajustes en la red y fortalecerla.











 text new page (beta)
text new page (beta)