El género Abies (abetos), así como la mayoría de las coníferas, tienen una morfología muy variada que responde a factores como altitud, precipitación, disponibilidad de nutrientes, entre otros (Hultine & Marshall 2000, Ostonen et al. 2007). Esto crea discrepancias sobre la identidad taxonómica de algunas especies descritas a lo largo de la Franja Volcánica Transmexicana y en la zona sur, justo en los límites entre México y Guatemala. Al respecto Liu (1971), Eguiarte & Furnier (1997), así como Scaltsoyiannes et al. (1999) sugieren que tal confusión taxonómica está relacionada con la hibridación frecuente entre estas especies, lo que ocasiona la interposición en la variación de algunos atributos de importancia taxonómica (Jaramillo-Correa et al. 2008, Semerikova et al. 2011). Además, los bosques de Abies tiene una distribución extremadamente dispersa pero localizada regionalmente, por lo que se pueden observar algunas poblaciones restringidas a un cerro, cañada o ladera (Rzedowski 2006). En conjunto la fragmentación, la hibridación y la distancia geográfica entre poblaciones pueden modificar las diferencias fenotípicas y con ello ampliar las confusiones taxonómicas (Strandby et al. 2009).
No obstante, las características morfológicas siguen siendo la manera usual de delimitar a las especies de abetos (Fernández & Nepamuceno 1989, Strandby et al. 2009, Vázquez-García et al. 2014, Papageorgiou et al. 2015, Jasińska et al. 2017). Muchos autores han descrito especies de coníferas con base en características morfológicas como los caracteres sexuales, ya que limitan el intercambio de gametos, o atributos específicos de las hojas como los canales resiníferos (Martínez 1963, Debreczy & Rácz 1995, Miller et al. 2014, Sekiewicz et al. 2016, Orlova et al. 2020). Adicionalmente, los análisis moleculares constituyen herramientas eficientes que esclarecen dudas taxonómicas generadas por la morfología, establecen relaciones genéticas y ayudan en la identificación de especies (Aguirre-Planter et al. 2000, Shao & Xiang 2015, Xiang et al. 2009, 2018).
El género Abies representa el segundo grupo más diverso dentro de la familia Pinaceae con aproximadamente 47 especies (Martínez 1948, Liu 1971, Farjon 2017). En México se han reportado entre ocho y 10 especies del género (Farjon & Rushforth 1989, Gernandt & Pérez-de la Rosa 2014, Farjon 2017). Los estudios genéticos basados en isoenzimas y marcadores de cloroplasto muestran baja diferenciación entre especies relacionada principalmente con distribuciones discontinuas, restringidas y fragmentadas a lo largo de gradientes montañosos (Parker et al. 1981, Eguiarte & Furnier 1997, Aguirre-Planter et al. 2000, Jaramillo-Correa et al. 2008, Strandby et al. 2009). Por lo que Cruz-Nicolás et al. (2021) proponen una menor cantidad de grupos (de siete a nueve) con base en el análisis filogenético de 45 individuos de nueve especies de Abies Mesoamericanos, concluyendo que la diversificación y endemismos en el género se deben a procesos alopátricos no adaptativos.
Una de las implicaciones de las dificultades taxonómicas basadas en caracteres morfológicos y la difícil delimitación de linajes específicos se presenta en la regulación de su extracción forestal. En un escenario favorable, estas especies se enlistarán bajo alguna categoría de amenaza, ya sea por lo restringido de su distribución o por asociaciones con otras especies de importancia para la conservación. Por ejemplo, Abies religiosa (Kunth) Schltdl. & Cham., se considera la única especie cuyos bosques albergan a la mariposa monarca (Danaus plexippus L.) durante su estancia en México (Sáenz-Romero et al. 2012). Además, prácticamente todas las especies de abetos mexicanos se encuentran amenazadas por el cambio climático debido a su origen neártico, y al parecer pocas especies se encuentran distribuidas en áreas naturales protegidas (Martínez-Méndez et al. 2016).
Al centro del país, existen localidades con identidades taxonómicas dudosas agrupadas bajo el nombre genérico de “Oyamel”, lo que facilita su extracción. Al existir problemas para identificar especies en estas zonas con alta demanda maderera, la posibilidad de extraer a una especie en peligro es alta. En el estado de Hidalgo, se cuentan con registros de presencia de tres especies de Abies: A. hidalgensis Debreczy, Rácz & Guízar, A. guatemalensis Rehder y A. religiosa (Hernández 1985, Debreczy & Rácz 1995).
Abies hidalgensis, es una especie listada bajo la categoría de Vulnerable D2 en la lista roja de especies amenazadas (www.iucnredlist.org). Sin embargo, no existe información sobre su distribución geográfica ni cuáles son las amenazas a sus poblaciones, aun no se han definido sus características dasométricas (altura y diámetro), e incluso algunos de sus caracteres taxonómicos fueron evaluados en material vegetal inmaduro (conos ovulados jóvenes). Farjon (2017) se refiere a A. hidalgensis como una variedad de Abies hickelii Flous & Gaussen; debido al número de canales resiníferos en sus hojas, altamente inusual en el género (4 a 7 en A. hidalgensis y 8 a 12 en A. hickelii), además ambas especies difieren en longitud e inserción de las brácteas de sus conos ovulados y en la longitud de las hojas. Tanto A. guatemalensis como A. hidalgensis se han reportado en las poblaciones de Tenango de Doria, y Agua Blanca de Iturbide, pero sin precisar las coordenadas geográficas (Hernández 1985, Debreczy & Rácz 1995). Debreczy & Rácz (1995) indican que A. guatemalensis y A. hidalgensis difieren en la densidad de hojas, siendo dos veces mayor en A. guatemalensis en una rama de sombra de igual longitud, así como en su ángulo de inserción, el número de canales resiníferos de sus hojas, dos en A. guatemalensis y de 5-7 en A. hidalgensis, el color de los conos ovulados, azul pruinoso en A. guatemalensis y verde pruinoso en A. hidalgensis, la forma de las escamas, ciatiformes en A. guatemalensis y flabeladas en A. hidalgensis, entre otros caracteres. Aguirre-Planter et al. (2000) evaluaron un ejemplar identificado como A. guatemalensis de Agua Blanca de Iturbide, Hidalgo, con marcadores de isoenzimas, pero no se agrupó con poblaciones del resto de la distribución de la especie, en tanto Cruz-Nicolás et al. (2021) estudiaron una muestra del mismo municipio mediante genotipado por secuenciación, pero identificada como A. hidalgensis, la cual tampoco se agrupo con las otras especies.
La distribución de A. religiosa es más amplia y mejor conocida en el estado de Hidalgo (Razo et al. 2013, Pérez-Miranda et al. 2017). Este taxón se diferencia de A. guatemalensis y A. hidalgensis en el número de canales resiníferos, la forma del ápice de la hoja, el largo de la bráctea y en la longitud de las hojas (Figura 1) (Farjon 2017)

Figura 1 Abies hidalgensis: A) vista general, B) cono ovulado, C) cono polínico, D) escama, E) bráctea, F) semilla. Abies sp: G) vista general, H) cono ovulado, I) cono polínico, J) escama, K) bráctea, L) semilla. A. religiosa: M) vista general, N) cono ovulado, O) cono polínico, P) escama, Q) bráctea, R) semilla.
El Instituto Nacional de Estadística, Geografía e Informática (INEGI 2017), reporta la extracción de 250 m3 y 191 m3 respectivamente, de madera en rollo de A. religiosa en Agua Blanca de Iturbide, Hidalgo. Sin embargo, esta especie no ha sido reportada en ese municipio lo que sugiere que el aprovechamiento mencionado corresponde a A. hidalgensis o A. guatemalensis.
Es necesario contar con herramientas eficaces para delimitar especies con mayor precisión, tal como los análisis multivariados ya que consideran la similitud relativa de cada caso utilizando varios atributos morfológicos con diferente naturaleza, algunos con mayor importancia evolutiva que otros. Además, es posible asignar individuos a un grupo concreto, en este caso especies de abetos. También es factible establecer una medida de divergencia relativa entre taxones, usando unidades de distancia que no se deforman por algoritmos ni son influenciadas por criterios establecidos a priori. Mejor aún, al contar con información morfológica y genética analizadas bajo los mismos principios de independencia, permiten corroborar la identidad taxonómica de las especies. Finalmente, este tipo de análisis proporcionan información esencial sobre los caracteres más confiables, tanto morfológicos como genéticos, que se deben considerar en un trabajo de sistemática (Cesaroni & Allegrucii 1991). Por todo lo anterior, los objetivos de este trabajo son a) determinar el grado de diferenciación de los Abies distribuidos en la región de estudio, considerando una aproximación morfométrica multivariada y un análisis genético y b) proponer una distribución geográfica de las poblaciones de A. hidalgensis con base en la prospección realizada.
Materiales y Métodos
Colecta de material biológico. Las poblaciones de A. hidalgensis y A. guatemalensis para el estado de Hidalgo, México fueron identificadas a través de los registros del Herbario Nacional de México (MEXU), Herbario CHAP y consultando datos en la página de GBIF (2018). Los registros encontrados, tanto en el sureste (Agua Blanca de Iturbide y Tenango de Doria) como en el noreste (Zimapán), han sido identificados como A. hidalgensis y como A. guatemalensis. Sin embargo, se le asignó la identidad de A. hidalgensis a las poblaciones de Agua Blanca de Iturbide y Tenango de Doria dada su proximidad geográfica con la descripción del holotipo. Las poblaciones registradas eran escasas, por lo que se realizó una prospección de campo para ubicar material suficiente, lo que requirió dos años de muestreos (2015 al 2017), dado lo aislado y depauperado de las poblaciones. Es de resaltar que en todas las localidades se identificó extracción forestal, lo que pone de manifiesto la preponderancia de este trabajo. Se resguardaron muestras de herbario tanto en MEXU como en el herbario del Centro de Investigaciones Biológicas de la UAEH, identificadas como A. hidalgensis.
Para realizar el estudio genético se consideraron las poblaciones de A. hidalgensis ubicadas en San Cornelio y Las Águilas, ambas en Agua Blanca de Iturbide y en el estudio morfológico se incluyó a las poblaciones de Tenango de Doria y Acaxochitlán. Para A. religiosa se consideraron las poblaciones de El Cerezo, Pachuca de Soto, El Xihuingo, Tepeapulco, Pueblo Nuevo y Mineral del Monte para el estudio genético y las poblaciones de Acoculco, Epazoyucan y Singuilucan para el estudio morfológico. Finalmente, debido a que la población de La Encarnación, Zimapán se encuentra aislada, no hay registros sobre más poblaciones del género en sitios cercanos y ya que previamente se identificó inciertamente como A. guatemalensis o A. hidalgensis, está se consideró como Abies sp. (Tabla 1).
Tabla 1 Individuos muestreados por especie y población en Hidalgo, México. m snm: metros sobre el nivel del mar; Ng: tamaño de muestra para la evaluación genética, Nf: tamaño de muestra para la evaluación morfológica.
| Especie | Localidad | Latitud (UTM) |
Longitud (UTM) |
m snm | Ng | Nf |
|---|---|---|---|---|---|---|
| Abies hidalgensis | Agua Blanca de Iturbide | 568144 | 2251648 | 2,311 | 60 | 180 |
| Zacacuautla, Acaxochitlán | 581170 | 2237122 | 2,180 | 90 | ||
| La Cruz de Tenango y Linda Vista, Tenango de Doria | 575346 | 2244301 | 2,318 | 30 | ||
| Abies sp. | La Encarnación, Zimapán | 477944 | 2307949 | 2,420 | 20 | 30 |
| A. religiosa | Pueblo Nuevo, Mineral del Monte | 527719 | 2230777 | 2,806-2,885 | 60 | |
| El Cerezo, Pachuca de Soto | 527721 | 2229763 | ||||
| El Xihuingo, Tepeapulco | 515281 | 2192823 | ||||
| Acoculco, Cuautepec de Hinojosa | 580672 | 2205845 | 2,806 | 90 | ||
| Xolostitla, Epazoyucan | 539303 | 2224384 | 2,885 | 90 | ||
| Tecocomulco, Singuilucan | 562926 | 2209250 | 2,880 | 90 |
Análisis morfológico. Se colectaron ramas estériles de la parte interna del tercio superior de las copas, asegurándose que fuera material de sombra para evitar incluir factores como insolación y orientación. Se tomaron aleatoriamente 30 hojas de dos años en la parte media de las ramas colectadas (Strandby et al. 2009, Xu et al. 2014, Jasińska et al. 2017). Además, se colectaron 30 conos ovulados de la sección superior de la copa por individuo. Las mediciones del largo y ancho del cono se realizaron con un Vernier marca Mitotuyo con precisión de centésimas de milímetro.
Se evaluaron 18 caracteres morfológicos (30 muestras por individuo), 15 cuantitativos y tres predictores categóricos (Tabla 2) (Martínez 1963, Fernández & Nepamuceno 1989, Strandby et al. 2009). Con excepción de las medidas sobre las estructuras reproductivas, los atributos foliares se midieron sobre fotografías tomadas con cámara de 10 megapixeles con el software ImageJ Launcher v. 1.4.2.67 (Kurt et al. 2016, Rasband 2018). Se realizó un Análisis de Funciones Discriminantes Generalizado (AFDG) considerando a las especies como grupo de asignación. Para ello se utilizó el programa STATISTICA v. 10 (www.statsoft.com), previa comprobación de la multinormalidad (prueba de Mardia) y la independencia de las matrices de covarianza (prueba de Bartlett) (Gómez-Gómez et al. 2003). Las variables Fa y Fb, no se incluyeron en el AFD ya que no aportan variación al modelo, debido a su naturaleza categórica.
Tabla 2 Características morfológicas consideradas en el Análisis de Funciones Discriminantes Generalizado para contrastar a las especies de Abies en Hidalgo, México.
| Tipo de Muestra | Característica | Clave |
|---|---|---|
| Hoja | Largo | Lh |
| Ancho | Ah | |
| *Forma del ápice emarginado (1), obtuso (2), agudo (3) | Fa | |
| *Orientación dísticas (1), subdísticas (2), arreglo espiral (3) | Oh | |
| Estomas abaxiales | Ea | |
| Número de canales resiníferos | Cr | |
| Cono ovulado | Largo | Lc |
| Ancho | Ac | |
| Largo de la escama | Le | |
| Ancho de la escama | Ae | |
| Largo de la bráctea | Lb | |
| Relación largo de la bráctea/ largo de la escama | Rbe | |
| *Forma de la bráctea Exerta (1), inclusa (2) | Fb | |
| Semillas | Largo | Ls |
| Largo del ala | La | |
| Relación largo de la semilla/ largo del ala | Rsa | |
| Cono polínico | Largo | Lcp |
| Ancho | Acp |
*Predictores categóricos. Se omiten características propuestas por Debreczy & Rácz como el color de los conos ovulados y la forma del ápice de las brácteas, debido a que no se contaban con muestras representativas de A. guatemalensis como contraste.
Los conos polínicos se evaluaron por separado debido a que se tenía una menor cantidad de muestras (un individuo por población) y menor cantidad de atributos a evaluar, pero su variación es importante para la identificación de grupos. Para ello se realizó una comparación de Kruskall-Wallis con una prueba posterior post-hoc de comparaciones múltiples de Dunnett. Además, se ajustó al valor de significancia P aplicando una corrección de Bonferroni para correlaciones múltiples que consiste en dividir el valor de alfa original (0.05) entre el número de correlaciones realizadas, con el software Past v. 4.5 (Hammer et al. 2021). Los caracteres analizados fueron largo (Lcp) y ancho (Acp) del cono polínico.
Análisis genético. La extracción del material genético se realizó con base en adecuaciones del método propuesto por Doyle & Doyle (1987). Se maceraron 2 g de tejido vegetal con nitrógeno líquido hasta obtener un polvo fino y se agregó 1 ml de buffer de extracción (Tris-HCl 100 mM pH 8, NaCl 1.4 M, EDTA 20 mM, CTAB 2 % y 1 µl de 2-β-mercaptoetanol) para seguir macerando. La mezcla fue centrifugada a 8,000 rpm × 8 min, el precipitado fue obtenido y lavado hasta en tres ocasiones con el buffer de extracción para posteriormente ser incubado a 37 °C × 1 h con 10 µl de RNAsa (1 mg/ml). A continuación, se agregó 10 µl de proteínasa K (10 mg/ml) e incubó nuevamente a 60 °C durante 1 h. Posteriormente se agregó 600 µl de fenol: cloroformo: alcohol isoamílico (25:24:1) y 250 µl de NaCl al 2 %, las muestras se agitaron a 300 rpm por una hora y posteriormente se centrifugaron a 10,000 rpm × 10 min. El sobrenadante se recuperó y precipitó con 2/3 partes del volumen final (300-500 µl) de isopropanol frío dejando reposar de 12-24 h a - 20 °C. Finalmente, el tubo se centrifugó a 12,000 rpm × 10 min, se eliminó el sobrenadante, se agregó 1 ml de etanol absoluto frío a la pastilla que se centrifugó a 14,000 rpm durante 10 min. Se retiró el etanol a la pastilla y se dejó secar para ser resuspendida en 50 µl de agua destilada estéril. El material genético se cuantificó por espectofotometría en un nanoespectofotómetro marca MAPADA.
Para la amplificación se utilizaron tres marcadores microsatélites nucleares desarrollados para A. guatemalensis por Rasmussen (2008) (Tabla 3). Los cebadores se emplearon a una concentración de 5 µM. La mezcla de reacción para la PCR por individuo analizado fue: 2 µl de DNA genómico (> 50 ng/µl), 1.6 µl de MgCl2 (25 mM), 0.7 µl de oligonuclétido anterior, 0.7 µl del posterior (5 µM), 0.3 µl de dNTP´s (10 mM), 2.4 µl de amortiguador (5x), 0.5 µl de dH2O y 0.3 µl (1U) de TaqDNA polimerasa Promega® para un total de 8 µl por tubo de reacción. Se realizó la técnica de PCR en un termociclador punto final Arktik de Thermo Scientific. El programa PCR se conformó de un paso de desnaturalización inicial de 9 minutos a 94 °C; ciclado en tres pasos repetido 30 veces que consiste en desnaturalización a 94 °C por 55 s, alineación 50 s a 57 °C (primer Ab07), 50 s a 59 °C (primer Ab 08), 50 s a 58 °C (primer Ab 12) y alineación a 72 °C por 55 s y por último la extensión final a 72 °C por 7 min. Los productos de PCR se visualizaron en geles de acrilamida al 15 %, corridos a 90 V por 90 min y teñidos con bromuro de etidio a una concentración de 10 mg/ml.
Tabla 3 Marcadores moleculares usados para el análisis de las especies de Abies en Hidalgo, México. Pb: pares de bases, Ta °C: temperatura de alineamiento.
| Locus | Primer Forward 5´- 3´ | Primer reverse 3´ - 5´ | Pb | Ta °C |
|---|---|---|---|---|
| Ab07 | ACTGGCATTTGTCGCATTC | CCTCGGAGGACAAGATTTGC | 219-257 | 57 °C |
| Ab08 | ATCGAGAGGCCCAGGTAGAC | GACATAGCTGATAGTGACGCAAC | 128-161 | 58 °C |
| Ab12 | AGGTTGTGTAAGCCCGTGTAG | CCCTTTGTTGATAGAGGGAAAC | 223-244 | 60 °C |
La cantidad de alelos fue depurada por probabilidad Poisson, considerando la eliminación de los alelos nulos al presentar frecuencias menores de 5 %, lo que permitió eliminar errores de genotipado, incluyendo alelos no amplificados, esto se realizó con el programa Micro-Checker v. 2.2.3 (Oosterhout et al. 2004). Con los datos obtenidos y utilizando el programa GenAlEx v. 6.5 (Peakall & Smouse 2005), se calcularon las frecuencias génicas, para determinar el promedio de muestras amplificadas por locus/localidad (N), el promedio de alelos por locus y por localidad (A), el número efectivo de alelos por locus y por localidad (
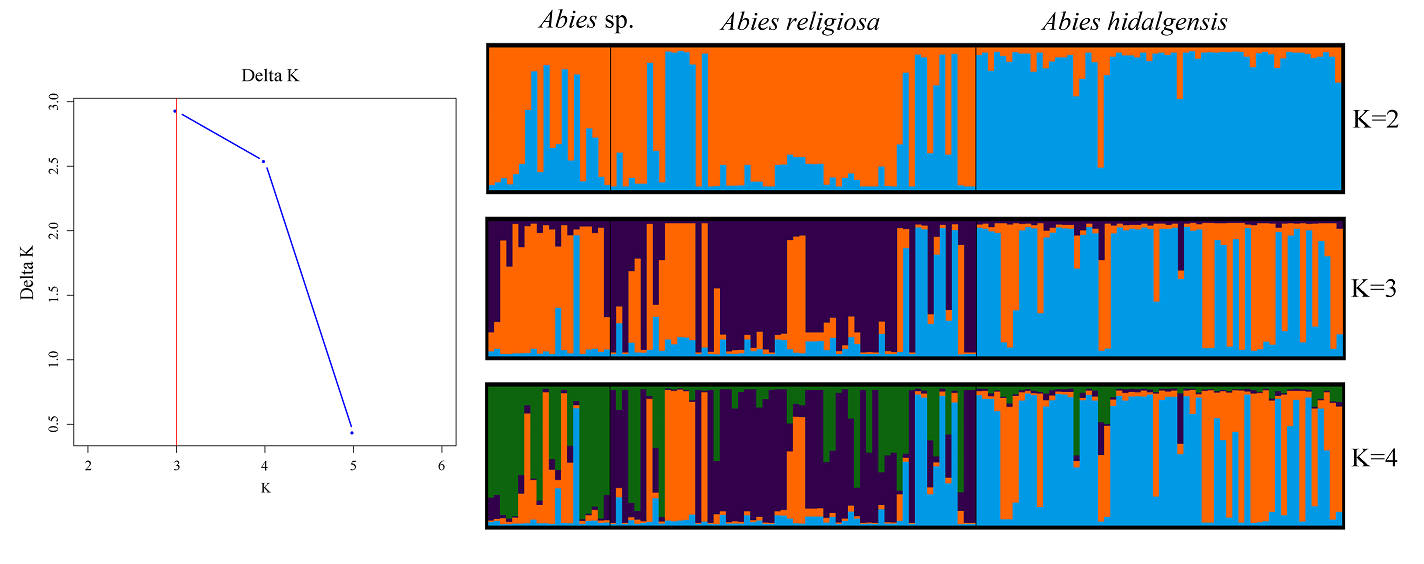
La estructura genética se evaluó con los estadísticos F de Wright (1951) (FIT, FIS, FST), para determinar la distribución de la variabilidad genética dentro del individuo, entre individuos dentro de las localidades y entre localidades analizadas, para lo que se empleó el mismo software. Se determinó el número de migrantes por generación (Nm) de forma pareada. Se realizó un Análisis de Varianza Molecular (AMOVA), asumiendo un modelo de alelos infinitos con 999 permutaciones. Para brindar un análisis sobre la distribución de la variación genética al interior de las poblaciones, se realizó un análisis de asignación bayesiana con el programa STRUCTURE v. 2.3.4 (Pritchard et al. 2000) a 50,000 iteraciones, con 50,000 repeticiones Markovianas, asumiendo un modelo “Admixture” considerando las frecuencias alélicas correlacionadas para una estimación de grupos desde K = 2 a K = 6 (número de localidades analizadas), considerando un valor de asignación Alpha global dado el modelo dependiente de asignación. El proceso fue repetido 10 veces para el cálculo del intervalo de confianza. El número de grupos genéticos se determinó con el modelo de ΔK de Evanno et al. (2005). El gráfico se realizó mediante la plataforma STRUCTURE SELECTOR (Li & Liu 2018).
Se realizó un Análisis de Funciones Discriminantes de Componentes Principales (AFDCP), considerando cada loci como una variable mediante el software STATISTICA v. 10 (www.statsoft.com). Este método descompone los datos genéticos en componentes principales como un modo de re-escalamiento métrico de las frecuencias y usa estos valores como entrada para un análisis de funciones discriminantes que permite el contraste entre grupos, en este caso especies. Lo que minimiza la variación al interior y maximiza el agrupamiento de forma que las distancias obtenidas son independientes del equilibrio de H-W, del efecto de la selección natural o del ligamiento (Jombart et al. 2010).
Así mismo, para determinar si existen cuellos de botella, se consideró una aproximación no paramétrica con respecto al exceso o deficiencia de individuos heterócigos en las poblaciones. El procedimiento asume que cada loci es dependiente, sin recambio, limitados y con la misma identidad entre las poblaciones y que los alelos se reparten aleatoriamente entre localidades en una distribución Poisson (modelo de mutación paso a paso o Stepwise Mutation Model -SMM), si existen diferencias con lo esperado, entonces se asume la deficiencia o exceso de heterócigos, este análisis se realizó mediante el programa Bottleneck v. 1.2.02 (Cornuet & Luikart 1996).
Mapa de distribución geográfica de Abies hidalgensis. Una vez finalizados los análisis morfológicos y genéticos, se realizó un mapa de la distribución geográfica de A. hidalgensis, considerando las poblaciones identificadas en estos análisis, así como información sobre ejemplares aislados localizados por prospección en campo.
Resultados
Análisis morfológico. Las especies A. hidalgensis y Abies sp. de la población de Zimapán difieren significativamente de A. religiosa de acuerdo con el AFDG (F (12, 585) = 362.851, P < 0.0019; distancia de Mahalanobis 82.39 y 45.26, respectivamente). La variación total del modelo se explica por dos factores, el primero (98.34 % de la varianza explicada) está relacionado principalmente con el ancho de la escama (Ae), relación largo de la bráctea/largo de la escama (Rbe) y largo de la bráctea (Lb), todos atributos del cono ovulado. El segundo factor (1.656 % de la varianza explicada) se relaciona principalmente con largo de la escama (Le), relación largo de la semilla/largo del ala (Rsa) y largo del ala (La; Tabla 4).
Tabla 4 Coeficientes canónicos estandarizados para cada función obtenida por el AFDG de las especies de Abies en Hidalgo, México; a partir de los datos morfológicos.
| Efecto | Factor 1 | Factor 2 |
|---|---|---|
| Largo de la hoja | -.0.064 | 0.422 |
| Ancho de la hoja | -0.12 | 0.206 |
| Largo del cono | 0.04 | -0.128 |
| Ancho del cono | 0.223 | -0.112 |
| Largo de la escama | 0.007 | -1.479* |
| Ancho de la escama | -0.654* | 0.519 |
| Relación largo de la bráctea/ largo de la escama | 0.611* | -0.670 |
| Largo de la bráctea | 0.630* | 1.15 |
| Relación largo de la semilla/ largo del ala | 0.245 | 1.520* |
| Largo de la semilla | 0.215 | 1.009 |
| Largo del ala | -0.295 | -1.36* |
| Estomas abaxiales | -0.205 | -0.037 |
| Número de canales resiníferos | -0.276 | -.0478 |
| Orientación de las hojas | 0.262 | -0.295 |
| Eigenvalue | 19.737 | 0.332 |
| Varianza acumulada | 98.344 % | 100 % |
* Las tres correlaciones significativas P < 0.05, más importantes.
El modelo es capaz de asignar correctamente los casos para A. hidalgensis (98 %) y A. religiosa (99.63 %), aunque con pobre discriminación para los individuos de Abies sp. (69.55 %) (Figura 2). Con estos resultados se pueden identificar los atributos con mayor discriminación entre especies, asociados principalmente con el cono ovulado (Tabla 5). Abies sp. y Abies hidalgensis tienen ápice de la hoja emarginado, en tanto que A. religiosa tiene ápice obtuso. Contrariamente, el número de canales resiníferos es un carácter diagnóstico, en el AFDG no resulta importante para discriminar puesto que tanto A. religiosa y Abies sp. comparten dos canales como estado de carácter, lo que reduce el porcentaje de asignación y aproxima morfológicamente ambas especies.
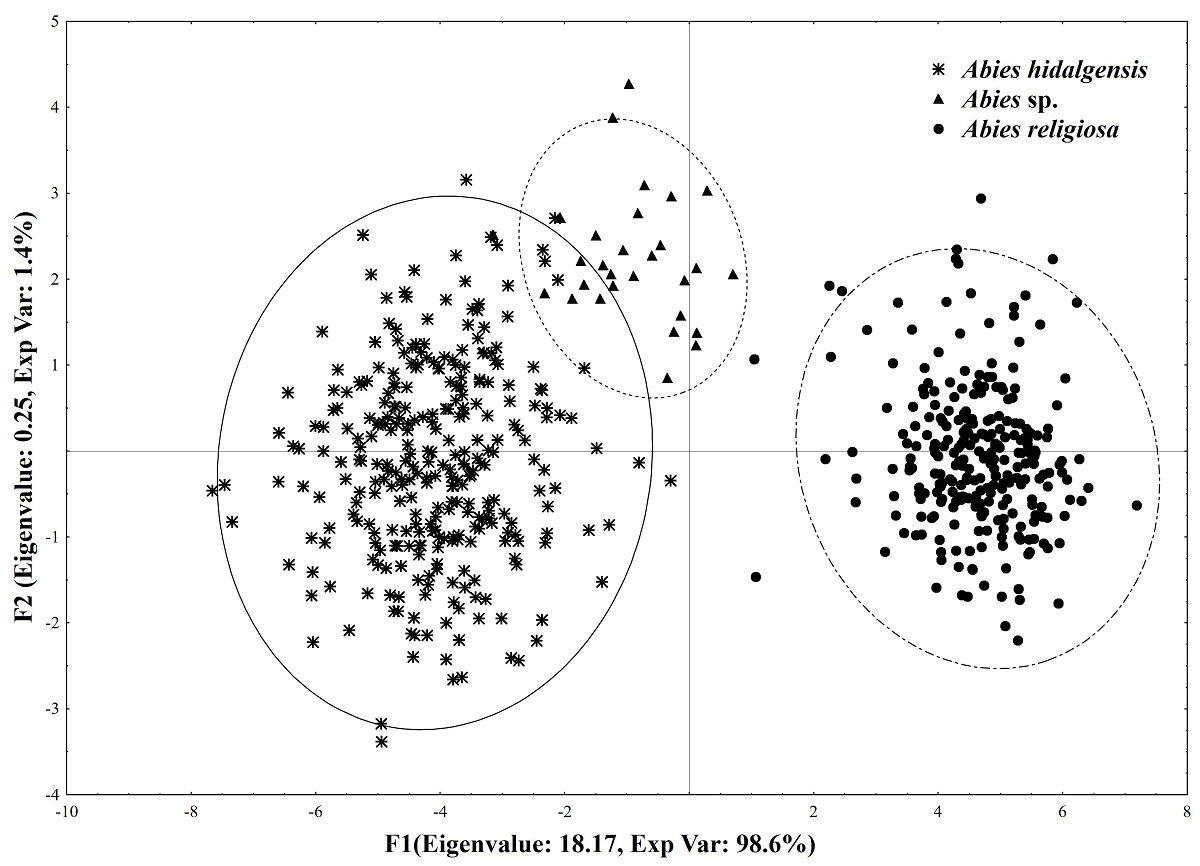
Figura 2 Ordenamiento por Análisis de Funciones Discriminantes para los caracteres morfológicos de los abetos de Hidalgo, México.
Tabla 5 Descriptores morfológicos de Abies hidalgensis, Abies sp. y A. religiosa.
| Especie | Abies hidalgensis | Abies sp. | A. religiosa |
|---|---|---|---|
| Largo de la hoja | 2.68±0.55 | 2.19±0.27 | 2.24±0.32 |
| Ancho de la hoja | 0.18±0.0007 | 0.18±0.0003 | 0.16±0.003 |
| Estomas abaxiales | 20.54±7.15 | 17.90±3.95 | 15.54±7.16 |
| Largo del cono | 8.27±1.95 | 8.00±1.11 | 10.44±1.77 |
| Ancho del cono | 3.67±0.12 | 3.64±0.29 | 4.65±0.27 |
| Largo de la escama | 2.37±0.06 | 2.15±0.06 | 2.22±0.13 |
| Ancho de la escama | 3.09±0.15 | 3.04±0.23 | 2.70±0.20 |
| Largo de la bráctea | 1.48±0.06 | 1.83±0.06 | 2.56±0.14 |
| Relación largo de la bráctea /largo de la escama | 0.62±0.01 | 0.85±0.002 | 1.16±0.01 |
| Largo de la semilla | 1.00±0.02 | 0.88±0.02 | 1.09±0.06 |
| Largo del ala | 1.09±0.02 | 1.09±0.02 | 1.11±0.04 |
| Relación largo de la semilla/largo del ala | 1.10±0.03 | 1.26±0.04 | 1.05±0.05 |
Modificado de Rosales (2018)
El ajuste del modelo AFDG al área bajo la curva (AUC) muestra que los atributos continuos evaluados, con excepción del largo del ala en las semillas, tienen una distribución gamma (AUC: 0.87, P = 0.42). La relación largo de la bráctea/largo de la escama (mayor en A. religiosa), así como la relación largo de la semilla/largo del ala (mayor en Abies sp.) tienen correlaciones no lineales significativas (r = 0.097 ± 0.024, P > 0.059).
Finalmente, en cuanto a los caracteres del cono polínico, el análisis de Kruskall-Wallis mostró que los conos de A. religiosa (Lcp 19.15 ± 3.50, Acp 7.11 ± 0.63 mm) son más grandes que los de A. hidalgensis (Lcp 16.49 ± 4.22, Acp 5.98 ± 0.9 mm) y Abies sp. (Lcp 15.22 ± 2.68, Acp 5.13 ± 0.4 mm) (χ2 = 53.26 y 13.7 respectivamente, en ambos casos P < 0.001), aunque con base en el valor de significancia de la prueba post-hoc, estas últimas dos especies no muestran diferencias significativas. Por lo tanto, en los análisis morfométricos A. hidalgensis no se separa de la población de Zimapán.
Análisis genético. Los marcadores no muestran alelos nulos y todos resultaron polimórficos (100 %) con un total de 31 alelos. Para facilitar la comparación y evitar sesgo por el tamaño de muestra, las poblaciones fueron agrupadas por especie, donde la más diversa fue Abies sp. ya que tiene mayor heterocigosis esperada y menor coeficiente de endogamia. Abies hidalgensis tiene indicadores de diversidad genética similares (Tabla 6). Esto corresponde con los valores de autofertilización (S) obtenidos, pues de igual manera esta especie mostró los valores más bajos.
Tabla 6 Descriptores genéticos de las especies de Abies analizadas en Hidalgo, México.
| Especie | N | %P | Na | Ne | I | Ho | He | F | S% |
|---|---|---|---|---|---|---|---|---|---|
| Abies hidalgensis | 60 | 100 | 8.1 ± 0.61 | 3.83 ± 0.95 | 1.45 ± 0.27 | 0.59 ± 0.14 | 0.70 ± 0.08 | 0.17 ± 0.11 | 27.16 |
| Abies sp. | 20 | 100 | 9 ± 0.57 | 5.37 ± 1.48 | 1.8 ± 0.18 | 0.68 ± 0.16 | 0.78 ± 0.04 | 0.15 ± 0.15 | 8.93 |
| A. religiosa | 60 | 100 | 8.6 ± 0.66 | 2.95 ± 0.64 | 1.43 ± 0.16 | 0.3 ± 0.048 | 0.63 ± 0.07 | 0.52 ± 0.03 | 68.75 |
N: número de individuos analizados, Na= Alelos por locus, Ne= Número efectivo de alelos por locus, I= Índice informativo de Shannon, Ho= Heterocigosis observada, He= Heterocigosis esperada, F= Índice de fijación S = porcentaje de autofertilización.
En cuanto a los niveles de diferenciación, la matriz pareada muestra que A. religiosa tiene una mayor distancia genética con respecto a las otras dos especies (Tabla 7). De acuerdo con el AMOVA, la mayor divergencia se encuentra entre loci al interior de los individuos, seguida de la diferencia entre individuos (Fst = 0.139, Fis = 0.302, Fit = 0.399; en todos los casos P = 0.001). La menor divergencia entre poblaciones se relaciona directamente con la similitud entre A. hidalgensis y Abies sp.
Tabla 7 Comparación pareada entre especies de Abies en Hidalgo, México. Sobre la diagonal: distancias de Nei; bajo la diagonal: número de migrantes por generación (Nm).
| Abies hidalgensis | Abies sp. | A. religiosa | |
| Abies hidalgensis | 0 | 0.4 | 0.486 |
| Abies sp. | 1.953 | 0 | 0.611 |
| A. religiosa | 1.755 | 1.374 | 0 |
Sin embargo, en el análisis de asignación, se identificaron tres grupos genéticos (Delta K = 3), repartidos de forma similar al interior de las especies, aunque es notorio que A. religiosa comparte variación con Abies sp. Resalta también que la distribución de grupos para A. hidalgensis es diferente en este análisis, con predominancia del grupo tres, así como Abies sp. tiene predominancia del grupo uno y A. religiosa del grupo dos (Figura 3).
El AFDCP mostró diferencias significativas entre las especies evaluadas (F (6-132) = 18.99 ± 7.065, P < 0.001; distancia de Mahalanobis “A. hidalgensis-A. religiosa = 23.44”, “A. hidalgensis - Abies sp. = 22.68”, “A. religiosa - Abies sp. =10.84”). La variación total del modelo se explica por dos factores, el primero (74.78 % de la varianza explicada) y el segundo (25.22 %) (Tabla 8). El modelo es capaz de asignar correctamente los casos para A. hidalgensis (70 %), A. religiosa (70 %), Abies sp. (90%), este último con mayor similitud con A. religiosa (Figura 4).
Tabla 8 Coeficientes canónicos estandarizados para cada función obtenida por el AFDCP de las especies de Abies en Hidalgo, México, a partir de los datos genéticos.
| Efecto | Factor 1 | Factor 2 |
|---|---|---|
| C1 | 0.779 | -0.564 |
| C2 | 0.841 | 0.431 |
| C3 | 0.341 | -0.535 |
| C4 | -0.493 | -0.542 |
| C5 | -0.107 | -0.088 |
| C6 | 0.010 | 0.214 |
| Eigenvalue | 1.335 | 0.450 |
| Varianza acumulada | 74.8% | 100 % |
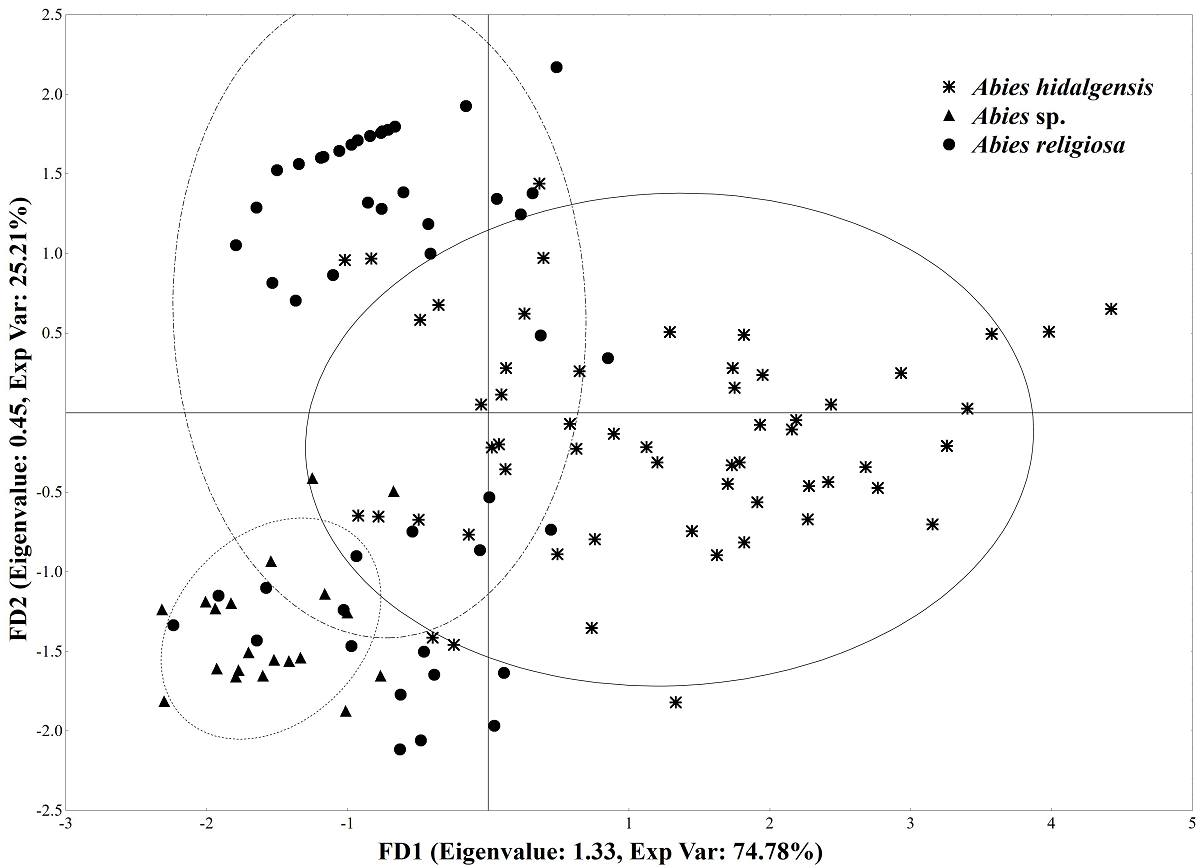
Figura 4 Ordenamiento por el Análisis de Funciones Discriminantes de Componentes Principales para los microsatélites de los abetos de Hidalgo México.
El análisis de cuellos de botella muestra que solamente A. religiosa ha sufrido una disminución significativa en su tamaño poblacional. El modelo de mutación SMM indica que los tres loci evaluados tienen diferencias significativas del equilibrio deriva-mutación para esta especie, lo que se asocia con cuellos de botella pasados (Tabla 9).
Tabla 9 Análisis de cuellos de botella para las poblaciones de Abies en Hidalgo, México. Se muestran los valores de probabilidad para las pruebas de Wilcoxon.
| Población | Modelo Deriva-Mutación | P Prueba Wilcoxon | ||
|---|---|---|---|---|
| P SMM | Deficiencia H | Exceso H | Exceso o deficiencia H | |
| Abies hidalgensis | 0.368 | 0.125 | 0.937 | 0.25 |
| Abies sp. | 0.064 | 0.188 | 0.87 | 0.375 |
| A. religiosa | > 0.0001 | > 0.05 | 1 | 0.125 |
Mapa de distribución geográfica de Abies hidalgensis. Abies hidalgensis se distribuye en las zonas sureste del estado de Hidalgo, con poblaciones remanentes escasas y ejemplares aislados en el norte de Puebla, identificados por prospección que amplían el área de distribución de la especie (Figura 5).
Discusión
Nuestros resultados muestran que existen dos especies de Abies en el estado de Hidalgo, localizadas geográficamente en las proximidades de la Faja Volcánica Transmexicana. A. religiosa tiene conos polínicos de mayor tamaño y brácteas de los conos ovulados más largas. Genéticamente, esta especie se puede ubicar en el centro del estado de Hidalgo, con algunas poblaciones en áreas naturales protegidas de las cuales se tienen registros de extracción, lo que ha generado cuellos de botella que afecta a todos los loci evaluados, asociado con el volumen de manera extraído anualmente (INEGI 2017). Por el contrario, lo que se había considerado como dos especies distintas en los municipios de Agua Blanca y Tenango de Doria (A. guatemalensis y A. hidalgensis), morfológicamente son similares y se concluye que todas las poblaciones de estos dos municipios y de Acaxochitlán corresponden a A. hidalgensis. Por otra parte, la población de La Encarnación (Zimapán) genéticamente es más similar a A. religiosa. De esta forma, lo que se había llamado A. guatemalensis en el centro de México, definitivamente no se trata de A. guatemalensis, sino de alguna otra variedad aún no definida, cercana morfológicamente a A. hidalgensis pero genéticamente a A. religiosa, aunque con algunos atributos morfológicos diagnósticos similares a A. religiosa, como el número de canales resiníferos.
En todos los casos, los rasgos diagnósticos para diferencias a estas especies en el centro de México están relacionados con las estructuras reproductivas. Sin embargo, la morfología del cono polínico ha sido poco utilizada en estudios taxonómicos (Martínez 1963, Vázquez-García et al. 2014, Farjon 2017). Dados los resultados obtenidos, se recomienda considerar dichas estructuras en futuras descripciones, tanto los conos ovulados como polínicos.
En cuanto a las semillas, la relación entre la longitud del ala y la longitud de la semilla ya han mostrado ser un carácter diagnóstico de utilidad (Strandby et al. 2009, Jasińska et al. 2017). Los resultados obtenidos apuntan a la importancia evolutiva de la dispersión para los abetos, ya que cualquier limitación del proceso incrementa la endogamia y puede condicionar la capacidad de colonización en una relación inversa con el peso, pero directa respecto a la longitud de las alas en las semillas (Cremer et al. 2012, Paluch & Marcin 2020). Nuestros resultados apuntan a que la mayor relación longitud del ala respecto a la semilla en A. hidalgensis, con relación a A. religiosa, podría incrementar su distancia de dispersión, por lo que tendría una menor endogamia, tal como se observa en los datos genéticos obtenidos, específicamente con el porcentaje de autofertilización estimado, casi tres veces mayor en A. religiosa respecto a A. hidalgensis. En abetos se ha documentado un efecto de nodricismo, normalmente sin competencia, inversamente relacionado con la distancia a las madres, efecto influenciado fuertemente por las condiciones ambientales (Houle 1995). La extracción en la zona central del estado de Hidalgo ha comprometido la supervivencia de semillas y la fecundidad por lo que se encuentra la menor diversidad en A. religiosa.
Cruz-Nicolás et al. (2021) identificaron que las poblaciones de A. guatemalensis de Chiapas y Guatemala conforman un clado bien definido y que la población identificada como esta especie localizada en San Luis Potosí, al norte de las poblaciones analizadas en nuestro trabajo; no se agrupa con estas, por lo que sugieren que se trata de una especie diferente no relacionada con A. guatemalensis, tal como nosotros sugerimos para la población de Zimapán. Cruz-Nicolás et al. (2021) también analizaron una población de A. hidalgensis que es hermana del clado en el que se agrupa al resto de los taxa de la Faja Volcánica Transmexicana (incluyendo a A. religiosa de Colima, Hidalgo, Michoacán, Querétaro y Veracruz), y a su vez la población de San Luis Potosí mencionada es hermana del clado de A. hidalgensis + FVT. Los resultados de Cruz-Nicolás et al. (2021) sustentan que A. guatemalensis forma un clado bien definido con poblaciones de Guatemala y Chiapas y que esta especie no se distribuye en el centro de México. Esto nos lleva a pensar que la especie estudiada en nuestro trabajo no es una variedad de A. hickelii como se había teorizado, sino que la menor cantidad de canales resiníferos (4 a 7 en A. hidalgensis y 8 a 12 en A. hickelii) (Farjon 2017), es un carácter emergente que no se manifiesta en A. flinckii, A. religiosa, A. jaliscana ni en A. guatemalensis procedente de la distribución original en el sureste mexicano. Aunado al número de canales resiníferos, la longitud de las semillas y el tamaño de las brácteas se pueden emplear como caracteres diagnósticos en caso de dudas taxonómicas. Estas estructuras permiten la separación definitiva entre las dos especies en conflicto taxonómico, donde A. hidalgensis se ubica geográficamente bien delimitada entre los estados de Hidalgo y Puebla, aunque con individuos aislados al norte de esta última entidad. Tal observación delimita el grado de amenaza para la especie pues su distribución resulta menor a 10 km2, lo que la coloca como En Peligro Crítico de acuerdo con el criterio B2 de la IUCN (www.iucnredlist.org).
Si bien, la profundidad y variación que muestran los microsatélites, no son propias para un estudio de sistemática, nos permiten afirmar que las especies analizadas no son iguales en cuanto a su variación poblacional, lo que cobra relevancia en el contexto de la conservación y manejo de los Abies del estado de Hidalgo. Particularmente al identificarse cuellos de botella en las poblaciones de A. religiosa. Si bien, el exceso de homócigos observado podría resultar también por endogamia o selección natural; consideramos que la selección no es capaz de modificar las proporciones alélicas de todos los loci analizados, ya que esta fuerza evolutiva actúa sobre locus únicos vinculados con la expresión de un rasgo ventajoso y en principio los microsatélites son neutrales. En cuanto a la endogamia, esta fuerza evolutiva elimina a los heterócigos sin modificar las frecuencias alélicas, situación que no se cumple en este modelo (Hedrick 2005). Al respecto Méndez-González et al. (2017) identificaron homocigosis asociable a selección disruptiva en poblaciones de A. religiosa en el Ajusco, pero usaron AFLPs como marcadores, cuya naturaleza incluye segmentos codificantes susceptibles de selección, obviamente esto también está relacionado con el mayor tamaño poblacional de A. religiosa que debería contrarrestar el efecto de los cuellos de botella identificados, por lo que se asume un efecto local, tanto de la endogamia como de la deriva.
En cuanto a las amenazas, se han mostrado diferentes situaciones de riesgo para los abetos mexicanos, incluyendo las actividades humanas relacionadas con la explotación maderera (Ramírez et al. 2003), el cambio climático (Sáenz-Romero et al. 2012), el cambio en el uso del suelo (Pineda-López et al. 2013) y los incendios forestales (Cerano-Paredes et al. 2021). En todos estos casos, las evaluaciones de riesgo se han centrado en A. religiosa dada su importancia económica y cobertura territorial, no obstante, bajo este nombre también se aprovechan poblaciones del resto de las especies de Abies, particularmente en la zona de estudio. La coexistencia geográficamente cercana entre A. hidalgensis y A. religiosa, su similitud morfológica para un ojo no entrenado y la necesidad económica de las comunidades, resulta en un riesgo mayor para la especie por lo que, cualquier actividad de extracción en la región, debe corroborar previamente la identidad taxonómica de la madera extraída. Desafortunadamente, la situación es crítica en el centro de Hidalgo, ya que las prospecciones realizadas en este trabajo permitieron identificar poblaciones de A. hidalgensis erradicadas a lo largo de tres años.
En conclusión, el aprovechamiento forestal en el estado de Hidalgo se ha realizado sobre las especies A. hidalgensis y A. religiosa bajo el nombre genérico de “Oyamel”, poniendo en un grave riesgo a la especie endémica en las poblaciones de La Encarnación, Tenango de Doria, Acaxochitlán y Agua Blanca de Iturbide. Además, en el estado solo encontramos a estas dos especies, por lo que los reportes de A. guatemalensis corresponden en realidad a A. hidalgensis al sureste de la distribución y a otra variedad aún no determinada en Zimapán. Finalmente, los estudios genéticos y morfológicos hacen posible identificar sin lugar a duda a las especies de abetos en Hidalgo, lo que es esencial para dirigir las acciones de conservación y manejo hacia especies con uso potencial, protegiendo a las poblaciones de especies endémicas y amenazadas ya mencionadas.











 nueva página del texto (beta)
nueva página del texto (beta)