El interés en los procesos de domesticación de plantas se ha visto renovado en las últimas décadas frente a los procesos de pérdida de agrobiodiversidad, de la cual dependen la agricultura, la alimentación y numerosos aspectos de la identidad cultural de las comunidades humanas (FAO 2019). Se ha calculado que en México y Centroamérica existen cerca de 6,500 especies de plantas nativas útiles, de las cuales cerca de 23.9 % cuentan con algún tipo de manejo (1,555 especies) y 17.8 % (1,148 especies) cuentan con algún grado de domesticación (Clement et al. 2021). Como “manejo” se entiende “aquellas actividades eminentemente humanas que involucran acciones deliberadas para transformar o mantener sistemas (o elementos o funciones de esos sistemas( poniendo en juego un grado elevado de conciencia o intencionalidad en tales transformaciones” (Casas & Parra 2016). El espectro de formas de manejo en estos sistemas (agroecosistemas y paisajes) incluye la recolección, la tolerancia (mantener plantas útiles en ambientes antropogénicos), el fomento (aumentar su densidad), la protección (eliminación de competidores/depredadores y la optimización del hábitat de plantas favorables), y el cultivo (propagar individuos en ambientes modificados para favorecer su desarrollo; Casas et al. 1997, Blancas et al. 2013).
Si se considera que el manejo implica decidir qué individuos han de sobrevivir y reproducirse (esto es, la selección humana), puede reconocerse que la domesticación tiene sus bases en el manejo y una población domesticada es aquella en la que el ser humano ha favorecido los individuos con características hereditarias morfológicas, fisiológicas, bioquímicas o fenológicas que satisfacen sus necesidades utilitarias o estéticas (Casas et al. 2007, Pickersgill 2007).
Las prácticas de selección, propagación y dispersión que suceden en una escala microevolutiva en el largo plazo generan cambios en las frecuencias de los fenotipos/genotipos favorecidos en las poblaciones, promoviendo la divergencia entre las poblaciones domesticadas y las silvestres, con impactos variables en sus patrones de diversidad genética (Meyer et al. 2012, Miller & Gross 2011). La diversidad genética en las poblaciones domesticadas puede disminuir por el manejo selectivo que genera cuellos de botella; por efectos fundadores asociados a la dispersión geográfica, por la propagación vegetativa y/o el favorecimiento de sistemas de reproducción autocompatibles (Doebley et al. 2006, Miller & Gross 2011, Zohary & Hopf 2012).
En contraparte, la diversidad genética puede aumentar gracias al flujo génico con parientes silvestres, la introducción de individuos provenientes de otras poblaciones, la coexistencia de distintas variedades y el favorecimiento de reclutas sexuales en algunas especies predominantemente clonales (Hughes et al. 2007, Martínez-Castillo et al. 2007, Parra et al. 2008, Brite 2021, Figueredo et al. 2015, Parra-Rondinel et al. 2021).
La filogeografía aborda el estudio de los procesos de diferenciación poblacional por medio del análisis del arreglo geográfico de los linajes de genes neutrales dentro y entre especies cercanamente emparentadas (Petit & Vendramin 2006, Avise 2009). Cuando se trata de taxa en domesticación, la filogeografía ayuda a reconocer relaciones de parentesco entre variedades, identificar parientes silvestres, reconstruir rutas de dispersión geográfica de germoplasma e identificar fenómenos demográficos influenciados por la selección y el manejo humano (Léotard et al. 2009, Arias et al. 2010, Gunn et al. 2011, Besnard et al. 2013).
Aunque existe abundante información sobre los patrones filogeográficos de especies anuales de relevancia agrícola mundial (v.g., Woodhouse & Hufford 2019), el conocimiento sobre especies perennes es limitado (Miller & Gross 2011, Gaut et al. 2015). Del total de especies de plantas nativas útiles registradas para México y Centroamérica, una proporción importante son árboles (28.6 %, 1,861 especies; Clement et al. 2021), muchos de ellos aprovechados y seleccionados en huertos familiares (Galluzzi et al. 2010, Ferrer et al. 2019).
Entre las especies comúnmente distribuidas en huertos del trópico mexicano se encuentran Crescentia alata Kunth y C. cujete L. (Bignoniaceae; nombre común: jícaro, tecomate). Estas especies producen frutos esféricos indehiscentes y duros, cuyos exocarpos son utilizados para elaborar recipientes para el transporte y el almacenamiento, así como utensilios domésticos y artesanías (especialmente las “jícaras”) (Gentry 1980).
Estudios filogeográficos previos en Crescentia cujete han mostrado que el manejo de sus poblaciones, asociado a su alto valor cultural, ha tenido un impacto significativo en la estructura filogeográfica de la especie tanto en México como en todo el Neotrópico (Moreira et al. 2017, Aguirre-Dugua et al. 2018). Este impacto se relaciona con la introducción, en tiempos precolombinos, de un linaje que en México existe sólo bajo cultivo y que ha conservado su identidad genética en los huertos familiares debido a la continua selección que favorece árboles que producen frutos grandes y redondos, propagados principalmente por vía vegetativa (Aguirre-Dugua et al. 2012).
Aunque la elaboración de recipientes con frutos de C. alata y C. cujete se realiza de forma similar, estas especies tienen una distribución contrastante y aún se desconoce si C. alata ha estado sujeta al mismo grado de selección. C. alata se distribuye en sabanas abiertas (pastizales), declives bajos y lechos de arroyos temporales en selvas bajas caducifolias y subperennifolias de la vertiente del Pacífico, desde Sonora a Chiapas y la Cuenca del Balsas, con una distribución disyunta en la Huasteca Potosina y el norte de Veracruz. Hacia el sur, su distribución continúa por la vertiente del Pacífico hasta Costa Rica. En cambio, C. cujete se distribuye en sabanas inundables y selvas bajas subperennifolias de la vertiente del Atlántico, desde el norte de Veracruz hasta la península de Yucatán, y continúa su distribución en Centro y Sudamérica hasta la cuenca amazónica de Perú y Bolivia (Gentry 1980, Rzedowski & Calderón de Rzedowski 1993, Pennington & Sarukhán 1998).
Estudios etnobotánicos muestran que C. alata tiene un alto valor cultural: los restos arqueológicos más antiguos de recipientes elaborados con sus frutos fueron hallados en las cuevas de El Gallo y La Chagüera, Morelos, con una antigüedad de 2,300 años antes del presente, mientras que el uso de sus frutos como recipientes fue mencionado muy tempranamente por los cronistas españoles (véase la Historia General y Natural de las Indias de Gonzalo Fernández de Oviedo, 1535) (Aguirre-Dugua & Casas, en prensa). Actualmente, el uso de tales recipientes está documentado en Sinaloa, Nayarit, San Luis Potosí, Veracruz, Guerrero y Oaxaca (Aguirre-Dugua & Casas, en prensa), y la pulpa de sus frutos tiene aplicaciones en la farmacopea tradicional, especialmente para tratar afecciones respiratorias (INI 1994, Santander-Cancino 1994). Crescentia alata figura entre las diez especies de mayor uso tradicional en las selvas bajas caducifolias de nuestro país (Boyás et al. 2001).
Este estudio tiene como objetivo evaluar la diversidad genética y la estructura filogeográfica de Crescentia alata, en relación con las prácticas de manejo que recibe en los huertos familiares donde se aprovechan sus frutos y su historia demográfica profunda. Se examinaron las siguientes preguntas: ¿Cuál es la estructura filogeográfica entre las poblaciones de Crescentia alata en México y Centroamérica, y cómo ésta se relaciona con los cambios climáticos del pasado? ¿Cuál es el origen del germoplasma presente en los huertos? ¿Cómo son los niveles de diversidad genética de las poblaciones de huertos y cómo se relacionan con las prácticas de manejo de los árboles?
En relación con el origen del germoplasma y los efectos del manejo se hipotetizaron dos escenarios: (1) La domesticación de C. alata se originó fuera del territorio mexicano, como aparentemente ocurrió en C. cujete y, en consecuencia, se observará un patrón de estructura genética marcada entre poblaciones silvestres y cultivadas, o bien (2) la selección humana ha ocurrido en el territorio mexicano y las poblaciones silvestres y cultivadas pertenecen a la misma poza génica. En relación con el manejo, se plantean dos hipótesis: 1) la propagación en las poblaciones cultivadas es por vía vegetativa y, por lo tanto, hay baja diversidad de haplotipos dentro de cada población, así como baja estructuración entre poblaciones, o 2) la propagación en las poblaciones cultivadas es por vía sexual y éstas contienen alta diversidad de haplotipos maternos, con alta estructuración entre poblaciones.
En relación con los cambios climáticos del pasado se espera encontrar un patrón filogeográfico asociado a una distribución de la especie más amplia a finales del Pleistoceno, lo cual se basa en la inferencia de una mayor distribución de sabanas y pastizales en ese periodo. La presencia de estos tipos de vegetación durante el periodo glaciar se ha deducido con base en condiciones de menor temperatura, mayor sequía y menor precipitación que en la actualidad, así como por una mayor abundancia de polen de la familia Poaceae (Leyden 1984, Markgraf 1989, Piperno 2006).
Materiales y métodos
Especie de estudio. Crescentia alata tiene hojas trifolioladas con pecíolo alado. Las flores crecen directamente en el tronco (caulifloría), son tubulares campanuladas, de hasta 6.5 cm de largo, son hermafroditas y polinizadas por murciélagos como Glossophaga soricina, Leptonycteris spp. y Phyllostomus discolor (Phyllostomidae) (Heithaus et al. 1975, Maldonado López 2008) (Figura 1).

Figura 1 Características generales de Crescentia alata. A) Árboles creciendo en un huerto en la Huasteca Potosina. B) Flores que se desarrollan en el tronco y ramas principales (caulifloría). C) Frutos esféricos y hojas trifolioladas. D) Jícaras utilizadas para consumir bebidas, elaboradas con el exocarpo, partiendo el fruto longitudinalmente por la mitad. E) Sonaja decorada proveniente de Pinotepa de Don Luis, Oaxaca, elaborada con el exocarpo del fruto entero.
El fruto es indehiscente con un exocarpo duro. Se ha propuesto que la indehiscencia de este género es resultado de la interacción con mamíferos frugívoros que formaron parte de la megafauna pleistocénica, así como una posible adaptación paralela a la dispersión por agua (hidrocoria) (Gentry 1974, Janzen & Martin 1982, Howe 1985). La especie es común en los pastizales inducidos (Cajas-Giron & Sinclair 2001, Bass 2004) debido al aprovechamiento histórico de las sabanas para la cría de ganado, así como gracias a su comportamiento heliófilo, su resistencia a los incendios (obs. pers.) y tendencia a comportarse como elemento frecuente de la vegetación secundaria.
Sitios y año de estudio. Se analizaron 243 individuos colectados en 28 sitios de México, así como muestras provenientes de Guatemala, Honduras y Costa Rica (35 sitios en total; Figura 2), colectados entre 2009 y 2011. Estas muestras corresponden a 43 individuos de huertos, 199 silvestres y uno proveniente de herbario (Tabla 1). Dieciséis de estos individuos fueron analizados en un estudio previo siguiendo la metodología aquí descrita (Aguirre-Dugua et al. 2018), incluyendo una muestra de Parmentiera aculeata utilizada como grupo externo (Ragsac et al. 2021) con el propósito de polarizar la red de haplotipos y facilitar la identificación de haplotipos ancestrales y derivados.

Figura 2 Distribución general de Crescentia alata en México y Centroamérica de acuerdo con los registros del Global Biodiversity Information Facility (doi.org/10.15468/dl.ru885y) y sitios colectados para el presente estudio (véase Tabla 1). Las letras azules señalan las provincias fisiográficas de acuerdo con INEGI (2001), a: Península de Baja California, b: Llanura Sonorense, c: Llanura Costera del Pacífico, d: Sierra Madre Occidental, e: Sierras y Llanuras del Norte, f: Mesa del Centro, g: Sierra Madre Oriental, h: Grandes Llanuras de Norteamérica, i: Llanura Costera del Golfo Norte, j: Eje Neovolcánico, k: Sierra Madre del Sur, l: Llanura Costera del Golfo Sur, m: Sierras de Chiapas y Guatemala, n: Cordillera Centroamericana, o: Península de Yucatán.
Tabla 1 Muestras de Crescentia alata analizadas en el presente estudio. H = individuos de huertos, S = individuos silvestres.
| Sitio | Estado | País | Individuos analizados | Latitud | Longitud | Vía de propagación1 | ||
|---|---|---|---|---|---|---|---|---|
| H | S | |||||||
| 1. Culiacán | Sinaloa | México | 5 | 25.0185 | -107.5318 | |||
| 2. Villa Unión | Sinaloa | México | 8 | 23.4782 | -107.0648 | |||
| 3. Acaponeta | Sinaloa | México | 7 | 22.5386 | -105.4632 | |||
| 4. Tequilita | Sinaloa | México | 6 | 21.1184 | -104.8247 | |||
| 5. Tomatlán | Jalisco | México | 9 | 19.9960 | -105.3148 | |||
| 6. Chamela | Jalisco | México | 13 | 19.4974 | -105.0390 | |||
| 7. Coahuayana huerto | Michoacán | México | 9 | 18.6961 | -103.6619 | T(3) | ||
| 8. Coahuayana silvestre | Michoacán | México | 2 | 18.7000 | -103.6900 | |||
| 9. Nocupétaro | Michoacán | México | 14 | 19.0969 | -101.2070 | |||
| 10. Chinapa | Michoacán | México | 18 | 19.3957 | -100.8751 | |||
| 11. Agua del Obispo | Michoacán | México | 14 | 18.6758 | -100.9136 | |||
| 12. San Ignacio | Morelos | México | 2 | 18.8232 | -99.0934 | - | ||
| 13. San Rafael | Morelos | México | 2 | 18.6406 | -99.0041 | - | ||
| 14. Tlaltizapán | Morelos | México | 2 | 18.6953 | -99.1142 | T(2) | ||
| 15. Tenango | Morelos | México | 10 | 18.7100 | -98.7500 | |||
| 16. Vergel | Morelos | México | 9 | 18.5675 | -99.0384 | |||
| 17. Antiguo Morelos | San Luis Potosí | México | 10 | 22.5318 | -99.0768 | |||
| 18. Ciudad Valles silvestre | San Luis Potosí | México | 6 | 22.0415 | -99.1103 | |||
| 19. Ciudad Valles huerto | San Luis Potosí | México | 4 | 21.0119 | -99.0859 | T (1) | ||
| 20. Concá | Querétaro | México | 10 | 21.4450 | -99.6356 | |||
| 21. Puente Nacional huerto | Veracruz | México | 4 | 19.3139 | -96.5176 | - | ||
| 22. Puente Nacional silvestre | Veracruz | México | 6 | 19.3066 | -96.5011 | |||
| 23. Tequisistlán | Oaxaca | México | 7 | 16.3992 | -95.6033 | T(2) | ||
| 24. San Mateo del Mar huerto | Oaxaca | México | 8 | 16.2100 | -94.9817 | T(3), S(3) | ||
| 25. San Mateo del Mar silvestre | Oaxaca | México | 2 | 16.2100 | -94.9800 | |||
| 26. El Morrito | Oaxaca | México | 8 | 16.7258 | -94.9800 | |||
| 27. Acala | Chiapas | México | 11 | 16.5097 | -92.8726 | |||
| 28. Trinitaria | Chiapas | México | 11 | 15.9749 | -92.0073 | |||
| 29. Azacualpilla | Guatemala | 3 | 14.7228 | -90.3616 | ||||
| 30. Teculután | Guatemala | 1 | 14.9949 | -89.7288 | ||||
| 31. Honduras C | Honduras | 5 | 14.7633 | -87.9581 | - | |||
| 32. Honduras J | Honduras | 7 | 14.4869 | -87.9872 | - | |||
| 33. Costa Rica Palo Verde | Costa Rica | 5 | 10.3834 | -85.3342 | ||||
| 34. Costa Rica J. Cavender | Costa Rica | 4 | ||||||
| 35. MEXU Cobar, García & Reyes AC436 | Guatemala | 1 | 14.9442 | -89.5286 | ||||
| Total | 43 | 200 | ||||||
1Sólo para los individuos de huertos y cuando fue posible obtener la información. Se señala entre paréntesis el número de individuos en cuestión. T = tolerado (nació solo o ya estaba en el terreno), V = sembrado por esqueje, S = sembrado por semilla.
Tipos de poblaciones. Los árboles muestreados fueron clasificados en dos categorías -de huertos y silvestres- con base en los siguientes criterios:
Huertos.- Árboles que se encuentran dentro de núcleos de población humana, ubicados en solares, traspatios o jardines familiares, dentro de terrenos de casa-habitación, dentro de terrenos sin construcciones, pero dentro del núcleo de población, terrenos rodeados de propiedades habitadas, en banquetas o en el borde de calles. Los árboles colectados en una misma localidad se consideraron como integrantes de una misma población.
Silvestres.- Árboles que se encuentran fuera de núcleos de población humana, ubicados en bordes de carreteras, áreas de vegetación natural como selvas bajas caducifolias, selvas medianas, sabanas, vegetación secundaria o manejada como pastizales derivados de actividades ganaderas. Debido a su comportamiento como componente de vegetación secundaria, es difícil establecer el verdadero estatus “silvestre” de las poblaciones. Se reconocieron como “silvestres” aquellas poblaciones que no presentaban evidencias de haber sido plantadas, y con signos de ser persistentes de forma autónoma, como por ejemplo por medio de la presencia de individuos jóvenes (población con estructura de edades) y evidencias de frugivoría por mamíferos silvestres como tlacuaches (Didelphis marsupialis L.), coatís (Nasua narica L.) y pecaríes (Dicotyles tajacu L.).
Se buscaron poblaciones de huerto geográficamente cercanas (< 10 km) a las poblaciones silvestres, mientras que las poblaciones silvestres fueron colectadas a una distancia mínima de 50 km entre ellas.
Vías de propagación. En los sitios donde se colectaron individuos de huertos se documentó la vía de propagación de los individuos. De acuerdo con las categorías de manejo descritas por Casas et al. (2007) se consideró que el árbol era “tolerado” cuando se trataba de un árbol que ya existía en el terreno o que nació “solo” y que los propietarios decidieron conservar; se consideró que el árbol era “sembrado” cuando los propietarios reportaron haberlo sembrado intencionalmente, ya sea por vía vegetativa usando esquejes, o por vía sexual usando semillas.
Amplificación de microsatélites de cloroplasto. Las hojas de los árboles muestreados fueron conservadas en nitrógeno líquido o deshidratadas en silica gel. Se extrajo el ADN total genómico con base en el protocolo de Doyle & Doyle (1987) basado en CTAB (bromuro de cetiltrimetil amonio) modificado con la adición de cloruro de sodio-Tris-EDTA. El ADN del ejemplar de herbario fue extraído con DNeasy Plant Mini Kit de Qiagen (Oxford, California, no. cat. 69106). Se amplificaron los microsatélites de cloroplasto con los diez oligonucleótidos universales publicados por Weising & Gardner (1999) marcados con fluorescencia, de los cuales seis mostraron variación en la muestra (Material suplementario, Tabla S1). Los microsatélites variables fueron simultáneamente amplificados en cada muestra en un volumen final de 5 μl utilizando el kit de PCR multiplex de Qiagen (no. cat. 206143) con la siguiente composición: mezcla multiplex 2.5 µl, coctel oligonucleótidos 1 µl, ADN (10ng/µl) 1 µl, H2O 0.5 µl; el coctel de oligonucleótidos contenía para el locus ccmp1 una concentración de oligos 3 mM, ccmp2 1.5 mM y para los loci ccmp4, 5, 6 y 10 una concentración de 1 mM. La amplificación se realizó con el siguiente programa: 95 °C 15 min; 35 ciclos, cada uno de 94 °C 30 s, 55.5 °C 1:30 min y 72 °C 1 min; y un paso final de 60 °C 30 min. Los productos de PCR fueron analizados en el AbiPrism 3100-Avant Genetic Analyser. El tamaño de cada microsatélite fue evaluado utilizando el software Peak Scanner v. 1.0 (Applied Biosystems, Waltham, Massachusetts) con un marcador de tamaños GeneScan 500 LIZ como referencia.
Para confirmar la repetibilidad del PCR se amplificaron en dos reacciones independientes 48 individuos pertenecientes a 14 sitios, y se utilizaron de uno a dos controles positivos en cada corrida. Asimismo, los tamaños de los microsatélites en cada muestra se registraron de manera transversal para disminuir la probabilidad de cometer errores en la lectura (i.e., primero un microsatélite en todas las muestras, luego el segundo microsatélite y así sucesivamente).
Análisis de datos. Los haplotipos fueron definidos como cada combinación particular de distintos alelos de microsatélites en cada locus. Las relaciones entre haplotipos fueron visualizadas por medio de una red de unión de medianas (median-joining network;Bandelt et al. 1999), la cual representa, a manera de red y en un solo gráfico, el conjunto de árboles posibles para el juego de datos analizado. Esta red se construyó con el software Network v. 4.6.1.2 (Bandelt et al. 1999, www.fluxus-engineering.com) bajo la modalidad ‘Y-chromosomal short tandem repeat’ que se refiere a haplotipos de herencia uniparental definidos por microsatélites de repeticiones sencillas que evolucionan una repetición a la vez (single-repeat mutational model). La distancia entre haplotipos se midió con base en un peso de 10 (default) para cada estado de carácter. Se definió el valor del parámetro ε = 10 para asegurar la inclusión en la red de todos los árboles más cortos posibles y se aplicó un paso adicional posterior de máxima parsimonia para eliminar vectores medianos superfluos.
Los niveles de diversidad genética se calcularon con base en el número total de haplotipos y por medio del índice de diversidad haplotípica h calculado con Arlequin v. 3.5.1.3 (Excoffier et al. 2005). Dicho índice puede interpretarse como la probabilidad de que dos haplotipos escogidos al azar dentro de una población sean distintos entre sí mediante la fórmula:
La estructuración de la diversidad en los sitios de huertos y sitios silvestres, así como en el total de sitios muestreados, fue evaluada mediante un análisis de varianza molecular (AMOVA, con Arlequin). La estructura filogeográfica fue evaluada con los estadísticos G ST y R ST calculados con PermutCpSSR v. 2.0 (Pons & Petit 1995, 1996). Mientras el estadístico G ST se basa exclusivamente en las frecuencias haplotípicas (alelos no ordenados), R ST incluye una matriz de pesos (en este caso, la diferencia en el número de repeticiones en los microsatélites) que refleja las relaciones genealógicas entre los haplotipos (esto es, los alelos están ordenados). Se puso a prueba la hipótesis nula R ST > G ST, , esto es, que los haplotipos emparentados están dentro de las mismas poblaciones (Pons & Petit 1996). Debido a restricciones en el cálculo de estos estadísticos se eliminaron los sitios con tamaños de muestra < 3 individuos, por lo que G ST y R ST se basaron en 29 sitios (22 silvestres y 7 de huertos; entre estos últimos se agrupó en una sola población los sitios SI, SR y Tiz del estado de Morelos debido a su cercanía geográfica).
Para reconocer grupos geográficos de poblaciones se utilizó el algoritmo de Monmonier, el cual coloca barreras entre poblaciones (y, por ende, define grupos a cada lado de la barrera) con base en las distancias genéticas que las separan (Manni et al. 2004). En este análisis la matriz de distancias entre poblaciones se basó en el número promedio de diferencias entre poblaciones de Nei (D) definido como
Finalmente, para contar con un escenario histórico de la especie previo al manejo humano se evaluó si las poblaciones silvestres presentan un patrón de aislamiento por distancia con una prueba de Mantel, utilizando como medida de distancia genética entre poblaciones el índice D mencionado y como distancia geográfica la distancia lineal entre ellas (en km). Para obtener un valor de significancia estadística del coeficiente de correlación entre matrices éstas se permutaron 1,000 veces (con el programa Arlequin). También se puso a prueba si la especie ha mantenido en estado silvestre tamaños poblacionales constantes o si ha experimentado crecimiento demográfico por medio del análisis de la distribución mismatch (la distribución de las diferencias pareadas entre haplotipos). La distribución mismatch suele ser multimodal en poblaciones que han mantenido un tamaño relativamente constante y unimodal en aquellas que han experimentado un crecimiento demográfico reciente (Rogers & Harpending 1992). El modelo de expansión demográfica puesto a prueba asume un crecimiento poblacional de tamaño N0 a tamaño N1 hace ( generaciones, después de lo cual la población alcanza un equilibrio demográfico (Rogers & Harpending 1992). Se utilizó el programa Arlequin para estimar el parámetro ( (siendo (=2ut, donde u es la tasa de mutación de la secuencia o haplotipo [expresada como u=m T µ, donde m T es la longitud de la secuencia en nucleótidos o número de loci de SSR del haplotipo, y µ es la tasa de mutación por nucleótido o por locus de SSR, respectivamente] y t es el número de generaciones) siguiendo la metodología de Schneider & Excoffier (1999). Se puso a prueba el modelo de expansión con una aproximación de bootstrap paramétrico de 10,000 repeticiones en el que se simulan árboles coalescentes acordes a una expansión demográfica con el valor de ( estimado a partir de los datos, recalculando cada vez el nuevo valor de (* y la distribución mismatch esperada. Las simulaciones permiten generar un intervalo de confianza de 95 % para el parámetro ( observado y evaluar el ajuste de la distribución mismatch observada a la distribución mismatch esperada utilizando como estadístico la suma de desviaciones al cuadrado (sum of squared differences o SSD) y el índice r de rugosidad de Harpending (Harpending 1994).
Para estimar el tiempo de la expansión demográfica se consideraron las tasas de mutación reportadas por Jakobsson et al. (2007) para microsatélites de cloroplasto de Arabidopsis thaliana. Tomando en cuenta la longitud de los microsatélites amplificados en el presente estudio (8 a 15 pb), se consideró el intervalo de 7 × 10-7 a 28 × 10-7 bases por año (Jakobsson et al. 2007). El tiempo a la expansión se estimó despejando la fórmula ( = 2ut, donde t = (/2u y u = mµ , donde µ es la tasa de mutación por nucleótido y m es el tamaño de la región genética estudiada (número de nucleótidos en el caso de la secuencia de una región de DNA, número de loci de SSRs en el caso de haplotipos de microsatélites) (Rogers & Harpending 1992). Nótese que si la tasa de mutación es expresada en años, en vez de generaciones, la estimación del tiempo a la expansión también se obtendrá en años, por lo que no es necesario conocer el tiempo de generación de la especie.
Modelos de distribución potencial. Se obtuvieron los registros de presencia de la especie en el Sistema Nacional de Información sobre Biodiversidad (SNIB) de la Comisión Nacional para el Conocimiento y Uso de la Biodiversidad (CONABIO), y los herbarios MEXU, MO, NY. Este listado se verificó manualmente para excluir registros ubicados dentro de núcleos de población y/o cultivados, así como registros duplicados a una resolución de 0.04º, lo cual generó un listado final de 315 registros (Material suplementario, Figura S1).
Para modelar la distribución de la especie en el presente y proyectar su distribución en el pasado usamos Maxent (Maximum Entropy Algorithm; Phillips et al. 2006). Maxent estima el nicho ecológico potencial de una especie por medio de la distribución probabilística de las variables ambientales que mejor describen las localidades donde se ha registrado la presencia del taxón, siendo capaz de distinguir entre sitios de presencia y sitios de fondo (Elith et al. 2011). La distribución se proyectó para cuatro periodos: presente, Holoceno Medio (HOLO, 6 mil años atrás), Último Máximo Glacial (UMG, 21 mil años atrás) y Último Interglacial (UIG, 130 mil años atrás). Los modelos fueron construidos con base en las variables bioclimáticas de WorldClim v. 1.4 (Hijmans et al. 2005), con una resolución de 2.5 minutos de arco. Las capas climáticas para HOLO y LGM se basaron en dos modelos alternativos de circulación: MIROC (Model for Interdisciplinary Research on Climate; Hasumi & Emori 2004) y CCSM (Kiehl & Gent 2004). Se estimaron coeficientes de correlación de Pearson entre todas las variables y se identificaron aquellas variables con coeficientes > 0.8 y se excluyeron aquellas con el factor de inflación de varianza más elevado. Para los modelos finales se seleccionaron nueve variables bioclimáticas (Material suplementario, Tabla S2).
El área accesible (M) para la especie se aproximó usando los polígonos espaciales de las eco-regiones terrestres (Olson et al. 2001) habitadas por la especie; esta área fue limitada usando un buffer espacial de 2º alrededor de los puntos de presencia. El área accesible para las proyecciones al pasado fue limitada usando un buffer espacial de 4º alrededor de los puntos de presencia. Los modelos de distribución se construyeron usando esta M para asegurar una cobertura de la dinámica temporal del rango de distribución y para disminuir la sobre-predicción asociada con la utilización de áreas más grandes.
Los modelos fueron construidos usando 20 réplicas con 10,000 pseudo-ausencias, un ajuste del modelo (RM) de 1.0 y con características cuadráticas ‘LHQT’; el ajuste del modelo fue determinado con el paquete ‘ENMeval’ (Muscarella et al. 2014) para R (R Core Team 2022) usando el método ‘randomkfolds’ con k= 20 y seis valores de RM (0.5, 1.0, 1.5, 2.0, 2.5, 3.0). Los modelos fueron calibrados utilizando una muestra aleatoria de 70 % de los registros de ocurrencia y fueron evaluados con el 30 % restante. Evaluamos los modelos con base en el área bajo la curva de operación del receptor (AUC) entre réplicas, considerando como válidas aquellas réplicas con valores superiores a 0.85 (Elith et al. 2006). El AUC promedio fue de 0.98 (0.001 s.d.). Las réplicas seleccionadas se combinaron para construir el modelo al presente y proyectarlo a las capas del pasado.
Resultados
Vías de propagación. Del total de sitios (9) e individuos (43) pertenecientes a huertos, se obtuvo información detallada sobre mecanismos de propagación practicados en 5 sitios y 11 individuos (56 % de los sitios, 26 % de los individuos; Tabla 1). De estos individuos, la mayoría fueron tolerados (8), pero algunos fueron sembrados por medio de semilla (3). No hubo registros de propagación mediante esquejes (propagación vegetativa o clonal). El patrón de tolerancia de árboles que nacen “espontáneamente” o “solos”, es decir, que ya estaban en el terreno cuando se decidió dejarlos en pie se presentó en distintas regiones geográficas como San Luis Potosí, Michoacán, Oaxaca y Morelos.
En el resto de la muestra (4 sitios, 32 árboles) las personas que brindaron la información desconocían el origen de los árboles, o bien éstos fueron muestreados con la autorización de los propietarios, pero sin que la entrevista pudiera llevarse a cabo.
Haplotipos de cpADN. Con base en los seis loci de microsatélites analizados se detectaron 17 haplotipos de cpADN en el total de la muestra. La Figura 3 representa las relaciones entre estos haplotipos con base en el método de redes de unión de medianas y su distribución geográfica.

Figura 3 A) Red de haplotipos de seis microsatélites de cpADN con base en el método de unión de medianas. Los puntos negros representan vectores medianos (haplotipos hipotéticos), las pequeñas barras transversales representan el número de cambios entre haplotipos. El tamaño de los círculos es proporcional a la frecuencia de cada haplotipo. Los haplotipos de C. cujete (cuadrados) son los reportados en un estudio previo (Aguirre-Dugua et al. 2018). B) Distribución geográfica de los haplotipos en los sitios muestreados. Las líneas divisorias corresponden a las provincias fisiográficas de México (véase Figura 2).
En la red de haplotipos de la Figura 3 se observa que los haplotipos característicos de C. alata forman un solo haplogrupo, a excepción de 10 individuos (4 % del total de la muestra) portadores del haplotipo A, el cual es característico de un linaje cultivado de C. cujete. Los individuos portadores del haplotipo A se distribuyen en el sitio de San Mateo del Mar, Oaxaca, y en huertos en Honduras. Dentro del haplogrupo principal de C. alata, los haplotipos más abundantes son el R, O, T y Q. De éstos, es posible inferir que los haplotipos O y R son ancestrales, debido a que el haplotipo O es compartido por los géneros Crescentia y Parmentiera, además de ubicarse entre el haplogrupo característico de C. cujete (haplotipos A, B, D, E) y el haplogrupo de C. alata, mientras que el haplotipo R, a un paso mutacional del O, es el más abundante en toda la muestra y se encuentra ubicado en el centro de la red de haplotipos (Figura 3A).
El haplotipo ancestral O se distribuye en los sitios de la vertiente al Golfo de México (Huasteca Potosina y centro de Veracruz) y en baja frecuencia en la Meseta Central de Chiapas. Los haplotipos R y S se observan en los sitios de la cuenca del río Balsas y costa central del océano Pacífico, mientras que los haplotipos Q2 a Q4 se encuentran en el límite norte de la distribución de la especie, en Nayarit y Sinaloa. Por otra parte, los haplotipos T, U y U1 corresponden a los sitios del extremo este de la cuenca del río Balsas, el Istmo de Tehuantepec y la Meseta Central de Chiapas. Las muestras centroamericanas se caracterizan por portar principalmente el haplotipo Q, acompañado de los haplotipos Q1 y Q4 en baja frecuencia en un sitio de Guatemala (Figura 3B).
Con respecto a la estructura espacial, el algoritmo de Monmonier definió tres conjuntos: Pacífico norte, centro de México y Oaxaca-Chiapas (Figura 4), y distinguió poblaciones únicas desde Chiapas hasta Centroamérica. Las poblaciones de huerto portadoras del haplotipo A característico de C. cujete se diferencian como poblaciones únicas (Oaxaca y Costa Rica). Al repetir el análisis considerando únicamente sitios silvestres se conserva el patrón de diferenciación del norte, centro y vertiente al Golfo, así como la separación de las poblaciones de Oaxaca y Chiapas, y las de Guatemala (Figura 4).

Figura 4 Barreras genéticas identificadas con el algoritmo de Monmonier entre A) todos los sitios, B) sólo los sitios silvestres. Las letras señalan en orden decreciente cada una de las barreras identificadas.
A nivel de la especie, se detectó un aislamiento por distancia significativo (R = 0.469, P < 0.01). Asimismo, se observó una distribución unimodal en el número de diferencias pareadas entre haplotipos, la cual se ajustó al modelo de expansión demográfica hace ( = 0.736 unidades de tiempo mutacional. Los dos estadísticos utilizados en este análisis señalaron que los datos observados se ajustan al modelo de expansión demográfica (SSD = 0.003, P = 0.48; r = 0.041, P = 0.73) (Figura 5). Con base en el intervalo de tasas de mutación considerado, el tiempo a la expansión t se estimó en 21,904 - 87,619 años atrás.
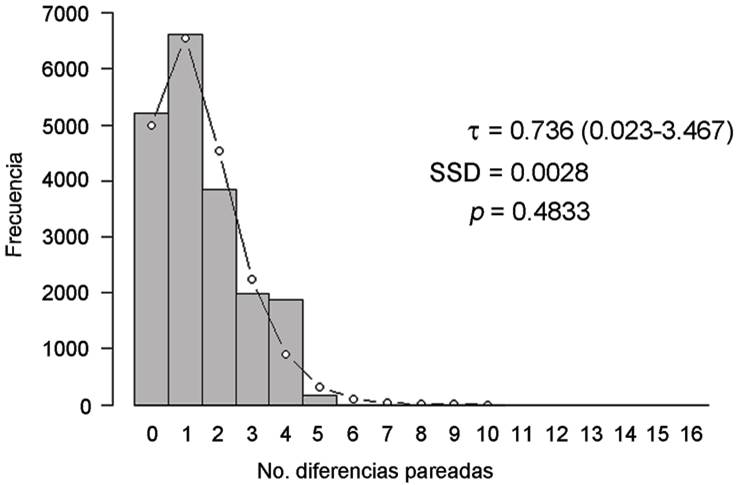
Figura 5 Análisis de distribución mismatch para las poblaciones silvestres de Crescentia alata. El histograma corresponde a las frecuencias observadas del número de diferencias pareadas entre haplotipos, mientras que las líneas, el valor del estadístico SSD y su ajuste corresponden a un modelo de expansión demográfica sucedido hace ( generaciones (entre paréntesis el intervalo de confianza al 95 % para el parámetro ( observado).
Al analizar la distribución de la diversidad, se encontró una subdivisión filogeográfica significativa de los haplotipos (R ST > G ST , Tabla 2), pero la subdivisión no se conservó al separar los datos en las dos categorías (huertos vs. poblaciones silvestres). Al comparar los sitios silvestres y los de huertos, los primeros presentaron mayor número de haplotipos que los segundos (16 vs. 7; en promedio 1.79 haplotipos por sitio vs. 1.5, respectivamente). Sin embargo, el índice h es mayor para los sitios de huertos que para los silvestres (h = 0.835 vs. h = 0.738, P < 0.01; Tabla 2), debido a que proporcionalmente el número de haplotipos detectado es mayor en relación con el tamaño de muestra (los huertos albergan 38 % de los haplotipos de las poblaciones silvestres con el 22 % del tamaño de muestra de éstas) y a que en los sitios silvestres existen numerosos haplotipos en baja frecuencia.
Tabla 2 Diversidad genética en Crescentia alata basada en 6 loci de microsatélites de cloroplasto. h = índice de diversidad haplotípica (Nei 1987); G ST = índice de estructuración con base en alelos no ordenados; RST = índice de estructuración con base en alelos ordenados (Pons & Petit 1996).
| Categoría | No. sitios | No. indivs. | No. haplotipos | h (sd) | GST (se)1 | RST (se)1 | P2 |
|---|---|---|---|---|---|---|---|
| Silvestres | 26 | 200 | 16 | 0.738 (0.028) | 0.645 (0.070) | 0.743 (0.090) | 0.102 |
| Huertos | 9 | 43 | 7 | 0.835 (0.022) | 0.655 (0.126) | 0.805 (0.171) | 0.135 |
| Total | 35 | 243 | 17 | 0.782 (0.021) | 0.650 (0.060) | 0.771 (0.093) | 0.036 |
1 Excluyendo sitios con tamaños de muestra < 3.
2 Valor de P para la prueba de hipótesis RST > GST .
Como se observa en la Figura 3B, los huertos albergaron haplotipos presentes en poblaciones silvestres simpátricas, lo cual indica el uso de germoplasma local en los huertos. Acorde con este patrón, el AMOVA mostró que sólo el 16 % de la varianza se distribuye entre poblaciones de huerto y silvestres, mientras que entre las poblaciones dentro de cada categoría se encuentra el 66 % de la varianza genética (Tabla 3). Pero a pesar de contar con los mismos haplotipos, los huertos cuentan con una estructuración ligeramente mayor (83 vs. 72 % de la variación entre poblaciones, respectivamente; Tabla 3), sobre todo si se considera la cercanía genealógica entre haplotipos (R ST = 0.805 vs. 0.743; Tabla 2). También se detectó un haplotipo único (T2) dentro de un sitio de huerto (Tequisistlán).
Tabla 3 Distribución de la varianza (%) en los distintos niveles del análisis de varianza molecular (AMOVA), considerando todas las poblaciones divididas en dos grupos (huertos y poblaciones silvestres) (a) y considerando cada grupo en particular (b y c).
| g.l. | Suma de cuadrados |
Componentes de la varianza |
% variación | |
|---|---|---|---|---|
| a) Todas las poblaciones muestreadas | ||||
| Entre grupos | 1 | 24.404 | 0.247 | 15.62 |
| Entre poblaciones dentro de grupos | 33 | 249.405 | 1.057 | 66.19 |
| Dentro de poblaciones | 208 | 57.393 | 0.276 | 17.47 |
| Total | 242 | 331.202 | 1.580 | |
| b) Huertos | ||||
| Entre poblaciones | 9 | 42.929 | 1.088 | 82.67 |
| Dentro de poblaciones | 33 | 7.536 | 0.228 | 17.33 |
| Total | 42 | 50.465 | 1.317 | |
| c) Silvestres | ||||
| Entre poblaciones | 24 | 98.082 | 0.493 | 72.25 |
| Dentro de poblaciones | 175 | 33.118 | 0.189 | 27.75 |
| Total | 199 | 131.200 | 0.682 |
Modelos de distribución. Los modelos indican cambios importantes en la distribución de C. alata durante los últimos 130 mil años. En la transición del UMG al Holoceno Medio se observa un desplazamiento de su distribución hacia el interior de la Cuenca del río Balsas y la costa Pacífico norte (Nayarit, Sonora y Sinaloa). En el Holoceno Medio se presentan condiciones adecuadas para la especie en el centro del Golfo de México y la Huasteca Potosina. Los haplotipos presentes en estas áreas indican que los linajes que colonizaron la cuenca del río Balsas provienen de la costa del océano Pacífico (haplotipo R) y del Istmo (T), mientras que los de la Huasteca y del Golfo podrían originarse en la región de Chiapas, a través del Istmo de Tehuantepec (haplotipo O).
Los modelos también sugieren que hay áreas que conservaron condiciones ambientales propicias para la presencia de la especie durante el UMG como la costa sur del océano Pacífico, el Istmo de Tehuantepec y la costa del océano Pacífico de Nicaragua (Figura 6).

Figura 6 Modelos de distribución potencial de Crescentia alata en el presente, en el Holoceno Medio y Último Máximo Glacial con base en dos modelos alternativos de circulación atmosférica (MIROC y CCSM) y el Último Inter Glacial (UIG). Las áreas en color verde representan la distribución de las condiciones climáticas adecuadas para la especie en el presente y el pasado. Se muestra el promedio obtenido de los valores de probabilidad para todas las réplicas (veinte), donde colores más oscuros representan valores promedio más altos de probabilidad.
Discusión
Las poblaciones de Crescentia alata analizadas indican que en esta especie existe una clara estructuración filogeográfica, asociada principalmente a la distribución del haplotipo ancestral R (en amarillo, cuenca del río Balsas y suroriente de México) y la diferenciación de áreas más pequeñas portadoras de otros haplotipos hacia el norte (Nayarit, Sonora y Sinaloa), al sur de México (Chiapas) y Centroamérica (Figura 4B), estas últimas caracterizadas por presentar haplotipos derivados. Los grupos mencionados coinciden con la definición de las provincias fisiográficas de la Sierra Madre del Sur, Llanura Costera del Pacífico y Cordillera Centroamericana (INEGI 2001).
En la escala geográfica, los grupos definidos por el algoritmo de Monmonier concuerdan con el patrón significativo de aislamiento por distancia, y dan cuenta del papel que juega el intervalo latitudinal que abarca la distribución de C. alata. Este patrón se ajusta también a una mayor estructuración entre poblaciones cuando se compara con su especie hermana, C. cujete (G ST = 0.645 vs. 0.384, Tabla 4; Aguirre-Dugua et al. 2018).
Tabla 4 Diversidad y estructuración de haplotipos de cpADN en algunas especies perennes domesticadas. SSRs: microsatélites; HD: diversidad haplotípica; h: índice de diversidad haplotípica de Nei (1987); G ST : índice haplotípico de diferenciación poblacional basado en alelos no ordenados (Pons & Petit 1996).
| Especie | Marcador | No. haplotipos | Diversidad haplotípica | Estructuración | Referencia |
|---|---|---|---|---|---|
| Olea europaea | SSRs | 4 | - | FST =0.720 | Besnard et al. 2002 |
| Vitis vinifera subsp. silvestris (silvestre) | SSRs | 5 8 |
HD=0.278 h=0.03-0.44 |
GST =0.631 GST =0.353 |
Grassi et al. 2006, Arroyo-García et al. 2006 |
| Vitis vinifera subsp. vinifera (cultivada) | SSRs | 6 8 |
HD=0.185-0.736 h=0.27-0.43 |
- GST =0.169 | Imazio et al. 2006, Arroyo-García et al. 2006 |
| Corylus avellana | - | 5 | - | GST =0.890 | Lascoux et al. 2004 |
| Crescentia cujete (silvestre) | SSRs | 15 | h =0.710 | GST =0.384 | Aguirre-Dugua et al. 2018 |
| Crescentia cujete (cultivada) | SSRs | 14 | h =0.546 | GST =0.518 | Aguirre-Dugua et al. 2018 |
| Crescentia alata (silvestre) | SSRs | 16 | h =0.7385 | GST =0.645 | Este estudio |
| Crescentia alata (cultivada) | SSRs | 7 | h =0.8350 | GST =0.655 | Este estudio |
La estructuración filogeográfica de la especie a lo largo de su intervalo latitudinal se asocia a los cambios en su distribución por las condiciones climáticas post-glaciales. Como se observa en la Figura 6, la especie se desplaza después del Último Máximo Glacial (de 21 mil años atrás hacia el presente) en dirección norte en ambas vertientes (Océano Pacífico y Golfo de México), y hacia el interior de la cuenca del río Balsas (Figura 6). Cabe hacer notar que, acorde con este patrón, las dos barreras geográficas más importantes separan a las poblaciones del extremo Pacífico norte (Sonora y Sinaloa, barrera a en Figura 5B), y a las poblaciones al norte y sur del Istmo de Tehuantepec (barrera b en Figura 5B). Sin embargo, estos cambios de distribución post-glaciales no estuvieron acompañados por un crecimiento demográfico. De acuerdo con nuestras estimaciones el crecimiento demográfico de la especie ocurrió antes, entre 21 y 87 mil años atrás, lo que corresponde al periodo entre el Último Inter Glacial (UIG) y el Último Máximo Glacial (UMG).
Las reconstrucciones paleoclimáticas basadas en registros bióticos y geológicos señalan que el UIG se caracterizó por una temperatura notablemente más alta que la actual (3 a 5 °C más en los polos), así como un aumento en el nivel del mar (Kopp et al. 2009), condiciones que redujeron la distribución de C. alata en el área que hoy es Centroamérica y la aumentaron en el norte de México (Figura 6). Al terminar el UIG, la temperatura fue disminuyendo hasta que se establecieron condiciones glaciales.
Durante el UMG la temperatura estaba entre 5 y 7.6 °C por debajo de la actual, con condiciones no solo más frías sino también más secas que las actuales (Hendy et al. 2016). Por lo tanto, el resultado de un crecimiento demográfico significativo durante el establecimiento de condiciones glaciales apoya nuestra hipótesis inicial de una mayor distribución de C. alata asociada al desarrollo de sabanas y pastizales en estas condiciones climáticas. Sin embargo, los modelos de distribución potencial para el UMG sugieren que este crecimiento demográfico no se acompañó de una ampliación del rango de distribución de la especie, sino más bien de un desplazamiento hacia el sur.
Asimismo, esta reconstrucción apoya nuestra hipótesis inicial, pero es un primer acercamiento a la historia de C. alata, ya que la estimación del tiempo a la expansión demográfica se basa en las tasas de mutación reportadas en la especie herbácea Arabidopsis thaliana (Brassicaceae) (Jakobsson et al. 2007). Las tasas de evolución molecular parecen estar asociadas a la duración del ciclo de vida, siendo más altas en plantas herbáceas en comparación con plantas arbóreas (aproximadamente 2.5 veces más altas, aunque dentro del mismo orden de magnitud; Smith & Donoghue 2008). Nuestras estimaciones deben por lo tanto tomarse como una aproximación general, en la que hay un intervalo amplio en la estimación debido a la incertidumbre en las tasas de mutación de los microsatélites empleados.
Después del Último Máximo Glacial existió una tendencia de aumento de la temperatura, de tal forma que durante el Holoceno Temprano y Medio (entre 6 y 9 mil años antes del presente) se registran condiciones de mayor precipitación y un aumento en la estacionalidad (es decir, en la precipitación de verano) debido al desplazamiento de la zona intertropical de convergencia (ITCZ por sus siglas en inglés) hacia el norte, aunque esta tendencia no es homogénea, pues existieron de dos a tres periodos breves de condiciones xéricas (Ruter et al. 2004, Correa-Metrio et al. 2012). El establecimiento de estas condiciones es el que da cuenta de la distribución actual de la especie en México y Centroamérica. Podemos entonces concluir que el área en que la especie perduró durante los últimos 130 mil años es la costa sur de lo que hoy es México, donde encontramos el haplotipo ancestral R (Figura 3).
A esta historia profunda podemos también añadir el efecto de las actividades antropogénicas en tiempos geológicamente recientes. Los registros palinológicos muestran una elevada proporción de Byrsonima y Poaceae en los últimos 3 a 4 mil años (Piperno 2006, Metcalfe 2006) asociados a polen de Zea mays, así como a restos de carbón que señalan incendios recurrentes (Leyden 1984, Leyden et al. 1998), utilizados no sólo en las prácticas agrícolas sino también para actividades de cacería (Piperno 2006, Montoya et al. 2011). Es muy probable que los grupos humanos, por medio de la acción del fuego y la deforestación, favorecieran la persistencia de los ambientes abiertos necesarios a Crescentia. Puig (1991) considera que las facies dominadas por Crescentia en la Huasteca son una degradación de bosques caducifolios por acción antrópica; mientras que Sarukhán (1968) propone que la mayoría de las sabanas de México tiene un origen secundario por su asociación con las actividades humanas.
La acción histórica sobre los ambientes abiertos donde se encuentran las poblaciones, a la cual se añade la incorporación de la especie a espacios antropogénicos como los huertos familiares, no parece haber modificado su estructura filogeográfica natural. La distribución de los haplotipos de cpADN muestra que los huertos en los que crece C. alata albergan haplotipos compartidos con poblaciones silvestres simpátricas (Figura 3B), lo que indica que las poblaciones locales son la fuente del germoplasma presente en los huertos y que ambas categorías de población forman parte de la misma poza génica. Esta observación cualitativa es reforzada por el análisis cuantitativo del AMOVA, en el cual se observa que la mayor parte de la variación genética se distribuye dentro de cada categoría de población, y no entre categorías. Este resultado coincide con la información recabada sobre la propagación de los árboles, que en su mayoría fueron tolerados (Tabla 1), lo cual probablemente facilita la incorporación de árboles que se desarrollan espontáneamente a partir de semillas de poblaciones vecinas. Este resultado señala que, a pesar de que el uso de los frutos de C. alata es similar al de su hermana C. cujete, su historia bajo manejo humano es claramente diferente.
Un primer factor que puede explicar este contraste es la diferencia en el rango de distribución geográfica y variabilidad morfológica de las especies. Crescentia cujete se distribuye desde México hasta el sur de la cuenca del río Amazonas, lo que implica que sus poblaciones crecen en un intervalo más amplio de condiciones ecológicas, con posible interacción con otras especies emparentadas (v.g., Crescentia amazonica; Moreira et al. 2017), y que éstas han sido expuestas a una mayor diversidad de contextos culturales que contribuyeron a su diversificación (v.g., Arango-Ulloa et al. 2009). En contraste, C. alata se distribuye sólo en México y Centroamérica, y la variación morfológica de sus frutos es mucho menor (obs. pers.). Un segundo factor posible es que la disponibilidad de linajes domesticados de C. cujete con frutos más grandes y redondos limitara el interés por seleccionar individuos locales de C. alata en esa dirección. Finalmente, otro aspecto a tomar en cuenta es que C. alata tiene usos más diversos, pues los frutos también son usados para elaborar instrumentos musicales (maracas) que requieren frutos pequeños y por sus propiedades medicinales; la madera también es altamente apreciada por su dureza y facilidad de trabajo (Santander-Cancino 1994). Esta diversidad de usos podría limitar la selección artificial específicamente dirigida a aumentar el tamaño de los frutos para usarlos como recipientes. Estas tres hipótesis (una especie con mayor distribución y variación morfológica tiene mayores probabilidades de contar con poblaciones domesticadas; la disponibilidad de un taxón domesticado reduce los esfuerzos de manejo y selección de otro taxón disponible; entre mayor número de usos, menor selección dirigida a un uso en particular) pueden evaluarse en otros grupos de plantas en los que existan especies o variedades cercanamente emparentadas que se usen de forma similar.
Además, la presencia de haplotipos compartidos con poblaciones silvestres locales sólo nos indica el origen del germoplasma cultivado, y no significa que C. alata no esté siendo sujeta a procesos de selección humana. La presencia común de la especie en áreas rurales, la tolerancia de individuos espontáneos que son conservados en vez de eliminados, así como las evidencias etnohistóricas y etnobotánicas (Aguirre-Dugua & Casas, en prensa), señalan que esta especie cuenta con alto valor cultural y puede por lo tanto estar sujeta a prácticas de manejo. Para evaluar estas prácticas es necesario analizar a escala local el grado de diferenciación morfológica entre poblaciones silvestres y de huerto, así como sus usos y relevancia cultural.
La importancia del manejo local de los árboles de C. alata presentes en los huertos se hace evidente en tres aspectos: la presencia del haplotipo A característico de C. cujete (Aguirre-Dugua et al. 2018), su alta diversidad genética y su contribución a la estructura filogeográfica global de la especie. En el primer aspecto, la presencia del haplotipo A en dos sitios de huertos indica la ocurrencia de eventos de introgresión que han tenido como consecuencia la captura de cloroplastos. En un estudio previo reportamos captura de haplotipos de cpADN de C. alata en C. cujete (Aguirre-Dugua et al. 2018), por lo que en el presente análisis la detección de haplotipos de C. cujete en C. alata (en la misma región geográfica, la costa oaxaqueña) señala que esta introgresión es bidireccional. Notoriamente, es en la población de San Mateo del Mar donde se documentó la propagación vía semilla (Tabla 1), lo que explicaría la incorporación de genotipos producto de polinización cruzada entre ambas especies.
Se ha encontrado evidencia sobre el papel del manejo humano histórico en la facilitación de estos procesos de flujo génico, centrales durante la adaptación y la diversificación de especies perennes en domesticación del continente americano y del euroasiático, en géneros como Leucaena (Fabaceae), Stenocereus (Cactaceae), Olea (Oleaceae), Phoenix (Arecaceae), Malus y Prunus (Rosaceae) (Hughes et al. 2007, Delplancke et al. 2012, Cruse-Sanders et al. 2013, Cornille et al. 2019, Flowers et al. 2019).
En el segundo aspecto, los huertos se caracterizan por presentar altos valores de diversidad haplotípica h, ya que en relación con el tamaño de muestra cuentan con un elevado número de haplotipos (incluyendo un haplotipo único) (Tabla 2). En el tercer aspecto, el índice R ST no es significativo si se toman en cuenta sólo las poblaciones silvestres, pero sí lo es si se agregan los huertos al juego de datos (Tabla 2); este resultado señala que los huertos cuentan con alta estructuración (i.e., no son homogéneos entre sí; Tabla 3) y que ésta no sólo es un reflejo de la estructura filogeográfica natural de la especie, sino que contribuye a reforzarla, ya que el cultivo se da de forma independiente a lo largo del área de distribución, sin intercambio entre zonas (véase Figura 4). Podemos por lo tanto concluir que los huertos actúan como reservorios de la diversidad de la especie, ya que en caso de que alguna población silvestre llegara a desaparecer, las poblaciones locales de huertos podrían actuar como fuente de germoplasma y, evolutivamente, mantener la identidad genética de las poblaciones de la región.
El papel de los huertos familiares en la conservación de diversidad genética de especies perennes templadas y tropicales ha sido documentado en taxa como el higo (Ficus; Achtak et al. 2010), la ciruela (Spondias; Miller & Schaal 2006), así como en Olea europaea (Oleaceae) o Corylus avellana (Betulaceae), taxa en los que el manejo humano es responsable de una mayor proporción de varianza distribuida entre poblaciones (Tabla 4). Este patrón señala la importancia de conservar la práctica de mantener estos árboles dentro de los huertos familiares en una amplia área geográfica para que éstos sigan actuando como reservorios de diversidad genética.
Material Suplementario
El material suplementario puede ser consultado aquí: https://doi.org/10.17129/botsci.3158











 nueva página del texto (beta)
nueva página del texto (beta)



