Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Botanical Sciences
versão On-line ISSN 2007-4476versão impressa ISSN 2007-4298
Bot. sci vol.93 no.3 México Set. 2015
https://doi.org/10.17129/botsci.247
Florística y taxonomía
Opuntia leiascheinvariana, una nueva especie de Cactaceae del estado de Hidalgo, México
Opuntia leiascheinvariana, a new species of Cactaceae from the state of Hidalgo, Mexico
César Ramiro Martínez–González1, Clemente Gallegos–Vazquez2, Isolda Luna–Vega1, 3 y Ricardo García–Sandoval1
1 Laboratorio de Biogeografía y Sistemática, Departamento de Biología, Facultad de Ciencias, Universidad Nacional Autónoma de México, Ciudad Universitaria, Delegación Coyoacán, 04510 México, D.F., México.
2 Centro Regional Universitario Centro Norte, Universidad Autónoma Chapingo, Cruz del Sur núm. 100, Colonia Constelación, El Orito, 98085, Zacatecas, Zacatecas, México.
3 Autora para correspondencia: luna.isolda@gmail.com
Recibido: 10 de septiembre de 2014.
Aceptado: 7 de noviembre de 2014.
Resumen
Se describe una nueva especie silvestre de Opuntia (L.)Mill. (Cactaceae) de frutos comestibles ácidos (xoconostles), conocida localmente como "xoconostle matizado", basados en micromorfología (areola, espina, glóquida, polen y semilla) y morfología externa. Se compara morfológicamente con otras tres especies silvestres distribuidas en la misma región: O. joconostle, O. oligacantha y O. matudae. Con base en el análisis filogenético de un gene de cloroplasto, un espaciador intergénico de cloroplasto y un marcador nuclear se reconoce a la especie como un grupo monofilético dentro del grupo de Opuntia sensu stricto.
Palabras clave: ITS, matK, micromorfología, Opuntia, plantas endémicas, relaciones filogenéticas, trnL–trnF, xoconostle.
Abstract
We describe a new species of a wild Mexican Opuntia (L.)Mill. (Cactaceae) producer of edible acid fruits (xoconostles), locally known as "xoconostle matizado", based on micromorphology attributes (epidermis, spines, gloquids, pollen and seed) and macromorphology. This species is compared with another three species of wild xoconostles distributed in the same region: O. joconostle, O. oligacantha, and O. matudae. Based on the phylogenetic analysis of one chloroplast genes, one plastid intergenic spacer and one nuclear molecular marker the new species was recognized as a monophyletic group inside Opuntia sensu stricto.
Key words: endemic plants, ITS, matK, micromorphology, trnL–trnF, Opuntia, phylogenetic relationships, xoconostle.
México constituye uno de los centros de diversidad más importantes de cactáceas y en él se alcanza la más alta diversidad a nivel continental (Bravo–Hollis, 1978; Hunt, 1999; Anderson, 2001). Cabe destacar la porción norte del país donde se ubica el desierto chihuahuense, una de las regiones con mayor riqueza de cactáceas, además de un alto endemismo a nivel específico y genérico (Godínez–Álvarez y Ortega–Baes, 2007). La zona árida queretano–hidalguense contiene uno de los principales ensambles de cactáceas endémicas, varias de las cuales están dentro de alguna categoría de riesgo dentro de la Norma Oficial Mexicana vigente (SEMARNAT, 2010) (Hernández et al., 2004; Sánchez–Martínez et al., 2006; Hernández–Oria et al., 2007). En las zonas semiáridas, matorrales xerófilos, pastizales y bosque tropical caducifolio del estado de Hidalgo se encuentran distribuidas varias de las especies del género Opuntia (L.) Mill. Se estima que en México crecen unas 93 especies de este género (Hunt, 1999), aunque este número ha variado dependiendo del autor, desde 58 (Britton y Rose, 1919–1923), 66 (Bravo–Hollis, 1937) y hasta 76 (Guzmán et al., 2003).
En México se designa como xoconostle (del náhuatl: xoco = ácido; noxtle = tuna) a los frutos ácidos (Bravo–Hollis, 1978), cuyas paredes gruesas son consumidas por diferentes grupos étnicos y son utilizadas en la medicina tradicional por su efecto hipoglicémico, para el control del colesterol y reducción de la obesidad (Gallegos–Vázquez et al., 2012). Diferentes autores reconocen entre ocho (García–Pedraza et al., 2005) y nueve especies de Opuntia con frutos ácidos (xoconostles) (Olivares–Orozco, 2003; Scheinvar et al., 2010). Otras dos especies de xoconostles fueron reconocidas por Arias et al. (1997) y Pérez et al. (2005) para la reserva de Tehuacán–Cuicatlán (Opuntia tehuacana S. Arias & U.Guzmán) y para Oaxaca (Opuntia olmeca Joel Pérez, J. Reyes & Brachet).
Al realizar un estudio de los nopales silvestres con frutos ácidos (xoconostles) en el estado de Hidalgo (Martínez–González, 2010), se encontraron poblaciones de una nueva especie endémica y de distribución restringida, conocida localmente como "xoconostle matizado". Parte del área de distribución de esta especie se encuentra alterada por actividades agrícolas y, debido a las actividades humanas ahí realizadas, se considera a esta especie como en riesgo.
Según los recientes estudios de Majure et al. (2012) y Majure y Puente (2014) basados en morfología y análisis filogenéticos, la tribu Opuntieae consiste de siete géneros, entre los cuales está el género Opuntia. Majure y Puente (2014) sostienen que la delimitación de especies dentro de la tribu Opuntieae, especialmente de Opuntia, es taxonómicamente muy problemática, debido principalmente: (1) al gran porcentaje de hibridación que existe, (2) la falta de conteos cromosómicos y (3) la falta de datos filogenéticos, entre otras. Las especies de Opuntia tienen un alto nivel de hibridación y subsecuente especiación vía poliploidía, lo que da como resultado que muchas especies sean polifiléticas. A este respecto, Segura et al. (2007) reportaron altos niveles de ploidía en 23 especies de Opuntia mexicanas, incluyendo varias especies de xoconostles.
El presente estudio tiene como objetivos: (a) describir esta especie de Opuntia como nueva para la ciencia, la cual puede reconocerse con base en características de las aréolas, espinas, glóquidas, epidermis y semillas, (b) comparar la nueva especie con otras morfológicamente similares que cohabitan en la zona y presentan frutos ácidos (xoconostles), con el fin de brindar elementos que coadyuven a la correcta determinación de ejemplares en campo y (c) ubicar filogenéticamente a la nueva especie con base en el análisis filogenético de dos marcadores plastídicos (matK y trnL–F) y un marcador nuclear (ITS), con respecto al muestreo taxonómico empleado por Hernández–Hernández et al. (2011) y Majure et al. (2012).
Área de estudio
El área de estudio se localiza en Villa de Tezontepec, Municipio de Villa Tezontepec, Hidalgo. Las coordenadas geográficas del área son 19° 52' 55" – 19° 53' 11'' N, 98° 49' 16'' – 98° 48' 53'' O, con una elevación promedio de 2,329 m. En esta área existe un clima BS1kw (w)(i')g, que de acuerdo con la clasificación de Köppen (1948) modificada por García (1973) corresponde a un clima semiseco, templado, con lluvia de verano, con un porcentaje de lluvia invernal menor a 5 %, isotermal y con el mes más caliente del año antes de junio. La vegetación predominante es el matorral xerófilo (sensu Rzedowski, 1978), con suelos sedimentarios.
Materiales y Métodos
Se revisó literatura sobre xoconostles de Opuntia, principalmente los siguientes trabajos: Bravo–Hollis (1978), Britton y Rose (1919–1923), Parfitt y Pinkava (1988), Pinkava (1996, 2002), Guzmán et al. (2003), Hunt (2006), Scheinvar (1974, 1981, 1982, 1985, 1987, 1999, 2002, 2004), Scheinvar y Rodríguez (2003), Scheinvar y Olalde–Parra (2008), Scheinvar y Manzanero (2009) y Scheinvar et al. (2010).
Se revisaron especímenes de herbario de xoconostles en las siguientes colecciones: Universidad Nacional Autónoma de México (MEXU), Instituto de Botánica Universidad de Guadalajara (IBUG), Instituto de Investigaciones de Zonas Desérticas de la Universidad Autónoma de San Luis Potosí (SLPM), Instituto Nacional de Estadística y Geografía (INEGI), Instituto de Ecología, Centro Regional del Bajío en Michoacán (IEB), Universidad Juárez Autónoma de Tabasco (UJAT), Museo Nacional de Historia Natural, Instituto Smithsoniano (US) y Jardín Botánico de Nueva York (NY).
La descripción morfológica fue realizada sobre material fresco recolectado por los autores, al igual que de las fotografías tomadas en campo. La descripción de la micromorfología se realizó en ejemplares frescos previamente tratados con solución FAA (formol, alcohol y ácido acético) durante 48–60 horas. Se fijaron aréolas, espinas, glóquidas, epidermis y semillas.
Las muestras se obtuvieron a partir de plantas provenientes de Villa de Tezontepec, estado de Hidalgo, México. Los métodos para preparar las muestras fueron los sugeridos por Martínez–González et al. (com pers.). El material se lavó con agua destilada para eliminar los excedentes de la solución FAA. Se deshidrataron con una serie diluciones de alcohol etílico (ETOH), del 50, 70, 95 y 100 %, en lapsos de 10 horas para cada dilución. Posteriormente se sumergieron en acetona durante 100 horas para eliminar ceras. Posteriormente se cambió la acetona por alcohol etílico absoluto. Con las estructuras ya limpias se realizó un secado por punto crítico y se montaron en los portamuestras para ser bañados con oro y fueron observadas en un Microscopio Electrónico de Barrido, marca Hitachi, modelo S–2460N. El polen recién colectado fue montado directamente en los portamuestras. Los valores micromorfométricos de las espinas se obtuvieron a partir de 50 medidas por especie utilizando el analizador de imágenes Image Tool versión 3.0 (Wilcox et al., 2002). Ocho espinas fueron recolectadas de diferentes individuos de la especie en estudio y de las otras tres especies productoras de xoconostles que viven en simpatría para llevar a cabo el análisis micromorfológico.
Extracción de DNA. El DNA genómico se extrajo a partir de 20–50 mg de tallo por el método de CTAB (Weising et al., 1995) con algunas modificaciones que permitieron eliminar de las muestras una mayor cantidad de compuestos fenólicos y polisacáridos. El DNA se cuantificó en un Nanodrop (Thermo, USA). De cada una de las muestras se prepararon diluciones a 20 ng para la amplificación de los genes.
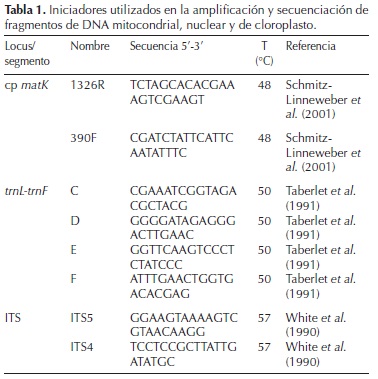
Amplificación de las secuencias. Para la amplificación de la región del ITS se utilizaron los iniciadores universales ITS5 e ITS4 (White et al., 1990), el espaciador intergénico trnL–trnF se utilizaron los iniciadores universales trnL–F (Taberlet et al., 1991) y el gen matK, que codifica una madurasa involucrada en el proceso de eliminación de intrones durante la maduración de transcritos multicistronicos del cloroplasto (Vogel et al., 1999), con los iniciadores universales 390F y 1326R (Schmitz–Linneweber et al., 2001). La mezcla de reacción para PCR se preparó en un volumen final de 25 µL conteniendo: buffer de la enzima 1 × Taq DNA polimerasa, 0.8 mM deoxinucleósido trifosfatos (0.2 mM cada uno), 100 ng DNA, 20 pmol de cada iniciador y 2 unidades de GoTaq DNA (Promega, USA). Las amplificaciones se realizaron con un ciclo inicial de desnaturalización a 96 °C por 2 min; 35 ciclos de desnaturalización a 94 °C por 1 min, hibridación con los iniciadores a 57, 50 y 48 °C respectivamente durante 1 min (Tabla 1), seguido de una extensión final de 5 min a 72 °C. Todas las reacciones de PCR se llevaron a cabo en un Peltier Thermal Cycler PTC–200 (BIORAD, México), las amplificaciones se verificaron por electroforesis en un gel de agarosa al 1.2 % preparado con 1 × TAE buffer (Tris Acetate–EDTA) y corrido a 87 V cm–3 durante 1 h. El gel se tiñó con Gel red (Biotium, USA) y las bandas se visualizaron en un transiluminador (Infinity 3000 Vilber Lourmat, Germany). Los productos amplificados se limpiaron con el kit de purificación Wizard SV gel y el sistema PCR clean up (Promega, EEUU), siguiendo las instrucciones del fabricante, se cuantificaron y se prepararon para la reacción de secuenciación usando el Bigdye terminator v. 3.1 (Applied Biosystem). Estos productos se secuenciaron en el Applied Biosystems modelo 3130XL (Applied BioSystems, EEUU).
Ensamble de las secuencias. Las secuencias de ambas hebras de cada uno de los genes se ensamblaron y editaron usando BioEdit versión 7.0.5 (Hall, 1999) para crear una secuencia consenso. Esta secuencia consenso se comparó con las depositadas en GenBank del National Center for Biotechnology Information (NCBI), empleando la herramienta BLASTN 2.2.19 (Zhang et al., 2000).
Análisis filogenético. La ubicación filogenética de la nueva especie en el clado Opuntia sensu lato fue corroborada mediante un análisis filogenético exploratorio realizado con base en el alineamiento de Hernández–Hernández et al. (2011, resultados no mostrados). Para investigar las relaciones filogenéticas de la nueva especie dentro del clado de Opuntia sensu stricto se elaboró un alineamiento con base en el muestreo taxonómico para especies no poliploides empleado por Majure et al. (2012). El muestreo taxonómico se ajustó para obtener máxima cobertura con los marcadores empleados: el gen del cloroplasto matK, el interespaciador nuclear ribosomal nrITS y el espaciador intergénico del cloroplasto trnL–F. A la matriz ajustada se le adicionaron tres secuencias de matK, tres secuencias de nrITS y tres secuencias de trnL–F provenientes de tres ejemplares designados como tipos nomenclaturales para O. leiascheinvariana (número de colecta 5, 6 y 7, C.R. Martínez–González), para un total de nueve secuencias generadas para este estudio (KM507350–KM507358). El alineamiento resultante fue analizado empleando los criterios de máxima parsimonia e inferencia Bayesiana. El análisis de máxima parsimonia se realizó con PAUP* 4b10 (Swofford, 2002) con 500 réplicas heurísticas, MAXTREE ajustado para auto–incremento y los parámetros nchuck = 2 y chuckscore = 100 ajustados para optimizar el uso de la memoria. Complementariamente se calculó el soporte de caracteres con una prueba de bootstrap no paramétrico (Felsenstein, 1985) con 500 réplicas, cada una con búsquedas heurísticas con 40 réplicas, MAXTREE ajustado para auto–incremento y los parámetros nchuck = 2 y chuckscore = 100 ajustados para optimizar la memoria. El análisis de inferencia Bayesiana se realizó con MrBayes 3.2.2 (Ronquist et al., 2012) declarando tres particiones correspondientes a los tres marcadores empleados, empleando la estrategia de salto reversible para la selección de modelos de sustitución (Huelsenbeck et al., 2004). La heterogeneidad de tasas se modeló con referencia a la distribución Gamma empleando cuatro categorías, adicionalmente se incluyó un parámetro para el porcentaje de sitios invariantes. Todos los parámetros fueron desligados entre particiones; la cadena de MCMC se ajustó para cuatro cadenas por duplicado durante 3.5 millones de generaciones con un porcentaje de burn–in del 25 %.
Resultados
Opuntia leiascheinvariana Martínez–González & Gallegos–Vázquez sp. nov.
Tipo: México. Hidalgo. Municipio Villa de Tezontepec, Villa de Tezontepec, 19° 53' 0.7'' N, 98° 48' 46'' O, 2,329 m s.n.m., César Ramiro Martínez–González 5 (Holotipo, MEXU; Isotipos, ARIZ, MO y CHAPA). Figuras 1, 2, 3 y 4.
Diagnosis. The new species is similar to the sympatric species Opuntia joconostle F.A.C. Weber ex Diguet and Opuntia matudae Scheinvar by the epidermis that is pubescent, but differs by the following characters: cladodes obovate, the terminal ones 18–28 × 11–18 cm, ca. 2.2 cm thick; areoles arranged in 11 and 13 series; spines 2 to 6 in all areoles, subcircular, porrect and divergent, white to slightly yellowish apex, emerging from the areole base; style white, stigma lobes 8–9, light green; pericarp oblong, with high tubercles; fruits obovate, with a shiny skin, reddish–pink and variegated green, with thick walls, acidic, white–pink with green and red streaks, edible; funicles semi–dry, tasteless; mature fruit persistent, observed on cladodes for 5–6 months or more.
Descripción morfológica. Planta arbustiva de aprox. 1 m de altura (Figura 1A). Tronco de 12 × 25 cm, corteza glabra, amarillenta. Cladodios obovados, de 18–28 × 11–18 × ca. 2.2 cm de espesor, verde–grisáceos, cubiertos de una capa cerosa, podarios ligeramente elevados (Figura 1B). Epidermis pubescente, estomas paracíticos (Figura 3E, F). Aréolas negruzcas, dispuestas en 11–13 series y distantes 1.6 cm entre sí, subcirculares, de 0.3–0.4 cm, con tricomas cortos (Figura 3A). Glóquidas amarillentas, cortas, dispuestas en los bordes de la aréola, retrobarbadas (Figura 3D). Espinas 2–6, dispuestas en todas las aréolas, setosas, aplanadas y encorvadas en la base, porrectas y divergentes, desiguales, de 0.6–2.4 cm de largo, blancas con el ápice ligeramente amarillento (Figura 3B). Cladodios juveniles con tubérculos subcirculares ligeramente prominentes, aréolas con tricomas cortos, rosados, las superiores con 1–2 tricomas setosos en la parte inferior de la aréola, blancos, hojas basales subuladas naciendo de la base de las areolas, erectas, color rojo–anaranjado, con ápice uncinado (Figura 1C). Botones florales agudos, con segmentos del perianto rojizos, pericarpelo oblongo, con tubérculos elevados, aréolas con tricomas largos, rosados y glóquidas marrón, algunas aréolas con cerdas muy largas, blancas con la base marrón o todas marrón, con hoja basal porrecta, rojiza (Figura 2A). Flores amarillentas con tonos blancos (Figura 2B), la fauce verdosa (Figura 2C), 6.5–7.9 cm de largo, pericarpelo oblongo, de ca. 4.7 × 2.7 cm, aréolas dispuestas en 7 series, distantes 0.4 cm entre sí, con escama basal crasa; segmentos exteriores del perianto oblongos, con ápice agudo, verdes en la base y el ápice rojo; segmentos interiores espatulados con el ápice recto u obtuso, mucronado, blancos con la base amarilla; estambres alcanzan 1/3 del largo del perianto, filamentos blancos con la base verdosa, anteras blancas; estilo claviforme, ca. 2.4 cm de largo, blanco, lóbulos del estigma 8–9, papilosos, de ca. 0.5 cm de largo, verdes con estría media rosada (Figura 2D). Granos de polen poligonales (Figura 4A), 103.86 µm de diámetro, 7353.64 µm2 de área, 15.91 µm de distancias entre cada apertura distal (poros) y con un diámetro de 17.86 µm (Figura 4B), 14–16 poros, con muros anchos de 1.61 µm y glabros (Figura 4C). Frutos obovados (Figura 2E), de 6–8.1 cm de largo, rosa–rojizos variegados de verde, con aréolas pequeñas, dispuestas en 6–7 series, distantes 0.9 cm y las aréolas distantes 0.7 cm entre sí, espinas ausentes, tricomas largos, grisáceos, glóquidas dispuestas en el centro, porrectas, color marrón, paredes anchas, de 0.5–1.4 cm, blanco–rosadas con estrías verdes y rojas; cicatriz floral hundida en 0.5 cm, estriada; funículos rosados, semisecos e insípidos (Figura 2F). Semillas lenticulares (Figura 4D), color crema, ca. 0.39 cm de diámetro, región hilo–micropilar basal (Figura 4E), arilo lateral irregular con células irregulares (Figura 4F).
Etimología. El nombre de esta especie es dedicado a la destacada cactóloga Dra. Léia Scheinvar, por sus valiosas aportaciones al diverso mundo de los nopales mexicanos.
Nombre común. Los lugareños la nombran "xoconostle matizado" por su color rosa–rojizo matizado de manchas verdes en el fruto.
Descripción micro–morfológica: Espinas con células epidérmicas lignificadas largas y anchas en la parte apical, 119.36 µm y 27.28 µm respectivamente (Figura 3B), parte media de la espina con células epidérmicas de una longitud de 105.40 µm y 22.32 µm de ancho, células epidérmicas ligeramente fisuradas con textura lisa (Figura 3C). Glóquidas con células epidérmicas retrobarbadas con ápices elevados (Figura 3D). Epidermis pubescente, tricomas cónicos y falcados, células epidérmicas con paredes anticlinales con textura lisa (Figura 3E) y estomas paracíticos (Figura 3F). Granos de polen poligonales, periporados, suprarreticulados (Figura 4A), 14 poros, con muros anchos y verrucosos. Semilla lenticular con arilo lateral irregular, región hilo–micropilar lateral, micrópilo y funículo inclusos, arilo lateral angosto e irregular (Figura 4E).
Análisis filogenético. El alineamiento analizado incluyó 52 especies y 1,951 caracteres totales, 1,787 caracteres constantes y 55 caracteres informativos para parsimonia. Los números de acceso de Genebank para las especies muestreadas se incluyen en el Apéndice 1. El análisis con máxima parsimonia de esta matriz resultó en 356 árboles de 201 pasos de longitud (indice de consistencia = 7209, indice de homoplasia = 0.2791, indice de consistencia rescalado = 0.8064, indice de retención = 0.9158). El consenso estricto de estos árboles no permitió reconocer la ubicación de la nueva especie debido a la falta de resolución (resultados no mostrados). El consenso de mayoría recupera a todos los representantes de O. leiascheinvariana como un grupo monofilético (con una frecuencia del 68 % de bootstrap) dentro de un clado que incluye a Opuntia pachyrrhiza H.M. Hern. y Opuntia stenopetala Engelm. (especies que pertenecen al grupo Basilares sensu Majure et al., 2012) con una frecuencia del 65 % en la prueba de bootstrap. Este clado es recuperado en el análisis de parsimonia sin ninguna afinidad reconocible debido a la severa falta de resolución, muy probablemente derivada de la falta de caracteres informativos para parsimonia. Análisis Bayesiano: después de 3.5 millones de generaciones, la desviación estándar entre las cadenas se estabilizó en 0.007, indicativo de que la MC3 alcanzó una fase estacionaria. Para confirmar que el tamaño de muestra era suficiente se examinó el archivo de parámetros en Tracer 1.6 (Rambaut et al., 2014) corroborándose que todos los parámetros tenían un tamaño de muestra estimado por arriba de 1,500. Las probabilidades posteriores (PP) obtenidas fueron calculadas en el consenso estricto producido por MrBayes e indicadas sobre el árbol de máxima credibilidad de clados (MCCT por su acrónimo en inglés; Heled y Bouckaert, 2013). En este caso en particular el uso de la MCCT permitió visualizar más fácilmente las relaciones inferidas por el análisis, aún cuando existe poca señal en la matriz y la resolución en general es deficiente (García–Sandoval, 2014). El análisis de inferencia Bayesiana recuperó a O. leiascheinvariana como un grupo monofilético con una probabilidad posterior de 0.95, dentro del mismo clado recuperado con parsimonia (que incluye a las Basilares Opuntia pachyrrhiza y O. stenopetala), con una probabilidad posterior de 0.95 (Figura 5).
Distribución y fenología. Hasta el momento sólo se tiene referencia de que Opuntia leiascheinvariana se distribuye únicamente en el Municipio Villa de Tezontepec, Hidalgo, México. Florece en marzo–abril y la fructificación en noviembre. Los frutos persisten durante 5–6 meses en los cladodios, de donde pueden ser consumidos por animales domesticados (bovinos) o silvestres. Las poblaciones silvestres de O. leiascheinvariana contribuyen a evitar la erosión del suelo y son utilizados como refugio por varias especies de aves, reptiles y pequeños mamíferos (observaciones directas del primer autor).
Conservación. Con base en las categorías de las Listas Rojas de la IUCN (IUCN, 2014), esta especie podría ser incluida como vulnerable (VU), por presentar una distribución geográfica restringida, por el riesgo al que está sometida debido a las distintas actividades humanas que ahí se desarrollan y los efectos del cambio climático actual. Esta especie se introdujo y está siendo cultivada en el Jardín Botánico del Instituto de Biología de la UNAM, en la "Colección Nacional de Especies Silvestres de Nopales Mexicanos", en el camellón OP–7 y con el registro CM5.
En la misma región se distribuyen otras tres especies silvestres de Opuntia con frutos ácidos (xoconostles): Opuntia joconostle F.A.C.Weber, Opuntia oligacantha Förster y Opuntia matudae Scheinvar, cuyas características morfológicas distintivas se comparan con la nueva especie (Tabla 2), diferenciándose ésta de las tres primeras por el mayor número de series de aréolas (a excepción de O. oligacantha), porque las anteras tienen filamentos blancos con la base verdosa, por el fruto rosa–rojizo variegado de verde y por las semillas lenticulares. Las características que las cuatro especies comparten son el hábito arbustivo, frutos ácidos, con paredes anchas y que no se desprenden del cladodio cuando maduran. Se ha observado que las aves no comen las paredes del fruto evitando la acidez y que las perforan en busca de los funículos de sabor insípido.
Conclusiones
La posición filogenética de esta especie dentro de Opuntia s.str. no es concluyente. El análisis de parsimonia no fue capaz de recuperar la misma topología reportada por Majure et al. (2012), probablemente debido a la falta de caracteres informativos (55/1,951 totales). Esta falta de señal filogenética fue previamente reportada para esta misma combinación de marcadores en este mismo nivel de estudio (Majure et al., 2012), por lo que la exploración de nuevos marcadores más informativos para este grupo sigue siendo necesaria. En contraste, el análisis de inferencia Bayesiana fue capaz de recuperar varios de los grupos reportados por Majure et al. (2012), aunque en algunos casos las relaciones entre ellos fueron diferentes debido a que diversos clados, reportados con soporte bajo o nulo en Majure et al. (2012) no fueron recuperados en nuestro análisis, debido posiblemente a la poca señal filogenética en el alineamiento.
A pesar de la baja proporción de caracteres informativos, los dos criterios de optimación empleados (parsimonia e inferencia Bayesiana) recuperaron al nuevo taxón como un grupo monofilético, estrechamente emparentado con especies del grupo de las Basilares. Si bien el objetivo del presente trabajo no es inferir las relaciones filogenéticas en Opuntia, los resultados obtenidos sustentan claramente a O. leiascheinvariana como una especie independiente dentro de Opuntia s.str.
Por otro lado cabe mencionar que Majure et al. (2012) y Majure y Puente (2014) reportan que muchas de las especies dentro del clado de las Basilares tienen cladodios pubescentes, lo cual es el caso de la especie descrita y resulta congruente con su ubicación en el árbol filogenético.
Según estos autores, las Basilares son el grupo más antiguo de especies del clado Norteamericano de Opuntia y los miembros de este clado han estado involucrados en la producción de múltiples híbridos, así como de especies poliploides, por lo que el primer autor del presente estudio actualmente lleva a cabo investigaciones para profundizar la caracterización genética de esta especie, con el fin de dilucidar con precisión su historia filogenética dentro de un grupo en que la evolución reticulada se ha documentado repetidamente (v.gr. Segura et al., 2007; Majure et al., 2012; Majure y Puente, 2014).
Agradecimientos
Este manuscrito fue mejorado sustancialmente a través de las constructivas sugerencias de Teresa Terrazas, Rodrigo Duno de Stefano y dos árbitros anónimos. Agradecemos los recursos otorgados y facilidades brindadas para la realización del presente trabajo, a la Red del Nopal perteneciente al Sistema Nacional de Recursos Fitogenéticos para la Alimentación y la Agricultura del Servicio Nacional de Inspección y Certificación de Semillas de la Secretaría de Agricultura, Ganadería, Desarrollo Rural, Pesca y Alimentación, a la línea prioritaria de investigación número cinco del Colegio de Posgraduados, Campus Montecillo, por las facilidades para la realización de los estudios moleculares. La M. en C. Berenit Mendoza tomó de fotografías bajo el microscopio electrónico de barrido del Instituto de Biología de la Universidad Nacional Autónoma de México.
Literatura citada
Anderson E.F. 2001. The Cactus Family. Timber Press. Portland. [ Links ]
Arias S., Gama S. y Guzmán U. 1997. Cactaceae A.L. Juss. Fascículo 14. Flora del Valle de Tehuacán–Cuicatlán. Universidad Nacional Autónoma de México, México.
Bravo–Hollis H. 1937. Las Cactáceas de México. Universidad Nacional Autónoma de México, México. [ Links ]
Bravo–Hollis H. 1978. Las Cactáceas de México. Vol. 1. Universidad Nacional Autónoma de México, México. [ Links ]
Britton N.L. y Rose J.N. 1919–1923. The Cactaceae. 4 vols. Carnegie Institute Washington Publications 248, Washington D.C. [ Links ]
Felsenstein J. 1985. Confidence limits on phylogenies: an approach using bootstrap. Evolution 39:783–791. [ Links ]
Gallegos–Vázquez C., Scheinvar L., Nuñez–Colín C.A. y Mondragón–Jacobo C. 2012. Morphological diversity of xoconostles (Opuntia spp.) or acidic cactus pears: a Mexican contribution to functional foods. Fruits 67:109–120. [ Links ]
García E. 1973. Modificaciones al Sistema de Clasificación Climática de Köppen (para adaptarlo a las condiciones de la Republica Mexicana). Instituto de Geografía, Universidad Nacional Autónoma de México, México. [ Links ]
García–Pedraza L.G., Reyes–Agüero J.A., Aguirre–Rivera J.R. y Pinos–Rodríguez J.M. 2005. Preliminary nutritional and organoleptic assessment of xoconostle fruit (Opuntia spp.) as a condiment or appetizer. Italian Journal of Food Science 17:333–340. [ Links ]
García–Sandoval R. 2014. Why some clades have low bootstrap frequencies and high Bayesian posterior probabilities. Israel Journal of Ecology and Evolution 60:41–44. [ Links ]
Godínez–Álvarez H. y Ortega–Baes P. 2007. Mexican cactus diversity: environmental correlates and conservation priorities. Boletín de la Sociedad Botánica de México 81:81–87. [ Links ]
Guzmán U., Arias S. y Dávila P. 2003. Catálogo de cactáceas mexicanas. Universidad Nacional Autónoma de México – Comisión Nacional para el conocimiento y Uso de la Biodiversidad, México. [ Links ]
Hall T.A. 1999. BioEdit: a user–friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series 41: 95–98. [ Links ]
Heled J. Yy Bouckaert R.R. 2013. Looking for trees in the forest: summary trees from posterior samples. BMC Evolutionary Biology 13: 221. [ Links ]
Hernández–Hernández T., Hernández H.M., De–Nova J.A., Puente R., Eguiarte L.E. y Magallón S. 2011. Phylogenetic relationships and evolution of growth form in Cactaceae (Caryophyllales, Eudicotyledoneae). American Journal of Botany 98:44–61. [ Links ]
Hernández H.M., Gómez–Hinostrosa C. y Goettsch B. 2004. Checklist of Chihuahuan Desert Cactaceae. Harvard Papers in Botany 9:51–68. [ Links ]
Hernández–Oria J.G., Chávez–Martínez R. y Sánchez–Martínez E. 2007. Factores de riesgo en las Cactaceae amenazadas de una región semiárida en el sur del desierto chihuahuense, México. Interciencia 32:728–734. [ Links ]
Huelsenbeck J.P., Larget B. y Alfaro M.E. 2004. Bayesian phylogenetic model selection using reversible jump Markov Chain Montecarlo. Molecular Biology and Evolution 21:1123–1133. [ Links ]
Hunt D. (comp.) 1999. CITES Cactaceae Checklist. Royal Botanical Gardens, Kew. [ Links ]
Hunt D.R., con Taylor N y Charles G. 2006. The New Cactus Lexicon. 2 vols. DH Press, Milborne Port. [ Links ]
IUCN. 2014. The IUCN Red List of Threatened Species. Version 2014.1 <www.iucnreslist.org> (consultado en febrero de 2014). [ Links ]
Köppen W.P. 1948. Climatología, con un estudio de los climas de la tierra. Fondo de Cultura Económica. México. [ Links ]
Majure L.C., Puente R., Griffith M.P., Judd W.S., Soltis P.S. y Soltis D.E. 2012. Phylogeny of Opuntia s.s. (Cactaceae): clade delineation, geographic origins, and reticulate evolution. American Journal of Botany 99:847–864. [ Links ]
Majure L. y Puente R. 2014. Phylogenetic relationships and morphological evolution in Opuntia s.str. and closely related members of tribe Opuntieae. Succulent Plant Research 8:9–30. [ Links ]
Martínez–González C.R. 2010. Especies de Opuntia Mill. (Cactaceae), productoras de xoconostle en Villa de Tezontepec, Hidalgo. Tesis de licenciatura. Universidad Nacional Autónoma de México, México, D.F. 119 pp. [ Links ]
Olivares–Orozco J.L., Zavaleta–Beckler P., Chimal–Hernández A., Montiel–Salero D., Fierro–Álvarez A. y Scheinvar L. 2003. Xoconostle: biología y manejo agronómico. Serie Académicos CBS No. 45. Dirección de Ciencias Biológicas y de la Salud. Universidad Autónoma Metropolitana–Unidad Xochimilco, México. [ Links ]
Parfitt B.D. y Pinkava D.J. 1988. Nomenclatural systematic reassessment of Opuntia engelmanii and Opuntia lindheimeri (Cactaceae). Madroño 35:342–349. [ Links ]
Pérez–Crisanto J., Reyes–Santiago J. y Brachet–Ize C. 2005. Opuntia olmeca, una nueva especie de la familia Cactaceae para el estado de Oaxaca, México. Cactáceas y Suculentas Mexicanas 50:89–95. [ Links ]
Pinkava D.J. 1996. Nomenclatural changes in Opuntia (Cactaceae). Haseltonia 4:103–104. [ Links ]
Pinkava D.J. 2002. On the evolution of the continental North American Opuntioideae. En: Hunt D. y Taylor N. Pp. 59–98. Studies in the Opuntioideae (Cactaceae). Succulent Plant Research. Vol 6. DH Books, Milborne Port. [ Links ]
Rambaut A., Suchard M.A., Xie D. y Drummond A.J. 2014. Tracer v1.6. <http://beast.bio.ed.ac.uk/Tracer> (consultado Diciembre 2014). [ Links ]
Ronquist F., Teslenko M., Van der Mark P., Ayres D.L., Darling A., Höhna S., Larget B., Liu L., Suchard M.A., Huelsenbeck J.P. 2012. MrBayes 3.2: efficient Bayesian phylogenetic inference and model choice across large model space. Systematic Biology 61:539–542. [ Links ]
Rzedowski J. 1978. Vegetación de México. Limusa, México. [ Links ]
Sánchez–Martínez E., Chávez–Martínez R.J., Hernández–Oria J.G. y Hernández–Martínez M.M. 2006. Especies de Cactáceas Prioritarias para la Conservación en la Zona Árida Queretano Hidalguense. Consejo de Ciencia y Tecnología del Estado de Querétaro, Querétaro, México. http://www.concyteq.edu.mx/concyteq/publicaciones/jb/pdf/conservacion_zona_QH.pdf (Consultado Diciembre de 2014). [ Links ]
Scheinvar L. 1974. Opuntia heliabravoana, una especie nueva de Cactaceae. Anales del Instituto de Biología, Universidad Nacional Autónoma de México, serie Botánica 45:75–86. [ Links ]
Scheinvar L. 1981. Especies, variedades y combinaciones nuevas de cactáceas del Valle de México. Phytologia 49:313–328. [ Links ]
Scheinvar L. 1982. Redescubrimiento de la Opuntia spinulifera Salm–Dyck. Anales del Instituto de Biología, Universidad Nacional Autónoma de México, serie Botánica 47:1–20. [ Links ]
Scheinvar L. 1985. Cactaceae. En: Rzedowski J. y Calderón de Rzedowski G. (comp. y eds.). La Flora Fanerogámica del Valle de México, vol. 2, Escuela Nacional de Ciencias Biológicas del Instituto Politécnico Nacional e Instituto de Ecología, A. C., México. [ Links ]
Scheinvar L. 1987. Redescubrimiento de Opuntia oligacantha Forster en el Valle de México y en la Altiplanicie Mexicana. Anales del Instituto de Biología, Universidad Nacional Autónoma de México, serie Botánica 57:109–122. [ Links ]
Scheinvar L. 1999. Opuntia zamudioi, una nueva especie de Querétaro. Cactáceas y Suculentas Mexicanas 44:88–93. [ Links ]
Scheinvar L. 2002. Opuntia stricta (Haw.) Haw. subsp. esparzae, una nueva subespecie de las dunas del río Concá, Arroyo Seco, Querétaro, México. Cactáceas y Suculentas Mexicanas 47:94–102. [ Links ]
Scheinvar L. 2004. Flora Cactológica del Estado de Querétaro. Diversidad y Riqueza. Fondo de Cultura Económica, México. [ Links ]
Scheinvar L. y Rodríguez A. 2003. Nueva subespecie de Opuntia streptacantha (Cactaceae) de la altiplanicie mexicana. Anales del Instituto de Biología, Universidad Nacional Autónoma de México, Serie Botánica 74:303–311. [ Links ]
Scheinvar L. y Olalde–Parra G. 2008. Tipificación de algunas cactáceas del estado de Guerrero. Cactáceas y Suculentas Mexicanas 53:36–50. [ Links ]
Scheinvar L. y Manzanero G. 2009. Opuntia chiangiana, una nueva especie de Cactaceae de Oaxaca, México. Novon: A Journal for Botanical Nomenclatura 19:222–228. [ Links ]
Scheinvar L., Olalde G., Filardo S. y Beckler P. 2010. Diez especies mexicanas productoras de xoconostles: Opuntia spp. y Cylindropuntia imbricata (Cactaceae). Universidad Nacional Autónoma de México, Universidad Autónoma del Estado de Hidalgo y Universidad Autónoma Metropolitana, México. [ Links ]
Schimitz–Linneweber C., Maier R.M., Alcaraz J–P., Cottet A., Herrmann R.G. y Mache R. 2001. The plastid chromosome of spinach (Spinacia oleracea): complete nucleotide sequence and gene organization. Plant Molecular Biology 45:307–315. [ Links ]
Segura S., Scheinvar L., Olalde G., Leblanc O., Filardo S., Muratalla A., Gallegos C. y Flores C. 2007. Genome sizes and ploidy levels in Mexican cactus pear species Opuntia (Tourn.) Mill. series Streptacanthae Britton et Rose, Leucotrichae DC., Heliabravoanae Scheinvar and Robustae Britton et Rose. Genetic Resources and Crop Evolution 54:1033–1041. [ Links ]
SEMARNAT (Secretaría de Medio Ambiente y Recursos Naturales). 2010. Norma Oficial Mexicana NOM–059–ECOL–2010: Protección Ambiental– Especies nativas de México de flora y fauna silvestres– Categorías de riesgo y especificaciones para su inclusión, exclusión o cambio–Especies en riesgo. Diario Oficial de la Federación, 30 de diciembre.
Swofford D.L. 2002. PAUP* Phylogenetic analysis using parsimony (*and other methods). Version 4. Sinauer Associates, Sunderland. [ Links ]
Taberlet P., Gielly L., Pautou G. y Bouvet J. 1991. Universal primers for amplification of three non–coding regions of chloroplast DNA. Plant Molecular Biology 17:1105–1109. [ Links ]
Vogel J., Börner T. y Hess W.R. 1999. Comparative analysis of splicing of the complete set of chloroplast group II introns in three higher plants mutants. Nucleic Acids Research 27:3866–3874. [ Links ]
Weising K., Atkinson R.G. y Gardner R.C. 1995. Genomic fingerprinting by microsatellite–primed PCR: a critical evaluation. PCR Methods Applications 4:249–255. [ Links ]
White T.J., Bruns T., Lee S. y Taylor J. 1990. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenies. En: Innis M.A., Gelfand D.H., Sninsky J.J. y White T. Eds. PCR Protocols: A Guide to Methods and Applications. Pp. 315–322, Academic Press, San Diego. [ Links ]
Wilcox C.D., Dove S., McDavis W. y Greer D. 2002. UTHSCSA. Image Tool version 3.0. University of Texas Health Center. San Antonio. <http://compdent.uthscsa.edu/dig/itdesc.html> (Consultado Diciembre del 2014) [ Links ]
Zhang Z., Schwartz S., Wagner L. y Miller W. 2000. A greedy algorithm for aligning DNA sequences. Journal of Computational Biology 7:203–214. [ Links ]














